Histone und Nukleosomen und ihr Einfluss auf die - StV Biologie ...
Histone und Nukleosomen und ihr Einfluss auf die - StV Biologie ...
Histone und Nukleosomen und ihr Einfluss auf die - StV Biologie ...
Erfolgreiche ePaper selbst erstellen
Machen Sie aus Ihren PDF Publikationen ein blätterbares Flipbook mit unserer einzigartigen Google optimierten e-Paper Software.
Bakkelaureatsarbeit<br />
PS Genexpression in Eukaryoten<br />
WS 2004/2005<br />
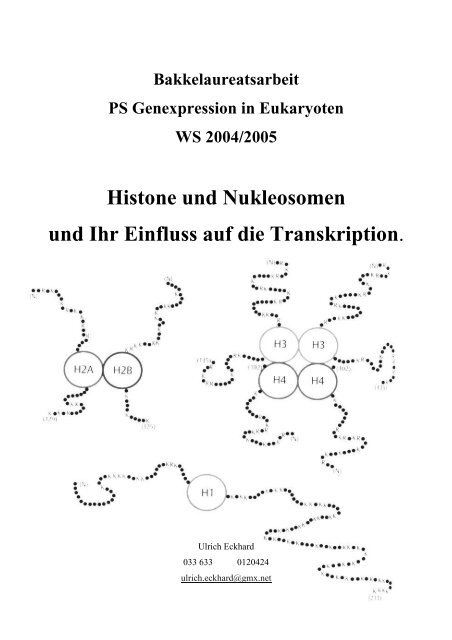
<strong>Histone</strong> <strong>und</strong> <strong>Nukleosomen</strong><br />
<strong>und</strong> Ihr <strong>Einfluss</strong> <strong>auf</strong> <strong>die</strong> Transkription.<br />
Ulrich Eckhard<br />
033 633 0120424<br />
ulrich.eckhard@gmx.net
1) Einleitung<br />
Die wichtigste Funktion der DNA ist das Übertragen von Genen, der Information, <strong>die</strong> alle Proteine spezi-<br />
fiziert, <strong>die</strong> ein Lebewesen <strong>auf</strong>bauen – einschließlich der Informationen, wann, in welchem Zelltyp <strong>und</strong> in<br />
welchen Mengen jedes Protein hergestellt werden soll.<br />
Die Genome der Eukaryoten sind dabei in mehrere Chromosomen <strong>auf</strong>geteilt, wobei Eukaryotische Chro-<br />
mosomen größer sind als prokaryotische, <strong>und</strong> auch einen höheren Grad an struktureller Organisation be-<br />
sitzen: z.B. enthalten <strong>die</strong> Genome der Hefe, der T<strong>auf</strong>liege <strong>und</strong> des Menschen etwa 4-, 40- <strong>und</strong> 1000mal<br />
soviel DNA wie das E. coli Genom.<br />
Tabelle 1 DNA-Gehalt verschiedener prokaryotischer <strong>und</strong> eukaryotischer Genome.<br />
Anmerkung: Die Werte gelten für das haploide Genom (Stryer S.1023)<br />
Organismus<br />
Anzahl der<br />
Basenpaare<br />
DNA-Länge<br />
(in mm)<br />
Anzahl der<br />
Chromosomen<br />
E. coli 4 x 10 6 1,4 1<br />
Hefe (S. cerevisiae) 1,4 x 10 7 4,6 16<br />
T<strong>auf</strong>liege (D. melangonaster) 1,7 x 10 8 56 4<br />
Mensch 2,9 x 10 9 990 23<br />
Jede Zelle des Menschen enthält z.B., von Ende zu Ende ausgestreckt, etwa 2 Meter DNA; während der<br />
<strong>die</strong> DNA enthaltende Kern nur einen Durchmessen von etwa 6 µm hat.<br />
Die komplexe Aufgabe der DNA-Verpackung wird durch spezialisierte Proteine erreicht, <strong>die</strong> an <strong>die</strong> DNA<br />
binden <strong>und</strong> sie in eine Folge von Windungen <strong>und</strong> Schleifen <strong>auf</strong>falten, <strong>die</strong> stufenweise eine höhere Orga-<br />
nisation bieten, <strong>und</strong> gleichzeitig verhindern, dass <strong>die</strong> DNA ein un<strong>auf</strong>lösbares Knäuel bildet. Trotzdem<br />
bleibt <strong>die</strong> DNA aber noch für viele Proteine leicht zugänglich, <strong>die</strong> sie verdoppeln, reparieren, <strong>und</strong> <strong>ihr</strong>e<br />
Gene zur Herstellung der Proteine nutzen.<br />
1.1) Das Chromatin<br />
Die DNA eukaryotischer Chromosomen liegt in vivo nie nackt vor. Sie ist fest mit einer Gruppe kleiner<br />
basischer Proteine, den <strong>Histone</strong>n verb<strong>und</strong>en, wobei <strong>die</strong>se sogar <strong>die</strong> Hälfte der Masse von Eukaryotench-<br />
romosomen ausmachen. Dieser Histon-DNA-(Nucleoprotein-)Komplex wird Chromatin genannt; Chro-<br />
matin besitzt eine kompakte Organisation, in der <strong>die</strong> meisten DNA-Sequenzen strukturell unzugänglich<br />
<strong>und</strong> funktionell inaktiv sind. Innerhalb <strong>die</strong>ser Masse liegt <strong>die</strong> geringere Zahl der aktiven, d.h. transkribier-<br />
1
2<br />
ten Sequenzen. Zusätzlich zum DNA-Faden <strong>und</strong> den <strong>Histone</strong>n, besteht Chromatin noch aus einer Kol-<br />
lektion von Nicht-Histon-Chromatin-Proteinen<br />
Während aber das Verhältnis von DNA zu <strong>Histone</strong>n in allen eukaryotischen Zellen gleich ist, <strong>und</strong> sich<br />
auch nicht wesentlich mit dem physiologischen Zustand der Zelle ändert, ist <strong>die</strong> Art, Menge, <strong>und</strong> Zu-<br />
sammensetzung der Nicht-Histon-Chromatin-Proteine von Zelltyp zu Zelltyp verschieden, <strong>und</strong> wechselt<br />
auch mit der genetischen Aktivität einer Zelle. Zu den Nicht-Histon-Chromatin-Proteinen gehören unter<br />
anderem auch Proteine des Transkriptionsapparates wie RNA-Polymerasen <strong>und</strong> positiver <strong>und</strong> negativer<br />
Transkriptionsfaktoren, deren Menge <strong>und</strong> Art sich mit dem physiologischen Zustand der Zelle ändert.<br />
Es gibt aber auch einige Arten von Nicht-Histon-Chromatin-Proteinen (sowohl in Tier- wie Pflanzenzel-<br />
len), <strong>die</strong> weitgehend unabhängig von der genetischen Aktivität im Zellkern vorkommen, wie z.B. <strong>die</strong> so<br />
genannten HMG-Proteine („high mobility group“), <strong>die</strong> hauptsächlich in zwei Gruppen von unterschieden<br />
werden:<br />
• HMG1 <strong>und</strong> HMG2: binden im Reagenzglasversuch bevorzugt an DNA mit ungewöhnlichen<br />
Strukturen, wie kreuzförmige DNA oder Stellen, wo <strong>die</strong> B-Form- in Z-Form-DNA übergeht. Im<br />
Chromatin kommen sie an der Linker-DNA zwischen den Nucleosomen vor.<br />
• HMG14 <strong>und</strong> HMG17. binden an Nucleosomen-Core-Partikel, <strong>und</strong> bewirken eine <strong>auf</strong>gelockerte<br />
Struktur des Chromatins, wodurch Gene transkribiert werden können.<br />
1.2) Die Nucleosomen sind <strong>die</strong> erste Stufe bei der Packung der DNA<br />
Die Gr<strong>und</strong>einheit des Chromatins besitzt bei allen Eukaryoten <strong>die</strong>selbe Struktur. Das Nucleosom enthält<br />
etwa 200bp DNA <strong>und</strong> ist in Form eines Oktamers aus kleinen basischen Proteinen organisiert, <strong>die</strong> eine<br />
perlenförmige Struktur bilden. Ein DNA-Strang von 200 Basenpaaren hätte in Lösung eine Länge von<br />
etwa 68nm, wohingegen <strong>die</strong>selbe DNA-Menge in ein Nucleosom mit einen Durchmesser von ~11nm<br />
passt. Das Packungsverhältnis (das Maß der Verdichtung) des Nucleosoms beträgt also ungefähr 6.<br />
Betrachtet man jedoch den Verdichtungsgrad der DNA in Chromosomen, kommt man <strong>auf</strong> eine Pa-<br />
ckungsverhältnis von etwa 10 4 (Gesamtlänge der 46 Metaphasechromosomen: ~200µm; DNA End-zu-<br />
End-Länge: ~1,8m). DNA im Interphasekern mit weniger dichtem Chromatin hat noch immer ein Pa-<br />
ckungsverhältnis von 10 2 bis 10 3 . Das bedeutet, dass noch weitere, höhere Verpackungsstufen existieren<br />
müssen.
1.3) Die höheren Verpackungsstufen<br />
Die zweite Organisationsstufe ist das Aufwickeln der Nucleosomenreihe<br />
in eine helicale Struktur, einer Art Spule (wurde als Solenoidmodell des<br />
Chromatins postuliert), so dass ein Faden von etwa 30 nm Stärke entsteht,<br />
den man sowohl im Interphasechromatin als auch in den mitotischen<br />
Chromosomen findet. Im Chromatin ergibt sich daraus ein DNA-<br />
Packungsverhältnis von etwa 40.<br />
Abb.2 Modell zur Chromatinpackung. Diese<br />
schematische Zeichnung zeigt einige der zahlreichen<br />
Ordungsprinzipien der Chromatinpackung,<br />
<strong>die</strong> postuliert wurden, um ein hochgradig kondensiertes<br />
mitotisches Chromosom entstehen zu<br />
lassen. (Alberts S.265)<br />
2) Evolution des Chromatins<br />
3<br />
Abb.1 Das Solenoedmodell des<br />
Chromatins mit 6 Nucleoso-men<br />
pro Helixwindung (Stryer S.1027)<br />
Das endgültige Packungsverhältnis wird durch <strong>die</strong> dritte Or-<br />
ganisationsstufe bestimmt, durch <strong>die</strong> Packung des Fadens<br />
selbst. Dadurch entsteht im Euchromatin schließlich ein Pa-<br />
ckungsverhältnis von etwa l0 3 . Das Euchromatin unterliegt<br />
jedoch zyklischen Veränderungen; durch das Verpacken in<br />
<strong>die</strong> Chromosomen während der Mitose wird ein Packungs-<br />
verhältnis von ungefähr 10 4 erreicht wird, ähnlich der Dich-<br />
tezunahme beim weitgehend inaktiven Heterochromatin.<br />
Jedoch darf nicht vergessen werden, dass bereits ab der Ebe-<br />
ne der 30nm Faser zahlreiche weitere Proteine (Nicht-<br />
Histon-Proteine) erforderlich sind: Entfernt man z.B. von<br />
Metaphasechromosomen <strong>die</strong> <strong>Histone</strong>, besitzen sie nach wie<br />
vor ein zentrales Proteingerüst, das von vielen langen DNA-<br />
Schleifen, <strong>die</strong> wahrscheinlich an das Proteingerüst geb<strong>und</strong>en<br />
sind, umgeben ist.<br />
Historisch werden <strong>Histone</strong> <strong>und</strong> Chromatin, obwohl Chromatin-like-packaging auch bei Archaean gefun-<br />
den wurden, immer in Zusammenhang mit Eukaryoten, als typisch eukaryotische Eigenschaft gebracht. In<br />
Prokaryoten vermutete man ebenfalls lange Zeit histon-like Proteine (wie HU <strong>und</strong> H-NS), <strong>die</strong> jedoch, wie<br />
sich später herausstellte, weder eine signifikante Homologie <strong>auf</strong> Sequenzebene, noch <strong>auf</strong> Ebene der Prote-<br />
infaltung, mit eukaryotischen <strong>Histone</strong>n <strong>auf</strong>wiesen.
4<br />
Behandelte man jedoch z.B. das Chromatinextrakt des Extremophilen Archaea Halobacterium salina-<br />
rum mit Proteinase K, verschwindet <strong>die</strong> zuvor beobachtbare Perlkettenschnur („beads-on-a-string“) Struk-<br />
tur des Chromatins, <strong>die</strong> den Nucleosomen von Eukaryoten ähnelt, <strong>und</strong> <strong>die</strong> nackten, intakten DNA-Stränge<br />
bleiben zurück.<br />
− Proteinase K<br />
+ Proteinase K<br />
Abb.3 Genomische DNA von Archaea ist organisiert in chromatinähnlichen Strukturen.<br />
Elektronenmikroskopische Betrachtung einer Chromosomenpräparation von Halobacterium salinarium<br />
(spät exponentielle Wachstumsphase). Die beobachteten Fasern (links) bestehen aus DNA <strong>und</strong> assozierten<br />
Proteinen. Behandlung mit Proteinase K lässt <strong>die</strong> nackten DNA-Stränge zurück (rechts). (Weinzierl S.278)<br />
Seit dem <strong>die</strong> genomische Sequenz des Archaea Methanococcus jannaschii bekannt ist, wurden z.B. nicht<br />
weniger als fünf histone-like-Genes gef<strong>und</strong>en.<br />
Die meisten Archaea leben unter ungewöhnlichen Wachstumsbedingungen <strong>und</strong> bewohnen für Menschen<br />
extreme Biotope („Extremophile“), wie kochend heiße Quellen (bis zu 113°C), schwefelsaure Schlamm-<br />
löcher (bis zu pH 0) oder hochkonzentrierten Salzlaken (bis zu 25% NaCl). Das sind Bedingungen, wie<br />
sie eventuell auch <strong>auf</strong> der noch jungen Erde zu finden waren, als sich das Leben zu entwickeln begann.<br />
Aus der Tatsache, dass <strong>Histone</strong> sowohl in Eukaryoten wie Archaea gef<strong>und</strong>en werden können, lässt sich<br />
schließen, dass <strong>die</strong> Evolution der <strong>Histone</strong> bereits kurz nach dem Ursprung des Lebens stattgef<strong>und</strong>en ha-<br />
ben muss, wahrscheinlich als Schutz vor <strong>die</strong>sen für <strong>die</strong> DNA unfre<strong>und</strong>lichen Bedingungen.<br />
Hyperthermophile Archaea wie Methanotermus wachsen im Labor optimal bei 83 °C, <strong>und</strong> das obwohl<br />
<strong>ihr</strong>e DNA sehr reich an A/T (67%) ist. In vivo spielen dabei drei Faktorern eine wesentliche Rolle: <strong>die</strong><br />
hohe intrazelluläre Konzentration an monovalenten Kationen (~950mM K + ), <strong>die</strong> Anwesenheit diverser<br />
Stoffwechselzwischenprodukte wie Zyklisches Diphosphoglycerat, <strong>und</strong> <strong>die</strong> Anwesenheit von <strong>Histone</strong>n.<br />
In vitro konnte gezeigt werden, dass unter low-salt Bedingungen <strong>die</strong> Schmelztemperatur der DNA durch<br />
<strong>die</strong> Anwesenheit von Archaea-<strong>Histone</strong>n von 60,5 <strong>auf</strong> 84,5 °C erhöht wird.<br />
<strong>Histone</strong> haben somit in der Evolution einen so genannten „switch of function“ durchgemacht. Während<br />
<strong>ihr</strong>e Haupt<strong>auf</strong>gabe bei den Archaea im Schutz der DNA vor Denaturierung liegt, fungieren sie bei Euka-
5<br />
ryoten als strukturbildende Proteine, <strong>die</strong> <strong>die</strong> Verpackung der – im Vergleich zu Bakterien riesigen Chro-<br />
mosomen – im Kern ermöglichen.<br />
3) Nucleosomen – sich wiederholende Einheiten des Chromatins<br />
Bereits 1974 entwickelte Roger Kornberg ein Modell, wonach Chromatin aus sich wiederholenden Ein-<br />
heiten besteht, wobei jede aus 200 DNA-Basenpaaren <strong>und</strong> je zwei Molekülen H2A, H2B, H3 <strong>und</strong> H4<br />
zusammengesetzt sei. Der größte Teil der DNA ist dabei um ein Core aus <strong>Histone</strong>n gewickelt; <strong>die</strong> übrige<br />
DNA verbindet als Linker benachbarte Nucleosomen <strong>und</strong> trägt somit zur Flexibilität der Chromatinfaser<br />
bei.<br />
3.1) <strong>Histone</strong><br />
Einfache biochemische Untersuchungen zeigen, dass in den Kernen aller Tier- <strong>und</strong> Pflanzenzellen fünf<br />
Histontypen vorkommen, nämlich <strong>die</strong> <strong>Histone</strong> H1, H2A, H2B, H3 <strong>und</strong> H4. Ihre Masse liegt im Bereich<br />
von 11 bis 21 kd. Nur in den Kernen von Hefezellen fehlt das Histon H1; <strong>die</strong> übrigen vier Histon-Typen<br />
sind aber wie bei allen anderen Eukaryoten vorhanden.<br />
Mit einigem Abstand betrachtet, haben alle <strong>Histone</strong> eine ähnliche Struktur mit einer zentralen, annähernd<br />
globulären Domäne <strong>und</strong> mit flexiblen aminoterminalen <strong>und</strong> carboxyterminalen Armen.<br />
Das Kennzeichen von <strong>Histone</strong>n ist der hohe Anteil an basischen Aminosäuren, Arginin <strong>und</strong> Lysin; dabei<br />
sind <strong>die</strong> basischen Aminosäuren nicht gleichmäßig über das Molekül verteilt, sondern kommen bevorzugt<br />
in den flexiblen Armen vor.<br />
Abb.5 Strukturformel<br />
Lysin<br />
Abb.6 Strukturformel<br />
Arginin<br />
Abb.4 <strong>Histone</strong>: Strukturschema. Alle <strong>Histone</strong> bestehen aus<br />
einer zentralen globulären Domäne <strong>und</strong> flexiblen Armen mit<br />
vielen positiv geladenen Aminosäuren: K = Lysin, R = Arginin,<br />
= andere Aminosäuren. Die <strong>Histone</strong> H2A <strong>und</strong> H2B<br />
lagern sich als Dimere, <strong>die</strong> <strong>Histone</strong> H3 <strong>und</strong> H4 als Tetramere<br />
aneinander (Knippers S.148))
Die Aminosäuresequenzen der <strong>Histone</strong> sind im L<strong>auf</strong>e der Evolution<br />
bemerkenswert konstant geblieben. So unterscheiden sich z.B. das<br />
Histon H4 aus Erbsenkeimlingen <strong>und</strong> Kalbsthymus in nur zwei von<br />
102 Resten, wobei <strong>die</strong> Änderungen geringfügig sind, da ähnliche Ami-<br />
nosäuren ausgetauscht wurden: Valin statt Isoleucin <strong>und</strong> Lysin statt<br />
Arginin. Das bedeutet: Die Aminsäuresequenz von H4 ist also in den<br />
geblieben. Auch H3 hat sich während <strong>die</strong>ser Periode kaum verändert.<br />
Abb.7<br />
Strukturformel<br />
Valin<br />
6<br />
Abb.8<br />
Strukturformel<br />
Isoleucin<br />
Die Aminosäuresequenzen von H3 aus Erbsenkeimlingen <strong>und</strong> Kalbsthymus unterscheiden sich in nur vier<br />
Positionen. Auch <strong>die</strong> Sequenzen der anderen Histon-Typen sind während der Evolution konserviert<br />
geblieben, allerdings nicht so ausgeprägt wie <strong>die</strong> der <strong>Histone</strong> H3 <strong>und</strong> H4. Am geringsten ist <strong>die</strong> Ähnlich-<br />
keit zwischen den Histon H1-Sequenzen verschiedener Tier- <strong>und</strong> Pflanzenarten. Aber insgesamt gilt, dass<br />
<strong>Histone</strong> zu den höchst konservierten Proteinen überhaupt gehören.<br />
Das bedeutet allerdings nicht, dass alle <strong>Histone</strong> eines gegebenen Typs im Chromatin identisch sind. Alle<br />
höheren Eukaryoten besitzen mehrere Gene für jedes Histon, wobei sich manche Gene von anderen des<br />
gleichen Histon-Typs durch kleine Unterschiede in den Ko<strong>die</strong>rungssequenzen unterscheiden, wodurch<br />
Histonsubtypen gebildet werden. Einige davon werden nur unter bestimmten Bedingungen gebildet, wie<br />
etwa ein als Histon H5 bezwichneter Histon-H1-Subtyp, der nur in den Kernen der hochdiffferenzierten<br />
Erythrocyten von Vögeln vorkommt, <strong>und</strong> dort zur dichten Packung des Chromatins <strong>und</strong> zur Blockade der<br />
genetischen Aktivität beiträgt.<br />
3.2) Nucleosomen<br />
Suspen<strong>die</strong>rt man Interphasekerne in einer Lösung mit niedriger lonenstärke, quellen sie <strong>auf</strong> <strong>und</strong> platzen,<br />
sodass <strong>die</strong> Chromatinfäden freigesetzt werden. In einigen Bereichen bestehen <strong>die</strong> Fäden aus sehr dicht<br />
gepacktem Material, in ausgestreckten Bereichen ist jedoch zu erkennen, dass sie separate Partikel enthal-<br />
ten, <strong>die</strong> Nucleosomen. In besonders ausgestreckten Bereichen sind <strong>die</strong> einzelnen Nucleosomen durch den<br />
dünnen Faden eines freien DNA-Doppelstranges (linker-DNA) verb<strong>und</strong>en (beads-on-a-string-structure<br />
bzw. Perlenschnurform). Die einzelnen Nucleosomen lassen sich durch Behandlung des Chromatins mit<br />
dem Enzym Micrococcus-Nuclease isolieren. Dabei handelt es sich um eine Endonuclease, <strong>die</strong> den DNA-<br />
Faden an der linker-DNA durchschneide, wobei <strong>die</strong> Partikel erst in Gruppen freigesetzt werden, <strong>und</strong> erst<br />
nach längerer Behandlung in Form einzelner Nucleosomen.
Abb.10 Chromatin nach Micrococcus-Nuclease Behandlung.<br />
Dabei werden einzelne Nucleosomen freigesetzt. Der<br />
eingezeichnete Strich entspricht 100nm (Lewin S.569)<br />
Abb.9 Chromatinaustritt aus lysierten Interphase-Zellkernen. Dabei<br />
besteht das Chromatin aus einer kompakt strukturierten Folge von Partikeln.<br />
Der eingezeichnete Strich entspricht 100nm (Lewin S.569)<br />
3.2.1) Behandlung von Chromatin mit Micrococcus-Nuclease <strong>und</strong> <strong>die</strong> Schlüsse daraus<br />
Eine Behandlung mit dem Enzym Mikrokokken-Nuclease greift <strong>die</strong> DNA zwischen den einzelnen Nucle-<br />
osomen eines Chromatin-Fadens an, lässt aber <strong>die</strong> unmittelbar <strong>auf</strong> dem Histon-Oktamer liegende DNA,<br />
unbeschädigt. Die Auftrennung der DNA Fragmente in einer Gelelektrophorese führt zu einer „DNA-<br />
Leiter", deren Einheitslänge, <strong>die</strong> sich aus den Abständen zweier Banden ergibt, r<strong>und</strong> 200bp beträgt. Die<br />
Tatsache, dass man in der 200-bp-Leiter mehr als 90% der Chromatin-DNA wiederfinden kann, bestätigt,<br />
dass DNA <strong>auf</strong> Reihen von Nucleosomen <strong>auf</strong>gewickelt ist, <strong>und</strong> fast <strong>die</strong> gesamte DNA in Nucleosomen<br />
organisiert ist. Dabei sind <strong>die</strong> Nucleosomen wahrscheinlich im nativen Zustand so dicht gepackt, dass <strong>die</strong><br />
DNA von einem Nucleosom direkt zum nächsten verläuft, <strong>und</strong> freie DNA in vivo kaum oder gar nicht<br />
vorkommt.<br />
Das erhaltene Fragmentmuster kann aber auch mit dem Zelltyp, innerhalb des Zelltyps (z.B. bei tandem-<br />
artig wiederholten Sequenzen wie den Clustern der 5S-RNA-Gene), mit dem Organismus (der Durch-<br />
schnitt liegt meistens zwischen 180 <strong>und</strong> 200bp, aber es gibt auch Extremfälle mit 154 bp – bei diversen<br />
Pilzen – oder mit bis zu 260bp bei Seeigelspermien) varrieren. Die Unterschiede lassen sich dabei <strong>auf</strong> <strong>die</strong><br />
linker-DNA zurückführen, <strong>die</strong> bei zunehmender Dauer der Nuclease-Behandlung abgebaut wird. Nur<br />
noch <strong>die</strong> Histon-Oktamere (aus je zwei Kopien von H2A, H2B, H3 <strong>und</strong> H4; den Core-<strong>Histone</strong>n) mit der<br />
direkt <strong>auf</strong>liegenden DNA (1,65 Windungen DNA) bleiben zurück; <strong>die</strong>ses DNA-Stück besteht unabhängig<br />
von der Herkunft des Chromatins aus 146 Basenpaaren, <strong>und</strong> wird als Core-particle des Nucleosoms be-<br />
7
8<br />
zeichnet, wobei bei der Überführung vollständiger Nucleosomen in Core-Partikel <strong>die</strong> H1 <strong>Histone</strong> verlo-<br />
ren gehen.<br />
Abb.11 Behandlung von Chromatin mit Micrococcen-Nuclease. Die Micrococcus-Nuclease schneidet zuerst<br />
zwischen den Nucleosomen. Mononucleosomen enthalten normalerweise etwa 200bp DNA. Die Verkürzung<br />
der Fragment vom Ende her verringert <strong>die</strong> DNA-Länge zuerst <strong>auf</strong> etwa 165bp (Nucleosom noch incl.<br />
Histon H1) <strong>und</strong> erzeugt schließlich Core-Partikel (ohne H1) mit 146bp DNA. (Lewin S. 573)<br />
Aufgr<strong>und</strong> <strong>die</strong>ser Analyse lässt sich <strong>die</strong> nucleosomale DNA in zwei Bereiche einteilen:<br />
• Die Core-DNA besitzt eine konstante Länge von 146bp <strong>und</strong> ist gegenüber einem Nucleaseabbau<br />
verhältnismäßig stabil.<br />
• Die Linker-DNA enthält den übrigen Teil der sich wiederholenden Sequenzeinheit, wobei <strong>die</strong><br />
Länge pro Nucleosom zwischen nur acht Basenpaaren <strong>und</strong> 114bp betragen kann.<br />
Die spezifische Größe des DNA-Fragments, das bei der ersten Reaktion der Micrococcus-Nuclease ent-<br />
steht, deutet dar<strong>auf</strong> hin, dass der Bereich, an dem das Enzym angreifen kann, <strong>auf</strong> einen Teil der Linker-<br />
DNA begrenzt ist, da sonst Fragmente verschiedenster Länge (von 146bp bis 260bp) entstehen würden.<br />
Erst wenn <strong>die</strong> Linker-DNA geschnitten wurde, wird anscheinend <strong>die</strong> übrige Region angreifbar <strong>und</strong> durch<br />
<strong>die</strong> weitere enzymatische Aktivität relativ schnell abgebaut.<br />
Ob eine Linker-DNA vorhanden ist, <strong>und</strong> wie lang sie ist, hängt von anderen Faktoren als den vier Core-<br />
<strong>Histone</strong>n ab. Experimente zur Rekonstituierung in vitro zeigen, dass <strong>Histone</strong> von sich aus <strong>die</strong> Fähigkeit<br />
besitzen, DNA in Form von Core-Partikeln zu strukturieren; sie bilden jedoch nicht zwangsläufig Nucleo-<br />
somen mit einer DNA-Einheitslänge, wie sie für den in vivo-Zustand charakteristisch sind.<br />
Insbesondere Nichthistonproteine <strong>und</strong> / oder Histon H1 beeinflussen <strong>die</strong> Länge der Linker-DNA, <strong>die</strong> in<br />
einer natürlichen Nucleosomenkette mit dem Histonoktamer assoziiert ist. Außerdem sind in vivo-beim<br />
Zusammenbau der Nucleosomen sogenannte „Assembly-Proteine" beteiligt, <strong>die</strong> jedoch selbst nicht zur<br />
Struktur des Nucleosoms gehören.
3.2.2) Die Core-Partikel<br />
Die Core-Partikel besitzen trotz <strong>ihr</strong>er geringeren Größe ähnliche Eigenschaften wie <strong>die</strong> Nucleosomen,<br />
Umfang <strong>und</strong> Größe sind ähnlich, sodass wahrscheinlich <strong>die</strong> gr<strong>und</strong>legende geometrische Struktur der Par-<br />
tikel <strong>auf</strong> den Wechselwirkungen zwischen der DNA <strong>und</strong> dem Proteinoktamer des Core-Partikels beruht.<br />
Bereits <strong>die</strong> elektronenmikroskopische Aufnahme eines Nucleosomen-Cor-Kristalls 1977 durch Finch et<br />
al. zeigte, dass <strong>die</strong>se Partikel homogen verteilt sind. Röntgenbeugungsanalysen <strong>und</strong> elektronenmikrosko-<br />
pische Untersuchungen <strong>die</strong>ser Kristalle durch Aaron Klug <strong>und</strong> John Finch ergaben in Folge für das Core-<br />
Partikel Dimensionen von 11 x 11 x 5,5 nm. Weitere Analysen ließen vermuten, dass sich ein H3- <strong>und</strong><br />
H4-Tetramer im Zentrum des Nucleosoms befindet, während je ein H2A-H2B Dimer an den Enden loka-<br />
lisiert ist. Dadurch wurden lang bekannte biochemische Bef<strong>und</strong>e bestätigt: Ließ man Nucleosomen in<br />
Lösungen hoher NaCl-Konzentration auseinander brechen, fand man neben der proteinfreien DNA als<br />
charakteristische Zerfallsprodukte Dimere der <strong>Histone</strong> H2A-H2B, sowie Tetramere aus je zwei Exempla-<br />
ren H3 <strong>und</strong> H4. Als Zwischenprodukte traten Hexamere <strong>auf</strong>, denen ein genau ein H2A-H2B-Dimer fehlte.<br />
Abb12. Elekronenmikroskopische Aufnahme eines<br />
Nucleosomen-Core-Kristalls. (Finch et al. 1977)<br />
Abb.13 Die Kristallstruktur des Histon-Core-Oktamers. H2A-H2B<br />
Dimer in blau. H32-H42 Tetramer in weiß. Oben: Aufsicht. Unten: Seitenansicht.<br />
Der mögliche DNA-Verl<strong>auf</strong> ist in der Aufsicht als dünner Schlauch<br />
(mit ¼ des DNA-Durchmessers) <strong>und</strong> in der Seitenansicht als parallele Linien<br />
in einem Bündel von 20 A Breite dargestellt. (Lewin S.581)<br />
Röntgenstruktur-Analysen von kristallisierten Core-Partikeln (Luger et al. 1997) ermöglichen einen ge-<br />
naueren Einblick in <strong>die</strong> Architektur von Nucleosomen. Die einzelnen Histon-Positionen wurden sichtbar,<br />
<strong>und</strong> es wurde deutlich, dass der globuläre Anteil der <strong>Histone</strong> aus einer Folge von α-Helices mit verbin-<br />
denden Schleifen entsteht. Charakteristisch ist dabei eine zentral gelegene lange α-Helix, <strong>die</strong> beiderseits<br />
von kürzeren α-Helices flankiert wird.<br />
9
10<br />
Dieses als Histon-Falte (histone fold) bekannte Strukturmotiv ist für <strong>die</strong> Kontakt<strong>auf</strong>nahme mit dem<br />
Partner-Histon verantwortlich. Darüberhinaus vermitteln <strong>die</strong> C-terminale Bereiche der H3 <strong>Histone</strong> den<br />
Kontakt zum Histon H3 im anderen H3/H4-Dimer <strong>und</strong> sind somit essentiell für <strong>die</strong> Tetramerisierung.<br />
Die Stabilität des Nucleosoms wird über Wechselwirkungen der globulären Anteile der <strong>Histone</strong> mit dem<br />
Phosphat<strong>die</strong>ster-Band in der kleinen Rinne der DNA (in Abständen von etwa 10bp) garantiert, während<br />
<strong>die</strong> flexiblen, basischen amino- <strong>und</strong> carboxyterminalen Histon-Arme über das Nucleosom hinausreichen,<br />
<strong>und</strong> mit den benachbarten Nucleosomen im Chromatin Wechselwirkungen eingehen können.<br />
Dies trifft insbesondere für den aminoterminalen Arm des Histons H4 zu, der sich mit unterschiedlichen<br />
Bindungseigenschaften – je nach Acetylierungsgrad seiner Lysin Reste – an ein Histon H2A/H2B-Dimer<br />
im nächst gelegenen Nucleosom lagert, <strong>und</strong> dadurch einen wesentlichen <strong>Einfluss</strong> <strong>auf</strong> <strong>die</strong> Anordnung der<br />
Nucleosomen im Chromatin hat.<br />
Abb.14 Die Struktur der aminoterminalen<br />
Schwänze des Core-Partikels ist nicht geklärt.<br />
Nur <strong>die</strong> globulären Anteile der <strong>Histone</strong><br />
liegen im Histonoktamer (Lewin S.582)<br />
Abb.15 <strong>Histone</strong> im Nucleosom. Diese Abbildung zeigt eine Ebene des Nucleosoms mit einer DNA Schleife von 73<br />
Basenpaaren. Es ist eine starke vereinfachung der Ergebnisse einer Röntgenstrukturanalyse kristallisierter Nucleosomen,<br />
damit <strong>die</strong> Wechselwirkung zwischen den einzelnen <strong>Histone</strong>n über <strong>die</strong> Histon-Falte <strong>und</strong> <strong>die</strong> Beziehung zwischen den<br />
<strong>Histone</strong>n <strong>und</strong> der DNA (graue Haken) besser zur Geltung kommen. (Knippers S.151)<br />
Eine abschließende Bemerkung zur nucleosomalen DNA. Sie ist mit durchschnittlich 10,2bp pro Helix-<br />
Windung stärker gew<strong>und</strong>en als proteinfreie DNA in Lösung (10,4-10,6bp pro Windung), weshalb es zu<br />
Verdrillungen <strong>und</strong> zur Ausbildung von Superhelices kommt, wenn Nucleosomen von „topologisch fixier-<br />
ter" DNA abgelöst werden. Im Übrigen ist der Wert von 10,2bp pro Windung ein Durchschnitt aller 14<br />
Windungen der nucleosomalen DNA; <strong>die</strong> Anzahl der Basenpaare pro Windung ist jedoch nicht konstant:<br />
sie ist am niedrigsten an den Ein- <strong>und</strong> Austrittsstellen (etwa 10bp pro Windung) <strong>und</strong> am höchsten in den<br />
drei zentralen Windungen (etwa 10,7bp pro Windung).
3.2.3) Das Histon H1<br />
H1 unterscheidet sich auch in seiner Stöchiometrie – ein Molekül pro Nucleosom verglichen mit zwei bei<br />
den anderen <strong>Histone</strong>n. Bemerkenswerterweise fand man im Gegensatz zu der Konstanz bei anderen<br />
<strong>Histone</strong>n mehrer H1-Typen, zudem wird H1 unmittelbar vor der Mitose phosphoryliert <strong>und</strong> nach der Mi-<br />
tose wieder dephosphoryliert, was vermuten lässt, dass <strong>die</strong>se kovalente Modifikation seine Fähigkeit steu-<br />
ert, <strong>die</strong> DNA zu verdichten.<br />
In vitro lassen sich <strong>die</strong> 10nm- <strong>und</strong> <strong>die</strong> 30nm Faser reversibel durch Veräb´nderung der Ionenstärke inein-<br />
ander überführen (Thoma et al. 1979), was dar<strong>auf</strong> hindeutet, dass <strong>die</strong> lineare Anordnung der Nucleoso-<br />
men im 10nm-Faden durch <strong>die</strong> Erhöhung der Ionenkonzentration <strong>und</strong> bei Anwesenheit von H1 zur 30nm-<br />
Struktur überspiralisiert wird. Diese Faltungen wird teilweise durch <strong>die</strong> Wechselwirkungen zwischen den<br />
Core-<strong>Histone</strong>n benachbarter Nucleosomen bestimmt, aber eine zentrale Funktion übernimmt wahrschein-<br />
lich das Histon Hl. Diesen Schluss wurde bereits vor mehr als 30 Jahren aus einfachen Experimenten ge-<br />
zogen: behandelte man Chromatin mit 0,5 M NaCl, hatte das <strong>die</strong> Ablösung von Histon Hl <strong>und</strong> zugleich<br />
eine Auflockerung des Chromatin zur Folge, während umgekehrt der Zusatz von Histon H1 das Chroma-<br />
tin verdichtete.<br />
Aber wo ist das Histon Hl lokalisiert? Beim Abbau von Chromatin geht es beim Verkürzen der Monome-<br />
ren (inclusive einem 165bp Stück-DNA) <strong>auf</strong> das Core-Partikel verloren, ohne jedoch <strong>die</strong> wesentliche<br />
Struktur des Nucleosoms selbst zu verändern. Das lässt dar<strong>auf</strong> schließen, dass Hl außerhalb des Partikels<br />
lokalisiert ist, <strong>und</strong> sich im Bereich der Linker-DNA befindet, <strong>die</strong> sich direkt an <strong>die</strong> Core-DNA anschließt.<br />
Dadurch könnte H1 <strong>die</strong> DNA im Nucleosom „versiegeln", indem es über Wechselwirkungen mit den<br />
H2A-Untereinheiten des Cores an <strong>die</strong> Stelle bindet, wo <strong>die</strong> DNA in das Nucleosom eintritt <strong>und</strong> es wieder<br />
verlässt.<br />
Abb. Schematische Darstellung eines Nucleosoms. Die DNA Doppelhelix ist<br />
um ein Histonoktamer gewickelt. Das Histon H1 lagert sich außen an <strong>die</strong>ses<br />
Core-Partikel <strong>und</strong> <strong>die</strong> Linker-DNA an <strong>und</strong> „versiegelt das Nucleosom.<br />
(Stryer S.1026)<br />
11
12<br />
Trotzdem ist <strong>die</strong> Lokalisation von Histon H1 noch nicht restlos geklärt, denn obwohl Hl für <strong>die</strong> Bil-<br />
dung der 30-nm-Struktur notwendig ist, ist H1 in 30nm Fäden schlechter zu finden als in 10nm Fäden, <strong>die</strong><br />
H1 noch enthalten. Auch durch Röntgenstrukturanalyse erzielte Beugungsdaten deuten mehr <strong>und</strong> mehr<br />
dar<strong>auf</strong> hin, dass H1 wahrscheinlich innen lokalisiert ist.<br />
3.2.4) Nucleosomenassembly<br />
Wie <strong>Histone</strong> mit der DNA bei der Nucleosomenbildung assozi-<br />
ieren, war schon immer eine schwierige Frage, <strong>die</strong> für einige<br />
Verwirrung sorgte. Bilden <strong>die</strong> <strong>Histone</strong> zuerst das Oktamer, um<br />
das sich <strong>die</strong> DNA später herumwickelt? Oder bindet ein H32-<br />
H42-Kern <strong>die</strong> DNA, <strong>und</strong> danach werden <strong>die</strong> H2A-H2B-Dimere<br />
hinzugefügt? Die Selbstformierung ist in vitro eine langsame<br />
<strong>und</strong> langwierige Reaktion, <strong>die</strong> dadurch eingeschränkt wird, dass<br />
<strong>die</strong> sich bildenden Partikel zur Präzipitation neigen, wobei in<br />
vitro beide Wege zur Formierung von Nucleosomen möglich<br />
sind.<br />
An der Assoziation der <strong>Histone</strong> mit der DNA sind in Folge<br />
Hilfsproteine beteiligt, <strong>die</strong> als „molekulare Anstandsdamen"<br />
(Chaperone) fungieren, indem sie an <strong>die</strong> <strong>Histone</strong> binden <strong>und</strong><br />
entweder einzelne <strong>Histone</strong> oder Histonkomplexe kontrolliert an<br />
<strong>die</strong> DNA übergeben. Das ist wahrscheinlich deshalb notwendig,<br />
da <strong>Histone</strong> als basische Proteine generell eine hohe Affinität zu<br />
Abb. Zwei mögliche Wege des Nucleosomenassembly.<br />
DNA kann in vitro<br />
einerseits dirket mit einem Histonoktamer<br />
in Wechselwirkung treten, oder<br />
aber sich zuerst mit dem H32-H42-<br />
Tetramer zusammenlagern, an das danach<br />
<strong>die</strong> H2A-H2B-Dimere angefügt<br />
werden. (Lewin S.584)<br />
DNA besitzen. Die Hilfsproteinen, wie z.B. Nl <strong>und</strong> Nucleoplasmin aus Xenopus Eiern, sind saure Protei-<br />
ne, <strong>die</strong> durch <strong>die</strong> Bindung an <strong>die</strong> <strong>Histone</strong> deren positive Nettoladung vermindern. Dadurch können Histo-<br />
ne Nucleosomen bilden, ohne dass sie in anderen kinetisch günstigen Zwischenstufen hängen bleiben.<br />
Versuche, Nucleosomen in vitro zu erzeugen, begannen mit der Analyse von Reaktionen zwischen freier<br />
DNA <strong>und</strong> <strong>Histone</strong>n. Neue Nucleosomen bilden sich in vivo jedoch nur, wenn DNA repliziert wird. Mit<br />
Extrakten aus menschlichen Zellen, welche <strong>die</strong> SV40-DNA replizieren <strong>und</strong> aus den Produkten Chromatin<br />
<strong>auf</strong>bauen, entwickelte man ein System, das <strong>die</strong> notwendigen Reaktionen simuliert. Die Chromatinbildung<br />
verläuft dabei bevorzugt mit replizierender DNA, wobei der Hilfsfaktor CAF-1, ein Komplex mit mehr<br />
als fünf Untereinheiten <strong>und</strong> einer Masse von 238 kD, erforderlich ist. CAF-1 wirkt stöchiometrisch, <strong>und</strong><br />
seine Funktion besteht darin, neusynthetisierte H3- <strong>und</strong> H4-<strong>Histone</strong> zu binden <strong>und</strong> an der Replikationsga-
13<br />
bel anzulagern. Die H2A-H2B Dimere treten später dazu. Die so gebildeten Nucleosomen enthalten in<br />
<strong>ihr</strong>er Wiederholungsstruktur 200 bp DNA, jedoch kein Hl-Histon; anscheinend ist H1 für <strong>die</strong> korrekten<br />
Abstände nicht notwendig.<br />
Bei der Vermehrung des Chromatins wird ein DNA-Strang repliziert, der bereits mit Nucleosomen asso-<br />
ziiert ist, <strong>und</strong> es entstehen zwei Tochterdoppelstränge. Aber was geschieht mit den existierenden Nucleo-<br />
somen? Dissoziieren <strong>die</strong> Oktamere in freie <strong>Histone</strong>, <strong>die</strong> später wieder verwendet werden, oder bleiben sie<br />
in zusammengesetzter Form erhalten?<br />
Lässt man zuerst Zellen in Gegenwart schwerer Aminosäuren wachsen, <strong>die</strong> Replikation jedoch in Anwe-<br />
senheit von leichten Aminosäuren durchführen, lassen sich alte <strong>und</strong> neue Oktamere – nach Quervernet-<br />
zung der Histonoktamere <strong>und</strong> anschließendem Zentrfugieren in einem Dichtegra<strong>die</strong>nten – eindeutig un-<br />
terscheiden, wenn bei der Replikation <strong>die</strong> Histonoktamere bestehen bleiben. Dann würde man einerseits<br />
<strong>die</strong> ursprünglichen Oktamere bei hoher Dichte, <strong>und</strong> andererseits <strong>die</strong> neuen Oktamere bei niedrigerer<br />
Dichte finden. Wenn jedoch <strong>die</strong> alten <strong>Histone</strong> freigesetzt, <strong>und</strong> mit neuen <strong>Histone</strong>n wieder zusammenge-<br />
fügt werden, besitzen <strong>die</strong> Oktamere nun eine mittlere Dichte. Experimentell zeigte sich, dass im Bereich<br />
hoher Dichte nur sehr wenig Material zu finden ist. Die Histonoktamere bleiben somit bei der Replikation<br />
offensichtlich nicht bestehen.<br />
Das Muster aus Zerlegen <strong>und</strong> Zusammensetzen der Nucleosomen ist bei weitem nicht geklärt. Mögli-<br />
cherweise dissoziieren <strong>die</strong> Oktamere vollständig in <strong>ihr</strong>e Histonbausteine. Es kann jedoch sein, dass <strong>die</strong><br />
Oktamere eines der H2A-H2B-Dimere oder beide verlieren, <strong>die</strong> zufällig durch neue oder alte Moleküle<br />
ersetzt werden. In <strong>die</strong>sem Fall bliebe das H32-H42-Tetramer erhalten. Vielleicht kommt es auch bei der<br />
Transkription zu einem ähnlichen Aufbrechen der Struktur. Das H32-H42-Tetramer könnte in der Lage<br />
sein, während der Replikation vorübergehend mit einem Einzelstrang zu assoziieren; möglicherweise<br />
bleibt es mit dem Leitstrang verb<strong>und</strong>en. Das würde bedeuten, dass zumindest einige Histonoktamere di-<br />
rekt an der DNA gebildet werden.<br />
3.2.5) Nucleosomen-Positionierung („nucleosome phasing“)<br />
In vitro lassen sich Nucleosomen unabhängig von der DNA-Sequenz rekonstituieren, was jedoch nicht<br />
bedeutet, dass <strong>die</strong> Nucleosomenbildung auch in vivo sequenzunabhängig erfolgt. Tatsächlich weisen di-<br />
verse experimentelle Ansätze <strong>auf</strong> eine Nucleosomenpositionierung hin.<br />
Eine Nucleosomenpositionierung ließe sich in der Theorie <strong>auf</strong> zwei Arten erreichen:
• Entweder inhärent: Jedes Nucleosom wird spezifisch an einer bestimmten DNA-Sequenz angela-<br />
gert; das hieße: <strong>die</strong> Histonoktamere könnten keine beliebige DNA-Sequenz binden<br />
• oder exogen: Das erste Nucleosom lagert sich bevorzugt an einer bestimmten Stelle in einem Be-<br />
reich ohne Nucleosomen an, <strong>und</strong> stellt einen Startpunkt für <strong>die</strong> folgenden Nucleosomen dar, <strong>die</strong><br />
dann Wiederholungseinheiten definierter Länge bilden.<br />
In vivo sind wahrscheinlich beide Prozesse beteiligt: wenn strukturelle Merkmale der DNA ausschlagge-<br />
bend sind, kommt es zu einer inhärenten Nucleosomenpositionierung. Wenn jedoch Muster <strong>auf</strong> Gr<strong>und</strong><br />
von Wechselwirkungen zwischen anderen Proteinen <strong>und</strong> der DNA <strong>und</strong> / oder den <strong>Histone</strong>n entstehen,<br />
sind sie exogen festgelegt.<br />
Bestimmte Strukturmerkmale der DNA beeinflussen <strong>die</strong> Plazierung der Histonoktamere. DNA neigt von<br />
sich aus dazu, sich in eine Richtung mehr zu krümmen als in eine andere. A/T-reiche Abschnitte lagern<br />
sich so, dass <strong>die</strong> kleine Furche zum Oktamer zeigt, während bei G/C-reichen Abschnitten <strong>die</strong> kleine Fur-<br />
che außen liegt. Auch kommen in der zentralen Superhelixwindung des Core-Bereiches keine langen Be-<br />
reiche aus dA/dT (mehr als acht Basenpaare) vor. Im Allgemein gilt, dass <strong>die</strong> Orientierung der DNA zum<br />
Nucleosom von der in einer bestimmten Richtung bevorzugten Krümmung der DNA abhängt.<br />
Abschließend ist zu sagen, dass aber auch Sequenzen, welche <strong>die</strong> Bildung ungewöhnlicher DNA-<br />
Strukturen hervorrufen können, zum Nucleosomen-Ausschluss führen können.<br />
4) Der <strong>Einfluss</strong> von Nucleosomen <strong>auf</strong> <strong>die</strong> Transkription<br />
Zahlreiche Beobachtungen sprechen für eine Beteiligung der Chromatin-Struktur an der Regulation gene-<br />
tischer Aktivität. So liegen etwa aktive Gene meist im Bereich des lockeren Euchromatins, während<br />
stumme Gene im dicht gepackten Heterochromatin vorkommen.<br />
4.1.) DNase-1-sensitives Chromatin <strong>und</strong> DNase-1-hypersensitive Stellen<br />
Während der größte Teil des Chromatins nichtexprimierte Gene (sowie andere Sequenzen) enthält, <strong>und</strong><br />
sich als relativ widerstandsfähig gegenüber DNase I zeigt, werden Gene spezifisch in denjenigen Gewe-<br />
ben gegenüber DNase I empfindlicher, in denen sie exprimiert werden.<br />
14
Mit Hilfe des Enzyms DNasel (schneidet sowohl ssDNA wie dsDNA) hat man eine bis zu zehnmal höhe-<br />
re Empfindlichkeit von transkribierten Chromatin-Abschnitten im Vergleich zu benachbart gelegenen,<br />
genetisch stummen, Abschnitten feststellen können, was eindeutig für eine <strong>auf</strong>gelockerte Struktur von<br />
transkribiertem, also genetisch aktivem Chromatin spricht.<br />
Neben <strong>die</strong>ser allgemeinen <strong>und</strong> relativ gering erhöhten Nuclease-Sensitivität von aktiven Genen, gibt es<br />
Stellen im Chromatin, <strong>die</strong> gegenüber einem Angriff von DNase I h<strong>und</strong>ert- bis tausendmal empfindlicher<br />
sind als gewöhnliches Chromatin; man spricht dann von DNase-I-hypersensitiven Stellen (DHS). Diese<br />
Stellen liegen fast auschließlich im Bereich der Promotoren <strong>und</strong> Enhancer aktiver Gene, wobei <strong>die</strong> Aus-<br />
bildung einer DHS durch das Ablösen oder Verdrängen von Nucleosomen erfolgt (vgl. auch Abb.23 Ver-<br />
änderungen der Chromatin-Struktur duch (De)Acetylierung), wobei Histon-Acetyl-Transfeasen wahr-<br />
scheinlich eine entscheidende Rolle spielen.<br />
4.2) Translationale <strong>und</strong> Rotations Positionierung der DNA am Nucleosom<br />
Die Positionierung der DNA <strong>auf</strong> den Nucleosomen lässt sich <strong>auf</strong> zwei Weisen beschreiben.<br />
• Die Position der DNA im Verhältnis zu den Nucleosomengrenzlinien wird als translationale Positi-<br />
onierung beschrieben. Dadurch wird festgelegt, welche Sequenzen in den Linker-Regionen zu lie-<br />
gen kommen, <strong>und</strong> welche direkt am Nucleosom.<br />
• Da <strong>die</strong> DNA an der Außenseite des Histonoktamers liegt, bleibt nach wie vor eine Seite frei zu-<br />
gänglich, während <strong>die</strong> andere vom Histonoktamer bedeckt wird. Somit kann eine DNA-Sequenz,<br />
abhängig von <strong>ihr</strong>er Position relativ zum Nucleosom, für Regulatorproteine zugänglich oder unzu-<br />
gänglich sein. Durch Verschieben der DNA um eine begrenzte Anzahl von Windungen (d.h. <strong>die</strong><br />
DNA rotiert relativ zur Proteinoberfläche) gelangen unterschiedliche Sequenzen an <strong>die</strong> Außenseite<br />
(Rotationspositionierung).<br />
Sowohl <strong>die</strong> translationale als auch <strong>die</strong> Rotationspositionierung können somit den Zugang zur DNA steu-<br />
ern, da einige Regulatoren z.B. nur an nucleosomenfreie DNA binden könne (translationale Positionie-<br />
rung entscheidend), andere zwar an in Nucleosomen verpackte DNA, aber nur, wenn <strong>die</strong> betreffende Se-<br />
quenz an der Nucleosomenoberfläche zu liegen kommt (richtige Rotationspositionierung notwendig).<br />
15
5) Histonmodifikationen <strong>und</strong> Ihr <strong>Einfluss</strong> <strong>auf</strong> <strong>die</strong> Transkription<br />
Einen entscheidenden <strong>Einfluss</strong> <strong>auf</strong> <strong>die</strong> Transkription haben Histonmodifikationen:<br />
Alle <strong>Histone</strong> werden im Gr<strong>und</strong>e posttranslational modifiziert, indem an <strong>die</strong> freien Reste bestimmter Ami-<br />
nosäuren zusätzliche chemische Gruppen kovalent geb<strong>und</strong>en werden. Zu einer Acetylierung <strong>und</strong> Methy-<br />
lierung kommt es an den freien (ε-)Aminogruppen des Lysins; dabei wird <strong>die</strong> positive Ladung der NH3 + -<br />
Gruppe beseitigt. An Arginin <strong>und</strong> Histidin kommt es ebenfalls zu einer Methylierung. Die Phosphorylie-<br />
rung der Hydroxylgruppen von Serin <strong>und</strong> Threonin, <strong>und</strong> ebenso <strong>die</strong> Phosphorylierung von Histidin er-<br />
zeugt jeweils eine negative Ladung in Form einer Phosphatgruppe. Weitere Histonmodifikationen sind<br />
<strong>die</strong> ADP-ribosylierung <strong>und</strong> <strong>die</strong> Ubiquitinilierung.<br />
Abb.19<br />
Strukturformel Serin<br />
Abb.18 Strukturformel<br />
Lysin<br />
Abb.21 Strukturformel<br />
Threonin<br />
Abb.20 Strukturformel<br />
Arginin<br />
Abb.22 Strukturformel<br />
Histidin<br />
Diese Modifikationen der aminoterminalen Domänen der <strong>Histone</strong> sind in der Regel temporär. Die Varia-<br />
tion der Ladung, der Fähigkeit zur Ausbildung von Wasserstoffbrücken <strong>und</strong> der Gestalt der <strong>Histone</strong> durch<br />
kovalente Modifikationen ist wichtig für <strong>die</strong> Packungsform der DNA, <strong>und</strong> somit für <strong>die</strong> Regulation <strong>ihr</strong>er<br />
Verfügbarkeit, sowohl für Replikation, wie Transkription.<br />
5.1) Acetylierung<br />
Acetylierte <strong>Histone</strong> findet man bevorzugt in aktiv transkribiertem Chromatin, <strong>und</strong> es ist sehr wahrschein-<br />
lich, dass <strong>die</strong> Acetylierung von <strong>Histone</strong>n eine Voraussetzung für <strong>die</strong> Aktivierung vieler Gene ist. Die<br />
Acetylierung von bis zu vier Lysin-Resten in den <strong>Histone</strong>n H2A, H2B, H3 <strong>und</strong> H4 wird von Histon-<br />
Acetyl-Transferasen bewerkstelligt; entfernt werden <strong>die</strong> Acetylgruppen wieder von Histon-Deacetylasen.<br />
Bereits 1964 stellte Vincent Allfrey, Rockefeller University, New York, als erster fest, dass <strong>die</strong> <strong>Histone</strong><br />
H3 <strong>und</strong> H4, aber auch <strong>die</strong> beiden anderen Core-<strong>Histone</strong> in genetisch aktiven Chromatin-Bereichen acety-<br />
liert sind (Allfrey et al. 1964), aber <strong>die</strong> große Bedeutung der Acetylierung bezüglich der Gen-Aktivierung<br />
wurde erst r<strong>und</strong> 30 Jahre später klar.<br />
16
17<br />
Welche Beziehung besteht nun aber zwischen der Acetylierung von <strong>Histone</strong>n <strong>und</strong> der Auflockerung<br />
der Chromatin-Struktur? Durch Acetylierung einer positiv geladenen Lysin-Seitengruppe, wird <strong>die</strong>se<br />
durch eine negativ geladenen Acetyl-Gruppe ersetzt, was sich in Folge <strong>auf</strong> <strong>die</strong> Bindungseigenschaften der<br />
flexiblen Domäne auswirkt, <strong>die</strong> unteranderem zur Kontakt<strong>auf</strong>nahme mit Nachbar-Nucleosomen, <strong>und</strong> so-<br />
mit an der Aufrecherhaltung einer übergeordneten Chromatin-Struktur (der 30nm Faser) beiträgt. Da-<br />
durch könnten <strong>die</strong> Kontakte zwischen benachbart gelegenen Nucleosomen <strong>auf</strong>gelöst werden, <strong>und</strong> es käme<br />
zu Veränderungen in der Chromatinstruktur.<br />
Abb.23 Veränderungen der Chromatin-Struktur<br />
duch (De)Acetylierung. Die Übertragung von Acetylgruppen<br />
(AC) <strong>auf</strong> <strong>Histone</strong> verursacht eine allgemeine<br />
Auflockerung des Chromatins, was sich unter<br />
anderem in einer höheren Empfindlichkeit gegenüber<br />
DNase I äußert. Chromatin-Remodeling ändert <strong>die</strong><br />
Lage oder Struktur einzelner Nucleosomen direkt im<br />
Strom<strong>auf</strong>wärtsbereich vor den Genen <strong>und</strong> führt zur<br />
Einrichtung von DNase-I-hypersensitiven Stellen.<br />
(Knippers S.389)<br />
Die obige Abbildung kann jedoch nur als ein Modell <strong>und</strong> eine Vereinfachung komplizierter Verhältnisse<br />
<strong>die</strong>nen, denn zur Zeit ist noch nicht klar, welche Konsequenzen Acetylierung an den unterschiedlichen<br />
Lysin-Resten der vier Core-<strong>Histone</strong>n haben. Sicher ist jedoch, dass acetyliertes Chromatin gegenüber<br />
DNase I <strong>und</strong> Micrococcus-Nuclease empfindlicher ist, <strong>und</strong> Acetylierung mit ähnlichen Veränderungen<br />
des Chromatins verb<strong>und</strong>en ist, wie sie auch bei der Genaktivierung <strong>auf</strong>treten.<br />
Die <strong>auf</strong>fälligste Veränderung der Histonmodifikationen ist <strong>die</strong> Unteracetylierung des Histons H4 im inak-<br />
tiven X-Chromosom weiblicher Säugern, was dar<strong>auf</strong> hindeutet, dass das Fehlen von Acetylgruppen für<br />
eine stärker verdichtete Struktur notwendig ist.<br />
5.2) Phosphorylierung<br />
Protein-Kinasen übertragen Phosphat-Gruppen z.B. <strong>auf</strong> Serin-Seitenketten der <strong>Histone</strong> H3 <strong>und</strong> H2A, oft<br />
als Reaktion <strong>auf</strong> Signale, <strong>die</strong> eine Zelle von außen empfängt. Besonders gut untersucht sind <strong>die</strong> Phospho-<br />
rylierungen des Histons Hl: H1 unterliegt einem Zyklus aus Phosphorylierung <strong>und</strong> Dephosphorylierung,
18<br />
jedoch unterscheidet sich <strong>die</strong> zeitliche Abstimmung vom Modifikationszyklus der übrigen <strong>Histone</strong>. Bei<br />
Säugerzellen in Kultur werden während der S-Phase eine oder zwei Phosphatgruppen eingeführt. Die<br />
wichtigste Phosphorylierungsreaktion ist jedoch das spätere Hinzufügen weiterer Phosphat-Gruppen wäh-<br />
rend der Mitose, sodass schließlich bis zu sechs Phosphatgruppen an der N-terminalen Domäne von H1<br />
vorhanden sind, <strong>die</strong> aber alle wieder am Ende der Zellteilung von Protein-Phosphatasen entfernt werden.<br />
Die M-Phasen-Kinase katalysiert <strong>die</strong> Phosphorylierung von Hl <strong>und</strong> bildet dadurch für <strong>die</strong> Mitose einen<br />
wichtigen Schalter.<br />
5.3) Methylierung<br />
Bei <strong>die</strong>ser Modifikation kommt es zu einer vermutlich nicht reversiblen Übertragung von Methyl-<br />
Gruppen <strong>auf</strong> <strong>die</strong> Amino-Gruppe in der Seitenkette von Lysinen <strong>und</strong> / oder Argininen der <strong>Histone</strong> H3 <strong>und</strong><br />
H4. Während sich Acetylierung <strong>und</strong> Phosphorylierung bestimmten genetischen Vorgängen zuordnen las-<br />
sen, ist <strong>die</strong> Funktion der Histon-Methylierung zur Zeit noch weitgehend ungeklärt; hauptsächlich deshalb,<br />
weil <strong>die</strong> zugehörigen Enzyme noch nicht bekannt sind. Außerdem kommt es bei Methylierungen im Ge-<br />
gensatz zu Acetylierungen <strong>und</strong> Phosphorylierungen zu keiner Änderung der Ladung an den Aminosäure-<br />
Seitenketten, wodurch eine elektrophoretische Auftrennung von methylierten <strong>und</strong> unmethylierten Histo-<br />
nen erschwert wird. Darüber hinaus können z.B. Lysin-Seitenketten mono-, di- oder trimethyliert werden,<br />
wodurch eine hohe Varibilität erreicht wird (Strahl and Allis 2000). Wahrscheinlich haben aber methy-<br />
lierte Lysinreste einen <strong>Einfluss</strong> <strong>auf</strong> <strong>die</strong> Phosphorylierung <strong>und</strong> Acetylierung anderer Aminosäuren (Chen<br />
et al. 1999).<br />
5.4) Der <strong>Histone</strong> Code<br />
Erst 2000 haben BD Strahl <strong>und</strong> CD Allis noch einmal <strong>auf</strong> den Punkt gebracht, dass Modifikationen von<br />
<strong>Histone</strong>n vielfältiger <strong>und</strong> komplexer sind, als oftmals angenommen, auch wenn sie nur <strong>auf</strong> dem begrenz-<br />
ten Raum der flexiblen aminoterminalen Domänen abspielen. Man betrachte dazu nur <strong>die</strong> simple Folge<br />
der Aminosäuren in den flexiblen Domänen der <strong>Histone</strong> H3 <strong>und</strong> H4 in Abb.24.<br />
Unter gegebenen physiologischen Bedingungen könnten der eine, unter anderen Bedingungen der andere<br />
oder beide oder schließlich <strong>die</strong> meisten Lysin-Reste acetyliert werden. Oder Bedingungen lösen <strong>die</strong> Ace-<br />
tylierung eines Lysin-Restes oder zweier oder dreier Lysin-Reste aus <strong>und</strong> dazu noch <strong>die</strong> Methylierung<br />
eines anderen Lysins <strong>und</strong> <strong>die</strong> Phosphorylierung eines Serins. Möglichkeiten für Variationen gibt es in
19<br />
unüberschaubar großer Zahl, <strong>und</strong> <strong>die</strong>se wird noch größer, wenn auch Veränderungen an den <strong>Histone</strong>n<br />
H2A <strong>und</strong> H2B in <strong>die</strong> Betrachtung einbezogen werden.<br />
Abb. Der N-terminus von humanem H3 <strong>und</strong> H4 im Einbuchstabencode. Die Roten Fahnen kennzeichnen Lysine,<br />
<strong>die</strong> acetyliert werden können. Sechsecke sind Stellen, <strong>die</strong> durch Histon-Methyl-Transferasen methyliert werden<br />
können, <strong>und</strong> Kreise bezeichnen Serinreste, <strong>die</strong> phosphoryliert werden können. Der Lysinrest 9 im Histon H3 kann<br />
sowohl acetyliert wie methyliert werden (Strahl and Allis 2000)<br />
Das Spektrum der möglichen Histon-Modifikationen ist ein Code, ein epigenetisches Markierungssys-<br />
tem, das zurzeit in seiner Komplexität noch nicht entschlüsselt werden kann, welches aber ein f<strong>und</strong>amen-<br />
tales Regulationssystem darstellt, <strong>und</strong> wahrscheinlich eine große Bedeutung bei der Umsetzung von Sig-<br />
nalen in genetische Aktivität, <strong>und</strong> somit <strong>auf</strong> das gesamte Zellschicksal hat (Jenuwein and Allis 2001).
6) Literaturliste<br />
6.1) Verwendete Lehrbücher<br />
Brown TA. Genomes, 2nd Edition. BIOS Scientific Publishers Ltd 2002<br />
Carey M, Smale ST. Transcriptional Regulation in Eukaryotes: Concepts, Strategies, and Techniques.<br />
Cold Spring Harbor Laboratory Press, Cold Spring Harbor, New York 2000<br />
Knippers R. Molekulare Genetik, 8. Auflage. Thieme Verlag Stuttgard New York 2001<br />
Lewin B. Genes VII. Oxford University Press & Cell Press 2000<br />
Stryer L. Biochemie, 4. Auflage. Spektrum Akademischer Verlag Heidelberg Berlin Oxford 1996<br />
Weinzierl ROJ. Mechanisms of Gene Expression. Imperial College Press 1999<br />
6.2.) Verwendete Papers<br />
Allfrey VG, Faulkner R, Mirsky AE (1964). Acetylation and methylation of histones and their possible<br />
role in the regulation of RNA synthesis. Proc Natl Acad Sci USA 51, 786-94.<br />
Chen D, Ma H, Hong H, Koh SS, Huang SM, Schurter BT, Aswad DW, Stallcup MR (1999). Regulation<br />
of transcription by a protein methyltransferase. Science 284, 2174-2177.<br />
Finch JT, Lutter LC, Rhodes D, Brown RS, Rushton B, Levitt M, Klug A (1977). Structure of nucleoso-<br />
me core particles of chromatin. Nature 269, 29-36.<br />
Jenuwein T and Allis CD (2001). Translating the <strong>Histone</strong> Code. Science 293, 1074-1080.<br />
Luger K, Mäder AW, Richmond RK, Sargent DF, Richmond TJ (1997). Crystal structure of the<br />
nucleosome core partikle at 2,8Å Resolution. Nature 389, 251-260.<br />
Kornberg RD (1974). Chromatin structure: a repeating unit of histones and DNA.<br />
Science 184, 868-871.<br />
Strahl BD and Allis CD (2000). The language of covalent histone modifications.<br />
Nature 403, 41-45.<br />
Thoma F, Koller TH, Klug A (1979). Involvement of <strong>Histone</strong> H1 in the Organisation of the Nucleosome<br />
and of the salt-dependent superstructures of Chromatin. J Cell Biol 83, 403-327.<br />
20