Caracterización Molecular de Razas Patogénicas de Fusarium ...
Caracterización Molecular de Razas Patogénicas de Fusarium ...
Caracterización Molecular de Razas Patogénicas de Fusarium ...
Create successful ePaper yourself
Turn your PDF publications into a flip-book with our unique Google optimized e-Paper software.
Universidad <strong>de</strong> Córdoba<br />
Instituto <strong>de</strong> Agricultura Sostenible, CSIC<br />
<strong>Caracterización</strong> <strong>Molecular</strong> <strong>de</strong> <strong>Razas</strong><br />
<strong>Patogénicas</strong> <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris<br />
y Análisis <strong>de</strong> su Diversidad Genética<br />
Doctorando: María <strong>de</strong>l Mar Jiménez Gasco<br />
Directores: Prof. Rafael M. Jiménez Díaz<br />
Dra. Encarnación Pérez Artés<br />
Córdoba, Diciembre <strong>de</strong> 2001
Universidad <strong>de</strong> Córdoba<br />
Instituto <strong>de</strong> Agricultura Sostenible, CSIC<br />
<strong>Caracterización</strong> <strong>Molecular</strong> <strong>de</strong> <strong>Razas</strong><br />
<strong>Patogénicas</strong> <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris<br />
y Análisis <strong>de</strong> su Diversidad Genética<br />
Memoria presentada para optar al Grado <strong>de</strong> Doctor por la<br />
Universidad <strong>de</strong> Córdoba, por la Ingeniero Agrónomo:<br />
María <strong>de</strong>l Mar Jiménez Gasco<br />
V. B:<br />
Los Directores <strong>de</strong> la Tesis<br />
Prof. Rafael M. Jiménez Díaz Dra. Encarnación Pérez Artés<br />
Catedrático <strong>de</strong> Patología Vegetal Científico Titular<br />
Universidad <strong>de</strong> Córdoba IAS-CSIC<br />
Córdoba, 2001
D. Rafael M. Jiménez Díaz, Catedrático <strong>de</strong> Patología Vegetal <strong>de</strong> la<br />
Escuela Técnica Superior <strong>de</strong> Ingenieros Agrónomos y <strong>de</strong> Montes <strong>de</strong> la<br />
Universidad <strong>de</strong> Córdoba, y Dña. Encarnación Pérez Artés, Científico<br />
Titular <strong>de</strong>l Instituto <strong>de</strong> Agricultura Sostenible <strong>de</strong>l CSIC;<br />
Co-directores <strong>de</strong> la Tesis Doctoral titulada ‘<strong>Caracterización</strong> molecular<br />
<strong>de</strong> razas patogénicas <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris y análisis<br />
<strong>de</strong> su diversidad genética’, realizada por al Ingeniero Agrónomo Dña.<br />
María <strong>de</strong>l Mar Jiménez Gasco,<br />
Certifican: Que la mencionada Tesis Doctoral ha sido realizada bajo<br />
nuestra dirección en los laboratorios e instalaciones <strong>de</strong>l<br />
Instituto <strong>de</strong> Agricultura Sostenible, y se encuentra finalizada<br />
para ser presentada y <strong>de</strong>fendida públicamente en la<br />
Universidad <strong>de</strong> Córdoba.<br />
Córdoba, 16 <strong>de</strong> Octubre <strong>de</strong> 2001<br />
Prof. Rafael M. Jiménez Díaz Dra. Encarnación Pérez Artés<br />
Catedrático <strong>de</strong> Patología Vegetal Científico Titular, IAS-CSIC
D. Luis López Bellido, Catedrático <strong>de</strong> Producción Vegetal y Director <strong>de</strong>l<br />
Departamento <strong>de</strong> Ciencias y Recursos Agrícolas y Forestales <strong>de</strong> la<br />
Universidad <strong>de</strong> Córdoba,<br />
Informa: Que el Consejo <strong>de</strong> Departamento, en su reunión <strong>de</strong>l 17 <strong>de</strong><br />
Octubre <strong>de</strong> 2001 ha acordado que la Tesis Doctoral presentada<br />
por Dña. María <strong>de</strong>l Mar Jiménez Gasco, con el título<br />
‘<strong>Caracterización</strong> molecular <strong>de</strong> razas patogénicas <strong>de</strong><br />
<strong>Fusarium</strong> oxysporum f. sp. ciceris y análisis <strong>de</strong> su<br />
diversidad genética’ y realizada bajo la dirección <strong>de</strong> D. Rafael<br />
M. Jiménez Díaz y Dña. Encarnación Pérez Artés, reúne las<br />
características necesarias para su lectura y <strong>de</strong>fensa.<br />
Córdoba, 17 <strong>de</strong> Octubre <strong>de</strong> 2001<br />
D. Luis López Bellido<br />
Catedrático <strong>de</strong> Producción Vegetal
Este Trabajo ha sido realizado en el Instituto <strong>de</strong> Agricultura Sostenible,<br />
Córdoba, gracias a la concesión <strong>de</strong> una Beca <strong>de</strong> Formación <strong>de</strong> Personal<br />
Investigador (FPI-MEC), y ha sido financiado por los Proyectos <strong>de</strong><br />
Investigación AGRE-CT91-0051, CICYT OLI96-2131 y CICYT AGF98-<br />
0872.
Biografía<br />
María <strong>de</strong>l Mar Jiménez Gasco nació en Logroño, La Rioja, en 1972.<br />
En Septiembre <strong>de</strong> 1990 inició sus estudios <strong>de</strong> Ingeniero Agrónomo en<br />
la Escuela Técnica Superior <strong>de</strong> Ingenieros Agrónomos y <strong>de</strong> Montes <strong>de</strong> la<br />
Universidad <strong>de</strong> Córdoba, obteniendo su Título en Mayo <strong>de</strong> 1997 al<br />
presentar el Trabajo Profesional Fin <strong>de</strong> Carrera titulado ‘<strong>Caracterización</strong> <strong>de</strong><br />
razas <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris mediante RAPD-PCR’, con la<br />
calificación máxima <strong>de</strong> Sobresaliente.<br />
En al Curso Académico 1993/94 realizó una estancia <strong>de</strong> seis meses<br />
como estudiante ERASMUS en la Universidad <strong>de</strong> Wageningen, Holanda,<br />
durante la cual participó en Investigaciones en el Departamento <strong>de</strong><br />
Nematología. Durante 1996 disfrutó <strong>de</strong> una Beca <strong>de</strong> ‘Introducción a la<br />
Investigación’ concedida por el Consejo Superior <strong>de</strong> Investigaciones<br />
Científicas, a <strong>de</strong>sarrollar en el Instituto <strong>de</strong> Agricultura Sostenible en<br />
Córdoba.<br />
Des<strong>de</strong> Septiembre <strong>de</strong> 1997 a Septiembre <strong>de</strong> 2001 ha disfrutado <strong>de</strong> una<br />
Beca <strong>de</strong> Formación <strong>de</strong> Personal Investigador (FPI-MEC) en el<br />
Departamento <strong>de</strong> Protección <strong>de</strong> Cultivos <strong>de</strong>l Instituto <strong>de</strong> Agricultura<br />
Sostenible. Las Investigaciones realizadas durante estos años han dado<br />
lugar a participación en Congresos nacionales e internacionales, así como a<br />
dos publicaciones en revistas incluidas en SCI (‘European Journal of Plant<br />
Pathology’, y ‘Plant Pathology’). A<strong>de</strong>más, durante estos años ha realizado<br />
varias estancias en Centros <strong>de</strong> Investigación extranjeros (1997, King’s<br />
College of London, Universidad <strong>de</strong> Londres; 1998, 1999, y 2000,<br />
Departamento <strong>de</strong> Patología Vegetal <strong>de</strong> la Universidad <strong>de</strong> Cornell, NY,<br />
EEUU).
A mi Madre, a mi Familia, a Vagelis
Agra<strong>de</strong>cimientos<br />
Quiero expresar mi más sincero agra<strong>de</strong>cimiento a los Directores <strong>de</strong> esta<br />
Tesis Doctoral por su inestimable ayuda en el <strong>de</strong>sarrollo experimental <strong>de</strong> la<br />
Investigación, así como durante la realización <strong>de</strong> este documento.<br />
Gracias a mis compañeros <strong>de</strong>l Departamento <strong>de</strong> Protección <strong>de</strong> Cultivos<br />
<strong>de</strong>l IAS, en especial a Susana Carrasco, Pablo Castillo, Juan Antonio<br />
Navas, y Blanca B. Landa, por estar siempre dispuestos a ayudarme, por<br />
sus consejos en todo momento, por las charlas <strong>de</strong>l café.<br />
Gracias al Prof. Michael G. Milgroom por acogerme en su laboratorio<br />
<strong>de</strong>l Departamento <strong>de</strong> Patología Vegetal <strong>de</strong> la Universidad <strong>de</strong> Cornell, por<br />
haber sido como un Director más, por las charlas científicas <strong>de</strong> los viernes<br />
por la tar<strong>de</strong> que tanto me han servido, por su entusiasmo y por<br />
escucharme. Gracias a tantos amigos <strong>de</strong> allí, en especial a Isabel Cristina<br />
McGuire, por mostrarme la preciosa Ithaca y por tantos consejos.<br />
Gracias a mis compañeros <strong>de</strong> laboratorio, en especial a Susana<br />
Carrasco, Gema Contreras y Antonio Valver<strong>de</strong>, por las risas, por su apoyo<br />
técnico y por haberme prestado esa tercera mano en tantos momentos.<br />
Gracias a todos mis amigos, en especial a Naty y a Carmen, por intentar<br />
sacarme <strong>de</strong>l laboratorio a una hora razonable.<br />
Gracias a Rocío, mi hermana, por mostrarme siempre tanto amor, y<br />
ayudarme tanto con la bibliografía...., a Rafa, a Rodrigo, y a toda mi familia,<br />
en especial a mi madre.<br />
Gracias a la persona que más confía en mí, que más me ha guiado, me<br />
ha enseñado, más entusiasmo me ha transmitido, mi Director, mi<br />
maestro....mi padre.<br />
And last but not least, thank you Vagelis, for helping me out with my<br />
poor self-confi<strong>de</strong>nce, and for always telling me that I am ‘the best’.
Resumen<br />
Resumen<br />
La Fusariosis Vascular <strong>de</strong>l garbanzo (Cicer arietinum L.), una <strong>de</strong> las<br />
enfermeda<strong>de</strong>s más importantes y limitantes <strong>de</strong> este cultivo en el mundo, es<br />
causada por <strong>Fusarium</strong> oxysporum Schlechtend.: Fr. f. sp. ciceris (Padwick)<br />
Matuo & K. Sato, un hongo anamórfico capaz <strong>de</strong> persistir<br />
prolongadamente en el suelo. El método más práctico y económico <strong>de</strong><br />
lucha contra esta enfermedad es la utilización <strong>de</strong> cultivares <strong>de</strong> garbanzo<br />
resistentes. Sin embargo, la eficiencia <strong>de</strong> éstos es amenazada por la<br />
existencia <strong>de</strong> razas patogénicas <strong>de</strong>l agente (razas 0, 1A, 1B/C, 2, 3, 4, 5, y<br />
6), que constituyen dos patotipos, <strong>de</strong>nominados Amarillez (razas 0 y 1B/C)<br />
y Marchitez (razas 1A a 6) en razón <strong>de</strong>l síndrome que causan en la planta<br />
huésped. La utilización eficiente <strong>de</strong> cultivares resistentes requiere<br />
información previa y precisa sobre las razas <strong>de</strong>l patógeno que prevalecen<br />
en las distintas áreas <strong>de</strong> cultivo, cuya i<strong>de</strong>ntificación y caracterización<br />
mediante pruebas <strong>de</strong> patogenicidad es laboriosa y costosa en material,<br />
espacio, y tiempo, y no es informativa sobre la relación y diversidad<br />
genética existente entre y <strong>de</strong>ntro <strong>de</strong> las razas <strong>de</strong> F. oxysporum f. sp. ciceris.<br />
Todo ello hace necesario que se investiguen procedimientos nuevos y<br />
alternativos basadas en las técnicas moleculares disponibles, para la<br />
<strong>de</strong>tección e i<strong>de</strong>ntificación rápida, precisa, consistente y fiable <strong>de</strong>l patógeno<br />
y <strong>de</strong> sus razas patogénicas. Los objetivos <strong>de</strong> esta Tesis Doctoral han sido<br />
<strong>de</strong>sarrollar un método, basado en secuencias <strong>de</strong> ADN polimórfico, que<br />
permita la i<strong>de</strong>ntificación <strong>de</strong> F. oxysporum f. sp. ciceris y <strong>de</strong> sus razas<br />
patogénicas mediante PCR específica, y el análisis <strong>de</strong> la variabilidad<br />
genética y <strong>de</strong> la relación evolutiva existente entre las distintas razas <strong>de</strong>l<br />
patógeno.<br />
La disponibilidad <strong>de</strong> marcadores moleculares <strong>de</strong> las razas <strong>de</strong> F.<br />
oxysporum f. sp. ciceris basados en ADN polimórfico se investigó mediante<br />
análisis RAPD-PCR <strong>de</strong> 99 aislados <strong>de</strong> F. oxysporum f. sp. ciceris<br />
representativos <strong>de</strong> las diferentes razas y orígenes geográficos <strong>de</strong>l patógeno.<br />
i
Resumen<br />
De un total <strong>de</strong> 40 iniciadores <strong>de</strong> secuencia arbitraria estudiados, cinco<br />
(OPI-09, OPI-18, OPF-06, OPF-10, y OPF-12) amplificaron fragmentos<br />
RAPD asociados a F. oxysporum f. sp. ciceris, así como a las razas 0, 1B/C, 5,<br />
y 6 <strong>de</strong>l patógeno. La aplicabilidad, reproducibilidad, consistencia y<br />
fiabilidad diagnóstica <strong>de</strong> dichos fragmentos <strong>de</strong> ADN fue confirmada<br />
mediante ‘experimentos ciegos’ <strong>de</strong> naturaleza biológica y molecular. El<br />
análisis UPGMA <strong>de</strong> los perfiles RAPD mostró que existe una alta<br />
similaridad genética entre los aislados <strong>de</strong> F. oxysporum f. sp. ciceris, y agrupó<br />
a éstos en ‘clusters’ que estuvieron correlacionados con el patotipo <strong>de</strong><br />
Amarillez o <strong>de</strong> Marchitez a la que pertenecen. Los aislados <strong>de</strong>l patotipo <strong>de</strong><br />
Amarillez constituyeron un grupo distintivo que incluye a los aislados <strong>de</strong><br />
las razas 0 y 1B/C; mientras que los aislados <strong>de</strong> Marchitez se ubicaron en<br />
otro gran ‘cluster’ que agrupa a los aislados <strong>de</strong> las razas 1A, 2, 3, 4, 5, y 6.<br />
Posteriormente, a partir <strong>de</strong> secuencias SCAR (‘Sequence Characterized<br />
Amplified Region’) proce<strong>de</strong>ntes <strong>de</strong> los fragmentos RAPD marcadores<br />
raciales inicialmente i<strong>de</strong>ntificados, se diseñaron iniciadores para PCR<br />
específica. Para ello, dichos fragmentos RAPD se clonaron y secuenciaron,<br />
y <strong>de</strong> las secuencias extremas <strong>de</strong> los fragmentos clonados se diseñaron los<br />
iniciadores <strong>de</strong> forma que contuviesen parte <strong>de</strong>l iniciador aleatorio RAPD<br />
que los originó. Estos iniciadores se utilizaron para amplificar ADN <strong>de</strong><br />
aislados representativos <strong>de</strong> todas las razas patogénicas <strong>de</strong> F. oxysporum f. sp.<br />
ciceris y <strong>de</strong> un amplio rango geográfico <strong>de</strong>l patógeno. De esta manera, se<br />
han diseñado cinco parejas <strong>de</strong> iniciadores para reacciones PCR específicas<br />
que permiten discriminar inequívocamente a aislados <strong>de</strong> F. oxysporum f. sp.<br />
ciceris <strong>de</strong>: (a) aislados <strong>de</strong> F. oxysporum no patogénicos <strong>de</strong> garbanzo; (b)<br />
aislados pertenecientes a otras formae speciales <strong>de</strong> F. oxysporum; y (c) otras<br />
especies <strong>de</strong> <strong>Fusarium</strong>. A<strong>de</strong>más, mediante análisis PCR específica utilizando<br />
los iniciadores ‘ad hoc’ ha sido posible diferenciar entre sí a los aislados <strong>de</strong><br />
cada una <strong>de</strong> las razas 0, 1A, 5, y 6 <strong>de</strong> F. oxysporum f. sp. ciceris, así como a<br />
éstos <strong>de</strong> otros aislados pertenecientes a otras razas <strong>de</strong>l patógeno.<br />
La diversidad fenotípica y geográfica observada en el patógeno condujo<br />
a plantear la hipótesis nula <strong>de</strong> un origen monofilético en F. oxysporum f. sp.<br />
ciceris. Dicha hipótesis se contrastó mediante la extensión y naturaleza <strong>de</strong> la<br />
variación existente en las secuencias <strong>de</strong> intrones <strong>de</strong> varios genes<br />
ii
Resumen<br />
conservados [factor <strong>de</strong> traducción y elongación 1α (EF1α), β-tubulina,<br />
histona 3, actina, y calmodulina] en aislados representativos <strong>de</strong> seis, 0, 1A,<br />
1B/C, 4, 5, y 6, <strong>de</strong> las ocho razas patogénicas <strong>de</strong>scritas. Los resultados<br />
mostraron que a pesar <strong>de</strong> la diversidad entre los aislados <strong>de</strong> este patógeno<br />
en cuanto su distribución geográfica, síndrome que inducen en la planta<br />
susceptible, y la patogenicidad cultivar-específica, todos poseen secuencias<br />
idénticas en los intrones <strong>de</strong> los cinco genes analizados. Adicionalmente, se<br />
<strong>de</strong>sarrollaron secuencias <strong>de</strong>l gen EF1α <strong>de</strong> un total <strong>de</strong> 17 aislados <strong>de</strong> F.<br />
oxysporum f. sp. ciceris, y <strong>de</strong> tres aislados <strong>de</strong> F. oxysporum no patogénicos <strong>de</strong><br />
garbanzo. El análisis PAUP (‘Phylogenetic Analysis Using Parsimony’) <strong>de</strong><br />
dichas secuencias y <strong>de</strong> las <strong>de</strong> 24 aislados representativos <strong>de</strong> otras 11 formae<br />
speciales <strong>de</strong> F. oxysporum obtenidas <strong>de</strong> GenBank, agrupó a los aislados <strong>de</strong> F.<br />
oxysporum f. sp. ciceris <strong>de</strong> forma distintiva respecto al resto <strong>de</strong> los aislados,<br />
<strong>de</strong>mostrando así su origen monofilético.<br />
La variabilidad genética entre aislados <strong>de</strong> una misma raza, y entre<br />
distintas razas <strong>de</strong> F. oxysporum f. sp. ciceris, así como la posible relación<br />
evolutiva entre ellas, se investigó mediante análisis <strong>de</strong> ‘fingerprints’ <strong>de</strong><br />
ADN generados con sondas <strong>de</strong> hibridación (FocB10, FocO2 y FocP18).<br />
Estas sondas se componen <strong>de</strong> ADN repetitivo i<strong>de</strong>ntificado en el genoma<br />
<strong>de</strong> F. oxysporum f. sp. ciceris en el curso <strong>de</strong> esta Tesis, y sus secuencias <strong>de</strong><br />
ADN presentan similaridad con elementos transponibles citados en la<br />
literatura fitopatológica. En particular, la secuencia <strong>de</strong> FocB10 presentó un<br />
80% <strong>de</strong> similaridad con el transposón Fot1 i<strong>de</strong>ntificado en F. oxysporum f.<br />
sp. melonis. El análisis <strong>de</strong> ‘fingerprints’ <strong>de</strong> ADN ha puesto <strong>de</strong> manifiesto<br />
que los aislados <strong>de</strong> F. oxysporum f. sp. ciceris presentan perfiles altamente<br />
correlacionados tanto con el síndrome que inducen en la planta huésped (i.<br />
e., patotipos <strong>de</strong> Amarillez y <strong>de</strong> Marchitez), como con su patogenicidad<br />
diferencial sobre líneas diferenciadoras <strong>de</strong> garbanzo (i. e., razas<br />
patogénicas). Tanto el análisis fenético <strong>de</strong> la similaridad (UPGMA) entre<br />
‘fingerprints’ <strong>de</strong> ADN, como su análisis cladístico (PAUP), distribuyeron a<br />
los aislados <strong>de</strong> F. oxysporum f. sp. ciceris en dos grupos que correspon<strong>de</strong>n,<br />
respectivamente, a los patotipos <strong>de</strong> Amarillez y <strong>de</strong> Marchitez. A<strong>de</strong>más, el<br />
grupo correspondiente al patotipo <strong>de</strong> Amarillez se compone <strong>de</strong> dos<br />
subgrupos en los que se incluyen los aislados <strong>de</strong> las razas 0 y 1B/C,<br />
iii
Resumen<br />
respectivamente. De igual manera, los aislados <strong>de</strong>l patotipo <strong>de</strong> Marchitez<br />
se distribuyeron en varios grupos que correspon<strong>de</strong>n, respectivamente, a las<br />
razas 1A/6, raza 5, y al grupo <strong>de</strong> razas 2, 3, y 4, estas últimas <strong>de</strong>scritas<br />
únicamente en la India. El análisis filogenético <strong>de</strong> los ‘fingerprints’ <strong>de</strong><br />
ADN mostró que los aislados <strong>de</strong> la raza 5 se encuentran más cercanos<br />
evolutivamente a las razas 2, 3, y 4 que al resto <strong>de</strong> las razas presentes en la<br />
Cuenca Mediterránea (razas 1A y 6). Aunque la raza 5 y las razas 2, 3, y 4<br />
se encuentran geográficamente separadas entre sí, son las más similares en<br />
cuanto a su virulencia sobre líneas diferenciadores.<br />
Se ha <strong>de</strong>sarrollado un método <strong>de</strong>l análisis <strong>de</strong>l ADN genómico <strong>de</strong> F.<br />
oxysporum f. sp. ciceris, basado en rep-PCR, alternativo a las hibridaciones<br />
tipo ‘Southern’ para generar ‘fingerprints’ <strong>de</strong> ADN. Para ello, hemos<br />
diseñado una pareja <strong>de</strong> iniciadores que amplifican las regiones <strong>de</strong> ADN<br />
existentes entre las copias <strong>de</strong>l elemento repetitivo FocB10 dispersas en el<br />
genoma <strong>de</strong> F. oxysporum f. sp. ciceris. Mediante el análisis rep-PCR se<br />
generaron patrones <strong>de</strong> amplificación que resultaron válidos para<br />
discriminar entre las razas <strong>de</strong> F. oxysporum f. sp. ciceris. A<strong>de</strong>más, el análisis<br />
UPGMA <strong>de</strong> los datos generados mediante este análisis puso <strong>de</strong> manifiesto<br />
que la información generada es equivalente a la proporcionada por<br />
hibridaciones ‘Southern’ obtenidas con el propio elemento repetitivo. En<br />
consecuencia, la metodología rep-PCR <strong>de</strong>sarrollada supone una<br />
herramienta muy a<strong>de</strong>cuada para el estudio <strong>de</strong> diversidad genética en las<br />
poblaciones <strong>de</strong> F. oxysporum f. sp. ciceris.<br />
Finalmente, nuestra hipótesis evolutiva en F. oxysporum f. sp. ciceris es<br />
que sus aislados <strong>de</strong>rivan <strong>de</strong> una población inicial fundadora pequeña<br />
(posiblemente un único individuo) que adquirió patogenicidad sobre Cicer<br />
spp. A<strong>de</strong>más, la subsiguiente diversificación <strong>de</strong>l patógeno en patotipos y<br />
razas patogénicas ha podido ser el resultado <strong>de</strong> la acumulación <strong>de</strong><br />
pequeños cambios genéticos, que refleja una larga historia <strong>de</strong> asociación<br />
con su planta huésped, el garbanzo, como especie cultivada. También<br />
postulamos que en F. oxysporum f. sp. ciceris unas razas se han originado a<br />
partir <strong>de</strong> otras, y que el avance evolutivo <strong>de</strong> las razas patogénicas va unido<br />
a una mayor virulencia <strong>de</strong> éstas sobre un conjunto <strong>de</strong> líneas<br />
diferenciadoras raciales. Así, la raza 0 es la menos virulenta <strong>de</strong> todas las<br />
iv
Resumen<br />
razas, y la más antigua <strong>de</strong> ellas en razón <strong>de</strong> la mayor diversidad genética<br />
que presenta. Por el contrario, la raza 5 es la más virulenta <strong>de</strong> las razas<br />
i<strong>de</strong>ntificadas hasta ahora en la Cuenca Mediterránea, la que menor<br />
diversidad genética posee, y a la que por tanto correspon<strong>de</strong>ría un origen<br />
evolutivo más reciente.<br />
v
Summary<br />
Summary<br />
<strong>Fusarium</strong> wilt of chickpea (Cicer arietinum L.), caused by <strong>Fusarium</strong> oxysporum<br />
Schlechtend.: Fr. f. sp. ciceris (Padwick) Matuo & K. Sato, is one of the<br />
most important diseases and limiting factors of chickpea production<br />
worldwi<strong>de</strong>. The pathogen is an anamorphic fungus which can survive in<br />
soil for long time. The most practical and cost-efficient method for<br />
control of the disease is the use of resistant cultivars, which efficiency is<br />
curtailed by the occurrence of pathogenic races. Eight pathogenic races<br />
<strong>de</strong>signated races 0, 1A, 1B/C, 2, 3, 4, 5, and 6 have been i<strong>de</strong>ntified, which<br />
can be classified into two pathotypes, yellowing (races 0 and 1B/C) and<br />
wilting (races 1A through 6), according to the disease syndrome that they<br />
induce in the plant. Characterization of pathogenic races and pathotypes in<br />
F. oxysporum f. sp. ciceris prevailing within a region is a key factor for the<br />
efficient use of resistant cultivars in the management of the disease.<br />
Characterization of pathogenic strains by means of biological pathotyping<br />
is costly in time, space, and resources and it is not informative of the<br />
genetic diversity and relationship existing among and within races and<br />
pathotypes. Therefore, there is a need of new, more informative methods<br />
for the rapid, consistent and reproducible i<strong>de</strong>ntification of the pathogen<br />
races. These new methods can be based on recently <strong>de</strong>veloped molecular<br />
biology techniques. The objectives of this Doctoral Dissertation were to<br />
<strong>de</strong>velop a method based on sequences of polymorphic DNA of use for<br />
the i<strong>de</strong>ntification of F. oxysporum f. sp. ciceris and characterization of its<br />
pathogenic races by means of specific PCR, and to analyze the genetic<br />
variability and evolutionary relationship that might exist among races of<br />
the pathogen.<br />
<strong>Molecular</strong> markers of F. oxysporum f. sp. ciceris races were first<br />
investigated by RAPD-PCR analysis using 40 arbitrary primers and 99<br />
isolates of the pathogen representatives of the eight races and of disease<br />
geographical range. Five (OPI-09, OPI-18, OPF-06, OPF-10, and OPF-<br />
12) out of the 40 primers produced RAPD amplicons associated with F.<br />
vii
Summary<br />
oxysporum f. sp. ciceris and with races 0, 1B/C, 5, and 6. Practicability,<br />
reproducibility, and fiability of these markers for diagnosis of the pathogen<br />
were confirmed by biological and molecular ‘blind trials’. UPGMA analysis<br />
of RAPD profiles showed a high genetic similarity among F. oxysporum f.<br />
sp. ciceris isolates, and grouped them in clusters that were correlated to<br />
Yellowing or Wilting pathotypes. Isolates belonging to the Yellowing<br />
pathotype formed a distinct cluster that inclu<strong>de</strong>d races 0 and 1B/C; while<br />
isolates belonging to the Wilting pathotype formed another cluster that<br />
inclu<strong>de</strong>d races 1A through 6. Sequence characterized amplified regions<br />
(SCARs) from the RAPD-race markers i<strong>de</strong>ntified served for the <strong>de</strong>sign of<br />
primers for specific PCR. To this aim, the amplified RAPD fragments<br />
were cloned and sequenced, and primers were <strong>de</strong>signed from both ends of<br />
the fragment sequence that contained part of the RAPD primer which<br />
originated the marker. The new primers <strong>de</strong>signed were used for specific<br />
PCR analyses using F. oxysporum f. sp. ciceris isolates representatives of all<br />
pathogenic races and of diverse geographical origin. Five primer pairs for<br />
specific PCR were <strong>de</strong>signed that allow consistent and reproducible<br />
discrimination of F. oxysporum f. sp. ciceris isolates from: a) F. oxysporum<br />
isolates nonpathogenic to chickpea; b) isolates of other F. oxysporum formae<br />
speciales; and c) isolates of other <strong>Fusarium</strong> spp. In addition, these specific<br />
PCR analyses using primers ‘ad hoc’ ma<strong>de</strong> it possible to differentiate<br />
among isolates o races 0, 1A, 5, and 6 of F. oxysporum f. sp. ciceris.<br />
The observed phenotypic and geographical diversity in F. oxysporum f.<br />
sp. ciceris suggested the null hypothesis that this pathogen is of<br />
monophyletic origin. This hypothesis was addressed by analyzing the<br />
extent and nature of intron sequence variation existing in the following<br />
conserved genes: elongation translation factor 1α (EF1α), β-tubulin,<br />
histone 3, actin and calmodulin, among isolates representative of races 0,<br />
1A, 1B/C, 4, 5, and 6 ad of diverse geographic origin. In spite of the wi<strong>de</strong><br />
diversity of pathogen isolates in the study, in terms of their geographic<br />
distribution, disease syndrome induced in the plant, and cultivar specific<br />
pathogenicity, all of them showed i<strong>de</strong>ntical sequences for each of the five<br />
genes of study.<br />
viii
Summary<br />
In addition, we generated the EF1α sequences of 17 F. oxysporum f. sp.<br />
ciceris isolates and three isolates of F. oxysporum nonpathogenic to chickpea.<br />
These sequences, together with those of 24 isolates representative of 11<br />
formae speciales of F. oxysporum obtained from GenBank were analyzed by<br />
means of PAUP (Phylogenetic Analysis Using Parsimony). All F. oxysporum<br />
f. sp. ciceris isolates were grouped together and distinctively from all other<br />
F. oxysporum isolates, <strong>de</strong>monstrating a monophyletic origin.<br />
The genetic variability among isolates of a race, and of different races,<br />
as well as the evolutionary relationship among the races, were investigated<br />
by means of analysis of DNA fingerprint. These fingerprints were<br />
generated with probes FocB10, FocO2, and FocP18 which contain<br />
repetitive DNA i<strong>de</strong>ntified within the F. oxysporum f. sp. ciceris genome and<br />
show DNA similarity with transposable elements referred in the literature.<br />
In particular, the FocB10 sequence showed 80% similarity with Fot1<br />
transposon from F. oxysporum f. sp. melonis. Analyses indicated that DNA<br />
fingerprinting was highly correlated with their nature in term of pathotype<br />
and race belonging. Both the phenetic analysis of similarity (UPGMA) and<br />
the cladistic analysis (PAUP) of fingerprints grouped F. oxysporum f. sp.<br />
ciceris isolates in two clusters which correlate with the Yellowing and<br />
Wilting pathotypes. Also, the cluster comprised of Yellowing isolates was<br />
formed by two subclusters which inclu<strong>de</strong>d race 0 and race 1B/C isolates,<br />
respectively. Similarly, the cluster including Wilting isolates comprised<br />
several subclusters which correspon<strong>de</strong>d with races 1A/6, race 5, and races<br />
2, 3, and 4. This latter group of races have been reported from India only.<br />
Phylogenetic analysis of DNA fingerprints showed that race 5 isolates are<br />
evolutionarily closer to races 2, 3, and 4 than to races 1A and 6 also<br />
present in the Mediterranean Basin. Although races 5 and races 2, 3, and 4<br />
are wi<strong>de</strong>ly separated apart, they are the most similar among F. oxysporum f.<br />
sp. ciceris races in terms of virulence to differential chickpea lines.<br />
In this research, we have also <strong>de</strong>veloped a rep-PCR-based method for<br />
analysis of genomic F. oxysporum f. sp. ciceris DNA, which is an alternative<br />
to DNA fingerprinting generated by Southern hybridization. To this aim,<br />
we <strong>de</strong>signed a primer pair that amplify DNA regions between copies of<br />
the repetitive element FocB10 dispersed within the F. oxysporum f. sp. ciceris<br />
ix
Summary<br />
genome. Rep-PCR analyses generated amplification profiles that<br />
discriminated among races of the pathogen. In addition, UPGMA analysis<br />
of data thus generated showed that the information provi<strong>de</strong>d by rep-PCR<br />
analysis is equivalent to that obtained by means of Southern hybridization<br />
using the same repetitive element. Therefore, the rep-PCR-based method<br />
<strong>de</strong>veloped is a useful and a<strong>de</strong>quate tool for studying genetic diversity<br />
within F. oxysporum f. sp. ciceris populations.<br />
Finally, our evolutionary hypothesis for F. oxysporum f. sp. ciceris is that<br />
isolates of this pathogen <strong>de</strong>rive from an initial, small foun<strong>de</strong>r population<br />
(possibly a single isolate) that acquired pathogenicity to Cicer spp. In<br />
addition, diversification of the pathogen in pathotypes and races may have<br />
resulted from accumulation of small genetic changes, which reflect a<br />
prolonged history of association with its cultivated host, the chickpea crop.<br />
We also postulate that races originate one from another, and that the<br />
evolutionary <strong>de</strong>velopment of new races implies higher virulence of them<br />
to chickpea differential lines. Thus, race 0 is the least virulent of all races<br />
and the ol<strong>de</strong>st among them because of the highest genetic diversity shown<br />
among isolates belonging to them. On the contrary, race 5, the most<br />
virulent of all races i<strong>de</strong>ntified in the Mediterranean Basin so far, is the race<br />
showing the least genetic diversity and would be the most recently<br />
evolved.<br />
x
ÍNDICE DE CONTENIDO<br />
Índice <strong>de</strong> Contenido<br />
INTRODUCCIÓN 1<br />
La Fusariosis Vascular <strong>de</strong>l garbanzo.......................................................... .1<br />
Importancia y distribución geográfica <strong>de</strong> la<br />
enfermedad..........................................................................................1<br />
Sintomatología.......................................................................................2<br />
Biología <strong>de</strong>l agente y patogénesis.......................................................3<br />
Variabilidad patogénica <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris .............4<br />
I<strong>de</strong>ntificación <strong>de</strong> razas <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris:<br />
Pruebas <strong>de</strong> Patogenicidad..................................................................7<br />
Control <strong>de</strong> la Fusariosis Vascular <strong>de</strong>l garbanzo mediante<br />
cultivares resistentes ..........................................................................8<br />
Objetivos......................................................................................................15<br />
CAPÍTULO I 19<br />
I<strong>de</strong>ntificación <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris<br />
y <strong>de</strong> las razas patogénicas 0, 1B/C, 5 y 6 mediante<br />
RAPD-PCR 19<br />
Introducción.................................................................................................19<br />
Materiales y Métodos..................................................................................22<br />
Aislados fúngicos................................................................................22<br />
Pruebas <strong>de</strong> Patogenicidad..................................................................26<br />
Extracción <strong>de</strong> ADN...........................................................................27<br />
Análisis RAPD....................................................................................28<br />
Análisis <strong>de</strong> datos .................................................................................29<br />
Resultados.....................................................................................................30<br />
Análisis RAPD <strong>de</strong> mezclas <strong>de</strong> ADN <strong>de</strong> aislados <strong>de</strong><br />
F. oxysporum f. sp. ciceris pertenecientes a una misma raza..........30<br />
xi
Índice <strong>de</strong> Contenido<br />
xii<br />
Análisis RAPD <strong>de</strong> ADN <strong>de</strong> aislados individuales <strong>de</strong><br />
cada raza............................................................................................31<br />
Confirmación <strong>de</strong> la caracterización racial mediante<br />
análisis RAPD...................................................................................32<br />
Análisis <strong>de</strong> los datos RAPD..............................................................34<br />
Discusión......................................................................................................38<br />
CAPÍTULO II 45<br />
Desarrollo <strong>de</strong> un método basado en el análisis<br />
PCR específica para la i<strong>de</strong>ntificación molecular<br />
<strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris y <strong>de</strong> las<br />
razas patogénicas 0, 1A, 5, y 6 45<br />
Introducción................................................................................................ 45<br />
Materiales y Métodos..................................................................................49<br />
Aislados fúngicos................................................................................49<br />
Extracción <strong>de</strong> ADN...........................................................................50<br />
Reacciones <strong>de</strong> amplificación RAPD................................................50<br />
Clonación <strong>de</strong> fragmentos RAPD .....................................................56<br />
Purificación <strong>de</strong> plásmido bacteriano................................................57<br />
Hibridación <strong>de</strong> las bandas clonadas con los perfiles <strong>de</strong><br />
amplificación RAPD y PCR específica.........................................58<br />
Hibridaciones ‘Southern’...................................................................60<br />
Secuenciación <strong>de</strong> los fragmentos RAPD clonados........................60<br />
Diseño <strong>de</strong> iniciadores específicos.....................................................61<br />
Reacciones <strong>de</strong> amplificación PCR....................................................61<br />
Resultados.....................................................................................................63<br />
Clonación <strong>de</strong> los fragmentos RAPD.. .............................................63<br />
Hibridaciones con el perfil RAPD...................................................64<br />
Hibridaciones ‘Southern’...................................................................68
Índice <strong>de</strong> Contenido<br />
Secuenciación <strong>de</strong> los fragmentos RAPD ........................................70<br />
Reacciones <strong>de</strong> amplificación PCR....................................................70<br />
Discusión.. ...................................................................................................79<br />
CAPÍTULO III 83<br />
Genealogías génicas <strong>de</strong>finen a <strong>Fusarium</strong> oxysporum f. sp. ciceris<br />
como un grupo monofilético 83<br />
Introducción .................................................................................................83<br />
Materiales y Métodos.................................................................................. 86<br />
Aislados fúngicos................................................................................86<br />
Amplificación <strong>de</strong> ADN y secuenciación.........................................88<br />
Análisis filogenético ...........................................................................89<br />
Resultados....................................................................................................90<br />
Discusión......................................................................................................92<br />
CAPÍTULO IV 99<br />
<strong>Caracterización</strong> <strong>de</strong> aislados <strong>de</strong> <strong>Fusarium</strong> oxysporum<br />
f. sp. ciceris mediante ‘fingerprinting’ genómico<br />
con secuencias <strong>de</strong> ADN repetitivo 99<br />
Introducción .................................................................................................99<br />
Materiales y Métodos................................................................................102<br />
Aislados fúngicos..............................................................................102<br />
Extracción <strong>de</strong> ADN.........................................................................103<br />
Hibridaciones ‘Southern’.................................................................103<br />
Análisis <strong>de</strong> datos ...............................................................................106<br />
Resultados..................................................................................................107<br />
Variabilidad genética entre razas <strong>de</strong> F. oxysporum<br />
f. sp. ciceris........................................................................................110<br />
xiii
Índice <strong>de</strong> Contenido<br />
xiv<br />
Variabilidad genética entre aislados <strong>de</strong> F. oxysporum<br />
f. sp. ciceris pertenecientes a la misma raza..................................112<br />
Discusión....................................................................................................116<br />
CAPÍTULO V 123<br />
Mejora <strong>de</strong> la metodología <strong>de</strong> análisis <strong>de</strong> poblaciones<br />
<strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris mediante<br />
PCR entre elementos <strong>de</strong> ADN repetitivo (rep-PCR) 123<br />
Introducción..............................................................................................123<br />
Materiales y Métodos................................................................................126<br />
Aislados fúngicos..............................................................................126<br />
Extracción <strong>de</strong> ADN.........................................................................126<br />
Diseño <strong>de</strong> iniciadores.......................................................................127<br />
Condiciones <strong>de</strong> amplificación rep-PCR ........................................127<br />
Análisis <strong>de</strong> datos ...............................................................................130<br />
Resultados..................................................................................................131<br />
Variabilidad en F. oxysporum f. sp. ciceris<br />
<strong>de</strong>terminada mediante análisis rep-PCR.....................................131<br />
Análisis <strong>de</strong> los datos rep-PCR ........................................................132<br />
Análisis comparativo entre los resultados <strong>de</strong> las<br />
hibridaciones ‘Southern’ y el análisis rep-PCR ..........................134<br />
Discusión....................................................................................................137<br />
DISCUSIÓN GENERAL 141<br />
CONCLUSIONES 149<br />
BIBLIOGRAFÍA CITADA 153
ÍNDICE DE FIGURAS<br />
Índice <strong>de</strong> Figuras<br />
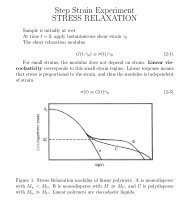
Figura 1. Amplificaciones RAPD generadas mediante el<br />
iniciador aleatorio OPF-16 utilizando mezclas <strong>de</strong> ADN<br />
<strong>de</strong> aislados pertenecientes a la misma raza (A), y ADN<br />
<strong>de</strong> aislados individuales (B) representativos <strong>de</strong> cada raza<br />
<strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris………………………………………..33<br />
Figura 2. Amplificaciones RAPD generadas mediante el<br />
iniciador aleatorio OPF-10 utilizando mezclas <strong>de</strong> ADN<br />
<strong>de</strong> aislados pertenecientes a la misma raza (A), y ADN<br />
<strong>de</strong> aislados individuales (B) representativos <strong>de</strong> cada raza<br />
<strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris………………………………………..35<br />
Figura 3. Dendrograma obtenido mediante el análisis<br />
UPGMA <strong>de</strong> los datos que generó el análisis RAPD <strong>de</strong>57<br />
aislados <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris y cuatro<br />
aislados <strong>de</strong> F. oxysporum no patogénicos <strong>de</strong> garbanzo,<br />
con los iniciadores OPI-01, OPI-09, OPI-18, OPF-06,<br />
OPF-10, OPF-12, y OPF-16 ............................................................................39<br />
Figura 4. Amplificación RAPD utilizando ADN <strong>de</strong><br />
aislados <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris con el<br />
iniciador aleatorio OPF-10 (A), e hibridación <strong>de</strong>l mismo<br />
perfil <strong>de</strong> amplificación con el fragmento RAPD clonado<br />
<strong>de</strong> 1,1 kb marcador <strong>de</strong> la raza 1B/C (clon FocD33)(B)................................65<br />
Figura 5. Hibridación ‘Southern’ <strong>de</strong>l ADN genómico<br />
<strong>de</strong> aislados <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris digerido<br />
con la enzima EcoRI, con la sonda FocL10 (0,9 kb –<br />
raza 5– OPF10)....................................................................................................69<br />
Figura 6. Amplificaciones PCR específicas <strong>de</strong> las razas<br />
0 (A), 5 (B), 6 (C), y 1A-6 (D) <strong>de</strong> <strong>Fusarium</strong> oxysporum f.<br />
sp. ciceris realizadas con los iniciadores basados en<br />
secuencias SCAR obtenidas a partir <strong>de</strong> fragmentos<br />
RAPD marcadores..............................................................................................73<br />
xv
Índice <strong>de</strong> Figuras<br />
Figura 7. Amplificación PCR específica utilizando<br />
ADN <strong>de</strong> <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris, <strong>de</strong> F.<br />
oxysporum no patogénicos <strong>de</strong> garbanzo, <strong>de</strong> otras formae<br />
speciales, y <strong>de</strong> diferentes <strong>Fusarium</strong> spp., y los iniciadores<br />
Foc0-12fwd y rev (A); e hibridación <strong>de</strong> los productos <strong>de</strong><br />
amplificación con el fragmento RAPD clonado <strong>de</strong> 1,5<br />
kb marcador <strong>de</strong> la forma specialis ciceris (clon Foc0-12) (B).............................75<br />
Figura 8. Cantidad <strong>de</strong> ADN <strong>de</strong>l aislado Foc-Tonini<br />
(raza 6) <strong>de</strong>tectada mediante la amplificación PCR<br />
específica utilizando los iniciadores Foc0-12fwd y rev,<br />
específicos <strong>de</strong> forma specialis ciceris .....................................................................77<br />
Figura 9. Uno <strong>de</strong> los tres árboles filogenéticos <strong>de</strong> igual<br />
máxima parsimonia inferidos a partir <strong>de</strong> la genealogía<br />
génica <strong>de</strong>l factor <strong>de</strong> traducción y elongación EF1α<br />
(EF1α), que <strong>de</strong>muestra la monofilia en <strong>Fusarium</strong><br />
oxysporum f. sp. ciceris ...........................................................................................93<br />
Figura 10. ‘Fingerprint’ <strong>de</strong> ADN genómico <strong>de</strong> aislados<br />
<strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris representativos <strong>de</strong><br />
todas las razas patogénicas <strong>de</strong>scritas (razas 0, 1A, 1B/C,<br />
2, 3, 4, 5, y 6), y <strong>de</strong> un amplio origen geográfico <strong>de</strong>l<br />
patógeno.............................................................................................................109<br />
Figura 11. Dendrograma UPGMA generado a partir <strong>de</strong><br />
la información obtenida mediante ‘fingerprints’ <strong>de</strong> ADN<br />
genómico <strong>de</strong> aislados <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris<br />
(razas 0, 1A, 1B/C, 2 a 6) y F. oxysporum no patogénicos<br />
(NP) <strong>de</strong> garbanzo con las sondas <strong>de</strong> ADN repetitivo<br />
FocB10, FocO2, y FocP18..................... ..............................................111<br />
Figura 12. Uno <strong>de</strong> dos árboles filogenéticos <strong>de</strong> igual<br />
máxima parsimonia generados mediante PAUP en base<br />
a la información generada mediante ‘fingerprints’ <strong>de</strong><br />
ADN genómico <strong>de</strong> aislados <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp.<br />
ciceris (razas 0, 1A, 1B/C, 2 a 6) y F. oxysporum no<br />
patogénicos <strong>de</strong> garbanzo (NP) generados con las sondas<br />
<strong>de</strong> ADN repetitivo FocB10, FocO2, y FocP18......................................115<br />
xvi
Índice <strong>de</strong> Figuras<br />
Figura 13. Amplificación rep-PCR <strong>de</strong> aislados <strong>de</strong><br />
<strong>Fusarium</strong> oxysporum f. sp. ciceris (razas 0, 1A, 1B/C, 2 a 6)<br />
y F. oxysporum no patogénicos (NP) <strong>de</strong> garbanzo....................................133<br />
Figura 14. Dendrograma UPGMA generado a partir <strong>de</strong><br />
la información obtenida mediante la amplificación rep-<br />
PCR <strong>de</strong> ADN <strong>de</strong> aislados <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp.<br />
ciceris (razas 0, 1A, 1B/C, 2 a 6) y F. oxysporum no<br />
patogénicos (NP) <strong>de</strong> garbanzo..............................................................135<br />
Figura 15. Dendrograma UPGMA generado a partir <strong>de</strong><br />
la información <strong>de</strong> ‘fingerprints’ obtenidos mediante<br />
hibridaciones ‘Southern’ <strong>de</strong> ADN genómico <strong>de</strong> aislados<br />
<strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris (razas 0, 1A, 1B/C, 2 a<br />
6) y F. oxysporum no patogénicos (NP) con el elemento<br />
repetitivo FocB10.........................................................................................137<br />
xvii
ÍNDICE DE TABLAS<br />
Índice <strong>de</strong> Tablas<br />
Tabla 1. Reacción <strong>de</strong> las líneas <strong>de</strong> garbanzo<br />
diferenciadoras a la inoculación con las distintas razas <strong>de</strong><br />
<strong>Fusarium</strong> oxysporum f. sp. ciceris..............................................................................9<br />
Tabla 2. Información geográfica y racial sobre los<br />
aislados <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris (Foc), y <strong>de</strong> F.<br />
oxysporum (Fo) no patogénicos <strong>de</strong> garbanzo, utilizados en<br />
el análisis RAPD-PCR........................................................................................23<br />
Tabla 3. Nombre y secuencia <strong>de</strong> los siete iniciadores<br />
seleccionados por su capacidad <strong>de</strong> producir<br />
polimorfismos <strong>de</strong> ADN informativos para la<br />
i<strong>de</strong>ntificación <strong>de</strong> F. oxysporum f. sp. ciceris y <strong>de</strong> sus razas<br />
patogénicas mediante el análisis RAPD...........................................................36<br />
Tabla 4. Análisis <strong>de</strong> la varianza molecular, AMOVA, <strong>de</strong><br />
los datos RAPDs obtenidos a partir <strong>de</strong> 57 aislados <strong>de</strong> F.<br />
oxysporum f. sp. ciceris agrupados según su patotipo<br />
(Amarillez, Marchitez), raza patogénica (razas 0, 1A,<br />
1B/C, 2, 3, 4, 5, y 6), y origen geográfico) .....................................................40<br />
Tabla 5. Información geográfica y racial sobre los<br />
aislados <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris (Foc), y <strong>de</strong> F.<br />
oxysporum (Fo) no patogénicos <strong>de</strong> garbanzo, utilizados en<br />
el análisis PCR específica ..................................................................................51<br />
Tabla 6. Aislados <strong>de</strong> otras formae speciales <strong>de</strong> <strong>Fusarium</strong><br />
oxysporum, así como <strong>de</strong> otras <strong>Fusarium</strong> spp. utilizados en<br />
el análisis PCR específica con aislados <strong>de</strong> F. oxysporum f.<br />
sp. ciceris.................................................................................................................54<br />
Tabla 7. Designación y características <strong>de</strong> los plásmidos<br />
bacterianos <strong>de</strong> ADN recombinante generados mediante<br />
la clonación <strong>de</strong> fragmentos RAPD marcadores <strong>de</strong><br />
<strong>Fusarium</strong> oxysporum f. sp. ciceris y <strong>de</strong> sus razas patogénicas.............................67<br />
xix
Índice <strong>de</strong> Tablas<br />
Tabla 8. Secuencias <strong>de</strong> los iniciadores, y condiciones <strong>de</strong><br />
amplificación para su utilización en análisis PCR<br />
específica para la i<strong>de</strong>ntificación <strong>de</strong> <strong>Fusarium</strong> oxysporum f.<br />
sp. ciceris y sus razas patogénicas 0, 1A, 1B/C, 5, y 6.....................................72<br />
Tabla 9. Información sobre la raza y origen geográfico<br />
<strong>de</strong> los aislados <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris (Foc) y<br />
<strong>Fusarium</strong> oxysporum (Fo) no patogénicos <strong>de</strong> garbanzo<br />
utilizados en el análisis <strong>de</strong> genealogías génicas ..............................................87<br />
Tabla 10. Secuencia <strong>de</strong> los iniciadores que amplifican<br />
intrones <strong>de</strong> los genes altamante conservados factor <strong>de</strong><br />
traducción y elongación 1α, β-tubulina, histona 3,<br />
actina, y calmodulina, utilizados en el análisis <strong>de</strong><br />
genealogías génicas en <strong>Fusarium</strong><br />
oxysporum f. sp. ciceris......................................................................................….89<br />
Tabla 11. Información geográfica y racial sobre los<br />
aislados <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris (Foc), y <strong>de</strong><br />
<strong>Fusarium</strong> oxysporum (Fo) no patogénicos <strong>de</strong> garbanzo<br />
utilizados en el análisis <strong>de</strong> ‘fingerprints’ <strong>de</strong> ADN........................................104<br />
Tabla 12. Información geográfica y racial sobre los<br />
aislados <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris (Foc), y <strong>de</strong><br />
<strong>Fusarium</strong> oxysporum (Fo) no patogénicos <strong>de</strong> garbanzo<br />
utilizados en el análisis <strong>de</strong> ‘fingerprints’ <strong>de</strong> ADN........................................128<br />
xx
INTRODUCCIÓN<br />
La Fusariosis Vascular <strong>de</strong>l garbanzo<br />
Importancia y distribución geográfica <strong>de</strong> la enfermedad<br />
Introducción<br />
La Fusariosis Vascular <strong>de</strong>l garbanzo (Cicer arietinum L.) causada por<br />
<strong>Fusarium</strong> oxysporum Schlechtend: Fr. f. sp. ciceris (Padwick) Matuo & K. Sato<br />
es, junto con la Rabia causada por Didymella rabiei Kovachevski [anamorfo<br />
Ascochyta rabiei (Pass.) Labrousse], la enfermedad más importante <strong>de</strong> las que<br />
afectan a la producción <strong>de</strong> garbanzo en España (Navas Cortés et al., 1998;<br />
2000; Trapero Casas y Jiménez Díaz, 1985).<br />
La enfermedad ha sido diagnosticada a<strong>de</strong>más en la mayoría <strong>de</strong> los países<br />
don<strong>de</strong> se cultiva el garbanzo, entre los que se encuentran Bangla<strong>de</strong>sh,<br />
Etiopía, India, Méjico, Pakistán, Siria y Túnez (Halila y Strange, 1996;<br />
Nene et al., 1984); Chile, Irán, Sudán (Haware et al., 1986a); Perú (Echandi,<br />
1970); Unión Soviética (Stepanova, 1971); Malavi (Kannaiyan, 1981);<br />
EEUU (California) (Bud<strong>de</strong>nhagen y Workneh, 1988; Phillips, 1988); Italia<br />
(Frisullo et al., 1989); Israel (Y. Katan, comunicación personal); y Líbano y<br />
Turquía (C. Akem, comunicación personal).<br />
La Fusariosis Vascular es uno <strong>de</strong> los principales factores limitantes <strong>de</strong>l<br />
cultivo <strong>de</strong> garbanzo en la región Mediterránea y el subcontinente Indio<br />
(Haware, 1990; Jiménez Díaz et al., 1989c; Nene y Reddy, 1987), pudiendo<br />
originar pérdidas anuales <strong>de</strong> rendimiento <strong>de</strong> 10-15% en la India (Singh y<br />
Dahiya, 1973) y España (Trapero Casas y Jiménez Díaz, 1985), y la<br />
<strong>de</strong>vastación completa <strong>de</strong>l cultivo en condiciones <strong>de</strong>terminadas (Halila y<br />
Strange, 1996).<br />
1
Introducción<br />
Sintomatología<br />
La Fusariosis Vascular <strong>de</strong>l garbanzo se caracteriza por dos síndromes,<br />
<strong>de</strong>nominados Amarillez y Marchitez, distinguibles entre sí tanto por los<br />
síntomas que los componen como por la cronología con que éstos se<br />
<strong>de</strong>sarrollan. Ambos síndromes son consecuencia <strong>de</strong> infecciones vasculares<br />
<strong>de</strong> la planta, y llevan asociada una coloración castaño oscura <strong>de</strong>l xilema y<br />
médula <strong>de</strong> la raíz, cuello y tallo <strong>de</strong> las plantas infectadas.<br />
El síndrome <strong>de</strong> Amarillez se caracteriza por el <strong>de</strong>sarrollo progresivo <strong>de</strong><br />
clorosis, amarillez y necrosis <strong>de</strong> foliolos, que progresan <strong>de</strong> forma acrópeta<br />
en la planta dando lugar a la <strong>de</strong>foliación <strong>de</strong> ésta. Generalmente, en<br />
inoculaciones artificiales las plantas no mueren antes <strong>de</strong> los 40 días <strong>de</strong>spués<br />
<strong>de</strong> la siembra en suelo infestado (Trapero Casas, 1983; Trapero Casas y<br />
Jiménez Díaz, 1985).<br />
El síndrome <strong>de</strong> Marchitez se caracteriza por un <strong>de</strong>sarrollo rápido <strong>de</strong><br />
flacci<strong>de</strong>z y clorosis ver<strong>de</strong> mate en las hojas, en ausencia <strong>de</strong> amarillez, que<br />
se extien<strong>de</strong> poco <strong>de</strong>spués al resto <strong>de</strong> la planta. La flacci<strong>de</strong>z es seguida por<br />
la <strong>de</strong>secación <strong>de</strong> hojas y tallos, los foliolos permanecen adheridos al raquis,<br />
y posteriormente la planta muere. En inoculaciones artificiales mediante<br />
siembra en suelo infestado por el patógeno, la muerte <strong>de</strong> plantas<br />
susceptibles ocurre generalmente antes <strong>de</strong> transcurridos los 20 días <strong>de</strong>s<strong>de</strong><br />
la siembra (Trapero Casas, 1983; Trapero Casas y Jiménez Díaz, 1985).<br />
Ambos síndromes han sido <strong>de</strong>scritos en la literatura fitopatológica<br />
asociados con la Fusariosis Vascular <strong>de</strong>l garbanzo, aunque el <strong>de</strong> Marchitez<br />
ha sido el más común y tradicionalmente reconocido en diversos países<br />
(Narasimhan, 1929; Nene, 1980; Raheja y Das, 1957). Por el contrario, el<br />
síndrome <strong>de</strong> Amarillez es el predominante en Andalucía, Castilla y León, y<br />
Extremadura (Alcalá Jiménez, 1995; Trapero Casas y Jiménez Díaz, 1986a,<br />
b).<br />
2
Biología <strong>de</strong>l agente y patogénesis<br />
Introducción<br />
F. oxysporum f. sp. ciceris es un hongo <strong>de</strong> suelo, necrotrofo obligado,<br />
patogénicamente especializado <strong>de</strong> especies <strong>de</strong> Cicer, a las que infecta<br />
vascularmente, y transmisible en semillas infectadas <strong>de</strong> C. arietinum, su<br />
huésped cultivado (Haware et al., 1978; 1986a; Nene y Haware, 1980). No<br />
obstante, F. oxysporum f. sp. ciceris pue<strong>de</strong> invadir asintomáticamente raíces<br />
<strong>de</strong> otras plantas, tanto leguminosas como no leguminosas, tales como:<br />
guisante (Pisum sativum L.), lenteja (Lens esculenta Moench), Cajanus cajan (L.)<br />
Millsp. (Haware y Nene, 1982a), altramuz blanco (Lupinus albus L.), habas<br />
(Vicia faba L.), judía (Phaseolus vulgaris L.), melón (Cucumis melo L.), patata<br />
(Solanum tuberosum L.), remolacha azucarera (Beta vulgaris L.), Chenopodium<br />
album L., y veza (Vicia sativa L.) (Cabrera <strong>de</strong> la Colina et al., 1987; Trapero<br />
Casas y Jiménez Díaz, 1985), que proporcionan al patógeno un medio <strong>de</strong><br />
sobrevivir parasíticamente durante periodos <strong>de</strong> tiempo en que el suelo<br />
permanece libre <strong>de</strong> su huésped cultivado. A<strong>de</strong>más, F. oxysporum f. sp. ciceris,<br />
al igual que los agentes causantes <strong>de</strong> otras Fusariosis Vasculares, posee la<br />
capacidad <strong>de</strong> sobrevivir inactivo en el suelo en forma <strong>de</strong> clamidosporas<br />
durante varios años en ausencia <strong>de</strong> plantas huésped (Allen, 1983); hasta 6<br />
años, según indican investigaciones realizadas en la India (Haware et al.,<br />
1986b).<br />
F. oxysporum f. sp. ciceris pue<strong>de</strong> invadir las plántulas <strong>de</strong> garbanzo <strong>de</strong><br />
cultivares tanto susceptibles como resistentes durante la germinación y<br />
emergencia, en los primeros días <strong>de</strong>spués <strong>de</strong> la siembra, sin necesidad <strong>de</strong><br />
heridas en los tejidos subterráneos <strong>de</strong> la joven planta. La penetración <strong>de</strong> la<br />
planta por el patógeno tiene lugar principalmente a través <strong>de</strong> los<br />
cotiledones, y en las zonas <strong>de</strong>l hipocotilo y epicotilo próximas a los<br />
cotiledones; y en menor proporción por zonas <strong>de</strong> la raíz principal, a<br />
excepción <strong>de</strong>l ápice radical (Jiménez Díaz et al., 1989a). Posteriormente, y<br />
tras el crecimiento <strong>de</strong>l hongo a través <strong>de</strong>l córtex radical, tiene lugar la<br />
colonización vascular <strong>de</strong> la planta <strong>de</strong>bido al crecimiento <strong>de</strong> las hifas a lo<br />
largo <strong>de</strong>l eje vegetal y al transporte <strong>de</strong> microconidias en los vasos <strong>de</strong>l<br />
xilema, seguido <strong>de</strong> la colonización <strong>de</strong> los vasos adyacentes <strong>de</strong>bido al<br />
crecimiento lateral <strong>de</strong>l micelio a través <strong>de</strong> los poros laterales <strong>de</strong> aquéllos.<br />
Inicialmente, la colonización vascular se produce mediante el crecimiento<br />
3
Introducción<br />
<strong>de</strong> hifas <strong>de</strong>lgadas en la raíz y base <strong>de</strong>l tallo <strong>de</strong> la plántula. Posteriormente,<br />
se <strong>de</strong>sarrollan hifas gruesas irregulares y ramificadas cuyo crecimiento<br />
profuso da lugar a la oclusión <strong>de</strong>l lumen <strong>de</strong> los vasos infectados (Jiménez<br />
Díaz, 1994; Jiménez Díaz et al., 1989a).<br />
La colonización vascular <strong>de</strong> la planta ocurre <strong>de</strong> forma similar por los<br />
aislados <strong>de</strong>l patógeno que causan el síndrome <strong>de</strong> Amarillez y por los que<br />
causan Marchitez. Sin embargo, los aislados causantes <strong>de</strong> Amarillez, menos<br />
virulentos que los <strong>de</strong> Marchitez, inva<strong>de</strong>n la planta más lentamente y con<br />
menor extensión. Para ambos tipos <strong>de</strong> aislados, los síntomas severos<br />
característicos se <strong>de</strong>sarrollan <strong>de</strong>spués <strong>de</strong> la colonización intensa y extensa<br />
<strong>de</strong> los haces xilemáticos <strong>de</strong> la raíz y base <strong>de</strong>l tallo por el hongo, y la<br />
distribución <strong>de</strong> éste a lo largo <strong>de</strong>l eje <strong>de</strong> la planta (Jiménez Díaz, 1994;<br />
Jiménez Díaz et al., 1989a).<br />
Variabilidad patogénica <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris<br />
Las poblaciones <strong>de</strong> F. oxysporum f. sp. ciceris contienen una variabilidad<br />
apreciable en lo concerniente a la capacidad patogénica <strong>de</strong> los aislados que<br />
las componen (Alcalá Jiménez, 1995; Haware y Nene, 1982b; Jiménez Díaz<br />
et al., 1989a, b). Por una parte, los aislados <strong>de</strong> F. oxysporum f. sp. ciceris<br />
pue<strong>de</strong>n ser caracterizados en dos patotipos por el síndrome <strong>de</strong> Amarillez<br />
(patotipo <strong>de</strong> Amarillez) o <strong>de</strong> Marchitez (patotipo <strong>de</strong> Marchitez) que<br />
inducen en cultivares <strong>de</strong> garbanzo susceptibles. A<strong>de</strong>más, superimpuesto<br />
sobre la caracterización patotípica, los aislados pertenecientes a un<br />
patotipo pue<strong>de</strong>n ser clasificados en razas patogénicas en base al patrón <strong>de</strong><br />
reacciones que originan en un conjunto <strong>de</strong> cultivares o líneas <strong>de</strong>nominados<br />
diferenciadores raciales. La primera <strong>de</strong>scripción <strong>de</strong> razas patogénicas en F.<br />
oxysporum f. sp. ciceris es <strong>de</strong>bida a Haware y Nene (1982b), quienes<br />
i<strong>de</strong>ntificaron las razas 1, 2, 3 y 4, según la interacción diferencial que<br />
obtuvieron al inocular 10 cultivares <strong>de</strong> garbanzo (‘Annigeri’, ‘BG-212’,<br />
‘Chafa’, ‘C-104’, ‘CPS-1’, ‘JG-62’, ‘JG-74’, ‘K-850 3/27’, ‘L-550’ y ‘WR-<br />
315’) con aislados <strong>de</strong>l patógeno proce<strong>de</strong>ntes <strong>de</strong> la India.<br />
4
Introducción<br />
La raza 1 se caracteriza por ser no patogénica sobre ‘JG-74’, ‘CPS-1’,<br />
‘BG-212’ y ‘WR-315’; mientras que la raza 2 es no patogénica únicamente<br />
sobre ‘WR-315’, la raza 3 es no patogénica únicamente sobre ‘JG-74’ y la<br />
raza 4 lo es sobre ‘JG-74’ y ‘WR-315’ (Haware y Nene, 1982b).<br />
Trabajos posteriores pusieron <strong>de</strong> manifiesto que <strong>de</strong>terminados aislados<br />
<strong>de</strong> F. oxysporum f. sp. ciceris proce<strong>de</strong>ntes <strong>de</strong> Andalucía son no patogénicos<br />
sobre el cv. JG-62 (Trapero Casas y Jiménez Díaz, 1985). Estos aislados<br />
inducen el síndrome <strong>de</strong> Amarillez en cultivares tipo ‘kabuli’ (semilla gran<strong>de</strong><br />
<strong>de</strong> color beige y forma <strong>de</strong> cabeza <strong>de</strong> carnero) locales; mientras que otros<br />
aislados proce<strong>de</strong>ntes <strong>de</strong> la misma región inducen el síndrome <strong>de</strong> Marchitez<br />
y resultaron ser altamente virulentos sobre la mayoría <strong>de</strong> los cultivares <strong>de</strong><br />
garbanzo (Trapero Casas y Jiménez Díaz, 1985).<br />
La inoculación artificial <strong>de</strong> seis (‘BG-212’, ‘C-104’, ‘CPS-1’, ‘JG-62’,<br />
‘JG-74’ y ‘WR-315’) <strong>de</strong> los 10 cultivares diferenciadores utilizados por<br />
Haware y Nene (1982b) y <strong>de</strong> ‘12-071/10054’, ‘ICCV-2’, ‘ICCV-4’, ‘PV-24’<br />
y ‘P-2245’, con 11 aislados <strong>de</strong> F. oxysporum f. sp. ciceris <strong>de</strong> Andalucía y con<br />
aislados representativos <strong>de</strong> las razas <strong>de</strong>scritas en la India, dio lugar a<br />
asignar dichos aislados a tres nuevas razas, <strong>de</strong>nominadas 0, 5 y 6 (Cabrera<br />
<strong>de</strong> la Colina et al., 1986; Jiménez Díaz et al., 1989b).<br />
La raza 0 es la menos virulenta <strong>de</strong> las razas <strong>de</strong>scritas y se caracteriza por<br />
ser no patogénica sobre el cultivar ‘<strong>de</strong>si’ (semilla pequeña, <strong>de</strong> color oscuro<br />
y perfil anguloso) JG-62, que es susceptible a todas las <strong>de</strong>más razas<br />
<strong>de</strong>scritas. La raza 0 es patogénica sobre ‘C-104’ y ‘12071/10054’. Hasta el<br />
momento, todos los aislados <strong>de</strong> la raza 0 estudiados inducen el síndrome<br />
<strong>de</strong> Amarillez, que se <strong>de</strong>sarrolla a los 30-40 días <strong>de</strong>spués <strong>de</strong> la inoculación<br />
por el método <strong>de</strong> siembra en suelo infestado contenido en macetas (Nene<br />
y Haware, 1980; Trapero Casas y Jiménez Díaz, 1985).<br />
La raza 5 induce el síndrome <strong>de</strong> Marchitez rápida en cultivares<br />
susceptibles, y se caracteriza por no ser patogénica en la línea ‘<strong>de</strong>si’ 12-<br />
071/10054, que es susceptible a la raza 0, pero patogénica en el resto <strong>de</strong><br />
los cultivares diferenciadores inoculados excepto ‘BG-212’ y ‘WR-315’.<br />
Los cvs. CPS-1 y JG-74 son mo<strong>de</strong>radamente susceptibles a la raza 5, por<br />
cuya infección <strong>de</strong>sarrollan un amarilleamiento foliar lento y progresivo que<br />
5
Introducción<br />
es distinguible <strong>de</strong> la amarillez causada por la raza 0 en cultivares<br />
susceptibles (Jiménez Díaz et al., 1989b).<br />
La raza 6, que induce una marchitez vascular similar a la originada por<br />
la raza 5 y las razas <strong>de</strong>scritas en la India, se caracteriza por ser patogénica<br />
sobre ‘C-104’, ‘ICCV-2’, ‘ICCV-4’ y ‘JG-62’, y no patogénica sobre ’12-<br />
071/10054’ (que es susceptible a la raza 1), ‘BG-212’, ‘CPS-1’ y ‘JG-74’<br />
(Jiménez Díaz et al., 1989b).<br />
Estudios posteriores han indicado que ninguna <strong>de</strong> las razas 0, 5 y 6 son<br />
patogénicas sobre ‘Annigeri’, ‘Chafa’, y ‘K-850 3/27’, que son susceptibles<br />
a las razas 1-4 (Jiménez Díaz, no publicado; Halila y Strange, 1996)<br />
La estructura <strong>de</strong> virulencia en poblaciones <strong>de</strong> F. oxysporum f. sp. ciceris<br />
fue reevaluada con una muestra <strong>de</strong> aislados <strong>de</strong>l patógeno más amplia que la<br />
estudiada en el periodo 1983-86 (Cabrera <strong>de</strong> la Colina et al., 1985; Jiménez<br />
Díaz et al., 1989b), incluyendo aislados <strong>de</strong> Andalucía, Extremadura y<br />
Castilla y León en España, California, Marruecos y Túnez. Los resultados<br />
confirmaron que los aislados <strong>de</strong> F. oxysporum f. sp. ciceris pue<strong>de</strong>n ser<br />
clasificados en uno <strong>de</strong> los patotipos <strong>de</strong> Amarillez o Marchitez <strong>de</strong> acuerdo<br />
con el síndrome que causan en cultivares susceptibles <strong>de</strong> garbanzo; y<br />
a<strong>de</strong>más indicaron que ambos patotipos existen en el sur y noroeste <strong>de</strong><br />
España, así como en California y posiblemente Marruecos (Alcalá Jiménez,<br />
1995; Jiménez Díaz et al., 1993a). Por el contrario, los aislados proce<strong>de</strong>ntes<br />
<strong>de</strong> Túnez pertenecen al patotipo <strong>de</strong> Amarillez, característica que ha sido<br />
recientemente confirmada por Halila y Strange (1996).<br />
Los estudios referidos permitieron i<strong>de</strong>ntificar las razas 0, 1, 5 y 6 entre<br />
los aislados investigados, y a su vez constatar que existen ligeras diferencias<br />
fenotípicas entre aislados <strong>de</strong> F. oxysporum f. sp. ciceris <strong>de</strong> una misma raza.<br />
Así, en dichos estudios se distinguieron a aislados proce<strong>de</strong>ntes <strong>de</strong><br />
California y caracterizados patogénicamente como raza 1, pero que<br />
inducen el síndrome <strong>de</strong> Amarillez (raza 1B/C), en contraposición con el<br />
resto <strong>de</strong> aislados <strong>de</strong> esta raza proce<strong>de</strong>ntes <strong>de</strong> California, España, India y<br />
Marruecos, los cuales inducen el síndrome <strong>de</strong> Marchitez (raza 1A)<br />
(Jiménez Díaz et al., 1993a).<br />
6
Introducción<br />
En consecuencia, en cultivares susceptibles <strong>de</strong> garbanzo los aislados<br />
pertenecientes a las razas 0, 1B/C inducen el síndrome <strong>de</strong> Amarillez<br />
<strong>de</strong>scrito anteriormente, mientras que aislados pertenecientes a las razas 1A,<br />
2, 3, 4, 5 y 6 causan el síndrome <strong>de</strong> Marchitez. A<strong>de</strong>más, la información<br />
más reciente indica que las razas 0, 1 (1A, y 1B/C), 5 y 6 <strong>de</strong> F. oxysporum f.<br />
sp. ciceris existen en España y California, mientras que las razas 0 y 1B/C<br />
existen en Siria, Túnez y Turquía; las razas 0, 1A y 6 en Israel; las razas 1A<br />
y 6 en Marruecos, y la raza 0 en el Líbano (Halila y Strange, 1996; Jiménez<br />
Díaz et al., 1993a; Jiménez Díaz, no publicado). Por el contrario, las razas 2, 3<br />
y 4 han sido <strong>de</strong>scritas únicamente en la India hasta el momento, don<strong>de</strong><br />
también existe la raza 1A (Jiménez Díaz, 1994).<br />
I<strong>de</strong>ntificación <strong>de</strong> razas <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris: Pruebas <strong>de</strong><br />
Patogenicidad<br />
La correcta i<strong>de</strong>ntificación <strong>de</strong> las variantes patogénicas <strong>de</strong> F. oxysporum f. sp.<br />
ciceris presentes en una <strong>de</strong>terminada zona <strong>de</strong> cultivo es un aspecto crucial<br />
con vistas a la utilización y <strong>de</strong>sarrollo mediante mejora genética <strong>de</strong><br />
cultivares con resistencia estable. De forma tradicional, la i<strong>de</strong>ntificación <strong>de</strong><br />
razas patogénicas en hongos fitopatógenos se ha llevado a cabo por el<br />
fenotipo <strong>de</strong> las interacciones planta-patógeno, en pruebas <strong>de</strong> patogenicidad<br />
basadas en la inoculación <strong>de</strong> líneas diferenciadoras <strong>de</strong> la planta huésped<br />
con aislados <strong>de</strong>l patógeno.<br />
Para la i<strong>de</strong>ntificación <strong>de</strong> razas <strong>de</strong> F. oxysporum f. sp. ciceris, en nuestro<br />
laboratorio se ha venido utilizando una modificación <strong>de</strong>l método <strong>de</strong>scrito<br />
por Haware y Nene (1982b), utilizando 10 líneas <strong>de</strong> garbanzo, que incluyen<br />
las empleadas por dichos autores a excepción <strong>de</strong> ‘Annigeri’, ‘Chafa’, ‘K-850<br />
3/27’ y ‘L-550’; y añadiendo las ‘P-2245’, ‘12-071/10054’, ‘ICCV-2’ e<br />
‘ICCV-4’ (Jiménez Díaz et al., 1989b) (Tabla 1).<br />
Para la realización <strong>de</strong> las pruebas <strong>de</strong> patogenicidad, la inoculación <strong>de</strong> las<br />
líneas diferenciadoras <strong>de</strong> garbanzo se lleva a cabo mediante la siembra <strong>de</strong><br />
semillas germinadas en suelo infestado artificialmente contenido en<br />
7
Introducción<br />
macetas. Con este propósito, los aislados <strong>de</strong> F. oxysporum f. sp. ciceris no<br />
<strong>de</strong>ben ser mantenidos repetidamente en medios <strong>de</strong> crecimiento sintéticos<br />
<strong>de</strong>bido a mutaciones que ocurren con frecuencia y que están asociadas con<br />
pérdida <strong>de</strong> virulencia. Por ello, los aislados son conservados en suelo<br />
estéril. El inóculo se incrementa en una mezcla <strong>de</strong> arena y harina <strong>de</strong> maíz<br />
(AMA) (9:1, p/p) durante 15 días a 25ºC. Posteriormente el AMA<br />
infestado se mezcla con suelo esterilizado en una proporción <strong>de</strong> 1:12<br />
(p/p). Una vez que el hongo está establecido en esta mezcla <strong>de</strong> suelo<br />
infestado, se siembran las semillas pregerminadas <strong>de</strong> garbanzo en macetas<br />
que contienen dicha mezcla. Este nivel <strong>de</strong> inóculo es suficiente para causar<br />
el 100% <strong>de</strong> mortalidad en ‘JG-62’, una línea <strong>de</strong> garbanzo altamente<br />
susceptible. La reacción <strong>de</strong> la planta a la infección se <strong>de</strong>termina mediante la<br />
severidad <strong>de</strong> los síntomas en una escala <strong>de</strong> 1 a 4 (1=no síntomas; 4=planta<br />
muerta), según el porcentaje <strong>de</strong> área foliar afectada a los 20 y 40 días<br />
<strong>de</strong>spués <strong>de</strong> la inoculación.<br />
Control <strong>de</strong> la Fusariosis Vascular <strong>de</strong>l garbanzo mediante cultivares resistentes<br />
La ecología <strong>de</strong>l patógeno y la epi<strong>de</strong>miología <strong>de</strong> la enfermedad indican que<br />
las epi<strong>de</strong>mias <strong>de</strong> Fusariosis Vascular <strong>de</strong>l garbanzo son <strong>de</strong> naturaleza<br />
monocíclica. En consecuencia, las estrategias más a<strong>de</strong>cuadas para su<br />
control son aquéllas que contribuyen a reducir la cantidad <strong>de</strong>l inóculo<br />
inicial en el suelo, a reducir su eficacia, o a ambas.<br />
La rotación <strong>de</strong> garbanzo con cultivos no huéspe<strong>de</strong>s pue<strong>de</strong> contribuir a<br />
reducir el inóculo <strong>de</strong> sus patógenos que resi<strong>de</strong>n en el suelo. Sin embargo,<br />
el control <strong>de</strong> la Fusariosis Vascular mediante la rotación <strong>de</strong> cultivos no es<br />
efectivo <strong>de</strong>bido a la capacidad <strong>de</strong>l patógeno <strong>de</strong> sobrevivir<br />
prolongadamente en el suelo mediante clamidosporas, así como <strong>de</strong><br />
infectarasintomáticamente otros cultivos <strong>de</strong> plantas leguminosas y no<br />
leguminosas (Cabrera <strong>de</strong> la Colina, 1986; Trapero Casas y Jiménez Díaz,<br />
1985). De forma similar, investigaciones sobre la utilización combinada <strong>de</strong><br />
tratamientos fungicidas <strong>de</strong> la semilla <strong>de</strong> cultivares <strong>de</strong> garbanzo <strong>de</strong> diferente<br />
susceptibilidad, indicaron que algunos fungicidas incrementan<br />
8
Introducción<br />
Tabla 1. Reacción <strong>de</strong> las líneas <strong>de</strong> garbanzo diferenciadoras a la inoculación con las<br />
distintas razas <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris a<br />
Línea<br />
Raza<br />
diferenciadora 0 1A 1B/C 2 3 4 5 6<br />
12-071/10054 S M S R R R R M<br />
JG-62 R S S S S S S S<br />
C-104 M M R/M S S S S M<br />
JG-74 R R R S R R M R<br />
CPS-1 R R R S M M M R<br />
BG-212 R R R S M M R R<br />
WR-315 R R R R S R R R<br />
ICCV-2 R R R S S S S M<br />
ICCV-4 R R R S S S S M<br />
P-2245 S S S S S S S S<br />
a Evaluada en una escala <strong>de</strong> severidad <strong>de</strong> 0-4 según el porcentaje <strong>de</strong> tejido<br />
foliar afectado (0=0%, 1=1-33%, 2=24-66, 3=67-100, 4=planta muerta) a<br />
los 40 días <strong>de</strong> la siembra en una mezcla <strong>de</strong> suelo infestado con AMA<br />
colonizado por el hongo. Las reacciones con severidad media 3 se<br />
consi<strong>de</strong>ran resistentes (R) y susceptibles (S), respectivamente. Las reacciones<br />
<strong>de</strong> severidad intermedia entre dichos valores se consi<strong>de</strong>ran mo<strong>de</strong>radamente<br />
susceptibles (M) (Halila y Strange, 1996; Haware y Nene, 1982b; Jiménez<br />
Díaz et al., 1989b).<br />
9
Introducción<br />
significativamente la emergencia <strong>de</strong> las plántulas y retrasan el comienzo <strong>de</strong><br />
las epi<strong>de</strong>mias en cultivares mo<strong>de</strong>radamente resistentes, pero no en<br />
cultivares altamente susceptibles como es el caso <strong>de</strong> ‘P-2245’. Sin embargo,<br />
ningún tratamiento fungicida proporcionó un nivel <strong>de</strong> control satisfactorio<br />
<strong>de</strong> la enfermedad (Jiménez Díaz y Trapero Casas, 1985).<br />
El método más práctico y económicamente eficiente para el control <strong>de</strong><br />
la Fusariosis Vascular es la utilización <strong>de</strong> cultivares resistentes (Nene y<br />
Reddy, 1987). Sin embargo, la efectividad <strong>de</strong> este método <strong>de</strong> control pue<strong>de</strong><br />
ser disminuida por la existencia <strong>de</strong> razas patogénicas <strong>de</strong> F. oxysporum f. sp.<br />
ciceris (Haware y Nene, 1982b; Jiménez Díaz et al., 1989b; 1993a). Como en<br />
otros patosistemas, el <strong>de</strong>sarrollo <strong>de</strong> genotipos <strong>de</strong> garbanzo resistentes a<br />
esta enfermedad pue<strong>de</strong> ser influido por factores como la genética <strong>de</strong> la<br />
resistencia en la planta y la variabilidad patogénica en el agente. Aún así, se<br />
han conseguido importantes avances en el <strong>de</strong>sarrollo <strong>de</strong> genotipos <strong>de</strong><br />
garbanzo resistentes o tolerantes a enfermeda<strong>de</strong>s individuales (Singh,<br />
1988). Sin embargo, en muchos casos, el germoplasma resistente a una<br />
enfermedad se ha mostrado altamente susceptible a otras enfermeda<strong>de</strong>s<br />
importantes <strong>de</strong>l cultivo.<br />
Estudios realizados en condiciones <strong>de</strong> campo indicaron que la<br />
resistencia a la Fusariosis Vascular en diferentes cultivares <strong>de</strong> garbanzo era<br />
monogénica con dominancia incompleta (Ayyar e Iyer, 1936), o<br />
monogénica y recesiva (López, 1974; Sidhu et al., 1983). Investigaciones<br />
más cuidadosas mostraron que la resistencia a la raza 1 <strong>de</strong> F. oxysporum f.<br />
sp. ciceris en los cvs. CPS-1 y WR-315 es conferida por un único alelo<br />
recesivo, que se encuentra en el mismo locus en ambos genotipos <strong>de</strong><br />
garbanzo (Kumar y Haware, 1982). Asimismo, Kumar y Haware (1982)<br />
sugirieron que en la resistencia a la raza 1A <strong>de</strong>l patógeno pudieran estar<br />
implicados otros genes mayores o complejos poligénicos, ya que los<br />
cruzamientos realizados entre líneas susceptibles y resistentes <strong>de</strong> garbanzo<br />
no mostraban la relación fenotípica esperada <strong>de</strong> 3 (susceptible): 1<br />
(resistente).<br />
Trabajos posteriores indicaron que el tiempo que transcurre hasta el<br />
<strong>de</strong>sarrollo <strong>de</strong> los síntomas en la planta es un componente fenotípico <strong>de</strong><br />
10
Introducción<br />
interés en el análisis <strong>de</strong> la reacción <strong>de</strong> genotipos <strong>de</strong> garbanzo a la<br />
inoculación con razas <strong>de</strong>l patógeno. Así, mientras en la marchitez <strong>de</strong> ‘JG-<br />
62’ (‘marchitez temprana’) los síntomas iniciales aparecen antes <strong>de</strong><br />
transcurridos los 20 días <strong>de</strong>s<strong>de</strong> la inoculación, en ‘C-104’ (‘marchitez<br />
tardía’) los síntomas iniciales son visibles pasados los 21 días <strong>de</strong>spués <strong>de</strong> la<br />
inoculación. A<strong>de</strong>más, esta diferencia es controlada por un único gen,<br />
siendo la marchitez temprana parcialmente dominante sobre la marchitez<br />
tardía (Upadhyaya et al., 1983a).<br />
Estudios <strong>de</strong> las segregaciones <strong>de</strong> cruzamientos entre los cvs. JG-62 y C-<br />
104 y las líneas completamente resistentes BG-212, CPS-1, P-436-2 y WR-<br />
315, así como <strong>de</strong> cruzamientos entre cultivares que mostraban marchitez<br />
tardía, ‘C-104’, ‘K-850 3/27’ y ‘H-208’, indicaron que la resistencia a la raza<br />
1A <strong>de</strong> F. oxysporum f. sp. ciceris parece ser controlada por al menos tres loci<br />
in<strong>de</strong>pendientes, <strong>de</strong>signados H1, H2 y H3 (Singh et al., 1987a, b; Upadhyaya<br />
et al., 1983b). Un alelo recesivo en homocigosis en cualquiera <strong>de</strong> los dos<br />
primeros loci, o el alelo dominante en el tercer locus, <strong>de</strong>termina un retraso<br />
en el <strong>de</strong>sarrollo <strong>de</strong> la marchitez; pero la existencia <strong>de</strong> dos <strong>de</strong> cualesquiera<br />
<strong>de</strong> los tres alelos <strong>de</strong>scritos operativos confiere resistencia completa a la<br />
raza 1A <strong>de</strong>l patógeno.<br />
Investigaciones posteriores por el mismo grupo <strong>de</strong> trabajo indicaron<br />
que la resistencia a la raza 2 <strong>de</strong> F. oxysporum f. sp. ciceris es regulada por un<br />
sistema genético similar al <strong>de</strong>scrito para la raza 1A. El estudio incluyó las<br />
segregaciones F 2 y F 3 <strong>de</strong>l cruzamiento ‘P-165’ (resistente) x ‘C-104’<br />
(susceptible) y las líneas CPS-1 y K-850, controles, respectivamente <strong>de</strong><br />
marchitez temprana y tardía (Gumber et al., 1995). Los resultados<br />
mostraron que la resistencia a la raza 2 <strong>de</strong>l patógeno es regulada por dos<br />
genes, uno <strong>de</strong> los cuales, A, <strong>de</strong>be estar en homocigosis recesiva y el otro,<br />
B, en homocigosis dominante o heterocigosis. Así, la reacción <strong>de</strong><br />
marchitez temprana ocurre en genotipos bb, in<strong>de</strong>pendientemente <strong>de</strong> A; y la<br />
<strong>de</strong> marchitez tardía ocurre en genotipos AB. Estos resultados tienen<br />
importantes implicaciones con respecto a la mejora <strong>de</strong>l garbanzo en cuanto<br />
a la resistencia a las razas 1A y 2 <strong>de</strong> F. oxysporum f. sp. ciceris, ya que pue<strong>de</strong><br />
obtenerse resistencia completa al patógeno mediante cruzamientos<br />
utilizando sólo parentales con resistencia <strong>de</strong>l tipo <strong>de</strong> marchitez tardía,<br />
11
Introducción<br />
aunque sean susceptibles (Singh et al., 1987a; van Rheenen et al., 1992).<br />
Dichos cultivares tienen generalmente buenas características agronómicas<br />
y pue<strong>de</strong>n producir rendimiento aún con ataques <strong>de</strong> la enfermedad,<br />
mientras que la muerte temprana <strong>de</strong> los cultivares <strong>de</strong> marchitez temprana<br />
excluye la producción <strong>de</strong> semilla por cultivos afectados (Gumber et al.,<br />
1995).<br />
Este tipo <strong>de</strong> estudios sobre la genética <strong>de</strong> la resistencia en la planta <strong>de</strong>be<br />
ser realizado para otras razas <strong>de</strong>l patógeno con fines comparativos, ya que<br />
es posible que algunos <strong>de</strong> los genes implicados en la resistencia a las razas<br />
1A y 2 <strong>de</strong> F. oxysporum f. sp. ciceris puedan ser efectivos también contra<br />
otras razas <strong>de</strong>l patógeno.<br />
Los progresos en la i<strong>de</strong>ntificación <strong>de</strong> fuentes <strong>de</strong> resistencia y en el<br />
<strong>de</strong>sarrollo <strong>de</strong> cultivares <strong>de</strong> alto rendimiento y resistentes a la Fusariosis<br />
Vascular han sido significativos (Singh, 1987; Singh y Reddy, 1991). En el<br />
ICRISAT (‘International Crop Research Institute for the Semiarid<br />
Tropics’) hay disponibles más <strong>de</strong> 150 líneas tanto <strong>de</strong>l tipo ‘<strong>de</strong>si’ como <strong>de</strong>l<br />
tipo ‘kabuli’, resistentes a la Fusariosis Vascular (Haware et al., 1989), así<br />
como otras fuentes <strong>de</strong> resistencia a la Fusariosis Vascular y a la<br />
Podredumbre <strong>de</strong> Raíz (Reddy et al., 1990). Dicha resistencia a la Fusariosis<br />
Vascular ha sido incorporada con éxito a cultivares <strong>de</strong> garbanzo ‘<strong>de</strong>si’ y<br />
‘kabuli’ <strong>de</strong> alto rendimiento, incluyendo ‘ICCV-2’, ‘ICCV-3’, ‘ICCV-4’ e<br />
‘ICCV-5’ (Kumar et al., 1985), e ‘ICCV-6’ (Ghanekar et al., 1990)<br />
proce<strong>de</strong>ntes <strong>de</strong> ICRISAT; ‘Surutato 77’, ‘Sonora 80’, ‘Gavilan’, ‘Kino’ y<br />
‘Tubutama’ <strong>de</strong> Méjico (Morales, 1986); ‘UC-15’ y ‘UC-27’ <strong>de</strong> California<br />
(Bud<strong>de</strong>nhagen et al., 1988; Helms et al., 1992) y ‘Andoum 1’ (Halila y<br />
Harrabi, 1990) <strong>de</strong> Túnez.<br />
En colaboración con el Programa <strong>de</strong> Mejora Genética <strong>de</strong>l Garbanzo <strong>de</strong><br />
ICARDA (‘International Center for Agricultural Research in the Dry<br />
Areas’) durante los años 1987-1990 se evaluó la resistencia a la Fusariosis<br />
Vascular <strong>de</strong> 2702 líneas <strong>de</strong> garbanzo ‘kabuli’ (el más frecuente en el Área<br />
Mediterránea y Oriente Próximo) en una parcela naturalmente infestada<br />
por las razas 0, 1A y 5 <strong>de</strong> F. oxysporum f. sp. ciceris en Santaella (Córdoba).<br />
Las líneas consi<strong>de</strong>radas más prometedoras un año fueron reevaluadas en<br />
años sucesivos, y las finalmente seleccionadas fueron inoculadas con las<br />
12
Introducción<br />
razas 0, 1A y 5 <strong>de</strong>l patógeno en condiciones controladas para confirmar la<br />
naturaleza <strong>de</strong> la reacción. Un total <strong>de</strong> 14 líneas fueron seleccionadas por su<br />
resistencia en campo. Cuando dichas líneas y 35 líneas adicionales se<br />
inocularon artificialmente con cada una <strong>de</strong> las tres razas <strong>de</strong>l patógeno,<br />
ocho <strong>de</strong> ellas fueron resistentes a las tres razas; 10 fueron resistentes a las<br />
razas 0 y 1A, y 16 líneas fueron resistentes sólo a la raza 0 (Jiménez Díaz et<br />
al., 1991; 1993a).<br />
Excepto en contados casos, las líneas <strong>de</strong> garbanzo ‘kabuli’ con<br />
resistencia a la Fusariosis Vascular generalmente son susceptibles a la<br />
Rabia causada por D. rabiei, lo cual limita consi<strong>de</strong>rablemente su utilidad en<br />
áreas geográficas don<strong>de</strong> ambas enfermeda<strong>de</strong>s son prevalentes. Por ello, se<br />
<strong>de</strong>sarrolló un programa <strong>de</strong> mejora genética en colaboración con el<br />
Departamento <strong>de</strong> Genética y Mejora Vegetal <strong>de</strong> la ETSIAM <strong>de</strong> la<br />
Universidad <strong>de</strong> Córdoba, el Departamento <strong>de</strong> Agronomía y Mejora <strong>de</strong>l<br />
CIFA-Córdoba y Koipesol S. A., que incluyó entre sus principales<br />
objetivos <strong>de</strong>sarrollar germoplasma <strong>de</strong> garbanzo ‘kabuli’ con resistencia<br />
combinada a las dos enfermeda<strong>de</strong>s. De dicho programa ha resultado la<br />
i<strong>de</strong>ntificación <strong>de</strong> cuatro líneas con semilla <strong>de</strong> interés comercial resistentes a<br />
la raza 5 <strong>de</strong> F. oxysporum f. sp. ciceris y mo<strong>de</strong>radamente resistentes a un<br />
aislado altamente virulento <strong>de</strong> D. rabiei; cuatro líneas resistentes a D. rabiei y<br />
mo<strong>de</strong>radamente resistentes a la raza 5; y una línea resistente tanto a la raza<br />
5 <strong>de</strong> F. oxysporum f. sp. ciceris como a D. rabiei (Navas et al., 1994).<br />
Asimismo, en colaboración con la Estación <strong>de</strong> Introducción <strong>de</strong><br />
Germoplasma Vegetal <strong>de</strong>l USDA en Washington, se evaluaron 52 entradas<br />
<strong>de</strong> 11 especies silvestres <strong>de</strong> Cicer por su resistencia a las razas 0 y 5 <strong>de</strong> F.<br />
oxysporum f. sp. ciceris en condiciones controladas, 47 <strong>de</strong> las cuales fueron<br />
también evaluadas por su resistencia a la raza 5 <strong>de</strong>l patógeno en<br />
condiciones <strong>de</strong> campo, en microparcelas artificialmente infestadas con<br />
aquélla. Mientras que frente a la raza 0, menos virulenta, se i<strong>de</strong>ntificó<br />
resistencia en C. bijugum K. H. Rech, C. canariense Santos Guerra & G. P.<br />
Lewis, C. chorassanicum (Bge) M. Popov., C. cuneatum Hochst. ex Rich., C.<br />
judaicum Boiss, y C. pinnatifidum Juab. & Spach; sólo C. bijugum, C. cuneatum y<br />
C. judaicum contienen resistencia a la raza 5. A<strong>de</strong>más, algunas entradas <strong>de</strong><br />
C. bijugum (PI 458550, PI 458552) y C. judaicum (PI 458559), resistentes a<br />
13
Introducción<br />
las razas 0 y 5 <strong>de</strong> F. oxysporum f. sp. ciceris, también son resistentes a D.<br />
rabiei (Kaiser et al., 1994).<br />
14
Objetivos<br />
Introducción<br />
Como se ha mencionado anteriormente, la estrategia <strong>de</strong> lucha contra la<br />
Fusariosis Vascular <strong>de</strong>l garbanzo más práctica y económicamente eficiente<br />
es la utilización <strong>de</strong> cultivares <strong>de</strong> garbanzo resistentes. La utilización<br />
eficiente <strong>de</strong> los cultivares <strong>de</strong> garbanzo disponibles con resistencia a la<br />
Fusariosis Vascular, requiere información previa y precisa sobre las razas<br />
<strong>de</strong>l patógeno que prevalecen en las distintas áreas <strong>de</strong> cultivo, ya que esta<br />
resistencia es generalmente operativa contra una o algunas <strong>de</strong> las razas <strong>de</strong><br />
F. oxysporum f. sp. ciceris <strong>de</strong>scritas (Jiménez Díaz et al., 1993b; Kaiser et al.,<br />
1994). La i<strong>de</strong>ntificación <strong>de</strong> razas <strong>de</strong>l patógeno mediante pruebas <strong>de</strong><br />
patogenicidad es costosa en espacio, tiempo, y recursos, y pue<strong>de</strong> ser<br />
influida por la variabilidad inherente al sistema experimental. A<strong>de</strong>más,<br />
dicho procedimiento <strong>de</strong> caracterización racial no proporciona información<br />
sobre las relaciones genéticas y la variabilidad existente entre, y <strong>de</strong>ntro <strong>de</strong>,<br />
las diferentes razas. Como consecuencia, tanto en éste, como en otros<br />
patosistemas similares en los que el agente sólo se reproduce asexualmente,<br />
se conoce muy poco sobre el origen <strong>de</strong> nuevas razas, o sobre si la<br />
extensión con que una raza ya conocida, i<strong>de</strong>ntificada en un nuevo lugar, es<br />
el resultado <strong>de</strong> una introducción o representa un origen in<strong>de</strong>pendiente <strong>de</strong><br />
dicha raza (Jiménez Díaz, 1994).<br />
Por estos motivos, a<strong>de</strong>más <strong>de</strong> la correcta i<strong>de</strong>ntificación <strong>de</strong>l agente<br />
causal <strong>de</strong> la enfermedad, es <strong>de</strong> suma importancia un mejor conocimiento<br />
<strong>de</strong> las relaciones genéticas existentes entre aislados <strong>de</strong>l patógeno (Kistler,<br />
1997). Todo ello hace que se hayan investigado vías alternativas, basadas<br />
en las técnicas moleculares disponibles, para la <strong>de</strong>tección e i<strong>de</strong>ntificación<br />
rápida, precisa y fiable <strong>de</strong> los patógenos, y <strong>de</strong> sus razas patogénicas, en<br />
particular <strong>de</strong> hongos mitospóricos.<br />
El gran <strong>de</strong>sarrollo <strong>de</strong> las técnicas <strong>de</strong> biología molecular que ha tenido<br />
lugar en la última década ha hecho posible que su aplicación alcance<br />
disciplinas como la Patología Vegetal (Bridge et al., 1998a, b). La aplicación<br />
<strong>de</strong> herramientas experimentales basadas en técnicas <strong>de</strong> biología molecular,<br />
tales como la reacción en ca<strong>de</strong>na <strong>de</strong> la polimerasa (PCR, ‘Polymerase<br />
Chain Reaction’) (Henson y French, 1993; Mullis y Faloona, 1987; Ward,<br />
15
Introducción<br />
1994) o la hibridación <strong>de</strong> ADN genómico con sondas <strong>de</strong> ADN específicas<br />
<strong>de</strong> una <strong>de</strong>terminada raza (Michelmore y Hulbert, 1987), pue<strong>de</strong> facilitar el<br />
<strong>de</strong>sarrollo <strong>de</strong> una metodología que permita la <strong>de</strong>tección e i<strong>de</strong>ntificación<br />
rápida y precisa <strong>de</strong> las razas <strong>de</strong> F. oxysporum f. sp. ciceris presentes en una<br />
zona <strong>de</strong> cultivo <strong>de</strong>terminada.<br />
De igual manera, este tipo <strong>de</strong> nuevas herramientas experimentales<br />
pue<strong>de</strong> facilitar el análisis <strong>de</strong> la diversidad genética existente en las<br />
poblaciones <strong>de</strong> F. oxysporum f. sp. ciceris, el nivel <strong>de</strong> similaridad entre<br />
aislados <strong>de</strong> una misma raza, y la posible relación evolutiva entre razas<br />
(Gordon y Martyn, 1997, Kistler, 1997). Este conocimiento representaría<br />
un avance significativo en la comprensión <strong>de</strong> la naturaleza genética e<br />
interrelaciones <strong>de</strong> variantes patogénicas <strong>de</strong> hongos que carecen <strong>de</strong><br />
reproducción sexual, y cuyas poblaciones <strong>de</strong>ben tener por tanto una<br />
estructura clonal <strong>de</strong>terminada (Taylor et al., 1999a, b). Con dicho<br />
conocimiento podría ser posible pre<strong>de</strong>cir los cambios que puedan ocurrir<br />
en la estructura racial existente en un suelo al someterlo al uso continuado<br />
<strong>de</strong> cultivares <strong>de</strong> garbanzo que presenten resistencia a una o alguna <strong>de</strong> las<br />
razas <strong>de</strong> F. oxysporum f. sp. ciceris.<br />
16<br />
Para ello, los objetivos <strong>de</strong> la presente Tesis Doctoral han sido:<br />
1. I<strong>de</strong>ntificar marcadores moleculares basados en ADN polimórfico<br />
asociados a las variantes patogénicas <strong>de</strong> F. oxysporum f. sp. ciceris, y el<br />
estudio <strong>de</strong> su utilidad diagnóstica para la caracterización racial <strong>de</strong><br />
aislados <strong>de</strong>l patógeno <strong>de</strong> diverso origen geográfico.<br />
2. Desarrollar una metodología basada en secuencias <strong>de</strong> ADN polimórfico<br />
amplificadas, para la i<strong>de</strong>ntificación <strong>de</strong> F. oxysporum f. sp. ciceris y <strong>de</strong> sus<br />
razas patogénicas mediante PCR específica.<br />
3. Analizar la variabilidad genética existente entre aislados pertenecientes a<br />
las distintas razas <strong>de</strong> F. oxysporum f. sp. ciceris, y <strong>de</strong> la relación genética<br />
entre ellas.<br />
4. Analizar las relaciones filogenéticas entre los aislados <strong>de</strong> F. oxysporum f.<br />
sp. ciceris.
Introducción<br />
En consecuencia, esta Tesis Doctoral se centra en dos temas<br />
principales: (1) la i<strong>de</strong>ntificación molecular específica <strong>de</strong> F. oxysporum f. sp.<br />
ciceris y <strong>de</strong> sus razas patogénicas; y (2) el estudio <strong>de</strong> su diversidad genética y<br />
<strong>de</strong> las relaciones inter e intra raciales. Los Capítulos I y II se centrarán en el<br />
<strong>de</strong>sarrollo <strong>de</strong> marcadores moleculares mediante la utilización <strong>de</strong> la PCR<br />
(Mullis y Faloona, 1987), que permitan la diferenciación específica <strong>de</strong> las<br />
razas <strong>de</strong> F. oxysporum f. sp. ciceris prevalentes en la Región Mediterránea. La<br />
estrategia seguida para la consecución <strong>de</strong> los dos primeros objetivos ha<br />
sido una búsqueda inicial <strong>de</strong> marcadores RAPD (‘Random Amplified<br />
Polymorphic DNA’) (Williams et al., 1990) asociados a una raza, mediante<br />
el análisis <strong>de</strong> una colección compuesta por aislados <strong>de</strong>l patógeno<br />
representativos <strong>de</strong> todas las razas, y <strong>de</strong> diverso origen geográfico (Capítulo<br />
I); y la posterior caracterización genética <strong>de</strong> los marcadores RAPD<br />
i<strong>de</strong>ntificados, su secuenciación, y la puesta a punto <strong>de</strong> la i<strong>de</strong>ntificación <strong>de</strong><br />
F. oxysporum f. sp. ciceris y sus razas mediante PCR (Capítulo II).<br />
Los Capítulos III, IV, y V se centrarán en el estudio <strong>de</strong> la variabilidad<br />
genética existente en F. oxysporum f. sp. ciceris, y a la relación genética entre<br />
sus razas patogénicas. El Capítulo III <strong>de</strong>mostrará un origen monofilético<br />
para la forma specialis ciceris. Los Capítulos IV y V exploran la variabilidad <strong>de</strong><br />
‘fingerprints’ <strong>de</strong> ADN generados, bien mediante sondas <strong>de</strong> ADN<br />
repetitivo (Capítulo IV), o mediante métodos basados en rep-PCR<br />
(Capítulo V).<br />
17
CAPÍTULO I<br />
Capítulo I<br />
I<strong>de</strong>ntificación <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris<br />
y <strong>de</strong> las razas patogénicas 0, 1B/C, 5 y 6 mediante<br />
RAPD-PCR<br />
Introducción<br />
El conocimiento <strong>de</strong> la naturaleza <strong>de</strong> la variación y distribución <strong>de</strong> las razas<br />
patogénicas <strong>de</strong> un agente fitopatógeno es un requisito crucial para el<br />
manejo eficiente <strong>de</strong> una enfermedad mediante cultivares resistentes, así<br />
como para i<strong>de</strong>ntificar cambios en la estructura racial o poblacional que<br />
puedan producirse en dicho agente (McDonald, 1997). En ciertos<br />
patosistemas, la i<strong>de</strong>ntificación <strong>de</strong> razas mediante pruebas <strong>de</strong> patogenicidad<br />
requiere <strong>de</strong> un coste y esfuerzo consi<strong>de</strong>rables, y proporciona una<br />
información mucho más limitada que aquélla que pue<strong>de</strong> ser generada<br />
mediante el uso <strong>de</strong> marcadores genéticos neutrales. Los marcadores<br />
genéticos selectivamente neutrales son especialmente útiles para la<br />
i<strong>de</strong>ntificación <strong>de</strong> razas en patógenos con reproducción estrictamente<br />
asexual, ya que el genoma <strong>de</strong>l patógeno se encuentra fijado al no<br />
producirse recombinación genética <strong>de</strong> origen sexual (Milgroom y Fry,<br />
1997).<br />
19
Capítulo I<br />
Las poblaciones naturales <strong>de</strong> <strong>Fusarium</strong> oxysporum Schlechtend.:Fr.<br />
contienen componentes fitopatógenos que causan enfermeda<strong>de</strong>s<br />
vasculares en una amplia gama <strong>de</strong> plantas cultivadas y no cultivadas.<br />
Mientras que la i<strong>de</strong>ntificación <strong>de</strong> la especie se basa en caracteres<br />
morfológicos <strong>de</strong>l hongo, los componentes patogénicos se clasifican en<br />
formae speciales en base a su gama <strong>de</strong> plantas susceptibles (Sny<strong>de</strong>r y Hansen,<br />
1940). A su vez, las formae speciales se divi<strong>de</strong>n en razas patogénicas según la<br />
patogenicidad específica <strong>de</strong> sus componentes sobre un grupo <strong>de</strong> líneas o<br />
cultivares diferenciadores <strong>de</strong> la planta huésped.<br />
F. oxysporum f. sp. ciceris (Padwick) Matuo & K. Sato, causante <strong>de</strong> la<br />
Fusariosis Vascular <strong>de</strong>l garbanzo (Cicer arietinum L.), ha sido clasificada en<br />
dos patotipos, <strong>de</strong>nominados <strong>de</strong> Amarillez (clorosis progresiva, coloración<br />
vascular, y muerte <strong>de</strong> la planta a los 40 días <strong>de</strong> la inoculación) y <strong>de</strong><br />
Marchitez (clorosis severa, flaci<strong>de</strong>z, coloración vascular, y muerte <strong>de</strong> la<br />
planta antes <strong>de</strong> los 20 días <strong>de</strong> la inoculación), según el síndrome que<br />
causan en cultivares susceptibles en inoculaciones artificiales mediante<br />
siembra en suelo infestado por el patógeno (Trapero Casas, 1983; Trapero<br />
Casas y Jiménez Díaz, 1985). A<strong>de</strong>más, se han <strong>de</strong>scrito ocho razas<br />
patogénicas (0, 1A, 1B/C, 2, 3, 4, 5, y 6) cuya i<strong>de</strong>ntificación se realiza<br />
tradicionalmente mediante pruebas <strong>de</strong> patogenicidad; <strong>de</strong> las cuales las razas<br />
0 y 1B/C pertenecen al patotipo <strong>de</strong> Amarillez y las 1A a 6 pertenecen al<br />
patotipo <strong>de</strong> Marchitez (Haware y Nene, 1982b; Jiménez Díaz et al., 1989b;<br />
1993a). Las pruebas <strong>de</strong> patogenicidad son laboriosas, costosas en tiempo<br />
(50-60 días), requieren infraestructuras importantes y pue<strong>de</strong>n ser influidas<br />
por la variabilidad inherente al sistema experimental. A<strong>de</strong>más, los datos<br />
sobre patogenicidad por si mismos no proporcionan información sobre la<br />
diversidad o relación genética existente entre y <strong>de</strong>ntro <strong>de</strong> las razas <strong>de</strong>l<br />
patógeno. Consecuentemente, se necesitan métodos nuevos y alternativos<br />
a las pruebas <strong>de</strong> patogenicidad que permitan la caracterización rápida e<br />
informativa <strong>de</strong> las razas <strong>de</strong> F. oxysporum f. sp. ciceris.<br />
La amplificación arbitraria <strong>de</strong> fragmentos <strong>de</strong> ADN polimórficos<br />
(RAPD, ‘Random Amplified Polymorphic DNA’) (Williams et al., 1990) ha<br />
sido aplicada extensamente para la <strong>de</strong>tección y caracterización genética <strong>de</strong><br />
hongos fitopatógenos (Brown, 1998; Miller, 1996), incluyendo la<br />
20
Capítulo I<br />
diferenciación <strong>de</strong> razas <strong>de</strong> varias formae speciales <strong>de</strong> F. oxysporum, i. e., formae<br />
speciales cubense (E. F. Smith) W. C. Sny<strong>de</strong>r & H. N. Hansen (Bentley et al.,<br />
1994), dianthi (Prill. & Delacr.) W. C. Sny<strong>de</strong>r & H. N. Hansen (Manulis et<br />
al., 1994; Migheli et al., 1998), pisi (Linf.) W. C. Sny<strong>de</strong>r & H. N. Hansen<br />
(Grajal Martín et al., 1993), y vasinfectum (Atk.) W. C. Sny<strong>de</strong>r & H. N.<br />
Hansen (Assigbetse et al., 1994). Esta técnica, basada en la reacción en<br />
ca<strong>de</strong>na <strong>de</strong> la polimerasa (PCR, ‘Polymerase Chain Reaction’) (Mullis y<br />
Faloona, 1987), utiliza iniciadores cortos (10 nucleótidos) <strong>de</strong> secuencia<br />
aleatoria. El análisis RAPD no requiere el conocimiento previo <strong>de</strong><br />
secuencias <strong>de</strong>l organismo en estudio, y presenta numerosas ventajas como<br />
su rapi<strong>de</strong>z y los escasos requerimientos en cantidad y calidad <strong>de</strong> ADN.<br />
A<strong>de</strong>más, los polimorfismos en nucleótidos individuales son más fácilmente<br />
i<strong>de</strong>ntificables mediante el análisis RAPD que mediante otro tipo <strong>de</strong><br />
análisis, por ejemplo el análisis <strong>de</strong> RFLPs (‘Restriction Fragment Length<br />
Polymorphism’). Ésto hace que los marcadores RAPD sean muy<br />
a<strong>de</strong>cuados para <strong>de</strong>tectar variabilidad intraespecífica (Keim et al., 1989), en<br />
especial en hongos tales como F. oxysporum que carecen <strong>de</strong> reproducción<br />
sexual, y cuyas poblaciones tienen estructura clonal (Taylor et al., 1999b).<br />
En trabajos anteriores, se utilizó el análisis RAPD mediante iniciadores<br />
<strong>de</strong> secuenciación o basados en secuencias conocidas <strong>de</strong> ADN ribosómico<br />
para caracterizar y diferenciar los patotipos <strong>de</strong> Amarillez y Marchitez en F.<br />
oxysporum f. sp. ciceris; sin embargo, dichos iniciadores no proporcionaron<br />
resultados satisfactorios en cuanto a la i<strong>de</strong>ntificación <strong>de</strong> razas patogénicas<br />
(Kelly et al., 1994). No obstante, los resultados <strong>de</strong> dicho estudio han<br />
mostrado ser <strong>de</strong> utilidad para la <strong>de</strong>tección <strong>de</strong>l patotipo <strong>de</strong> Marchitez en<br />
planta y en suelo (García Pedrajas et al., 1999; Kelly et al., 1998).<br />
Los objetivos <strong>de</strong> las investigaciones contenidas en este Capítulo han<br />
sido: (1) <strong>de</strong>mostrar si las diferentes razas patogénicas <strong>de</strong> F. oxysporum f. sp.<br />
ciceris pue<strong>de</strong>n ser distinguidas mediante el análisis RAPD utilizando otros<br />
iniciadores distintos <strong>de</strong> los informativos sobre patotipo; y (2) analizar las<br />
relaciones genéticas y variabilidad existente entre aislados representativos<br />
<strong>de</strong> estas razas.<br />
21
Capítulo I<br />
Materiales y Métodos<br />
Aislados fúngicos<br />
Se han utilizado 99 aislados <strong>de</strong> F. oxysporum f. sp. ciceris representativos <strong>de</strong><br />
las ocho razas patogénicas <strong>de</strong>scritas y <strong>de</strong> un amplio origen geográfico <strong>de</strong>l<br />
patógeno, así como tres aislados no patogénicos <strong>de</strong> F. oxysporum obtenidos<br />
<strong>de</strong> raíces <strong>de</strong> plantas sanas <strong>de</strong> garbanzo (Tabla 2). Todos los aislados <strong>de</strong> F.<br />
oxysporum f. sp. ciceris, a excepción <strong>de</strong> los proce<strong>de</strong>ntes <strong>de</strong> Israel, Líbano,<br />
Siria, Túnez, y Turquía (Grupo D en Tabla 2), fueron caracterizados<br />
racialmente mediante pruebas <strong>de</strong> patogenicidad en trabajos anteriores<br />
(Jiménez Díaz et al., 1993a; Kelly et al., 1994).<br />
Los 99 aislados fúngicos proce<strong>de</strong>n <strong>de</strong> la micoteca <strong>de</strong>l Departamento <strong>de</strong><br />
Protección <strong>de</strong> Cultivos, Instituto <strong>de</strong> Agricultura Sostenible, CSIC,<br />
Córdoba, España. Los aislados Foc–9601 a –9606 proce<strong>de</strong>n <strong>de</strong> Túnez y<br />
fueron proporcionados por el Dr. M. H. Halila (‘Institute Nationale <strong>de</strong> la<br />
Recherche Agronomique’, Ariana, Túnez). Los aislados Foc–9620 a –9632,<br />
–cc21C, –cc22D, –cc62R, y –cc63K, proce<strong>de</strong>ntes <strong>de</strong> Israel, fueron cedidos<br />
por el Prof. J. Katan (‘Department of Plant Pathology and Microbiology,<br />
The Hebrew University of Jerusalem’, Israel). Los aislados etiquetados<br />
como L, Sy, y T, proce<strong>de</strong>n <strong>de</strong> Líbano, Siria y Turquía, respectivamente, y<br />
fueron proporcionados por el Dr. C. Akem (ICARDA, Aleppo, Siria).<br />
Todos los aislados se han mantenido como cultivos monoconídicos en<br />
suelo estéril, en tubos <strong>de</strong> ensayo, a 4ºC, en oscuridad. Los cultivos activos<br />
<strong>de</strong> los aislados se obtuvieron a partir <strong>de</strong>l subcultivo <strong>de</strong> los aislados<br />
almacenados a 4ºC, en agar patata-<strong>de</strong>xtrosa (PDA) a 25ºC y un<br />
fotoperiodo <strong>de</strong> 12 h <strong>de</strong> luz fluorescente y cercana a ultravioleta (NUV) a<br />
36 µEm -2 s -1 .<br />
Para la extracción <strong>de</strong> ADN fúngico se utilizó micelio liofilizado <strong>de</strong> los<br />
aislados. A tal fin, se transfirió 1 ml <strong>de</strong> suspensión <strong>de</strong> conidias (5 x 10 6<br />
conidias/ml) a un matraz con 100 ml <strong>de</strong> caldo <strong>de</strong> patata-<strong>de</strong>xtrosa (PDB).<br />
Los cultivos fueron incubados durante 4 días en un agitador orbital<br />
22
Capítulo I<br />
Tabla 2. Información geográfica y racial sobre los aislados <strong>de</strong> <strong>Fusarium</strong><br />
oxysporum f. sp. ciceris (Foc), y <strong>de</strong> F. oxysporum (Fo) no patogénicos <strong>de</strong><br />
garbanzo, utilizados en el análisis RAPD-PCR<br />
Raza<br />
Aislado(s) a Origen b Patogenicidad Análisis RAPD c<br />
GRUPO A<br />
Foc-7932, -82115, -8317, -8601,<br />
-8602, -8604, -8733, -8901, -8911,<br />
-8912, -8913, -8914, -90111,<br />
-9198, -91100, -91114<br />
España 0 NT<br />
Foc-1987-B EEUU 0 NT<br />
Foc-USA 1633-2RIJ EEUU 1A NT<br />
Foc-1992 R1N India 1A NT<br />
Foc-8905, -8924 España 6 NT<br />
Foc-1987 T EEUU 6 NT<br />
GRUPO B<br />
Foc-7802, -7952, -7982, -8207,<br />
-82108, -82113, -8310, -8413,<br />
-8415, -8717, -9018 JG62,<br />
-9018 PV1, -91105, -91108<br />
España 0 0<br />
Foc-8250 d España NP NP<br />
Foc-1987-W17, -USA 3-1 JG62 EEUU 1B/C 1B/C<br />
Foc-7989 India 1A *<br />
Foc-9166 e, -9168 Marruecos 1A *<br />
Foc-8605, -1992 R2N India 2 2<br />
Foc-8606, -1992 R3N India 3 3<br />
Foc-8607, -1992 R4N India 4 4<br />
Foc-8012, -8257, -8408, -8508, -9035 España 5 5<br />
Foc-USA 1-1 JG62, -1987-W6-1 EEUU 5 5<br />
Foc-9164 Marruecos 6 6<br />
Foc-8272, -8720, -9023, -9093 PV1 España 6 6<br />
GRUPO C<br />
Foc-8503f, -9032 España 0 0 (W)<br />
Foc-9165<br />
Marruecos 1A * (W)<br />
23
Capítulo I<br />
Tabla 2. (Continuación) Información geográfica y racial sobre los aislados<br />
<strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris (Foc), y <strong>de</strong> F. oxysporum (Fo) no<br />
patogénicos <strong>de</strong> garbanzo, utilizados en el análisis RAPD-PCR<br />
Aislado(s) a<br />
24<br />
Raza<br />
Origen b Patogenicidad Análisis RAPD c<br />
Foc-9027 PV1e España 1A * (W)<br />
Foc-USA SD-8415 EEUU 1A * (W)<br />
Foc-9094 JG62 España 5 5 (W)<br />
Foc-USA 6-1 JG62, -USA 14201 EEUU 5 5 (W)<br />
Foc-9170 Marruecos 6 6 (W)<br />
Foc-Tonini<br />
GRUPO D<br />
EEUU 6 6 (W)<br />
Foc-cc21C, -cc62R, -cc63K Israel 0 0 (U)<br />
Foc-L96-5 a -L96-7, -L96-9 a -L96-11 Líbano 0 0 (U)<br />
Foc-Sy96-14-1, -Sy96-18-3, -Sy96-19-1 Siria 0 0 (U)<br />
Foc-9601, -9603 a -9606 Túnez 0 0 (U)<br />
Foc-T96-1-3 Turquía 0 0 (U)<br />
Foc-cc22D Israel 1B/C 1B/C (U)<br />
Foc-9602 Túnez 1B/C 1B/C (U)<br />
Foc-Sy96-10-1 Siria 1B/C 1B/C (U)<br />
Foc-T96-1-1, -T96-1-2, -T96-2-1,<br />
-T96-2-2<br />
Turquía 1B/C 1B/C (U)<br />
Foc-9620, -9622, -9628, -9632 Israel 6 6 (U)<br />
GRUPO E<br />
Fo-9009, -9081, -90105 España NP NP<br />
a Los aislados en los grupos A a D fueron caracterizados racialmente<br />
mediante: A, análisis RAPD utilizando mezclas <strong>de</strong> ADN (ver Materiales y<br />
Métodos: estrategia 1); B, análisis RAPD usando mezclas <strong>de</strong> ADN y ADN<br />
individualizado (ver Materiales y Métodos: estrategia 2); C, análisis RAPD<br />
en un ‘ensayo ciego’; D, pruebas <strong>de</strong> patogenicidad y análisis RAPD en<br />
‘ensayos ciegos’ dobles. Grupo E: aislados <strong>de</strong> F. oxysporum no patogénicos<br />
<strong>de</strong> garbanzo.<br />
b Los aislados <strong>de</strong> EEUU (California), España, y Marruecos proce<strong>de</strong>n <strong>de</strong> la<br />
micoteca <strong>de</strong>l Departamento <strong>de</strong> Protección <strong>de</strong> Cultivos, Instituto <strong>de</strong><br />
Agricultura Sostenible, CSIC, Córdoba. Los aislados <strong>de</strong> la India fueron<br />
proporcionados por el Dr. M. P. Haware, ICRISAT, Hy<strong>de</strong>rabad, India. Los
Capítulo I<br />
aislados proce<strong>de</strong>ntes <strong>de</strong> Túnez fueron proporcionados por el Dr. M. H.<br />
Halila, Institute Nacionale <strong>de</strong> la Recherche Agronomique, Ariana, Túnez.<br />
Los aislados <strong>de</strong> Israel fueron cedidos por el Profesor J. Katan,<br />
Departamento <strong>de</strong> Patología Vegetal y Microbiología, The Hebrew<br />
University of Jerusalem, Israel. Los aislados originarios <strong>de</strong> Líbano, Siria y<br />
Turquía fueron proporcionados por el Dr. C. Akem, ICARDA, Aleppo,<br />
Siria.<br />
c NT = no estudiado; NP = no patogénico <strong>de</strong> garbanzo; U, W = la i<strong>de</strong>ntidad<br />
racial <strong>de</strong> estos aislados era <strong>de</strong>sconocida (U), o conocida con anterioridad a<br />
los análisis RAPD pero no fue revelada al operador hasta la terminación <strong>de</strong><br />
los análisis (W). El asterisco indica que no se encontraron marcadores<br />
RAPD para la raza 1A.<br />
d Aislado clasificado originalmente como raza 0 (Cabrera <strong>de</strong> la Colina, 1986)<br />
pero confirmado posteriormente como aislado <strong>de</strong> F. oxysporum no<br />
patogénico <strong>de</strong> garbanzo (Kelly et al., 1994).<br />
e Aislados clasificados originalmente como pertenecientes a la raza 1A <strong>de</strong> F.<br />
oxysporum f. sp. ciceris, pero caracterizados como raza 6 en pruebas <strong>de</strong><br />
patogenicidad duplicadas (Alcalá Jiménez, 1995).<br />
f Aislado clasificado originalmente como raza 5 (Cabrera <strong>de</strong> la Colina, 1986)<br />
pero confirmado en el presente trabajo como aislado perteneciente a la raza<br />
0 <strong>de</strong> F. oxysporum f. sp. ciceris.<br />
25
Capítulo I<br />
a 125 rpm, 25ºC y 12 h <strong>de</strong> luz. El micelio se recolectó mediante filtrado a<br />
través <strong>de</strong> gasa estéril, se lavó con abundante agua estéril, y <strong>de</strong>spués fue<br />
liofilizado, y almacenado a –20ºC.<br />
Pruebas <strong>de</strong> Patogenicidad<br />
Los aislados <strong>de</strong> F. oxysporum f. sp. ciceris no caracterizados con anterioridad<br />
a este estudio (Grupo D, en Tabla 2) se caracterizaron racialmente<br />
mediante pruebas <strong>de</strong> patogenicidad, en la realización <strong>de</strong> ‘ensayos ciegos’ <strong>de</strong><br />
este trabajo. Para ello, se utilizaron las líneas <strong>de</strong> garbanzo diferenciadoras<br />
raciales P-2245, JG-62, BG-212, C-104, JG-74, CPS-1, WR-315, Annigeri,<br />
Chafa, K850 12-071/10054, y ICCV-4 (Haware y Nene, 1982b; Jiménez<br />
Díaz et al., 1993a).<br />
El inóculo se incrementó en una mezcla <strong>de</strong> arena y harina <strong>de</strong> maíz<br />
(AMA) (Trapero Casas y Jiménez Díaz, 1985) durante 2 semanas a 25ºC, y<br />
un fotoperiodo <strong>de</strong> 12 h <strong>de</strong> luz fluorescente y NUV a 36 µEm -2 s -1 . El AMA<br />
infestado se mezcló con suelo esterilizado en autoclave (arena: limo: turba,<br />
1:1:1, v/v) en una proporción <strong>de</strong> 1:12 (p/p).<br />
Las semillas se <strong>de</strong>sinfestaron superficialmente, se germinaron, y se<br />
sembraron en macetas <strong>de</strong> arcilla <strong>de</strong> 15 cm <strong>de</strong> diámetro (cuatro plantas por<br />
maceta) rellenas <strong>de</strong> la mezcla <strong>de</strong> suelo infestada. Las plantas testigo se<br />
cultivaron en una mezcla <strong>de</strong> AMA no infestado y suelo esterilizado en<br />
autoclave. Las plantas se incubaron en una cámara <strong>de</strong> cultivo ajustada a 24<br />
± 1ºC, 60-90 % humedad relativa, y un fotoperiodo <strong>de</strong> 14 h <strong>de</strong> luz<br />
fluorescente a 36 µEm -2 s -1 . A<strong>de</strong>más, las plantas se fertilizaron<br />
semanalmente con 100 ml <strong>de</strong> una solución nutritiva (Hoagland y Arnon,<br />
1950).<br />
Las plantas se observaron diariamente respecto <strong>de</strong>l <strong>de</strong>sarrollo <strong>de</strong> la<br />
sintomatología <strong>de</strong> la enfermedad. La reacción <strong>de</strong> las plantas a la infección<br />
se evaluó en base a la severidad <strong>de</strong> los síntomas en una escala <strong>de</strong> 0-4 según<br />
el porcentaje foliar afectado (0=0%, 1=1-33%, 2=34-66%, 3=67-100%,<br />
4=planta muerta), a intervalos <strong>de</strong> 7 días, durante 2 meses. Valores<br />
26
Capítulo I<br />
inferiores a 1 se consi<strong>de</strong>raron propios <strong>de</strong> reacción <strong>de</strong> resistencia, y valores<br />
superiores a 3 se consi<strong>de</strong>raron reacciones susceptibles. Los valores <strong>de</strong><br />
enfermedad intermedios fueron consi<strong>de</strong>rados reacciones mo<strong>de</strong>radamente<br />
susceptibles. Una vez terminados los experimentos, se comprobó la<br />
existencia <strong>de</strong> infección vascular en plantas sintomáticas y asintomáticas<br />
mediante el aislamiento <strong>de</strong> <strong>Fusarium</strong> a partir <strong>de</strong> segmentos <strong>de</strong> tallos. Para<br />
ello, los trozos <strong>de</strong> tejido se colocaron sobre agar-jugoV8-oxgall-PCNB<br />
(VOPA), medio selectivo <strong>de</strong> <strong>Fusarium</strong> (Bouhot y Rouxel, 1971), y el<br />
crecimiento polar <strong>de</strong>l hongo <strong>de</strong>s<strong>de</strong> la superficie <strong>de</strong> corte <strong>de</strong>l tallo indicó la<br />
infección vascular <strong>de</strong>l tallo. Se realizaron dos experimentos, cada uno <strong>de</strong><br />
los cuales tuvo un diseño en bloques completos al azar, con tres<br />
repeticiones (maceta con cuatro plantas) para cada combinación aisladolínea<br />
diferenciadora.<br />
Extracción <strong>de</strong> ADN<br />
Para cada aislado fúngico se extrajo ADN genómico a partir <strong>de</strong> 50 mg <strong>de</strong><br />
micelio liofilizado y molido. El ADN se purificó mediante el método<br />
<strong>de</strong>scrito por Rae<strong>de</strong>r y Broda (1985), con ligeras modificaciones. El micelio<br />
molido fue resuspendido en una solución tampón <strong>de</strong> extracción (200 mM<br />
Tris HCl [pH 8,5]; 25 mM NaCl; 25 mM EDTA; 0,5% SDS). La mezcla se<br />
homogenizó cuidadosamente con fenol equilibrado y una mezcla <strong>de</strong><br />
cloroformo : alcohol isoamílico (24:1), y se centrifugó durante 1 h a 13.000<br />
rpm (4ºC). La fase acuosa se transfirió a un nuevo tubo y se sometió a un<br />
tratamiento con ARNasa. Posteriormente, se añadió un volumen <strong>de</strong><br />
cloroformo: alcohol isoamílico (24:1), y se centrifugó <strong>de</strong> nuevo durante 15<br />
min a 13.000 rpm y 4ºC. La fase superior se transfirió a un nuevo tubo, el<br />
ADN se precipitó con isopropanol frío, y a continuación se centrifugó<br />
durante 30 min a 13.000 rpm. El ADN se resuspendió en 100 µl <strong>de</strong> agua<br />
químicamente ultrapura (UHQ), y se almacenó a –20ºC. Con el fin <strong>de</strong><br />
estimar la calidad y concentración <strong>de</strong> las muestras <strong>de</strong> ADN, se analizaron<br />
alícuotas <strong>de</strong> éstas en geles al 0,7% <strong>de</strong> agarosa y tampón Tris-acetato-<br />
EDTA (TAE; 40 mM Tris-acetato y 1mM EDTA, pH 8,0). Las muestras<br />
27
Capítulo I<br />
<strong>de</strong> ADN se diluyeron a una concentración <strong>de</strong> 50-100 ng/µl en agua estéril<br />
para las reacciones <strong>de</strong> PCR.<br />
Análisis RAPD<br />
Las reacciones RAPD (Williams et al., 1990) se realizaron utilizando 40<br />
iniciadores <strong>de</strong> 10 nucleótidos correspondientes a los ‘kits’ OPF y OPI<br />
(Operon Technology, Alameda, CA, EEUU). Las mezclas <strong>de</strong> reacción (25<br />
µl) estuvieron compuestas por 0,5 µM <strong>de</strong> iniciador, 200 µM <strong>de</strong> cada dNTP,<br />
2,5 µl <strong>de</strong> 10 x tampón <strong>de</strong> reacción (50 µM KCl, 10 mM Tris-HCl, pH 9,0<br />
[25 ºC], 1% v/v triton X-100), 2 U <strong>de</strong> Taq ADN Polimerasa (Promega,<br />
Madison, WI, EEUU), 1,5 mM MgCl 2, y 50-100 ng <strong>de</strong> ADN fúngico.<br />
Las amplificaciones se realizaron en varios termocicladores diferentes:<br />
Perkin-Elmer 2400, Perkin-Elmer 9600 (Perkin-Elmer, Norwalk, CT,<br />
EEUU) y PTC 100 (MJ Research Inc., Watertown, MA, EEUU). El ciclo<br />
<strong>de</strong> temperaturas consistió en 4 min <strong>de</strong> <strong>de</strong>snaturalización inicial a 94ºC, 30<br />
ciclos <strong>de</strong> 1 min <strong>de</strong> <strong>de</strong>snaturalización a 94ºC, 1 min <strong>de</strong> complementación<br />
entre el ADN mol<strong>de</strong> y el iniciador a 37ºC, y 3 min <strong>de</strong> polimerización a<br />
72ºC; el ciclo final fue <strong>de</strong> 6 min <strong>de</strong> extensión a 72ºC para producir<br />
fragmentos completos <strong>de</strong> ADN <strong>de</strong> doble ca<strong>de</strong>na. La temperatura entre la<br />
complementación y la polimerización aumentó a 0,6ºC/s.<br />
Los productos <strong>de</strong> amplificación fueron separados mediante<br />
electroforesis en geles <strong>de</strong> agarosa al 1,5% y 1,5 V·cm -1 , teñidos con<br />
bromuro <strong>de</strong> etidio, y visualizados bajo luz ultravioleta. Como marcador <strong>de</strong><br />
peso molecular se utilizó el ‘0,1-kb DNA lad<strong>de</strong>r XIV’ (Roche Diagnostics,<br />
Mannheim, Alemania). Todas las reacciones <strong>de</strong> amplificación fueron<br />
repetidas al menos dos veces y siempre incluyeron controles negativos (sin<br />
ADN mol<strong>de</strong>).<br />
La realización <strong>de</strong>l análisis RAPD estuvo compuesta por dos estrategias<br />
(1 y 2) con el objeto <strong>de</strong> facilitar la selección <strong>de</strong> los iniciadores que<br />
produjesen los perfiles <strong>de</strong> amplificación más informativos. La estrategia 1<br />
consistió en el análisis <strong>de</strong> mezclas <strong>de</strong> ADN <strong>de</strong> aislados pertenecientes a<br />
28
Capítulo I<br />
una misma raza. Para ello, se mezclaron cantida<strong>de</strong>s iguales <strong>de</strong> ADN <strong>de</strong><br />
aislados pertenecientes a una misma raza <strong>de</strong> F. oxysporum f. sp. ciceris. De<br />
esta manera se analizaron un total <strong>de</strong> 60 aislados <strong>de</strong>l patógeno distribuidos<br />
en ocho mezclas <strong>de</strong> ADN. Dichas mezclas <strong>de</strong> ADN estuvieron<br />
compuestas por 32 aislados <strong>de</strong> la raza 0, cinco <strong>de</strong> la raza 1A, dos <strong>de</strong> cada<br />
una <strong>de</strong> las razas 1B/C, 2, 3, y 4, siete <strong>de</strong> la raza 5 y ocho <strong>de</strong> la raza 6 (Tabla<br />
2). Cada mezcla <strong>de</strong> ADN se amplificó utilizando 40 iniciadores Operon.<br />
Todos los iniciadores que generaron fragmentos RAPD asociados a<br />
alguna <strong>de</strong> las razas <strong>de</strong> F. oxysporum f. sp. ciceris fueron seleccionados para el<br />
posterior análisis individualizado <strong>de</strong> los aislados componentes <strong>de</strong> las<br />
mezclas <strong>de</strong> ADN (estrategia 2). El objetivo <strong>de</strong> la estrategia 2 fue confirmar<br />
que los marcadores RAPD i<strong>de</strong>ntificados en las mezclas <strong>de</strong> ADN <strong>de</strong> cada<br />
raza eran igualmente amplificados en cada uno <strong>de</strong> los aislados<br />
componentes <strong>de</strong> dichas mezclas. En esta segunda estrategia <strong>de</strong> estudio se<br />
analizaron un total <strong>de</strong> 38 aislados F. oxysporum f. sp. ciceris (Tabla 2).<br />
Análisis <strong>de</strong> datos<br />
Un total <strong>de</strong> siete iniciadores, cuatro pertenecientes al grupo OPF (OPF-06,<br />
-10, -12, -16) y tres al grupo OPI (OPI-01, -09, -18), amplificaron<br />
polimorfismos consistentes e informativos en los análisis RAPD. Con el<br />
objeto <strong>de</strong> estudiar la relación genética entre aislados pertenecientes a una<br />
misma raza, así como entre razas, se analizó la información RAPD<br />
generada a partir <strong>de</strong> ADN individual <strong>de</strong> aislados F. oxysporum f. sp. ciceris<br />
presentes en las mezclas <strong>de</strong> ADN <strong>de</strong> cada raza, así como un grupo <strong>de</strong><br />
aislados <strong>de</strong>l ‘ensayo ciego’. A tal fin, se construyó una matriz binaria<br />
resultante <strong>de</strong> combinar los datos generados por el análisis RAPD <strong>de</strong> 57<br />
aislados F. oxysporum f. sp. ciceris y cuatro aislados F. oxysporum no<br />
patogénicos con los siete iniciadores. En la matriz se representó la<br />
presencia o ausencia <strong>de</strong> los fragmentos amplificados como ‘1’ ó ‘0’,<br />
respectivamente. Las bandas <strong>de</strong> ADN <strong>de</strong> igual movilidad en el gel (i. e., <strong>de</strong><br />
igual peso molecular) fueron consi<strong>de</strong>radas idénticas. El análisis <strong>de</strong><br />
similaridad entre aislados se realizó mediante el programa NTSYSpc2.0<br />
29
Capítulo I<br />
(Rohlf, 1988) utilizando el ‘unweighted paired group method with<br />
arithmetic averages’ (UPGMA), que se basó en el coeficiente <strong>de</strong><br />
similaridad <strong>de</strong> Jaccard (Sneath and Sokal, 1973). Este coeficiente consi<strong>de</strong>ra<br />
únicamente la coinci<strong>de</strong>ncia en la presencia <strong>de</strong> bandas, y no en su ausencia,<br />
con lo cual muestra una imagen más precisa <strong>de</strong> la similaridad entre<br />
aislados.<br />
Los datos RAPD se utilizaron también para realizar el análisis <strong>de</strong><br />
Varianza <strong>Molecular</strong> (AMOVA, ‘Analysis of <strong>Molecular</strong> Variance’)<br />
(Excoffier et al., 1992), a fin <strong>de</strong> i<strong>de</strong>ntificar y analizar la variación genética<br />
entre aislados. Esta metodología convierte la matriz <strong>de</strong> distancias<br />
fenotípicas en el equivalente análisis <strong>de</strong> varianza, permitiendo así la<br />
estimación <strong>de</strong> los componentes <strong>de</strong> la varianza <strong>de</strong> los haplotipos RAPD,<br />
mediante la partición <strong>de</strong> la varianza total entre la que correspon<strong>de</strong> a la<br />
variación entre poblaciones y la correspondiente a la variación<br />
intrapoblacional. El nivel <strong>de</strong> significación <strong>de</strong> los valores AMOVA se<br />
calculó mediante procedimientos permutacionales no paramétricos.<br />
Resultados<br />
Análisis RAPD <strong>de</strong> mezclas <strong>de</strong> ADN <strong>de</strong> aislados <strong>de</strong> F. oxysporum f. sp.<br />
ciceris pertenecientes a una misma raza<br />
El análisis RAPD inicial utilizando mezclas <strong>de</strong> ADN <strong>de</strong> aislados<br />
pertenecientes a una misma raza <strong>de</strong>l patógeno ha resultado una estrategia<br />
muy eficiente en este trabajo ya que ha permitido estudiar <strong>de</strong> un gran<br />
número <strong>de</strong> combinaciones aislado-iniciador. En ningún caso hubo<br />
amplificación en las reacciones que sirvieron <strong>de</strong> control. Para la mayor<br />
parte <strong>de</strong> los iniciadores, los perfiles <strong>de</strong> amplificación RAPD fueron<br />
reproducibles entre experimentos tanto cuando se utilizó ADN extraído<br />
<strong>de</strong>l mismo micelio liofilizado, como cuando se utilizó ADN <strong>de</strong> micelio<br />
obtenido <strong>de</strong> cultivos nuevos <strong>de</strong> los aislados F. oxysporum f. sp. ciceris.<br />
Aquellos iniciadores para los que los perfiles <strong>de</strong> amplificación RAPD no<br />
30
Capítulo I<br />
fueron reproducibles fueron <strong>de</strong>scartados. Los perfiles RAPD fueron<br />
igualmente reproducibles cuando las amplificaciones fueron realizadas con<br />
distintos termocicladores, y por distintos operadores <strong>de</strong>l mismo<br />
laboratorio y <strong>de</strong> diferente laboratorio, i. e., ‘Microbial Physiology Group’,<br />
‘King’s College’, Universidad <strong>de</strong> Londres.<br />
De los 40 iniciadores utilizados para amplificar las mezclas <strong>de</strong> ADN, 12<br />
(OPF-05, -06, -08, -10, -12, -16, -18, -19; y OPI-01, -09, -18, -19)<br />
produjeron 31 bandas <strong>de</strong> ADN polimórficas. En los perfiles <strong>de</strong><br />
amplificación realizados con dichos iniciadores se i<strong>de</strong>ntificaron marcadores<br />
RAPD putativos para todas las razas F. oxysporum f. sp. ciceris <strong>de</strong> este<br />
trabajo, a excepción <strong>de</strong> la raza 1A. Para esta raza 1A, el análisis <strong>de</strong> las<br />
mezclas <strong>de</strong> ADN produjo bandas <strong>de</strong> 1,2 y 2,7 kb que también fueron<br />
amplificadas en la mezcla <strong>de</strong> ADN <strong>de</strong> aislados <strong>de</strong> la raza 6 <strong>de</strong>l patógeno.<br />
De igual manera, la banda <strong>de</strong> 1,5 kb generada por OPF-12 a partir <strong>de</strong><br />
ADN <strong>de</strong> cualquiera <strong>de</strong> las razas fue consi<strong>de</strong>rada marcadora <strong>de</strong> la forma<br />
specialis ciceris.<br />
Análisis RAPD <strong>de</strong> ADN <strong>de</strong> aislados individuales <strong>de</strong> cada raza<br />
Con el objeto <strong>de</strong> confirmar la especificidad <strong>de</strong> las bandas RAPD<br />
marcadoras <strong>de</strong> raza seleccionadas inicialmente <strong>de</strong>l análisis <strong>de</strong> mezclas <strong>de</strong><br />
ADN, se realizaron análisis RAPD utilizando ADN individual <strong>de</strong> los<br />
aislados componentes <strong>de</strong> dichas mezclas, y los 12 iniciadores que<br />
generaron los marcadores inicialmente.<br />
Siete <strong>de</strong> los 12 iniciadores amplificaron un total <strong>de</strong> 17 bandas <strong>de</strong> ADN,<br />
generando perfiles <strong>de</strong> amplificación RAPD iguales para todos los aislados<br />
<strong>de</strong> cada raza (Fig. 1 y 2). Esta consistencia confirmó la especificidad <strong>de</strong> las<br />
bandas <strong>de</strong> ADN marcadoras <strong>de</strong> las razas 0, 1B/C, 2, 3, 4, 5, y 6 (Tabla 3).<br />
El aislado Foc–8250, representante <strong>de</strong> la raza 0, fue una excepción ya que<br />
produjo un perfil RAPD completamente diferente a los obtenidos <strong>de</strong> los<br />
otros 31 aislados <strong>de</strong> dicha raza, in<strong>de</strong>pendientemente <strong>de</strong>l iniciador o el<br />
ADN utilizados (ADN mezcla o ADN individual) (Fig. 1 y 2). Esta<br />
discrepancia fue confirmada mediante la realización <strong>de</strong> nuevos ensayos <strong>de</strong><br />
31
Capítulo I<br />
patogenicidad, que indicaron que Foc–8250 es un aislado <strong>de</strong> F. oxysporum<br />
no patogénico <strong>de</strong> garbanzo (Kelly et al., 1994).<br />
Confirmación <strong>de</strong> la caracterización racial mediante análisis RAPD<br />
La utilidad <strong>de</strong>l análisis RAPD para la i<strong>de</strong>ntificación <strong>de</strong> razas <strong>de</strong> F. oxysporum<br />
f. sp. ciceris fue validada mediante ‘ensayos ciegos’, en los que se utilizaron<br />
10 nuevos aislados <strong>de</strong>l patógeno seleccionados <strong>de</strong> forma arbitraria <strong>de</strong> entre<br />
los disponibles en nuestra colección <strong>de</strong> cultivos. Estos aislados (<strong>de</strong>signados<br />
‘W’ en la Tabla 2) habían sido caracterizados racialmente con anterioridad<br />
mediante pruebas <strong>de</strong> patogenicidad. Asimismo, se incluyeron en los<br />
análisis tres aislados <strong>de</strong> F. oxysporum no patogénicos <strong>de</strong> garbanzo. Todos<br />
estos aislados fueron caracterizados racialmente mediante análisis RAPD, y<br />
posteriormente se comparó dicha caracterización con su i<strong>de</strong>ntidad racial<br />
<strong>de</strong>terminada con bioensayos.<br />
Los análisis RAPD i<strong>de</strong>ntificaron correctamente la raza a la cual<br />
pertenecían los aislados <strong>de</strong> F. oxysporum f. sp. ciceris estudiados. Las bandas<br />
RAPD que se señalan a continuación son diagnósticas para las siguientes<br />
razas <strong>de</strong>l patógeno: raza 0, 0,9 kb (OPI-09); raza 1B/C, 0,53 kb (OPI-09),<br />
0,53 kb (OPI-18), 1,9 kb (OPF-06), 0,51 kb y 1,1 kb (OPF-10); raza 2, 0,9<br />
kb (OPF-12); raza 3, 2,0 kb (OPF-06); raza 4, 0,95 kb (OPF-10); raza 5, 0,9<br />
kb (OPF-10); y raza 6, 1,3 kb (OPI-09) (Fig. 1 y 2). La banda RAPD 1,5 kb<br />
(OPF-12) fue confirmada como diagnóstica <strong>de</strong> la forma specialis ciceris (Tabla<br />
3).<br />
El aislado Foc–8503 había sido i<strong>de</strong>ntificado como raza 5, previamente,<br />
mediante bioensayos pero los análisis RAPD dieron como resultado un<br />
perfil <strong>de</strong> amplificación RAPD característico <strong>de</strong> la raza 0. Posteriores<br />
ensayos <strong>de</strong> patogenicidad con este aislado indicaron que causa el típico<br />
síndrome <strong>de</strong> Amarillez (datos no mostrados) e i<strong>de</strong>ntificaron a este aislado<br />
como perteneciente a la raza 0.<br />
32<br />
El análisis RAPD <strong>de</strong> los tres aislados <strong>de</strong> F. oxysporum no patogénicos
2,6<br />
1,<br />
1,<br />
0,<br />
A B<br />
M 1 2 3 4 5 6 7 8<br />
M 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25<br />
Capítulo I<br />
0,65<br />
kb<br />
Figura 1. Amplificaciones RAPD generadas mediante el iniciador aleatorio OPF-<br />
16 utilizando mezclas <strong>de</strong> ADN <strong>de</strong> aislados pertenecientes a la misma raza (A), y<br />
ADN <strong>de</strong> aislados individuales (B) representativos <strong>de</strong> cada raza <strong>de</strong> <strong>Fusarium</strong><br />
oxysporum f. sp. ciceris. Los números a la izquierda son los pesos moleculares (kb)<br />
<strong>de</strong>l ‘0,1kb DNA lad<strong>de</strong>r’ (Roche Diagnostics) (carril M). El número situado a la<br />
<strong>de</strong>recha correspon<strong>de</strong> al peso molecular <strong>de</strong> la banda marcadora <strong>de</strong> la raza 0. A,<br />
carriles 1-8, mezclas <strong>de</strong> ADN <strong>de</strong> las razas 0, 1B/C, 1A, 2, 3, 4, 5, y 6. B, carriles:<br />
9-19, aislados <strong>de</strong> la raza 0 Foc–7802, –9018 JG62, –82108, –8717, –7952, –8207,<br />
–9032, –9601, –91108, –9604, –82113; 20-22, aislados <strong>de</strong> la raza 1B/C Foc–USA<br />
3-1JG62, –1987 W17; 23-26, aislados <strong>de</strong> la raza 1A Foc–7989, raza 5 Foc–8012,<br />
raza 6 Foc–9093 PV1, y aislado <strong>de</strong> F. oxysporum no patogénico <strong>de</strong> garbanzo Fo–<br />
9009, respectivamente.<br />
33
Capítulo I<br />
indicó que éstos presentaron ciertas similarida<strong>de</strong>s con los aislados <strong>de</strong> F.<br />
oxysporum f. sp. ciceris <strong>de</strong> la raza 0. Los fragmentos RAPD <strong>de</strong> 0,39 kb<br />
(iniciador OPF-12) y 0,65 kb (iniciador OPF-16) (Fig. 1) que habían<br />
i<strong>de</strong>ntificado a la raza 0 en análisis previos, fueron también amplificados<br />
con estos iniciadores a partir <strong>de</strong> ADN <strong>de</strong> algunos <strong>de</strong> los aislados no<br />
patogénicos <strong>de</strong> F. oxysporum.<br />
Con objeto <strong>de</strong> reconfirmar la vali<strong>de</strong>z <strong>de</strong>l análisis RAPD para la<br />
i<strong>de</strong>ntificación racial <strong>de</strong> los aislados <strong>de</strong> F. oxysporum f. sp. ciceris, se llevaron a<br />
cabo ‘ensayos ciegos dobles’ <strong>de</strong> 29 aislados no caracterizados racialmente<br />
proce<strong>de</strong>ntes <strong>de</strong> Israel, Líbano, Siria, Túnez y Turquía (Grupo D en Tabla<br />
2). Cada aislado fue caracterizado racialmente mediante el análisis RAPD<br />
utilizando los iniciadores OPI-01, OPI-09, OPI-18, OPF-06, OPF-10,<br />
OPF-12, y OPF-16; y mediante pruebas <strong>de</strong> patogenicidad sobre líneas<br />
diferenciadoras.<br />
Los resultados <strong>de</strong> ambos análisis indicaron un 100% <strong>de</strong> coinci<strong>de</strong>ncia en<br />
la i<strong>de</strong>ntificación racial <strong>de</strong> cada aislado. Los 21 aislados <strong>de</strong> Líbano, Siria,<br />
Túnez, y Turquía pertenecen al patotipo <strong>de</strong> Amarillez, <strong>de</strong> los cuales 16<br />
fueron <strong>de</strong> la raza 0 y cinco <strong>de</strong> la raza 1B/C (Tabla 2). De igual manera, <strong>de</strong><br />
los ocho aislados proce<strong>de</strong>ntes <strong>de</strong> Israel, cuatro pertenecen al patotipo <strong>de</strong><br />
Amarillez (tres <strong>de</strong> la raza 0 y uno <strong>de</strong> la raza 1B/C) y cuatro fueron<br />
clasificados como patotipo <strong>de</strong> Marchitez y fueron caracterizados como<br />
raza 6 (Tabla 2).<br />
Análisis <strong>de</strong> los datos RAPD<br />
Los análisis RAPD generaron un total <strong>de</strong> 160 fragmentos <strong>de</strong> ADN. El<br />
análisis UPGMA <strong>de</strong> los datos RAPD distribuyó los aislados F. oxysporum f.<br />
sp. ciceris en tres grupos principales (‘clusters’) (I, II, III) cada una <strong>de</strong> los<br />
cuales compartió aproximadamente un 72% <strong>de</strong> similaridad. A<strong>de</strong>más, estos<br />
‘clusters’ estuvieron altamente correlacionados con la capacidad <strong>de</strong> los<br />
aislados <strong>de</strong> inducir el síndrome <strong>de</strong> Amarillez o <strong>de</strong> Marchitez (Fig. 3). Por el<br />
34
2,6<br />
1,<br />
1,<br />
0,<br />
A<br />
M 1 2 3 4 5 6 7 8<br />
B<br />
M 9 11 13 15 17 19 21 23 25 27 29<br />
Capítulo I<br />
0,9 kb<br />
Raza<br />
Figura 2. Amplificaciones RAPD generadas mediante el iniciador aleatorio OPF-<br />
10 utilizando mezclas <strong>de</strong> ADN <strong>de</strong> aislados pertenecientes a la misma raza (A), y<br />
ADN <strong>de</strong> aislados individuales (B) representativos <strong>de</strong> cada raza <strong>de</strong> <strong>Fusarium</strong><br />
oxysporum f. sp. ciceris. Los números a la izquierda son los pesos moleculares (kb)<br />
<strong>de</strong>l ‘0,1kb DNA lad<strong>de</strong>r’ (Roche Diagnostics) (carril M). El número situado a la<br />
<strong>de</strong>recha correspon<strong>de</strong> al peso molecular <strong>de</strong> la banda marcadora <strong>de</strong> la raza 5. A,<br />
carriles 1-8, mezclas <strong>de</strong> ADN <strong>de</strong> las razas 0, 1B/C, 1A, 2, 3, 4, 5, y 6. B, carriles:<br />
9-12, aislados <strong>de</strong> la raza 0 Foc–7802, –91108, –9018 JG62, –9601; 13-16, aislados<br />
<strong>de</strong> la raza 1A Foc–9027 PV1, –9165, –9166, –7989; 17-23, aislados <strong>de</strong> la raza 5<br />
Foc–8012, –8508, –9035, –USA 1-1 JG62, –USA 14201, –9094 JG62, –1987 W6-<br />
1; 24-27, aislados <strong>de</strong> la raza 6 Foc–Tonini, –9023, –9164, –9093 PV1; y 28-29,<br />
aislados <strong>de</strong> F. oxysporum no patogénicos <strong>de</strong> garbanzo Fo–8250 y –9009,<br />
respectivamente.<br />
35
Capítulo I<br />
Tabla 3. Nombre y secuencia <strong>de</strong> los siete iniciadores seleccionados por su<br />
capacidad <strong>de</strong> producir polimorfismos <strong>de</strong> ADN informativos para la<br />
i<strong>de</strong>ntificación <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris y <strong>de</strong> sus razas patogénicas<br />
mediante el análisis RAPD a<br />
36<br />
Nombre Secuencia (5’–3’)<br />
Bandas <strong>de</strong> ADN (kb)<br />
polimórfico amplificadas Raza patogénica<br />
OPI-01 ACCTGGACAC 1,0 1A<br />
1,2 0, 1A, 1B/C, 6<br />
2,7 1A, 6<br />
OPI-09 TGGAGAGCAG 0,53 1B/C<br />
0,9 0<br />
1,0 6<br />
1,4 6<br />
OPI-18 TGCCCAGCCT 0,53 1B/C<br />
1,0 2, 3, 4<br />
OPF-06 GGGAATTCGG 1,9 1B/C<br />
2,0 3<br />
OPF-10 GGAAGCTTGG 0,51 1B/C<br />
0,95 4<br />
0,9 5<br />
1,1 1B/C<br />
OPF-12 ACGGTACCAG 0,39 b 0<br />
0,9 2<br />
1,5 F. o. ciceris c<br />
OPF-16 GGAGTACTGG 0,65 b 0<br />
a Los análisis RAPD se realizaron utilizando ADN individualizado <strong>de</strong> aislados<br />
presentes en el ADN mezcla <strong>de</strong> una raza.<br />
b También amplificada en los análisis RAPD utilizando ADN <strong>de</strong> algunos<br />
aislados <strong>de</strong> F. oxysporum no patogénicos <strong>de</strong> garbanzo.<br />
c Banda RAPD i<strong>de</strong>ntificada como marcadora <strong>de</strong> la forma specialis ciceris por ser<br />
amplificada a partir <strong>de</strong> ADN <strong>de</strong> aislados pertenecientes a todas las razas <strong>de</strong><br />
F. oxysporum f. sp. ciceris, pero no <strong>de</strong> aislados <strong>de</strong> F. oxysporum no patogénicos<br />
<strong>de</strong> garbanzo.
Capítulo I<br />
contrario, los aislados <strong>de</strong> F. oxysporum no patogénicos <strong>de</strong> garbanzo no se<br />
agruparon con los aislados <strong>de</strong> F. oxysporum f. sp. ciceris. Todos los aislados<br />
<strong>de</strong> la raza 0, que causan el síndrome <strong>de</strong> Amarillez, constituyeron un<br />
‘cluster’ (I) con una similaridad media aproximada entre aislados <strong>de</strong>l 82%.<br />
Los aislados <strong>de</strong> la raza 1B/C, que también inducen el síndrome <strong>de</strong><br />
Amarillez, se agruparon juntos formando otro ‘cluster’ (II). Todos los<br />
aislados pertenecientes al patotipo <strong>de</strong> Amarillez, que incluye a los aislados<br />
<strong>de</strong> las razas 0 y 1B/C, compartieron un 73% <strong>de</strong> similaridad media.<br />
Todos los aislados causantes <strong>de</strong> síndrome <strong>de</strong> Marchitez, que compren<strong>de</strong><br />
a los aislados <strong>de</strong> las razas 1A, 2, 3, 4, 5, y 6, formaron un tercer ‘cluster’<br />
(III) que compartió aproximadamente un 82% <strong>de</strong> similaridad media. Estos<br />
aislados <strong>de</strong> marchitez estuvieron distribuidos en varios subgrupos con una<br />
similaridad <strong>de</strong>l 80% al 100%. Los aislados <strong>de</strong> las razas 2, 3, 4, proce<strong>de</strong>ntes<br />
<strong>de</strong> la India constituyeron grupos distinguibles entre sí y separados <strong>de</strong> las<br />
razas 1A, 5, y 6, <strong>de</strong>ntro <strong>de</strong>l ‘cluster’ <strong>de</strong> aislados <strong>de</strong> Marchitez (Fig. 3). No<br />
existió asociación alguna entre la distribución <strong>de</strong> los aislados en el<br />
<strong>de</strong>ndrograma generado a partir <strong>de</strong> los datos RAPD y el origen geográfico<br />
<strong>de</strong> los aislados, a excepción <strong>de</strong> la indicada para los aislados <strong>de</strong> la India.<br />
La variación genética <strong>de</strong> F. oxysporum f. sp. ciceris según la asignación <strong>de</strong><br />
los aislados a un patotipo (Amarillez, Marchitez) y su origen geográfico se<br />
estudió mediante el análisis AMOVA (Tabla 4). Los resultados <strong>de</strong>l<br />
AMOVA mostraron que las diferencias entre aislados pertenecientes a<br />
cada uno <strong>de</strong> los dos patotipos fueron altamente significativas (P < 0,001).<br />
Dentro <strong>de</strong>l grupo <strong>de</strong> aislados <strong>de</strong>l patotipo <strong>de</strong> Amarillez, las diferencias<br />
genéticas entre las razas 0 y 1B/C fueron también altamente significativas,<br />
siendo un 54% <strong>de</strong> la variación total atribuible a las diferencias entre razas.<br />
El AMOVA también indicó que no hubo diferencias significativas entre<br />
aislados que inducen el síndrome <strong>de</strong> Marchitez (razas 1A, 5 y 6)<br />
proce<strong>de</strong>ntes <strong>de</strong> la región Mediterránea y <strong>de</strong> EEUU. Sin embargo, si hubo<br />
diferencias altamente significativas (P < 0,001) entre el grupo <strong>de</strong> aislados<br />
<strong>de</strong>l patotipo <strong>de</strong> Marchitez proce<strong>de</strong>ntes <strong>de</strong> la India (razas 1A, 2, 3 y 4) y los<br />
proce<strong>de</strong>ntes <strong>de</strong> la región Mediterránea y EEUU conjuntamente (razas 1A,<br />
5 y 6). Asimismo, el AMOVA indicó que un 36% <strong>de</strong> la variación genética<br />
total era <strong>de</strong>bida a diferencias entre los aislados pertenecientes al patotipo<br />
37
Capítulo I<br />
<strong>de</strong> Marchitez (Tabla 4), y esta proporción aumentó cuando se excluyó <strong>de</strong>l<br />
análisis la raza 1A <strong>de</strong> la India (datos no mostrados).<br />
Discusión<br />
La i<strong>de</strong>ntificación rápida y precisa <strong>de</strong> las razas <strong>de</strong> F. oxysporum f. sp. ciceris es<br />
<strong>de</strong> crucial importancia para el <strong>de</strong>sarrollo y utilización eficiente <strong>de</strong> cultivares<br />
<strong>de</strong> garbanzo resistentes a la Fusariosis Vascular. Nuestro objetivo principal<br />
en el trabajo contenido en este Capítulo fue <strong>de</strong>terminar si las diferentes<br />
razas <strong>de</strong> este patógeno podían ser distinguidas entre sí mediante el análisis<br />
RAPD. Los resultados alcanzados respaldan dicha hipótesis, ya que se han<br />
i<strong>de</strong>ntificado fragmentos <strong>de</strong> ADN amplificados por iniciadores arbitrarios<br />
en análisis RAPD que son diagnósticos <strong>de</strong> F. oxysporum f. sp. ciceris, así<br />
como <strong>de</strong> las razas 0, 1B/C, 5, y 6 <strong>de</strong>l patógeno.<br />
La aplicabilidad general y fiabilidad <strong>de</strong>l método han sido confirmadas al<br />
i<strong>de</strong>ntificar correctamente, mediante ‘ensayos ciegos’, la raza patogénica a la<br />
que pertenecían aislados <strong>de</strong> F. oxysporum f. sp. ciceris caracterizados<br />
patogénicamente con anterioridad a este trabajo, así como aislados no<br />
caracterizados previamente. El bajo número <strong>de</strong> aislados representativos <strong>de</strong><br />
las razas 2, 3, y 4 disponible para el estudio ha impuesto limitaciones en la<br />
confirmación <strong>de</strong> la capacidad diagnóstica <strong>de</strong> los marcadores asociados a<br />
dichas razas. En cambio, el mayor número <strong>de</strong> aislados <strong>de</strong> las razas 0,<br />
1B/C, 5, y 6 que se han analizado, apoyan la consistencia y utilidad <strong>de</strong> los<br />
marcadores RAPD i<strong>de</strong>ntificados para la caracterización <strong>de</strong> estas razas.<br />
En los últimos años, alguno trabajos realizados en otras formae speciales<br />
<strong>de</strong> F. oxysporum, similares al aquí presentado, no han tenido éxito al tratar<br />
<strong>de</strong> relacionar marcadores RAPD y razas patogénicas <strong>de</strong>l agente (Woo et al.,<br />
1996). En cambio, existen otros trabajos que han <strong>de</strong>mostrado la utilidad <strong>de</strong><br />
la técnica RAPD para este propósito (Assigbetse et al., 1994; Grajal-Martín<br />
38
0.00 0.25 0.50 0.75<br />
7802<br />
7952<br />
8207<br />
8413<br />
9032<br />
9605<br />
9606<br />
9603<br />
8503<br />
7982<br />
82108<br />
8310<br />
8415<br />
82113<br />
91105<br />
9018 PV1<br />
9018 JG62<br />
9601<br />
8717<br />
91108<br />
9604<br />
9602<br />
1987-W17<br />
USA3-1 JG62<br />
9027 PV1<br />
Tonini<br />
9165<br />
9093PV1<br />
9164<br />
9170<br />
9166<br />
7989<br />
9168<br />
8720<br />
9620<br />
9023<br />
9622<br />
9628<br />
9632<br />
8257<br />
8408<br />
8508<br />
USA1-1 JG62<br />
1987-W6-1<br />
USA14201<br />
9035<br />
9094 JG62<br />
8272<br />
USA6-1 JG62<br />
8012<br />
USA-SD-8415<br />
8605<br />
1992R2N<br />
8606<br />
1992R3N<br />
8607<br />
1992R4N<br />
9081<br />
90105<br />
8250<br />
9009<br />
1.00<br />
Raza<br />
patogénica Origen<br />
Raza<br />
patogénica geográfico<br />
Origen<br />
geográfico<br />
0<br />
0<br />
0<br />
0<br />
0<br />
0<br />
0<br />
0<br />
0<br />
0<br />
0<br />
0<br />
0<br />
0<br />
0<br />
0<br />
0<br />
0<br />
0<br />
0<br />
0<br />
1B/C<br />
1B/C<br />
1B/C<br />
1A<br />
6<br />
1A<br />
6<br />
6<br />
6<br />
1A<br />
1A<br />
1A<br />
6<br />
6<br />
6<br />
6<br />
6<br />
6<br />
5<br />
5<br />
5<br />
5<br />
5<br />
5<br />
5<br />
5<br />
6<br />
5<br />
5<br />
1A<br />
2<br />
2<br />
3<br />
3<br />
4<br />
4<br />
NP<br />
NP<br />
NP<br />
NP<br />
España<br />
España<br />
España<br />
España<br />
España<br />
Túnez<br />
Túnez<br />
Túnez<br />
España<br />
España<br />
España<br />
España<br />
España<br />
España<br />
España<br />
España<br />
España<br />
Túnez<br />
España<br />
España<br />
Túnez<br />
Túnez<br />
EEUU<br />
EEUU<br />
España<br />
EEUU<br />
Marruecos<br />
España<br />
Marruecos<br />
Marruecos<br />
Marruecos<br />
India<br />
Marruecos<br />
España<br />
Israel<br />
Israel<br />
Israel<br />
Israel<br />
Israel<br />
España<br />
España<br />
España<br />
EEUU<br />
EEUU<br />
EEUU<br />
España<br />
España<br />
España<br />
EEUU<br />
España<br />
EEUU<br />
India<br />
India<br />
India<br />
India<br />
India<br />
India<br />
España<br />
España<br />
España<br />
España<br />
Capítulo I<br />
Amarillez Marchitez<br />
Cluster I CII Cluster III<br />
Figura 3. Dendrograma obtenido mediante el análisis UPGMA <strong>de</strong> los datos que<br />
generó el análisis RAPD <strong>de</strong> 57 aislados <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris y cuatro<br />
aislados <strong>de</strong> F. oxysporum no patogénicos <strong>de</strong> garbanzo, con los iniciadores OPI-01,<br />
OPI-09, OPI-18, OPF-06, OPF-10, OPF-12, y OPF-16. La escala inferior<br />
correspon<strong>de</strong> a la similaridad entre aislados basada en el coeficiente <strong>de</strong> Jaccard.<br />
NP=no patogénico <strong>de</strong> garbanzo.<br />
39
Capítulo I<br />
Tabla 4. Análisis <strong>de</strong> la varianza molecular, AMOVA, <strong>de</strong> los datos RAPDs<br />
obtenidos a partir <strong>de</strong> 57 aislados <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris<br />
agrupados según su patotipo (Amarillez, Marchitez), raza patogénica (razas<br />
0, 1A, 1B/C, 2, 3, 4, 5, y 6), y origen geográfico<br />
Fuente <strong>de</strong> Variación a<br />
Grados <strong>de</strong> Cuadrado Componente <strong>de</strong><br />
libertad medio la varianzab Valor P c<br />
Amarillez vs Marchitez 1 2,043 0,0712 (52,2)
Capítulo I<br />
et al., 1993; Manulis et al., 1994; Migheli et al., 1998). Nuestro trabajo indica<br />
una clara correlación entre los perfiles RAPD i<strong>de</strong>ntificados y la<br />
caracterización patogénica <strong>de</strong> las razas 0, 1B/C, 5, y 6 <strong>de</strong> F. oxysporum f. sp.<br />
ciceris mediante bioensayos.<br />
La falta <strong>de</strong> éxito en encontrar un marcador RAPD <strong>de</strong> los aislados <strong>de</strong> la<br />
raza 1A, así como la i<strong>de</strong>ntificación <strong>de</strong> ciertas bandas RAPD presentes en<br />
las razas 1A y 6, pue<strong>de</strong> ser explicado por la existencia <strong>de</strong> una gran<br />
similaridad genética entre dichas razas. Un caso similar fue <strong>de</strong>scrito por<br />
Woo et al. (1996), quienes i<strong>de</strong>ntificaron aislados <strong>de</strong> F. oxysporum f. sp.<br />
phaseoli Kendr. & Zin<strong>de</strong>r <strong>de</strong> diversa patogenicidad que presentaban<br />
patrones RAPD similares. Las razas 1A y 6 <strong>de</strong> F. oxysporum f. sp. ciceris<br />
presentan patrones <strong>de</strong> virulencia similares en cultivares <strong>de</strong> garbanzo<br />
diferenciadores (Halila y Strange, 1996; Jiménez Díaz et al., 1993a), por lo<br />
que existe la posibilidad <strong>de</strong> una caracterización racial errónea <strong>de</strong> estos<br />
aislados, <strong>de</strong>bida a algún fallo durante la realización <strong>de</strong> las pruebas <strong>de</strong><br />
patogenicidad. Análisis RAPD adicionales, utilizando un mayor número <strong>de</strong><br />
aislados <strong>de</strong> la raza 1A <strong>de</strong>l patógeno, así como nuevos iniciadores RAPD,<br />
podrían resolver esta cuestión. Asimismo, también es posible que estas dos<br />
razas patogénicas aún no se encuentren lo suficientemente separadas entre<br />
sí <strong>de</strong>s<strong>de</strong> un punto <strong>de</strong> vista evolutivo, para haber <strong>de</strong>sarrollado marcadores<br />
diagnósticos y patogénicos in<strong>de</strong>pendientes.<br />
Los marcadores RAPD i<strong>de</strong>ntificados en este trabajo ofrecen<br />
aplicaciones importantes para la epi<strong>de</strong>miología y manejo <strong>de</strong> la Fusariosis<br />
Vascular <strong>de</strong>l garbanzo. Esta utilidad ha sido probada mediante la<br />
i<strong>de</strong>ntificación correcta <strong>de</strong> dos aislados (Foc–8250 y –8503) cuyos perfiles<br />
RAPD no correspondieron con la caracterización racial esperada. El<br />
aislado Foc–8250 fue el menos virulento <strong>de</strong> los aislados <strong>de</strong> la raza 0<br />
estudiados por Cabrera <strong>de</strong> la Colina (1986). Posteriormente, repetidas<br />
pruebas <strong>de</strong> patogenicidad sobre líneas <strong>de</strong> garbanzo susceptibles, en<br />
condiciones estandarizadas, revelaron tanto la ausencia <strong>de</strong> síntomas <strong>de</strong><br />
Amarillez como la ausencia <strong>de</strong> infección en la planta (Kelly et al., 1994).<br />
Por lo tanto, tanto las pruebas <strong>de</strong> patogenicidad como la ausencia <strong>de</strong> los<br />
marcadores RAPD <strong>de</strong> la raza 0 indicaron que Fo–8250 es un aislado no<br />
patogénico <strong>de</strong> garbanzo. De forma similar, Foc–8503 fue caracterizado<br />
41
Capítulo I<br />
originalmente mediante bioensayos como un aislado <strong>de</strong> la raza 5, pero los<br />
análisis RAPD, así como pruebas <strong>de</strong> patogenicidad repetidas,<br />
rei<strong>de</strong>ntificaron a este aislado como perteneciente a la raza 0.<br />
Un beneficio neto adicional <strong>de</strong> este estudio ha sido la posibilidad <strong>de</strong><br />
discriminar las razas 0 y 1B/C mediante el análisis RAPD, ya que dichas<br />
razas inducen el síndrome <strong>de</strong> Amarillez en las pruebas <strong>de</strong> patogenicidad.<br />
En trabajos anteriores, los análisis RAPD utilizando los iniciadores KS, P2,<br />
y P6, no pudieron diferenciar entre esta dos razas, aunque si las asignaron<br />
al patotipo <strong>de</strong> Amarillez (Kelly et al., 1994). Los casos reflejados son<br />
ejemplos que resaltan los problemas que pue<strong>de</strong>n presentarse en las pruebas<br />
<strong>de</strong> patogenicidad, y que pue<strong>de</strong>n ser eliminados mediante el uso <strong>de</strong><br />
marcadores RAPD diagnósticos.<br />
El análisis <strong>de</strong> ‘clusters’ <strong>de</strong> los datos RAPD mostró una alta similaridad<br />
genética entre los aislados <strong>de</strong> F. oxysporum f. sp. ciceris. Todos los aislados<br />
que inducen el síndrome <strong>de</strong> Amarillez constituyen un grupo distintivo, que<br />
correspon<strong>de</strong> a los aislados <strong>de</strong> las razas 0 y 1B/C. Por el contrario, los<br />
aislados <strong>de</strong> Marchitez se ubicaron en otro gran ‘cluster’, agrupando a los<br />
aislados <strong>de</strong> las razas 1A, 2, 3, 4, 5, y 6. Esta división en diferentes grupos<br />
<strong>de</strong> los aislados <strong>de</strong> F. oxysporum f. sp. ciceris es consistente con la <strong>de</strong>scrita por<br />
Kelly et al. (1994), quienes utilizaron sólo tres iniciadores RAPD y una<br />
parte <strong>de</strong>l mismo grupo <strong>de</strong> aislados <strong>de</strong>l patógeno que se ha utilizado en este<br />
trabajo. A<strong>de</strong>más, los resultados <strong>de</strong>l análisis <strong>de</strong> ‘clusters’ son también<br />
consistentes con los <strong>de</strong>l análisis AMOVA. Por lo tanto, po<strong>de</strong>mos concluir<br />
que F. oxysporum f. sp. ciceris es un grupo clonal <strong>de</strong> aislados patógenos con<br />
una fuerte estructura racial, en el cual la correlación entre los marcadores<br />
genéticos neutrales y la patogenicidad pue<strong>de</strong> ser utilizada para inferir<br />
conclusiones sobre la relación genética entre razas.<br />
Estudios anteriores <strong>de</strong>mostraron que todas las razas <strong>de</strong> F. oxysporum f.<br />
sp. ciceris presentan el mismo patrón RFLP <strong>de</strong>l ADN mitocondrial (Pérez<br />
Artés et al., 1995), pertenecen al mismo grupo <strong>de</strong> compatibilidad vegetativa<br />
(VCG) (Nogales Moncada, 1997), y poseen el mismo elemento repetitivo<br />
(Kelly, 1996). A<strong>de</strong>más, el aislado Foc–USA 3-1 JG62 <strong>de</strong> la raza 1B/C<br />
proce<strong>de</strong>nte <strong>de</strong> California pue<strong>de</strong> formar heterocariontes estables (i. e.,<br />
vegetativamente compatible) con aislados <strong>de</strong> F. oxysporum no patogénicos<br />
42
Capítulo I<br />
obtenidos <strong>de</strong> raíces <strong>de</strong> plantas sanas <strong>de</strong> garbanzo (Nogales Moncada,<br />
1997). Este hecho sugiere que pue<strong>de</strong> producirse el intercambio genético<br />
asexual entre aislados <strong>de</strong> diferentes razas, y también entre aislados<br />
patogénicos y no patogénicos.<br />
Correll (1991) propuso varios mo<strong>de</strong>los para explicar la relación entre<br />
formae speciales, razas, y VCGs en F. oxysporum, con la asunción <strong>de</strong> que, en su<br />
origen, la especie estuvo constituida por una población parasítica, no<br />
patogénica, y diversa en VCGs, <strong>de</strong> la cual pudieron producirse mutaciones<br />
en aislados que dieron lugar a la adquisición <strong>de</strong> la patogenicidad específica.<br />
Nuestros resultados apoyan dicha hipótesis, y parecen correspon<strong>de</strong>r al<br />
patrón <strong>de</strong> diversidad entre razas y VCGs <strong>de</strong>l mo<strong>de</strong>lo III <strong>de</strong> Correll (1991),<br />
i. e., diferentes razas <strong>de</strong> una forma specialis originadas a partir <strong>de</strong>l mismo<br />
VCG. En nuestro trabajo hemos observado que los aislados <strong>de</strong> la raza 0 <strong>de</strong><br />
F. oxysporum f. sp. ciceris y algunos aislados <strong>de</strong> F. oxysporum no patogénicos<br />
<strong>de</strong> garbanzo comparten bandas <strong>de</strong> ADN que son amplificadas por varios<br />
iniciadores <strong>de</strong> Operon, en coinci<strong>de</strong>ncia con Pérez Artés et al. (1996)<br />
quienes utilizaron diferentes aislados <strong>de</strong> F. oxysporum y los iniciadores KS,<br />
P2, y P6 (Kelly et al., 1994). Hibridaciones ‘Southern’ utilizando como<br />
sonda el fragmento RAPD específico <strong>de</strong> Amarillez clonado, <strong>de</strong>mostraron<br />
la existencia <strong>de</strong> cierta similitud en la secuencia <strong>de</strong> fragmentos <strong>de</strong> ADN<br />
amplificados <strong>de</strong> aislados <strong>de</strong> F. oxysporum no patogénicos <strong>de</strong> garbanzo y<br />
aislados <strong>de</strong>l patotipo <strong>de</strong> Amarillez <strong>de</strong> F. oxysporum f. sp. ciceris (Pérez Artés<br />
et al., 1996). El hecho <strong>de</strong> que los aislados <strong>de</strong> F. oxysporum f. sp. ciceris<br />
pertenecientes a un mismo VCG compartan una alta similaridad genética,<br />
pero pertenezcan a razas diferentes, sugiere que estos aislados fueron<br />
clones, o se originaron <strong>de</strong> un parental común, aunque en el presente se<br />
encuentren separados geográficamente y sean patogénicamente diversos<br />
(Leslie, 1993).<br />
La raza 0 <strong>de</strong> F. oxysporum f. sp. ciceris no ha sido i<strong>de</strong>ntificada en el<br />
subcontinente Indio, pero es la más común <strong>de</strong> las i<strong>de</strong>ntificadas en la región<br />
Mediterránea (Halila y Strange, 1996; Jiménez Díaz et al., 1989b; 1993a;<br />
Jiménez Díaz, no publicado). La raza 0 es no patogénica sobre el cv. JG-62,<br />
susceptible universal <strong>de</strong> las razas causantes <strong>de</strong> Marchitez, y es patogénica<br />
principalmente en cultivares <strong>de</strong> garbanzo ‘kabuli’ (Jiménez Díaz et al.,<br />
43
Capítulo I<br />
1989b; 1993a). Los garbanzos ‘kabuli’ se cultivan <strong>de</strong> forma tradicional y<br />
generalizada en la región Mediterránea, mientras que los cultivares ‘<strong>de</strong>si’<br />
son cultivados principalmente en el subcontinente Indio, y la mayoría son<br />
resistentes a la raza 0 <strong>de</strong>l patógeno. Las razas 2, 3, y 4, i<strong>de</strong>ntificadas hasta<br />
ahora sólo en la India, son las más virulentas <strong>de</strong> las ocho razas <strong>de</strong>scritas<br />
(Halila y Strange, 1996; Haware y Nene, 1982b; Jiménez Díaz et al., 1989b;<br />
1993a), y las que más distan genéticamente <strong>de</strong>l resto <strong>de</strong> las razas <strong>de</strong> F.<br />
oxysporum f. sp. ciceris (Fig. 3, Tabla 4). Esta aparente correlación entre las<br />
razas patogénicas <strong>de</strong>l agente y los cultivares <strong>de</strong>l huésped, podría explicarse<br />
si las razas fuesen grupos <strong>de</strong> aislados geográficamente separados adaptados<br />
al germoplasma local.<br />
En conclusión, la metodología RAPD constituye una herramienta<br />
diagnóstica po<strong>de</strong>rosa para la i<strong>de</strong>ntificación <strong>de</strong> las principales razas <strong>de</strong> F.<br />
oxysporum f. sp. ciceris presentes en la región Mediterránea. Los marcadores<br />
moleculares que hemos i<strong>de</strong>ntificados podrán ser utilizados para estudiar la<br />
distribución <strong>de</strong> dichas razas, y facilitar así el empleo eficiente <strong>de</strong> los<br />
cultivares resistentes disponibles. Estos marcadores ayudarán a la<br />
<strong>de</strong>tección temprana <strong>de</strong> la posible introducción <strong>de</strong> razas, así como a la<br />
<strong>de</strong>tección <strong>de</strong> los cambios que puedan ocurrir en la frecuencia relativa <strong>de</strong> las<br />
distintas razas en una población, como repuesta a la utilización <strong>de</strong><br />
cultivares <strong>de</strong> garbanzo resistentes. Finalmente, las bandas <strong>de</strong> ADN<br />
marcadoras con probada capacidad diagnóstica podrán ser clonadas y<br />
secuenciadas, con el objeto <strong>de</strong> <strong>de</strong>sarrollar sondas e iniciadores específicos<br />
<strong>de</strong> raza, para uso en hibridaciones ‘dot-blot’ o PCR específica,<br />
respectivamente.<br />
44
CAPÍTULO II<br />
Capítulo II<br />
Desarrollo <strong>de</strong> un método basado en el análisis PCR<br />
específica para la i<strong>de</strong>ntificación molecular <strong>de</strong><br />
<strong>Fusarium</strong> oxysporum f. sp. ciceris y <strong>de</strong> las razas<br />
patogénicas 0, 1A, 5, y 6<br />
Introducción<br />
Tradicionalmente, la i<strong>de</strong>ntificación <strong>de</strong> hongos fitopatógenos se ha basado<br />
en la observación y comparación <strong>de</strong> caracteres morfológicos <strong>de</strong> los<br />
especímenes; sin embargo, en cada uno <strong>de</strong> los niveles taxonómicos, i. e.,<br />
especie, subespecie, variedad, forma specialis y raza, cada vez es menos<br />
numeroso el tipo <strong>de</strong> caracteres morfológicos que se consi<strong>de</strong>ran válidos<br />
para la diferenciación entre dichos grupos. A<strong>de</strong>más, mientras que<br />
subespecies y varieda<strong>de</strong>s suelen diferir en algunos aspectos morfológicos,<br />
formae speciales y razas difieren generalmente en la gama <strong>de</strong> especies y <strong>de</strong><br />
cultivares <strong>de</strong> éstas, respectivamente, sobre las que son patogénicas, pero no<br />
pue<strong>de</strong>n ser distinguidas entre si por su morfología (Coddington y Gould,<br />
1992).<br />
La taxonomía <strong>de</strong> <strong>Fusarium</strong> es un tema complejo que se encuentra en<br />
constante proceso <strong>de</strong> revisión y actualización (Nelson et al., 1981; 1983). F.<br />
oxysporum Schlechtend.:Fr. es un hongo anamórfico que incluye tanto<br />
45
Capítulo II<br />
aislados fitopatogénicos como no patogénicos. Las formas patogénicas<br />
causan enfermeda<strong>de</strong>s vasculares y podredumbres corticales, y se agrupan<br />
en formae speciales en base a su gama <strong>de</strong> plantas susceptibles. A<strong>de</strong>más,<br />
<strong>de</strong>ntro <strong>de</strong> una forma specialis pue<strong>de</strong> existir variación en la virulencia que sus<br />
miembros manifiestan sobre cultivares <strong>de</strong> su especie, dando lugar a la<br />
<strong>de</strong>signación <strong>de</strong> patotipos y razas patogénicas (Kistler, 1997).<br />
F. oxysporum f. sp. ciceris (Padwick) Matuo & K. Sato es paradigmática<br />
entre las formae speciales <strong>de</strong> F. oxysporum en términos <strong>de</strong> diversidad<br />
patogénica. Así, los componentes <strong>de</strong> las poblaciones naturales <strong>de</strong> este<br />
patógeno <strong>de</strong> suelo pue<strong>de</strong>n ser diferenciados en los patotipos <strong>de</strong> Amarillez<br />
y Marchitez, por las características <strong>de</strong>l síndrome patológico que <strong>de</strong>terminan<br />
en cultivares <strong>de</strong> garbanzo susceptibles en inoculaciones artificiales por<br />
siembra en suelo infestado; i. e., Amarillez: clorosis foliar progresiva,<br />
coloración vascular y muerte <strong>de</strong> la planta a los 40 días <strong>de</strong> la inoculación;<br />
Marchitez: clorosis severa, flaci<strong>de</strong>z, coloración vascular y muerte antes <strong>de</strong><br />
los 20 días <strong>de</strong> la inoculación (Trapero Casas, 1983; Trapero Casas y<br />
Jiménez Díaz, 1985). Superpuesta sobre la diferenciación en patotipos, los<br />
componentes <strong>de</strong> F. oxysporum f. sp. ciceris pue<strong>de</strong>n ser asignados a una <strong>de</strong><br />
ocho razas patogénicas (razas 0, 1A, 1B/C, 2, 3, 4, 5, y 6) según la reacción<br />
diferencial <strong>de</strong> líneas diferenciadoras en inoculaciones estandarizadas, en<br />
ambiente controlado (Haware y Nene, 1982b; Jiménez Díaz et al., 1989b;<br />
1993a).<br />
En consecuencia y en ausencia <strong>de</strong> métodos alternativos, la<br />
caracterización <strong>de</strong> aislados <strong>de</strong> F. oxysporum f. sp. ciceris, como los <strong>de</strong> otras<br />
formae speciales <strong>de</strong> F. oxysporum, requiere <strong>de</strong> la realización <strong>de</strong> bioensayos que<br />
conllevan la inoculación <strong>de</strong>l huésped para reproducir la sintomatología<br />
característica <strong>de</strong> la enfermedad. Los procedimientos experimentales para<br />
llevar a cabo dichos bioensayos son relativamente simples y<br />
económicamente practicables, pero requieren tiempo, disponibilidad <strong>de</strong><br />
recursos e instalaciones experimentales, y operarios especializados.<br />
Asimismo, el resultado <strong>de</strong> este tipo <strong>de</strong> metodologías <strong>de</strong>pen<strong>de</strong> en cierta<br />
manera <strong>de</strong>l juicio subjetivo <strong>de</strong>l investigador y, consecuentemente, <strong>de</strong> su<br />
experiencia en la realización <strong>de</strong> diagnósticos visuales. A<strong>de</strong>más, en nuestra<br />
experiencia en la Fusariosis Vascular <strong>de</strong>l garbanzo, aislados no patogénicos<br />
46
Capítulo II<br />
<strong>de</strong> F. oxysporum obtenidos <strong>de</strong> plantas <strong>de</strong> garbanzo sintomáticas pue<strong>de</strong>n ser<br />
i<strong>de</strong>ntificados erróneamente como F. oxysporum f. sp. ciceris <strong>de</strong>bido a fallos<br />
inherentes al sistema experimental (i. e., incubación <strong>de</strong> plantas inoculadas<br />
en ambiente ina<strong>de</strong>cuado, inóculo insuficiente, senescencia prematura <strong>de</strong> las<br />
plantas, etc.). Morfológicamente, los aislados <strong>de</strong> F. oxysporum f. sp. ciceris<br />
son indistinguibles <strong>de</strong> aquellos aislados no patogénicos <strong>de</strong> F. oxysporum que<br />
se encuentran infectando el córtex radical <strong>de</strong> garbanzo, pero que no son<br />
capaces <strong>de</strong> invadir el sistema vascular, y por tanto no causan enfermedad.<br />
La i<strong>de</strong>ntificación consistente, rápida y precisa <strong>de</strong> las variantes<br />
patogénicas <strong>de</strong>l agente es clave para el <strong>de</strong>sarrollo y utilización eficiente <strong>de</strong><br />
cultivares resistentes <strong>de</strong>l huésped, que en muchos casos es el medio <strong>de</strong><br />
lucha más eficiente contra enfermeda<strong>de</strong>s <strong>de</strong> plantas. Por ello, existe una<br />
gran <strong>de</strong>manda <strong>de</strong> métodos simples, fiables, y rápidos para la <strong>de</strong>tección y<br />
diferenciación <strong>de</strong> formas patogénicas especializadas <strong>de</strong> F. oxysporum, que<br />
no estén basados en caracteres morfológicos ni patogénicos.<br />
La reacción en ca<strong>de</strong>na <strong>de</strong> la polimerasa (PCR, ‘Polymerase Chain<br />
Reaction’) ha sido utilizada ampliamente y con éxito para la i<strong>de</strong>ntificación<br />
<strong>de</strong> hongos fitopatógenos <strong>de</strong> importancia, como Verticillium dahliae Kleb. y<br />
V. albo-atrum Reinke & Berthold (Car<strong>de</strong>r y Barbara, 1994; Nazar et al.,<br />
1991; Pérez-Artés et al., 2000), Phytophthora spp., etc. (Henson y French,<br />
1993; Martin et al., 2000; Miller, 1996), así como para diferenciar F.<br />
culmorum (W. G. Sm.) Sacc., F. graminearum Schwäbe, y F. avenaceum (Fr.)<br />
Sacc. (Schilling et al., 1996).<br />
Las estrategias utilizadas convencionalmente, para <strong>de</strong>sarrollar un<br />
método <strong>de</strong> i<strong>de</strong>ntificación <strong>de</strong> especies <strong>de</strong> hongos fitopatógenos basado en la<br />
PCR, ha sido el diseño <strong>de</strong> iniciadores basados en secuencias polimórficas<br />
<strong>de</strong> regiones <strong>de</strong> ADN altamente conservadas entre aquéllas, tales como las<br />
regiones ITS (‘Internal Transcribed Spacer’) e IGS (‘Intergenic Spacer’) <strong>de</strong>l<br />
ADN ribosómico (Henson y French, 1993; Ward, 1994). Aunque estas<br />
estrategias han tenido éxito para la i<strong>de</strong>ntificación <strong>de</strong> especies (Bunting et al.,<br />
1996; Lee et al., 1993; Lévesque et al., 1994; Nazar et al., 1991), no ocurre<br />
así en cuanto a su uso para la diferenciación <strong>de</strong> grupos o variantes<br />
subespecíficos, tales como formae speciales o razas patogénicas (White et al.,<br />
1990). En cambio, otra <strong>de</strong> las estrategias utilizadas para <strong>de</strong>sarrollar<br />
47
Capítulo II<br />
marcadores específicos, basada en el aislamiento y secuenciación <strong>de</strong><br />
fragmentos <strong>de</strong> ADN polimórfico amplificados mediante RAPD (‘Random<br />
Amplified Polymorphic DNA’), sí permite <strong>de</strong>tectar y utilizar<br />
polimorfismos asociados a dichos grupos subespecíficos. La utilización <strong>de</strong><br />
fragmentos RAPD secuenciados (SCARs, ‘Sequence Characterized<br />
Amplified Regions’) fue aplicada por primera vez por Paran y Michelmore<br />
(1993) para i<strong>de</strong>ntificar resistencia a Mildiu (Bremia lactucae Regel) en lechuga<br />
(Lactucae sativa L.).<br />
El objetivo <strong>de</strong> las investigaciones contenidas en este Capítulo fue<br />
<strong>de</strong>sarrollar un método diagnóstico rápido, eficaz, sensible y fiable que<br />
posibilite la <strong>de</strong>tección selectiva <strong>de</strong> la forma specialis ciceris, patogénica en<br />
garbanzo, así como la i<strong>de</strong>ntificación y diferenciación <strong>de</strong> las razas<br />
patogénicas <strong>de</strong> F. oxysporum f. sp. ciceris presentes en la Cuenca<br />
Mediterránea.<br />
En investigaciones anteriores (Capítulo I), estudiamos la diversidad<br />
genética <strong>de</strong> una colección mundial <strong>de</strong> aislados <strong>de</strong>l patógeno representativos<br />
<strong>de</strong> todas las razas patogénicas, mediante el análisis RAPD-PCR. Dichas<br />
investigaciones dieron lugar a la i<strong>de</strong>ntificación <strong>de</strong> iniciadores arbitrarios<br />
que generan marcadores RAPD diagnósticos <strong>de</strong> F. oxysporum f. sp. ciceris,<br />
así como <strong>de</strong> las razas 0, 1B/C, 5, y 6 <strong>de</strong>l patógeno. La amplificación que<br />
generan dichos iniciadores ha <strong>de</strong>mostrado ser sumamente útil para la<br />
diferenciación <strong>de</strong>l patógeno y <strong>de</strong> sus razas; sin embargo, <strong>de</strong>bido a la<br />
naturaleza aleatoria <strong>de</strong> los marcadores RAPD, la reproducibilidad <strong>de</strong> la<br />
técnica podría ser influida por <strong>de</strong>terminados factores, incluyendo la fuente<br />
y procedimiento <strong>de</strong> extracción <strong>de</strong>l ADN fúngico, la existencia <strong>de</strong><br />
contaminantes, la amplificación <strong>de</strong> secuencias <strong>de</strong> ADN diferentes pero <strong>de</strong>l<br />
mismo tamaño que el fragmento marcador, etc.<br />
Para obviar la posibilidad <strong>de</strong> las dificulta<strong>de</strong>s referidas anteriormente,<br />
hemos <strong>de</strong>sarrollado un método <strong>de</strong> análisis PCR específica que ha requerido<br />
el diseño <strong>de</strong> iniciadores basados en las secuencias <strong>de</strong> ADN <strong>de</strong> dichas<br />
bandas marcadoras RAPD. El <strong>de</strong>sarrollo <strong>de</strong> tales iniciadores y la tecnología<br />
<strong>de</strong> PCR específica facilitaría eventualmente la <strong>de</strong>tección <strong>de</strong> F. oxysporum f.<br />
sp. ciceris y sus razas patogénicas en tejido vegetal infectado y en suelo. En<br />
el presente Capítulo <strong>de</strong> esta Tesis Doctoral estudiamos la posibilidad <strong>de</strong><br />
48
Capítulo II<br />
utilizar SCARs para <strong>de</strong>sarrollar marcadores basados en PCR específicas, a<br />
partir <strong>de</strong> marcadores RAPD informativos. Para la consecución <strong>de</strong> dicho<br />
objetivo fue necesario: (i) clonar los fragmentos RAPD asociados a cada<br />
raza <strong>de</strong> F. oxysporum f. sp. ciceris; (ii) caracterizar las secuencias <strong>de</strong> ADN<br />
contenidas en los fragmentos clonados, para lo cual es necesario asegurar<br />
la especificidad <strong>de</strong> éstos mediante hibridaciones con el perfil RAPD y con<br />
ADN genómico <strong>de</strong> las razas digerido con enzimas <strong>de</strong> restricción; (iii)<br />
secuenciar dichos fragmentos; y (iv) diseñar iniciadores a partir <strong>de</strong> las<br />
regiones extremas y <strong>de</strong>mostrar su vali<strong>de</strong>z mediante la realización <strong>de</strong> análisis<br />
<strong>de</strong> PCR específica.<br />
Materiales y Métodos<br />
Aislados fúngicos<br />
Se han utilizado 76 aislados <strong>de</strong> F. oxysporum f. sp. ciceris representativos <strong>de</strong><br />
las ocho razas patogénicas (0, 1A, 1B/C, 2, 3, 4, 5, y 6) <strong>de</strong>scritas y <strong>de</strong> un<br />
amplio origen geográfico <strong>de</strong>l patógeno (Tabla 5). De igual manera, se han<br />
incluido en el estudio 20 aislados <strong>de</strong> F. oxysporum obtenidos <strong>de</strong> raíces sanas<br />
<strong>de</strong> garbanzo y no patogénicos <strong>de</strong> éste (Tabla 5), seis aislados pertenecientes<br />
a otras formae speciales <strong>de</strong> F. oxysporum (Tabla 6), y 58 aislados <strong>de</strong> 47 especies<br />
<strong>de</strong> <strong>Fusarium</strong> diferentes a F. oxysporum (Tabla 6). Todos los aislados <strong>de</strong> F.<br />
oxysporum f. sp. ciceris habían sido caracterizados racialmente en trabajos<br />
anteriores (Jiménez Díaz et al., 1989b; 1993a; Kelly et al., 1994; Capítulo I).<br />
Los 96 aislados <strong>de</strong> F. oxysporum patogénicos y no patogénicos <strong>de</strong><br />
garbanzo, así como los seis pertenecientes a otras formae speciales, proce<strong>de</strong>n<br />
<strong>de</strong> la colección <strong>de</strong>l Departamento <strong>de</strong> Protección <strong>de</strong> Cultivos, Instituto <strong>de</strong><br />
Agricultura Sostenible, CSIC, Córdoba, y los restantes aislados fueron<br />
amablemente cedidos por el Dr. J. F. Leslie, Departamento <strong>de</strong> Patología<br />
Vegetal, Universidad <strong>de</strong> Kansas, Manhattan, KS, EEUU. Todos los<br />
aislados se han mantenido almacenados como cultivos monoconídicos en<br />
suelo estéril en tubos <strong>de</strong> ensayo, a 4ºC, en oscuridad. De dichos cultivos se<br />
49
Capítulo II<br />
obtuvieron cultivos en crecimiento activo mediante métodos <strong>de</strong>scritos<br />
anteriormente (Pág. 22, Capítulo I).<br />
Para la extracción <strong>de</strong> ADN fúngico se utilizó micelio liofilizado <strong>de</strong> los<br />
aislados. A tal fin, el micelio se obtuvo por dos métodos: (1) mediante<br />
cultivo en caldo <strong>de</strong> patata-<strong>de</strong>xtrosa (PDB) y posterior filtrado, lavado y<br />
liofilización <strong>de</strong>l micelio recogido (Pág. 26, Capítulo I); o (2) mediante<br />
cultivo <strong>de</strong>l hongo sobre una lámina <strong>de</strong> celofán estéril (121 ºC, 20 min, en<br />
autoclave) colocada sobre agar patata-<strong>de</strong>xtrosa (PDA). La lámina <strong>de</strong><br />
celofán permite el tránsito <strong>de</strong> nutrientes hacia el micelio fúngico en<br />
crecimiento, mientras que el tamaño <strong>de</strong> sus poros es <strong>de</strong>masiado pequeño<br />
para permitir el crecimiento <strong>de</strong> las hifas fúngicas a su través. De esta<br />
manera, se pudo recolectar gran cantidad <strong>de</strong> micelio puro <strong>de</strong> los aislados<br />
fúngicos mediante el raspado <strong>de</strong> la colonia. Posteriormente, este micelio<br />
fue sometido a los procesos <strong>de</strong> liofilización, y molienda, quedando listo<br />
para proce<strong>de</strong>r a la extracción <strong>de</strong> su ADN.<br />
Extracción <strong>de</strong> ADN<br />
El ADN genómico <strong>de</strong> cada aislado se purificó a partir <strong>de</strong> micelio<br />
liofilizado y molido, según el método <strong>de</strong> Rae<strong>de</strong>r y Broda (1985) con ligeras<br />
modificaciones (Pág. 27-28, Capítulo I). Las muestras <strong>de</strong> ADN se<br />
diluyeron hasta una concentración <strong>de</strong> 5-10 ng/µl para realizar los análisis<br />
PCR posteriores.<br />
Reacciones <strong>de</strong> amplificación RAPD<br />
Las reacciones <strong>de</strong> amplificación RAPD se realizaron <strong>de</strong> forma idéntica a la<br />
<strong>de</strong>scrita anteriormente (Pág. 28, Capítulo I) utilizando los iniciadores <strong>de</strong> 10<br />
oligonucleótidos OPF-10, OPF-12 y OPI-09 (Operon Technology,<br />
Alameda, CA, EEUU) (Tabla 3, Pág. 36, Capítulo I). Las condiciones <strong>de</strong><br />
50
Capítulo II<br />
Tabla 5. Información geográfica y racial sobre los aislados <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris (Foc), y <strong>de</strong> F. oxysporum (Fo)<br />
no patogénicos <strong>de</strong> garbanzo, utilizados en el análisis PCR específica<br />
Aislado Origen geográfico a Raza patogénica b Aislado Origen geográfico a Raza patogénica b<br />
Foc-7802 España 0 Foc-9606 Túnez 0<br />
Foc-7952 España 0 Foc-T3 Túnez 0<br />
Foc-8207 España 0 Foc-cc21C Israel 0<br />
Foc-82108 España 0 Foc-cc62R Israel 0<br />
Foc-82113 España 0 Foc-cc63K Israel 0<br />
Foc-8503 España 0 Foc-L96-5 Líbano 0<br />
Foc-8733 España 0 Foc-L96-6 Líbano 0<br />
Foc-8912 España 0 Foc-L96-7 Líbano 0<br />
Foc-9018 PV1 España 0 Foc-L96-9 Líbano 0<br />
Foc-9018 JG62 España 0 Foc-L96-10 Líbano 0<br />
Foc-9032 España<br />
0 Foc-L96-11 Líbano 0<br />
Foc-90111 España 0 Foc-Sy 96-14-1 Siria 0<br />
Foc-91100 España 0 Foc-Sy 96-18-3 Siria 0<br />
Foc-91015 España 0 Foc-Sy 96-19-1 Siria 0<br />
Foc-91108 España 0 Foc-T 96-1-3 Turquía 0<br />
Foc-91114 España 0 Foc-USA 3-1JG62 EEUU 1B/C<br />
Foc-9601 Túnez 0 Foc-1987-W17 EEUU 1B/C<br />
Foc-9603 Túnez 0 Foc-9602 Túnez 1B/C<br />
Foc-9604 Túnez 0 Foc-Sy 96-10-1 Siria 1B/C<br />
Foc-9605 Túnez 0 Foc-T 96-1-1 Turquía 1B/C<br />
51
Capítulo II<br />
Tabla 5. (Continuación) Información geográfica y racial sobre los aislados <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris (Foc), y <strong>de</strong> F.<br />
oxysporum (Fo) no patogénicos <strong>de</strong> garbanzo, utilizados en el análisis PCR específica<br />
52<br />
Aislado Origen gepgráfico a Raza patogénica b Aislado Origen geográfico a Raza patogénica b<br />
Foc-T 96-1-2 Turquía 1B/C Foc-9094 PV1 España 5<br />
Foc-T 96-2-1 Turquía 1B/C Foc-9094 JG62 España 5<br />
Foc-T 96-2-2 Turquía 1B/C Foc-USA1-1JG62 EEUU<br />
5<br />
Foc-cc22D Israel 1B/C Foc-USA W6-1 EEUU 5<br />
Foc-7989 India 1A Foc-USA14201 EEUU 5<br />
Foc-9027 PV1c España<br />
1A Foc-8272 España 6<br />
Foc-9166 c Marruecos<br />
1A Foc-8905 España 6<br />
Foc-9168 Marruecos 1A Foc-8924 España 6<br />
Foc-8605 India 2 Foc-9023 España 6<br />
Foc-1992 R2N India 2 Foc-9093 PV1 España 6<br />
Foc-8606 India<br />
3 Foc-9164 Marruecos 6<br />
Foc-1992 R3N India 3 Foc-9170 Marruecos 6<br />
Foc-8607 India<br />
4 Foc-Tonini EEUU 6<br />
Foc-1992 R4N India 4 Foc-9620 Israel 6<br />
Foc-8012 España<br />
5 Foc-9622 Israel 6<br />
Foc-8257 España 5 Foc-9628 Israel 6<br />
Foc-8408 España 5 Foc-9632 Israel 6<br />
Foc-8508 España 5 Fo-8250 España NP<br />
Foc-9035 España 5 Fo-9009 España NP
Capítulo II<br />
Tabla 5. (Continuación) Información geográfica y racial sobre los aislados <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris (Foc), y <strong>de</strong> F.<br />
oxysporum (Fo) no patogénicos <strong>de</strong> garbanzo, utilizados en el análisis PCR específica<br />
Aislado Origen geográfico a Raza patogénica b Aislado Origen geográfico a Raza patogénica b<br />
Fo-9081<br />
Fo-90101<br />
Fo-90105<br />
Fo-9169<br />
Fo-91117<br />
Fo-Fsp4<br />
Fo-Fsp7<br />
Fo-Fsp8<br />
Fo-Fsp9<br />
España NP Fo-420 Italia NP<br />
España NP Fo-422 Italia NP<br />
España NP Fo-425 Italia NP<br />
Marruecos NP Fo-442 Italia NP<br />
España NP Fo-448 Italia NP<br />
Argelia NP Fo-457 Italia NP<br />
Argelia NP Fo-506 Italia NP<br />
Argelia NP Fo-526 Italia NP<br />
Argelia NP Fo-C4P Italia NP<br />
a<br />
Los aislados <strong>de</strong> Argelia, EEUU (California), España, Italia y Marruecos proce<strong>de</strong>n <strong>de</strong> la micoteca <strong>de</strong>l Departamento <strong>de</strong> Protección<br />
<strong>de</strong> Cultivos, Instituto <strong>de</strong> Agricultura Sostenible, CSIC, Córdoba. Los aislados <strong>de</strong> la India fueron proporcionados por el Dr. M. P.<br />
Haware, ICRISAT, Hy<strong>de</strong>rabad, India. Los aislados proce<strong>de</strong>ntes <strong>de</strong> Túnez fueron proporcionados por el Dr. M. H. Halila, Institute<br />
Nacionale <strong>de</strong> la Recherche Agronomique, Ariana, Túnez; los aislados <strong>de</strong> Israel fueron cedidos por el Profesor J. Katan,<br />
Departamento <strong>de</strong> Patología Vegetal y Microbiología, The Hebrew University of Jerusalem, Israel; los aislados originarios <strong>de</strong><br />
Líbano, Siria y Turquía fueron proporcionados por el Dr. C. Akem, ICARDA, Aleppo, Siria.<br />
b Raza <strong>de</strong>terminada mediante pruebas <strong>de</strong> patogenicidad en líneas <strong>de</strong> garbanzo diferenciadoras (Alcalá-Jiménez, 1995; Jiménez-Gasco<br />
et al., 2001). NP = no patogénicos <strong>de</strong> garbanzo.<br />
c<br />
Aislados caracterizados por Alcalá Jiménez (1995) como pertenecientes a la raza 6 <strong>de</strong> F. oxysporum f. sp. ciceris en pruebas <strong>de</strong><br />
patogenicidad duplicadas.<br />
53
Capítulo II<br />
Tabla 6. Aislados <strong>de</strong> diferentes formae speciales <strong>de</strong> <strong>Fusarium</strong> oxysporum, así<br />
como <strong>de</strong> otras <strong>Fusarium</strong> spp. utilizados en el análisis PCR específica <strong>de</strong><br />
<strong>Fusarium</strong> oxysporum f. sp. ciceris y <strong>de</strong> las razas patogénicas 0, 1A, 5, y 6<br />
<strong>Fusarium</strong> spp. a Aislado<br />
F. oxysporum Schlechtend.:Fr.<br />
f. sp. lycopersici (Sacc.) Sny<strong>de</strong>r & Hansen<br />
f. sp. melonis (Leach & Currence) Sny<strong>de</strong>r &<br />
Hansen Fom-8701, -9016<br />
f. sp. niveum (E. F. Sm.) Sny<strong>de</strong>r & Hansen Fon-8805, Fon-8822<br />
f. sp. phaseoli Kendr. & Sny<strong>de</strong>r DR85<br />
F. acuminatum Ellis & Kelerm. 11442<br />
F. acutatum Nirenberg & O’Donnell 10769<br />
F. armeniacum (Forbes et al.) Burgess & Summerell 11623<br />
F. andiyazi 4647<br />
F. anthophilum (A. Braun) Wollenw. 11560<br />
F. avenaceum (Fr.) Sacc. 11440<br />
F. babinda Summerell, Rugg & Burgess 11478<br />
F. begoniae Nirenberg & O’Donnell 10767<br />
F. brevicatenulatum Nirenberg & O’Donnell 10756<br />
F. bulbicola Nirenberg & O’Donnell 10759<br />
F. chlamydosporum Wollenw. & Reiking 11397<br />
F. circinatum Nirenberg & O’Donnell 10766, H-10847, H-10850<br />
F. compactum (Wollenw.) Gordon 11624<br />
F. concentricum Nirenberg & O’Donnell 10765<br />
F. crookwellense Burgess, Nelson, & Tousson 11451<br />
F. culmorum (W. G. Smith) Sacc. 11427<br />
F. <strong>de</strong>cemcellulare Brick 11411<br />
F. <strong>de</strong>nticulatum Nirenberg & O’Donnell 10763<br />
F. dimerum Penzig 11425<br />
F. equiseti (Corda) Sacc. 11439<br />
F. globosum Rhee<strong>de</strong>r et al. 11554<br />
F. guttiforme Nirenberg & O’Donnell 10764<br />
F. lactis Pirotta & Riboni 10757<br />
F. lateritium Nees 11403<br />
F. longipes Wollenw. & Reiking 11428<br />
54
Capítulo II<br />
Tabla 6. (Continuación) Aislados <strong>de</strong> diferentes formae speciales <strong>de</strong> <strong>Fusarium</strong><br />
oxysporum, así como <strong>de</strong> otras <strong>Fusarium</strong> spp. utilizados en el análisis PCR<br />
específica <strong>de</strong> F. oxysporum f. sp. ciceris y <strong>de</strong> las razas patogénicas 0, 1A, 5, y 6<br />
<strong>Fusarium</strong> spp. a Aislado<br />
F nelsonii Marasas & Logrieco<br />
11564<br />
F. nisikadoi Nirenberg & Aoki 10758<br />
F. nygamai Burgess & Trimboli 5111<br />
F. phyllophilum Nirenberg & O’Donnell 10768<br />
F. poae (Peck) Wollenw. 11470<br />
F. polyphialidicum Marasas, Nelson, Tousson & van Wijk 11414<br />
F. proliferatum (Matsushima) Nirenberg 11558, D-4853, D-4854<br />
F. pseudoanthophilum Nirenberg et al. 10755<br />
F. pseudocircinatum O’Donnell & Nirenberg 10761<br />
F. pseudograminearum O´Donnel & Aoki 11435<br />
F. pseudonygamai O’Donnell & Nirenberg 10762<br />
F. ramigenum O’Donnell & Nirenberg 10670<br />
F. sacchari (Butler) W. Gams 278, B-3852, B-3853<br />
F. scirpi Lambotte & Fautr. 11409<br />
F. semisectum Berk. & Rav. 11432<br />
F. solani (Martius) Sacc. 11420<br />
F. sporotrichioi<strong>de</strong>s Sherb. 11552<br />
F. subglutinans (Wollenw. & Reinking) Nelson et al. 11544, E-990, E-2192<br />
F. thapsinum Klittich et al. F-4093, F-4094<br />
F. torulosum (Berk. & Curt.) Nirenberg 11419<br />
F. tricinctum (Corda) Sacc. 11566<br />
F. verticillioi<strong>de</strong>s (Sacc.) Nirenberg 11556, A-149, A-999<br />
a Los aislados <strong>de</strong> F. oxysporum proce<strong>de</strong>n <strong>de</strong> la micoteca <strong>de</strong>l Departamento <strong>de</strong><br />
Protección <strong>de</strong> Cultivos, Instituto <strong>de</strong> Agricultura Sostenible, CSIC, Córdoba.<br />
Los aislados <strong>de</strong> las diferentes especies <strong>de</strong> <strong>Fusarium</strong> fueron proporcionados por<br />
el Dr. J. F. Leslie, Department of Plant Pathology, Kansas State University,<br />
Manhattan, KS, EEUU.<br />
55
Capítulo II<br />
reacción fueron iguales a las <strong>de</strong>scritas anteriormente, salvo porque se<br />
utilizó 0,6 U <strong>de</strong> EcoTaq ADN Polimerasa (EcoGen, Madrid, España) por<br />
reacción. Las amplificaciones se realizaron en los termocicladores Perkin-<br />
Elmer 2400 y Perkin-Elmer 9600 (Perkin-Elmer, Norwalk, CT, EEUU).<br />
Posteriormente, los fragmentos RAPD fueron separados mediante<br />
electroforesis en geles <strong>de</strong> agarosa y visualizados bajo luz ultravioleta, como<br />
se ha <strong>de</strong>scrito anteriormente (Pág. 28, Capítulo I)<br />
Clonación <strong>de</strong> fragmentos RAPD<br />
Los fragmentos RAPD asociados a alguna <strong>de</strong> las razas <strong>de</strong> F. oxysporum f.<br />
sp. ciceris se retiraron <strong>de</strong>l gel <strong>de</strong> agarosa y se purificaron mediante el ‘kit’<br />
Qiaex II (Qiagen, Hil<strong>de</strong>n, Alemania), siguiendo las instrucciones <strong>de</strong>l<br />
fabricante. La estrategia seguida fue la clonación T/A, que se basa en que<br />
la gran mayoría <strong>de</strong> las enzimas ADN polimerasas generan fragmentos <strong>de</strong><br />
ADN con extremo 3’-A; con lo cual el método más eficaz <strong>de</strong> clonación es<br />
unir dichos fragmentos amplificados a vectores bacterianos linearizados<br />
que presentan extremos 3’-T. Para ello se utilizaron los plásmidos<br />
bacterianos pGem-T (Promega, Madison, WI, EEUU) o pCR2.1-TOPO<br />
vector (‘TOPO TA Cloning kit’, Invitrogen, Groningen, Holanda), y<br />
posteriormente se transformaron células supercompetentes DH5α<br />
(Sambrook et al., 1989) o TOP 10F’ (‘TOPO TA Cloning Kit’, Invitrogen,<br />
Groningen, Holanda) <strong>de</strong> Escherichia coli.<br />
Las colonias bacterianas que albergaban plásmido recombinante fueron<br />
i<strong>de</strong>ntificadas por la tonalidad blanca que adquirieron en el medio Luria-<br />
Bertani (LB) ampicilina/X-gal/IPTG, en contraste con la coloración azul<br />
que presentan las colonias bacterianas con plásmido sin inserto, <strong>de</strong>bido a la<br />
α complementación (Sambrook et al., 1989). Para obtener los clones<br />
bacterianos que albergaban los plásmidos con el inserto correcto se realizó<br />
el siguiente proceso: (1) se seleccionaron entre 30 y 50 colonias blancas<br />
por cada transformación; (2) se purificó ADN plasmídico a partir <strong>de</strong> 1,5<br />
56
Capítulo II<br />
ml <strong>de</strong> cada cultivo bacteriano incubado durante la noche a 37 ºC en medio<br />
LB líquido, mediante el método <strong>de</strong> lisis por ebullición (Sambrook et al.,<br />
1989); (3) se seleccionaron un conjunto <strong>de</strong> clones potencialmente<br />
a<strong>de</strong>cuados en base a su migración en gel <strong>de</strong> agarosa; (4) <strong>de</strong> ese primer<br />
conjunto seleccionado se eligieron entre cuatro y seis clones según su<br />
perfil <strong>de</strong> restricción con las enzimas EcoRI, BamHI, XbaI, ApaI, PstI, y SacI<br />
(Roche Diagnostics, Mannheim, Alemania; Pharmacia KLB, Upsala,<br />
Suecia); y (5) cada uno <strong>de</strong> los clones seleccionados fue utilizado como<br />
sonda en experimentos <strong>de</strong> hibridación con el perfil RAPD generado por el<br />
iniciador que amplificó el fragmento original.<br />
Purificación <strong>de</strong> plásmido bacteriano<br />
El plásmido bacteriano se purificó por tres métodos diferentes, i. e.,<br />
mediante lisis por ebullición, lisis alcalina, y ‘Qiagen Plasmid Minikit’<br />
(Qiagen, Hil<strong>de</strong>n, Alemania). En el método <strong>de</strong> lisis por ebullición se<br />
centrifugaron (5 min, 6.000 rpm) 1,5 ml <strong>de</strong> cultivo bacteriano en medio LB<br />
líquido con ampicilina (incubado en agitación durante la noche a 37 ºC), y<br />
el precipitado se resuspendió en 350 µl <strong>de</strong> solución STET (8% sacarosa,<br />
0,5% Triton-X, 50 mM EDTA, 10 mM Tris, pH 8,0). Posteriormente, se<br />
añadieron a la mezcla 25 µl <strong>de</strong> lisozima fresca (10 mg/ml, en 10 mM<br />
Tris·HCl [pH 8,0]), y ésta se incubó a 100 ºC durante 40 s exactos.<br />
Después, la mezcla se centrifugó (10 min, 13.000 rpm) para eliminar los<br />
restos <strong>de</strong> células bacterianas, al sobrenadante acuoso se añadieron 40 µl <strong>de</strong><br />
2,5 M acetato sódico (pH 5,2) y 420 µl <strong>de</strong> isopropanol puro, y<br />
seguidamente se centrifugó (5 min, 13.000 rpm) para precipitar el ADN. El<br />
ADN precipitado se resuspendió en 40 µl <strong>de</strong> H 2O UHQ.<br />
Para la purificación <strong>de</strong> ADN plasmídico mediante lisis alcalina, el<br />
precipitado obtenido por centrifugación <strong>de</strong> 3 ml <strong>de</strong> cultivo bacteriano en<br />
medio LB líquido con ampicilina (incubado en agitación durante la noche,<br />
a 37 ºC) se resuspendió en 200 µl <strong>de</strong> solución tampón GTE (50 mM<br />
Glucosa, 25 mM Tris [pH 8,0], 10 mM EDTA). Después, a la suspensión<br />
se añadieron secuencialmente 300 µl <strong>de</strong> una solución fresca <strong>de</strong> 0,2 M<br />
57
Capítulo II<br />
NaOH :1% SDS, y 300 µl <strong>de</strong> 3 M acetato sódico, con 5 min <strong>de</strong> incubación<br />
a 0 ºC <strong>de</strong>spués <strong>de</strong> cada adición. Seguidamente, la mezcla se centrifugó (10<br />
min, 13.000 rpm), y se trató el sobrenadante con ARNasa. El sobrenadante<br />
tratado se extrajo con cloroformo : alcohol isoamílico (24:1), se precipitó<br />
el ADN plasmídico con isopropanol, y se centrifugó 30 min a 13.000 rpm.<br />
El ADN se resuspendió en 30 µl <strong>de</strong> H 2O UHQ.<br />
Por último, el ADN plasmídico <strong>de</strong>stinado a la secuenciación fue<br />
purificado mediante el ‘Qiagen Plasmid Minikit’ siguiendo las instrucciones<br />
<strong>de</strong>l fabricante.<br />
Hibridación <strong>de</strong> las bandas clonadas con los perfiles <strong>de</strong> amplificación<br />
RAPD y PCR específica<br />
Los fragmentos RAPD clonados en vectores bacterianos se utilizaron<br />
como sondas <strong>de</strong> ADN en hibridaciones con el perfil RAPD generado por<br />
el iniciador que originó cada fragmento transferido a membranas <strong>de</strong> nylon.<br />
Estas hibridaciones se llevaron a cabo con cuatro fines: (1) confirmar que<br />
el clon bacteriano seleccionado albergaba el inserto <strong>de</strong> estudio, y no otro<br />
diferente, como resultado <strong>de</strong> la escisión incorrecta <strong>de</strong> ADN <strong>de</strong>l perfil<br />
RAPD; (2) confirmar que existe homología entre las bandas <strong>de</strong>l mismo<br />
peso molecular amplificadas en aislados pertenecientes a la misma raza; (3)<br />
confirmar que esa misma banda RAPD no está siendo amplificada en<br />
aislados <strong>de</strong> otras razas a intensidad no <strong>de</strong>tectable en gel <strong>de</strong> agarosa; y (4)<br />
<strong>de</strong>sestimar la posibilidad <strong>de</strong> que exista homología entre el fragmento<br />
RAPD en proceso <strong>de</strong> estudio, y otros fragmentos <strong>de</strong> diferente peso<br />
molecular originados por el mismo iniciador aleatorio. Para ello, los<br />
perfiles <strong>de</strong> amplificación RAPD <strong>de</strong> un grupo <strong>de</strong> aislados representativos <strong>de</strong><br />
todas las razas patogénicas y orígenes geográficos <strong>de</strong>l patógeno, separados<br />
en geles <strong>de</strong> agarosa tales como los <strong>de</strong>scritos con anterioridad, fueron<br />
transferidas por capilaridad en condiciones alcalinas (Sambrook et al.,<br />
1989) a membranas Zeta-Probe Blotting Membranes (Bio-Rad Lab. S.A.,<br />
Madrid).<br />
58
Capítulo II<br />
Para la producción <strong>de</strong> las sondas <strong>de</strong> ADN, se obtuvo primero el inserto<br />
correspondiente mediante digestión <strong>de</strong>l plásmido bacteriano con las<br />
enzimas <strong>de</strong> restricción que tenían su secuencia <strong>de</strong> corte en la región<br />
‘polylinker’ <strong>de</strong>l vector. Seguidamente, el inserto y el vector se separaron<br />
electroforéticamente, y el inserto se escindió <strong>de</strong>l gel <strong>de</strong> agarosa mediante el<br />
‘kit’ Quiaex II, y se marcó. El marcaje se realizó por métodos no<br />
radiactivos, mediante la incorporación <strong>de</strong> DIG-11-dUTP [digoxigenina-3-<br />
O– metilcarbonil – amino– caproil– 5-(3-aminoalil)-ridina5’trifosfato]con<br />
iniciadores aleatorios utilizando el ‘DNA labelling kit’ (Roche Diagnostics,<br />
Mannheim, Alemania).<br />
Los tratamientos <strong>de</strong> prehibridación e hibridación se realizaron en<br />
condiciones altamente astringentes (68 ºC) según lo <strong>de</strong>scrito por<br />
Sambrook et al. (1989). El revelado <strong>de</strong> las membranas <strong>de</strong> hibridación se<br />
realizó mediante métodos quimioluminiscentes utilizando el ‘kit’ <strong>de</strong><br />
revelado ‘DIG Luminescent Detection Kit’ (Roche Diagnostics,<br />
Mannheim, Alemania) con CSPD (Roche Diagnostics, Mannheim,<br />
Alemania), siguiendo las instrucciones <strong>de</strong>l fabricante. Las membranas<br />
reveladas se expusieron a películas BioMax Light (Eastman Kodak<br />
Company, Rochester, NY, EEUU) durante un tiempo <strong>de</strong> 15 a 30 min, a<br />
temperatura ambiente, procediendo seguidamente al revelado <strong>de</strong> las<br />
películas siguiendo las instrucciones <strong>de</strong>l fabricante.<br />
De igual manera, se realizaron hibridaciones <strong>de</strong> las mismas sondas <strong>de</strong><br />
ADN <strong>de</strong>scritas anteriormente con los productos <strong>de</strong> análisis PCR<br />
específica. Estos productos fueron generados por los iniciadores para PCR<br />
diseñados en esta investigación para la i<strong>de</strong>ntificación específica <strong>de</strong> F.<br />
oxysporum f. sp. ciceris y <strong>de</strong> las razas patogénicas 0, 1A, 1B/C, 5, y 6. El<br />
proceso se realizó <strong>de</strong> forma idéntica al <strong>de</strong>scrito anteriormente para la<br />
hibridación <strong>de</strong> los perfiles <strong>de</strong> amplificación RAPD.<br />
59
Capítulo II<br />
Hibridaciones ‘Southern’<br />
Las sondas <strong>de</strong> ADN generadas como se ha <strong>de</strong>scrito anteriormente también<br />
se utilizaron para realizar hibridaciones con ADN total <strong>de</strong> aislados <strong>de</strong> una<br />
raza digerido con la enzima <strong>de</strong> restricción EcoRI. Para ello, se digirieron<br />
entre 3 y 5 µg <strong>de</strong> ADN con 15 a 25 U <strong>de</strong> EcoRI incubando a 37ºC durante<br />
12-14 h. Los fragmentos generados por la digestión se separaron<br />
electroforéticamente en geles <strong>de</strong> agarosa al 0,8 % <strong>de</strong> 15 x 25 cm, en<br />
tampón TAE, a 1 V·cm -1 durante 12-14 h, y posteriormente se<br />
transfirieron a membranas <strong>de</strong> nylon Zeta-Probe Blotting Membranes<br />
mediante vacío (VacuGene XL y VacuGene Pump, Pharmacia LKB,<br />
Upsala, Suecia). Los tratamientos <strong>de</strong> prehibridación e hibridación se<br />
realizaron en condiciones <strong>de</strong> alta astringencia (68 ºC) (Sambrook et al.,<br />
1989). El revelado <strong>de</strong> las membranas <strong>de</strong> hibridación se realizó como se ha<br />
indicado anteriormente. Una vez reveladas, las membranas se expusieron a<br />
películas BioMax Light durante un tiempo variable, <strong>de</strong>s<strong>de</strong> 2 a 12 h. El<br />
revelado <strong>de</strong> las películas se realizó como ha sido <strong>de</strong>scrito en el apartado<br />
anterior.<br />
Secuenciación <strong>de</strong> los fragmentos RAPD clonados<br />
Los insertos <strong>de</strong> cada uno <strong>de</strong> los plásmidos seleccionados, correspondientes<br />
a cada una <strong>de</strong> las bandas RAPD asociadas a las razas <strong>de</strong> F. oxysporum f. sp.<br />
ciceris, fueron secuenciados por los servicios <strong>de</strong> secuenciación <strong>de</strong> la<br />
Universidad <strong>de</strong> Cornell, Ithaca, NY, EEUU, y <strong>de</strong> NBT (Newbiotechnic,<br />
S.A., Sevilla, España). Las dos ca<strong>de</strong>nas <strong>de</strong> ADN <strong>de</strong> las bandas marcadoras<br />
se secuenciaron utilizando un secuenciador ABI 377 <strong>de</strong> Applied<br />
Biosystems, con los iniciadores universales M13 forward (-21) y reverse,<br />
así como con iniciadores internos a la secuencia, y posterior solapamiento<br />
hasta completar la totalidad <strong>de</strong>l inserto. Los cromatogramas fueron<br />
visualizados y alineados con el programa informático Sequencher 3.1.1.<br />
(GenCo<strong>de</strong>s, Ann Arbor, MI, EEUU). Se realizó una búsqueda <strong>de</strong><br />
homología <strong>de</strong> las secuencias mediante el programa BLASTN 2.1.1<br />
60
Capítulo II<br />
(Altschul et al., 1997) <strong>de</strong>l servicio NCBI (‘National Center for<br />
Biotechnology Information’; www.ncbi.nlm.nih.gov).<br />
Diseño <strong>de</strong> iniciadores específicos<br />
Los iniciadores para PCR específica se diseñaron mediante el programa <strong>de</strong><br />
PrimerSelect 3.11 <strong>de</strong>l paquete <strong>de</strong> ‘software DNA Star’ (Madison, WI,<br />
EEUU). Las secuencias seleccionadas para construir los iniciadores<br />
estuvieron basadas en las regiones más externas <strong>de</strong> los fragmentos RAPD<br />
clonados, incluyendo parte <strong>de</strong>l iniciador aleatorio RAPD que las originó.<br />
Los iniciadores para PCR específica fueron sintetizados comercialmente<br />
por GENSET S. A. (Paris, Francia). Las secuencias <strong>de</strong> cada uno <strong>de</strong> estos<br />
iniciadores se presentan en la Tabla 8.<br />
Reacciones <strong>de</strong> amplificación PCR<br />
Las reacciones <strong>de</strong> amplificación PCR se pusieron a punto<br />
in<strong>de</strong>pendientemente para cada par <strong>de</strong> iniciadores. Las condiciones <strong>de</strong><br />
amplificación PCR específica para la i<strong>de</strong>ntificación <strong>de</strong> F. oxysporum f. sp.<br />
ciceris, así como <strong>de</strong> cada una <strong>de</strong> la raza 0, 1B/C, 6, y 1A-6 fueron las que<br />
siguen a continuación. Las mezclas <strong>de</strong> reacción (25 µl) estuvieron<br />
compuestas por 0,2 µM <strong>de</strong> cada iniciador, 200 µM <strong>de</strong> cada dNTP, 2,5 µl <strong>de</strong><br />
10 x tampón <strong>de</strong> reacción [166 mM (NH 4) 2SO 4; 670 mM Tris-HCl (pH 8,0,<br />
25ºC); 0,1% Tween-20], 0,6 U <strong>de</strong> EcoTaq ADN Polimerasa (EcoGen,<br />
Madrid, España), 1,0 mM MgCl 2, y 10-25 ng <strong>de</strong> ADN fúngico. Las<br />
amplificaciones se realizaron en los termocicladores Perkin-Elmer 2400 y<br />
Perkin-Elmer 9600. Los ciclos <strong>de</strong> temperaturas consistieron en 2 min <strong>de</strong><br />
<strong>de</strong>snaturalización inicial a 94ºC, 25 a 28 ciclos <strong>de</strong> 30 s <strong>de</strong> <strong>de</strong>snaturalización<br />
a 94ºC, 1 min <strong>de</strong> complementación a 58ºC para los iniciadores específicos<br />
<strong>de</strong> F. oxysporum f. sp. ciceris y <strong>de</strong> 61ºC para los <strong>de</strong> las razas 0, 1B/C, 6, y 1A-<br />
6, y 30 s <strong>de</strong> polimerización a 72ºC; y un último paso <strong>de</strong> 4 min <strong>de</strong> extensión<br />
61
Capítulo II<br />
a 72ºC para producir fragmentos completos <strong>de</strong> ADN <strong>de</strong> doble ca<strong>de</strong>na<br />
(Tabla 8).<br />
Para la amplificación PCR específica utilizando ADN genómico <strong>de</strong><br />
aislados <strong>de</strong> la raza 5 se utilizó la modalidad ‘touch-down’ <strong>de</strong> la PCR (Don<br />
et al., 1991). Dicho método consistió en un aumento <strong>de</strong> la temperatura <strong>de</strong><br />
complementación <strong>de</strong> 10ºC en el primer ciclo <strong>de</strong> PCR (71ºC), y la<br />
disminución consecutiva <strong>de</strong> 1ºC en cada ciclo <strong>de</strong> amplificación, durante los<br />
10 primeros ciclos, hasta conseguir la temperatura <strong>de</strong> especificidad óptima<br />
(61ºC), seguido <strong>de</strong> 15 ciclos normales <strong>de</strong> PCR a dicha temperatura óptima<br />
como se ha <strong>de</strong>scrito anteriormente. Esta estrategia tiene por objeto<br />
aumentar tanto la especificidad <strong>de</strong> la reacción como el rendimiento <strong>de</strong> la<br />
amplificación. La composición <strong>de</strong> las mezclas <strong>de</strong> reacción fue la misma que<br />
la <strong>de</strong>scrita para el resto <strong>de</strong> los pares <strong>de</strong> iniciadores, excepto por la<br />
utilización <strong>de</strong> 1 U <strong>de</strong> EcoTaq ADN polimerasa.<br />
Los productos <strong>de</strong> amplificación fueron separados mediante<br />
electroforesis en geles al 1% <strong>de</strong> agarosa, como se ha <strong>de</strong>scrito<br />
anteriormente. Como marcador <strong>de</strong> peso molecular se utilizó el 0,1-kb<br />
DNA lad<strong>de</strong>r XIV (Roche Diagnostics, Mannheim, Alemania).<br />
Todas las reacciones <strong>de</strong> amplificación se repitieron al menos dos veces,<br />
y siempre incluyeron controles negativos (sin ADN mol<strong>de</strong>). A<strong>de</strong>más, para<br />
establecer la cantidad mínima <strong>de</strong> ADN <strong>de</strong>tectable por los iniciadores y<br />
visible en gel <strong>de</strong> agarosa, se realizaron amplificaciones utilizando ADN a<br />
concentraciones <strong>de</strong> 200, 150, 100, 50, 25, 10, 5, 1, 0,1, 0,01, 10 –3 , 10 –4 y 10 -<br />
5 ng en la mezcla <strong>de</strong> reacción. Las condiciones específicas para la<br />
amplificación selectiva <strong>de</strong>l ADN <strong>de</strong> los aislados <strong>de</strong> F. oxysporum f. sp. ciceris<br />
pertenecientes a cada una <strong>de</strong> las razas 0, 1B/C, 5, 6, y 1A-6, así como <strong>de</strong> la<br />
forma specialis ciceris, se presentan recogidas en la Tabla 8.<br />
62
Resultados<br />
Capítulo II<br />
La estrategia seguida para diseñar iniciadores útiles para la i<strong>de</strong>ntificación<br />
específica <strong>de</strong> F. oxysporum f. sp. ciceris, así como para diferenciar entre sus<br />
razas patogénicas 0, 1A, 1B/C, 5, y 6, se ha basado en la utilización <strong>de</strong><br />
secuencias polimórficas SCAR generadas mediante análisis RAPD con<br />
iniciadores arbitrarios. Para ello, los fragmentos RAPD asociados a la forma<br />
specialis ciceris y a cada una <strong>de</strong> sus razas (Capítulo I) se clonaron y<br />
secuenciaron, y <strong>de</strong> las secuencias se diseñaron nuevos iniciadores. A partir<br />
<strong>de</strong> los fragmentos <strong>de</strong> ADN clonados se <strong>de</strong>sarrollaron sondas marcadas<br />
con digoxigenina, con las cuales se realizaron hibridaciones con el perfil<br />
RAPD original y con el ADN genómico digerido mediante enzimas <strong>de</strong><br />
restricción. Posteriormente, se diseñaron iniciadores para PCR específica y<br />
se comprobó la capacidad diagnóstica <strong>de</strong> los productos <strong>de</strong> reacción.<br />
Clonación <strong>de</strong> los fragmentos RAPD<br />
Para la obtención <strong>de</strong> los fragmentos RAPD marcadores raciales, y <strong>de</strong><br />
interés en este estudio, se utilizaron los siguientes aislados <strong>de</strong> F. oxysporum<br />
f. sp. ciceris: raza 0, Foc-9018 JG62; raza 1B/C, Foc-USA3-1JG62; raza 2,<br />
Foc-8605; raza5, Foc-9035; y raza 6, Foc-9093 PV1 (Tabla 7). Los<br />
fragmentos clonados fueron los siguientes: raza 0, 0,39 kb (OPF-12), y 0,9<br />
kb (OPI-09); raza 1B/C, 0,53 kb (OPI-09), y 1,1 kb (OPF-10); raza 2, 1,0<br />
kb (OPF-12); raza 5, 0,9 kb (OPF-10); raza 6, 1,0 kb, y 1,4 kb (OPI-09).<br />
A<strong>de</strong>más, se realizaron clonaciones <strong>de</strong> la banda <strong>de</strong> 1,5 kb amplificada por<br />
OPF-12 y marcadora <strong>de</strong> F. oxysporum f. sp. ciceris, que se obtuvo <strong>de</strong> un<br />
aislado <strong>de</strong> la raza 0 (Foc-7802). Los fragmentos referidos fueron separados<br />
y purificados <strong>de</strong>l gel <strong>de</strong> agarosa, y clonados en un vector bacteriano (Tabla<br />
7).<br />
En todos los casos se obtuvo una variedad consi<strong>de</strong>rable <strong>de</strong> insertos<br />
(entre tres y seis tipos), cuyas diferencias fueron advertidas <strong>de</strong>bido a ligeras<br />
variaciones en el peso molecular <strong>de</strong>l inserto, o a los distintos patrones <strong>de</strong><br />
63
Capítulo II<br />
restricción que se generaron en las digestiones con las enzimas EcoRI,<br />
BamHI, XbaI, ApaI, PstI, y SacI. Estas diferencias en los patrones <strong>de</strong><br />
restricción puso <strong>de</strong> manifiesto la existencia <strong>de</strong> diversos insertos formados<br />
por distintas secuencias <strong>de</strong> ADN, como resultado <strong>de</strong> la clonación <strong>de</strong> un<br />
fragmento RAPD purificado <strong>de</strong>l gel <strong>de</strong> agarosa. La hibridación <strong>de</strong> cada<br />
uno <strong>de</strong> estos insertos con el perfil generado por el iniciador que originó<br />
cada fragmento RAPD permitió seleccionar el clon a<strong>de</strong>cuado. Los<br />
experimentos <strong>de</strong> hibridación confirmaron la existencia <strong>de</strong> secuencias<br />
diferentes, ya que se i<strong>de</strong>ntificaron clones que generaron señal <strong>de</strong><br />
hibridación con bandas <strong>de</strong> igual peso molecular, no visibles en el gel <strong>de</strong><br />
agarosa. Se seleccionaron un total <strong>de</strong> nueve clones bacterianos, cuya<br />
nomenclatura, vector bacteriano, y características <strong>de</strong>l inserto están<br />
recogidos en la Tabla 7.<br />
Hibridaciones con el perfil RAPD<br />
Se realizaron experimentos <strong>de</strong> hibridación con los perfiles <strong>de</strong> amplificación<br />
RAPD generados con cada uno <strong>de</strong> los iniciadores OPF-10, OPF-12 y<br />
OPI-09, utilizando como sonda <strong>de</strong> ADN cada una <strong>de</strong> las bandas RAPD<br />
clonadas. Esto se realizó con los siguientes objetivos: (1) confirmar que el<br />
clon bacteriano seleccionado albergaba el inserto <strong>de</strong> estudio, y no otro<br />
diferente, como resultado <strong>de</strong> la escisión incorrecta <strong>de</strong> ADN <strong>de</strong>l perfil<br />
RAPD; (2) confirmar que existe homología entre las bandas <strong>de</strong>l mismo<br />
peso molecular amplificadas en aislados pertenecientes a la misma raza; (3)<br />
confirmar que esa misma banda RAPD no está siendo amplificada en<br />
aislados <strong>de</strong> otras razas a intensidad no <strong>de</strong>tectable en gel <strong>de</strong> agarosa; y (4)<br />
<strong>de</strong>sestimar la posibilidad <strong>de</strong> que exista homología entre el fragmento<br />
RAPD en proceso <strong>de</strong> estudio, y otros fragmentos <strong>de</strong> diferente peso<br />
molecular originados por el mismo iniciador aleatorio.<br />
64
1,1<br />
2,6<br />
1,<br />
1,<br />
0,<br />
A<br />
M 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19<br />
B<br />
Capítulo II<br />
Figura 4. Amplificación RAPD utilizando ADN <strong>de</strong> aislados <strong>de</strong> <strong>Fusarium</strong><br />
oxysporum f. sp. ciceris con el iniciador aleatorio OPF-10 (A), e hibridación <strong>de</strong>l<br />
mismo perfil <strong>de</strong> amplificación con el fragmento RAPD clonado <strong>de</strong> 1,1 kb<br />
marcador <strong>de</strong> la raza 1B/C (clon FocD33) (B). Los números a la izquierda<br />
correspon<strong>de</strong>n a los pesos moleculares (kb) <strong>de</strong>l ‘0,1 kb lad<strong>de</strong>r XIV’ (Roche<br />
Diagnostic). Carriles: 1-3, aislados <strong>de</strong> la raza 0 Foc–8207, –9018 JG62, –7952; 4,<br />
5, aislados <strong>de</strong> la raza 1B/C Foc–USA 3-1JG62, –1987-W17; 6, 7, aislados <strong>de</strong> la<br />
raza 1A Foc–7989, –9166; 8, 9, aislados <strong>de</strong> la raza 2 Foc–8605, –1992R2N; 10,<br />
11, aislados <strong>de</strong> la raza 3 Foc–8606, –1992R3N; 12, 13, aislados <strong>de</strong> la raza 4 Foc–<br />
8607, –1992R4N; 14-16, aislados <strong>de</strong> la raza 5 Foc–8012, –9035, –USA1-1 JG62;<br />
17, 18, aislados <strong>de</strong> la raza 6 Foc–9023, –Tonini; 19, 20, aislados <strong>de</strong> F. oxysporum<br />
no patogénicos <strong>de</strong> garbanzo, Fo–9009 y –90105, respectivamente.<br />
65
Capítulo II<br />
Los resultados mostraron que en todos los casos hubo 100% <strong>de</strong><br />
homología, indicada por la alta intensidad <strong>de</strong> la señal <strong>de</strong> hibridación entre<br />
las bandas RAPD amplificadas en aislados <strong>de</strong> la misma raza, incluso en<br />
aquéllos que son <strong>de</strong> distinto origen geográfico. Dicha homología no se<br />
observó en aislados pertenecientes a otras razas, excepto por el clon<br />
FocC19. El clon FocC19 (0,39 kb–raza 0–OPF12) produjo señal <strong>de</strong><br />
hibridación en la misma posición en el gel (y por tanto, con un fragmento<br />
amplificado <strong>de</strong>l mismo peso molecular) en las amplificaciones <strong>de</strong> aislados<br />
<strong>de</strong> otras razas <strong>de</strong> F. oxysporum f. sp. ciceris distintas a raza 0, así como con el<br />
aislado <strong>de</strong> F. oxysporum Fo-90105 no patogénico <strong>de</strong> garbanzo. Aunque<br />
dicha banda no fue <strong>de</strong>tectada visualmente en el perfil <strong>de</strong> amplificación<br />
RAPD generado por OPF-12 en otras razas, este resultado <strong>de</strong>muestra que<br />
la misma secuencia no sólo está presente en los genomas <strong>de</strong> dichas razas,<br />
sino que está siendo amplificada a niveles no <strong>de</strong>tectables visualmente en<br />
gel, en aislados <strong>de</strong> otras razas.<br />
Los clones FocD33 (1,0 kb–raza 1B/C–OPF10), FocL10 (0,9 kb–raza<br />
5–OPF10), y FocO2 (1,0 kb–raza 6–OPI09) hibridaron únicamente con la<br />
banda RAPD marcadora racial correspondiente, i. e., las amplificadas<br />
únicamente en los aislados <strong>de</strong> las razas 1B/C, 5, y 6, respectivamente (Fig.<br />
4). En cambio, para el resto <strong>de</strong> los clones se <strong>de</strong>tectó señal <strong>de</strong> hibridación<br />
con otras bandas <strong>de</strong> diferente peso molecular amplificadas en aislados <strong>de</strong><br />
otras razas, pero en ningún caso con bandas <strong>de</strong>l mismo peso molecular.<br />
Otro resultado <strong>de</strong>stacable en estos experimentos fue la hibridación cruzada<br />
entre los clones FocM15 (0,9 kb–raza 0–OPI-09) y FocP18 (1,4 kb–raza<br />
6–OPI-09), aunque existió gran diferencia en la intensidad <strong>de</strong> la señal con<br />
respecto a las hibridaciones homólogas (datos no mostrados). Resultados<br />
posteriores en este estudio confirmaron que una parte <strong>de</strong> las secuencias <strong>de</strong><br />
dichos clones es idéntica.<br />
66
Capítulo II<br />
Tabla 7. Designación y características <strong>de</strong> los plásmidos bacterianos <strong>de</strong><br />
ADN recombinante generados mediante la clonación <strong>de</strong> fragmentos<br />
RAPD marcadores <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris y <strong>de</strong> sus razas<br />
patogénicas<br />
Clon<br />
bacteriano Vector a<br />
Aislado utilizado<br />
Fragmento<br />
RAPD clonado b<br />
FocB10 pGem-T Foc-8605 1,0 kb–R2–OPF12<br />
FocC19 pGem-T Foc-9018 JG62 0,39 kb–R0–OPF-12<br />
FocD33 pGem-T Foc-USA 3-1 JG62 1,1 kb–R1B/C–OPF-10<br />
FocL10 pCR2.1 Foc-9035 0,9 kb–R5–OPF-10<br />
FocM15 pCR2.1 Foc-9018 JG62 0,9 kb–R0–OPI-09<br />
FocN5 pCR2.1 Foc-USA 3-1 JG62 0,53 kb–R1B/C–OPI-09<br />
FocO2 pCR2.1 Foc-9093 PV1 1,0 kb–R6–OPI-09<br />
FocP18 pCR2.1 Foc-9093 PV1 1,4 kb–R6–OPI-09<br />
Foc0-12 pCR2.1 Foc-9018 JG62 1,5 kb–Foc–OPF-12<br />
a Vectores bacterianos comerciales pGem-T (Promega, Madison, WI, EEUU), y<br />
pCR2.1 (TOPO TA Cloning Kit, Invitrogen, Groningen, Holanda).<br />
b Peso molecular aproximado <strong>de</strong>l fragmento RAPD clonado, raza <strong>de</strong> F.<br />
oxyspmorum f. sp. ciceris a la que i<strong>de</strong>ntifica, e iniciador aleatorio que lo generó,<br />
respectivamente.<br />
67
Capítulo II<br />
Hibridaciones ‘Southern’<br />
La naturaleza <strong>de</strong>l ADN contenido en los fragmentos RAPD clonados se<br />
estudió por el patrón que éstos generaron al utilizarlos como sondas en<br />
hibridaciones con ADN genómico digerido con EcoRI. Generalmente, los<br />
patrones <strong>de</strong> hibridación se clasifican en tres clases (Goodwin et al., 1992):<br />
(1) altamente repetitivos, i. e., que presentan un fondo (‘smear’) <strong>de</strong><br />
hibridación indistinguible, o más <strong>de</strong> 20 bandas distintivas; (2)<br />
mo<strong>de</strong>radamente repetitivos, i. e., que presentan entre 5 y 20 bandas <strong>de</strong><br />
hibridación; y (3) <strong>de</strong> copia única o bajo número <strong>de</strong> copias, i. e., aquéllos<br />
cuyos patrones <strong>de</strong> hibridación presentan sólo una a cuatro bandas.<br />
La mayoría <strong>de</strong> las sondas utilizadas en nuestro estudio pue<strong>de</strong>n ser<br />
consi<strong>de</strong>radas como sondas altamente repetitivas, ya que generaron<br />
patrones <strong>de</strong> hibridación que contenían <strong>de</strong> 20 a 30 bandas claramente<br />
distinguibles. La única excepción a este hecho fue el clon FocL10 (0,9 kb–<br />
R5–OPF-10), que hibridó con una o dos <strong>de</strong> las bandas <strong>de</strong> digestión<br />
generadas, según la enzima <strong>de</strong> restricción y según el aislado fúngico<br />
utilizados. Así, Foc10L hibridó sólo con dos <strong>de</strong> las bandas en el ADN <strong>de</strong><br />
aislados <strong>de</strong> la raza 5 <strong>de</strong> F. oxysporum f. sp. ciceris digerido con EcoRI;<br />
mientras que en el resto <strong>de</strong> los aislados sólo se observó señal <strong>de</strong><br />
hibridación con una banda <strong>de</strong> diferente tamaño (Figura 5). Aún así, puesto<br />
que este clon presentó polimorfismos bien en los lugares <strong>de</strong> corte <strong>de</strong> la<br />
enzima o bien en la secuencia homóloga a FocL10, no se <strong>de</strong>sestimó su<br />
posible utilidad para posteriores análisis.<br />
De igual manera, se <strong>de</strong>tectaron numerosos polimorfismos en los<br />
patrones <strong>de</strong> hibridación generados por las secuencias <strong>de</strong> ADN repetitivo.<br />
En general, se observó variabilidad en dichos patrones entre aislados<br />
pertenecientes a las distintas razas <strong>de</strong> F. oxysporum f. sp. ciceris, y entre<br />
aislados <strong>de</strong> la misma raza <strong>de</strong>l patógeno. Las sondas producidas a partir <strong>de</strong><br />
FocM15 y FocP18, que habían originado señal <strong>de</strong> hibridación cruzada con<br />
los perfiles <strong>de</strong> amplificación RAPD, generaron patrones <strong>de</strong> hibridación<br />
con el ADN genómico digerido con EcoRI completamente diferentes. A<br />
continuación, se secuenciaron todos los fragmentos RAPD clonados.<br />
68
8,<br />
7,<br />
4,<br />
2,<br />
1,<br />
M C 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18<br />
Capítulo II<br />
Figura 5. Hibridación ‘Southern’ <strong>de</strong>l ADN genómico <strong>de</strong> aislados <strong>de</strong> <strong>Fusarium</strong><br />
oxysporum f. sp. ciceris digerido con la enzima EcoRI, con la sonda FocL10 (0,9 kb–<br />
raza5–OPF-10). Los números a la izquierda correspon<strong>de</strong>n al peso molecular (kb)<br />
aproximado <strong>de</strong> las bandas <strong>de</strong> ADN. Carriles: M, marcador <strong>de</strong> peso molecular; C,<br />
control <strong>de</strong> hibridación; 1, 2, aislados <strong>de</strong> la raza 0 Foc–8207, –82108; 3, aislado <strong>de</strong><br />
la raza 1B/C Foc–1987-W17; 4, aislado <strong>de</strong> la raza 2 Foc–1992R2N; 5, aislados <strong>de</strong><br />
la raza 4 Foc–1992R4N; 6-8, aislados <strong>de</strong> la raza 1A Foc–7989, –9166, –9027 PV1;<br />
9-13, aislados <strong>de</strong> la raza 5 Foc–8012, –USA1-1 JG62, –USAW6-1, –9035, –9094<br />
JG62; 14, 16, aislados <strong>de</strong> la raza 6 Foc–Tonini, –9093 PV1; 15, 17, y 18, aislados<br />
<strong>de</strong> F. oxysporum no patogénicos <strong>de</strong> garbanzo, Fo–8250, –9009 y –90105,<br />
respectivamente.<br />
69
Capítulo II<br />
Secuenciación <strong>de</strong> los fragmentos RAPD<br />
Se secuenciaron un total <strong>de</strong> nueve clones (Tabla 7). No se <strong>de</strong>tectó<br />
homología significativa alguna con secuencias presentes en GenBank para<br />
los fragmentos representados en los clones FocN5 y Foc0-12. Sin<br />
embargo, la secuencia insertada en el clon FocL10 presentó homologías<br />
con varias proteínas transportadoras <strong>de</strong> urea, permeasas, y otras proteínas<br />
<strong>de</strong> transporte i<strong>de</strong>ntificadas en hongos.<br />
Las secuencias <strong>de</strong> los clones FocB10, FocM15, FocO2, y FocP18<br />
presentaron similitu<strong>de</strong>s con secuencias relacionadas con transposones. En<br />
particular, la secuencia <strong>de</strong> FocB10 presentó un 80% <strong>de</strong> similaridad con el<br />
transposón Fot1 i<strong>de</strong>ntificado en F. oxysporum f. sp. melonis (Leach &<br />
Currence) W. C. Sny<strong>de</strong>r & H. N. Hans. (Daboussi et al., 1992).<br />
La secuencia <strong>de</strong> FocO2 presentó un 52% <strong>de</strong> similaridad con una<br />
proteína similar a una transposasa i<strong>de</strong>ntificada en F. oxysporum (Okuda et<br />
al., 1998); y las <strong>de</strong> FocP18 y FocM15 con transposasas putativas <strong>de</strong> otros<br />
organismos.<br />
Las secuencias <strong>de</strong> FocM15 y FocP18, que habían originado señal <strong>de</strong><br />
hibridación cruzada con los productos <strong>de</strong> amplificación RAPD,<br />
presentaron 247 bp idénticas entre si, <strong>de</strong> un total <strong>de</strong> 0,9 kb y 1,4 kb,<br />
respectivamente. Sin embargo, el resto <strong>de</strong> las secuencias <strong>de</strong> ambos clones<br />
fueron completamente diferentes. Ésto explica la obtención <strong>de</strong> señal <strong>de</strong><br />
hibridación cruzada con la amplificación RAPD generada por OPI-09.<br />
Debido a que al menos uno <strong>de</strong> los extremos <strong>de</strong> ambos clones eran<br />
diferentes, la utilidad potencial <strong>de</strong> estas secuencias para diseñar iniciadores<br />
que amplifiquen SCARs no fue <strong>de</strong>sestimada.<br />
Reacciones <strong>de</strong> amplificación PCR<br />
Para el diseño <strong>de</strong> los iniciadores específicos se seleccionaron los clones<br />
FocD33, FocL10, FocM15, FocN5, FocO2, FocP18, y Foc0-12 (Tabla 7).<br />
En total se diseñaron siete pares <strong>de</strong> iniciadores para reacciones PCR<br />
70
Capítulo II<br />
específica (Tabla 8). Todos los iniciadores fueron diseñados a partir <strong>de</strong> las<br />
secuencias extremas <strong>de</strong> los fragmentos clonados, <strong>de</strong> forma que<br />
contuviesen parte <strong>de</strong>l iniciador aleatorio RAPD que los originó. Estos<br />
iniciadores se utilizaron para amplificar ADN <strong>de</strong> aislados representativos<br />
<strong>de</strong> todas las razas patogénicas <strong>de</strong> F. oxysporum f. sp. ciceris y <strong>de</strong> un amplio<br />
rango geográfico <strong>de</strong>l patógeno.<br />
Todos los pares <strong>de</strong> iniciadores amplificaron los fragmentos <strong>de</strong>l tamaño<br />
esperado. Los pares <strong>de</strong> iniciadores FocR1B/C-D33fwd y FocR1B/C-<br />
D33rev; y FocR6-P18fwd y FocR6-P18rev, que se diseñaron para<br />
i<strong>de</strong>ntificar a las razas 1B/C y 6 <strong>de</strong> F. oxysporum f. sp. ciceris,<br />
respectivamente, amplificaron fragmentos <strong>de</strong> ADN <strong>de</strong> la misma intensidad<br />
con el ADN <strong>de</strong> aislados <strong>de</strong> cada una <strong>de</strong> las razas <strong>de</strong>l patógeno (datos no<br />
mostrados), por lo cual fueron <strong>de</strong>sestimados.<br />
En cambio, los iniciadores FocR0-M15fwd y FocR0-M15rev<br />
amplificaron un fragmento <strong>de</strong> 900 bp únicamente en los aislados <strong>de</strong> la raza<br />
0 <strong>de</strong> F. oxysporum f. sp. ciceris y con igual intensidad en todos ellos, pero no<br />
se produjeron amplicones en el resto <strong>de</strong> las razas, ni en aislados <strong>de</strong> F.<br />
oxysporum no patogénicos <strong>de</strong> garbanzo (Fig. 6A). Hibridaciones <strong>de</strong>l<br />
fragmento <strong>de</strong> 900 bp con los productos <strong>de</strong> amplificación PCR utilizando<br />
dichos iniciadores y ADN <strong>de</strong> aislados representativos <strong>de</strong> cada raza <strong>de</strong>l<br />
patógeno, generaron señal únicamente en los aislados <strong>de</strong> la raza 0 (datos<br />
no mostrados). Ello significa que esta secuencia <strong>de</strong> 900 bp no es<br />
amplificada en ninguna otra raza distinta <strong>de</strong> la 0, ni siquiera a niveles no<br />
visuales en gel <strong>de</strong> agarosa.<br />
De igual manera, los iniciadores FocR6-O2fwd y FocR6-O2rev<br />
amplificaron un fragmento <strong>de</strong> 1000 bp únicamente en los aislados <strong>de</strong> la<br />
raza 6 <strong>de</strong> F. oxysporum f. sp. ciceris, pero no en el resto <strong>de</strong> las razas <strong>de</strong>l<br />
patógeno ni en aislados <strong>de</strong> F. oxysporum no patogénicos <strong>de</strong> garbanzo (Fig.<br />
6C); y dicha amplificación fue asimismo corroborada mediante ensayos <strong>de</strong><br />
hibridación (datos no mostrados). Los iniciadores FocR1B/C-N5fwd y<br />
FocR1B/C-N5rev, diseñados a partir <strong>de</strong> la secuencia <strong>de</strong> FocN5 (0,53 kb–<br />
R1B/C–OPI09), amplificaron un único fragmento <strong>de</strong> 553 bp en los<br />
71
Capítulo II<br />
Tabla 8. Secuencias <strong>de</strong> los iniciadores, y condiciones <strong>de</strong> amplificación para su utilización en análisis PCR específica para la i<strong>de</strong>ntificación <strong>de</strong><br />
<strong>Fusarium</strong> oxysporum f. sp. ciceris y sus razas patogénicas 0, 1A, 1B/C, 5, y 6<br />
72<br />
Iniciador a Secuencia (5’-3’) b Raza<br />
Tamaño <strong>de</strong>l<br />
fragmento<br />
Condiciones <strong>de</strong><br />
amplificación c<br />
Foc-0-12fwd GGCGTTTCGCAGCCTTACAATGAAG F. o. ciceris 1,5 kb Tª complementación 58 ºC<br />
Foc-0-12rev GACTCCTTTTTCCCGAGGTAGGTCAGAT<br />
28 ciclos <strong>de</strong> PCR<br />
FocR0-M15fwd GGAGAGCAGGACAGCAAAGACTA R0 0,9 kb Tª complementación 61 ºC<br />
FocR0-M15rev GGAGAGCAGCTACCCTAGATACACC<br />
28 ciclos <strong>de</strong> PCR<br />
FocR1B/C-N5fwd GAGAGCAGGGTCAGCGTAGATAG R1B/C 0,5 kb Tª complementación 61 ºC<br />
FocR1B/C-N5rev GCAGCAGAAGAGGAAGAAAATGTA<br />
25 ciclos <strong>de</strong> PCR<br />
FocR5-L10fwd GGAAGCTTGGCATGACATAC R5 0,9 kb PCR ‘touch-down’<br />
FocR5-L10rev AAGCTTGGGCACCCTCTT<br />
Tª complementación 71-61ºC<br />
FocR6-O2fwd GAGCAGTCAATGGCAATGG R6 1,0 kb Tª complementación 61 ºC<br />
FocR6-O2rev AGAGCAGGGTCAGCGTAGATA<br />
28 ciclos <strong>de</strong> PCR<br />
FocR6-P18fwd GGAGAGCAGTAGAGTTACAGCAGTATT R1A, R6 1,5 kb Tª complementación 61 ºC<br />
FocR0-M15rev GGAGAGCAGCTACCCTAGATACACC<br />
25 ciclos <strong>de</strong> PCR<br />
a La nomenclatura <strong>de</strong> los iniciadores: raza <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris a la que i<strong>de</strong>ntifican y clon bacteriano en el que se basó su<br />
secuencia, a excepción <strong>de</strong> los iniciadores Foc0-12fwd y rev que amplifican específicamente ADN <strong>de</strong> la forma specialis ciceris, y los<br />
iniciadores FocR6-P18fwd y FocM15rev que amplifican ADN <strong>de</strong> las razas 1A y 6 cuando son utilizados conjuntamente.<br />
b En el diseño <strong>de</strong> los iniciadores para PCR específica se utilizaron las secuencias <strong>de</strong> los fragmentos RAPD clonados, <strong>de</strong> manera que<br />
contuvieron parte o la totalidad <strong>de</strong>l iniciador aleatorio que los generó (secuencia subrayada), a excepción <strong>de</strong> los iniciadores Foc0-12<br />
fwd y rev cuyas secuencias fueron algo más internas.<br />
c Los ciclos <strong>de</strong> PCR normales consistieron en: 2 min-94ºC + 25 ó 28 ciclos [30s-94ºC + 1 min-Tª complementación + 30s-72ºC] +<br />
4 min-72ºC. El ciclo <strong>de</strong> PCR ‘touch-down’ (Don et al., 1991) estuvo compuesto por: 2 min-94ºC + 10 [30s-94ºC + 1 min- 71ºC-<br />
61ºC + 30s-72ºC] + 15 [30s-94ºC + 1 min- 61ºC + 30s-72ºC] + 4 min-72ºC.
2,6<br />
1,<br />
1,<br />
0,<br />
2,6<br />
1,<br />
1,<br />
0,<br />
Capítulo II<br />
A M 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 B M 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18<br />
C D<br />
M 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 M 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18<br />
Figura 6. Amplificaciones PCR específica <strong>de</strong> las razas 0 (A), 5 (B), 6 (C), y 1A-6<br />
(D) <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris realizadas con los inciadores basados en<br />
secuencias SCAR obtenidas a partir <strong>de</strong> los fragmentos RAPD marcadores. Los<br />
números a la izquierda correspon<strong>de</strong>n a los pesos moleculares (kb) <strong>de</strong>l ‘0,1 kb<br />
lad<strong>de</strong>r XIV’ (Roche Diagnostic). A, y C, carriles: 1-3, aislados <strong>de</strong> la raza 0 Foc–<br />
7802, 82113, –91108; 4, 5, aislados <strong>de</strong> la raza 1B/C Foc–USA 3-1JG62, –1987-<br />
W17; 6-8, aislados <strong>de</strong> las razas 2, 3, y 4 Foc Foc–8605, –8606, –8607; 9-11,<br />
aislados <strong>de</strong> la raza 1A Foc–7989, –9166, –9164; 12-14, aislados <strong>de</strong> la raza 6 Foc–<br />
Tonini, –9093 JG62, –9023; 15-16, aislados <strong>de</strong> la raza 5 Foc–8012, –USA 14201;<br />
y 17-18, aislados <strong>de</strong> F. oxysporum no patogénicos <strong>de</strong> garbanzo, Fo–90101 y –<br />
90105, respectivamente. B, carriles: 1-3, aislados <strong>de</strong> la raza 0 Foc–7802, 82113, –<br />
91108; 4, 5, aislados <strong>de</strong> la raza 1B/C Foc–USA 3-1JG62, –1987-W17; 6-8,<br />
aislados <strong>de</strong> las razas 2, 3, y 4 Foc Foc–8605, –8606, –8607; 9-10, aislados <strong>de</strong> la<br />
raza 1A Foc–7989, –9166; 11-12, aislados <strong>de</strong> la raza 6 Foc–Tonini, –9023; 13-17,<br />
aislados <strong>de</strong> la raza 5 Foc–8012, –9035, –USA W6-1, –USA 1-1 JG62, –USA<br />
14201; 18, 19, aislados <strong>de</strong> F. oxysporum no patogénicos <strong>de</strong> garbanzo, Fo–90101 y –<br />
9081, respectivamente. D, carriles: 1-5, aislados <strong>de</strong> la raza 1A Foc–7989, –9164, –<br />
9166, –9168, 9027 PV1; 6-12, aislados <strong>de</strong> la raza 6 Foc–Tonini, –9093 JG62, –<br />
9023, –9170, –8272, – 8905, –8924; 13-18, aislados <strong>de</strong> cada una <strong>de</strong> las razas 0,<br />
1B/C, 2, 3, 4, y 5, Foc–7802, –USA 3-1JG62, –8605, –8606, –8607, –8012; 19,<br />
aislado <strong>de</strong> F. oxysporum no patogénico <strong>de</strong> garbanzo Fo–90105, respectivamente.<br />
73
Capítulo II<br />
aislados <strong>de</strong> la raza 1B/C proce<strong>de</strong>ntes <strong>de</strong> California. Sin embargo, dichos<br />
iniciadores no amplificaron dicha banda cuando se utilizaron aislados <strong>de</strong> la<br />
raza 1B/C <strong>de</strong> otras zonas geográficas (i. e., Turquía).<br />
Los iniciadores FocR5-L10fwd y FocR5-L10rev amplificaron un<br />
fragmento <strong>de</strong> 938 bp visible únicamente en los aislados <strong>de</strong> la raza 5 <strong>de</strong> F.<br />
oxysporum f. sp. ciceris; sin embargo, los ensayos <strong>de</strong> hibridación revelaron<br />
que dicha secuencia es amplificada a muy baja intensidad en el resto <strong>de</strong> las<br />
razas <strong>de</strong>l patógeno (datos no mostrados). A fin <strong>de</strong> aumentar la<br />
especificidad e intensidad <strong>de</strong> la amplificación con ADN <strong>de</strong> los aislados <strong>de</strong><br />
la raza 5 como mol<strong>de</strong>, se utilizó la modalidad <strong>de</strong> ciclo <strong>de</strong>nominado ‘touchdown’<br />
PCR <strong>de</strong>scrita anteriormente. Esta modalidad <strong>de</strong> PCR mejoró<br />
significativamente el rendimiento <strong>de</strong> la amplificación, generando un<br />
producto <strong>de</strong> mayor intensidad sin que se produjera amplificación alguna en<br />
el resto <strong>de</strong> las razas <strong>de</strong> F. oxysporum f. sp. ciceris (Fig. 6B).<br />
Para la i<strong>de</strong>ntificación específica <strong>de</strong> aislados <strong>de</strong> F. oxysporum f. sp. ciceris, y<br />
su diferenciación <strong>de</strong> aislados <strong>de</strong> F. oxysporum no patogénicos <strong>de</strong> garbanzo,<br />
se diseñaron los iniciadores Foc0-12fwd y Foc0-12rev. Estos iniciadores<br />
amplificaron un único fragmento <strong>de</strong> ADN <strong>de</strong> 1,5 kb solamente en aislados<br />
<strong>de</strong> F. oxysporum f. sp. ciceris, in<strong>de</strong>pendientemente <strong>de</strong> la raza a la que<br />
pertenecían o <strong>de</strong> su origen geográfico, y sin presentar diferencias en la<br />
intensidad <strong>de</strong> la amplificación (Fig. 7). Sin embargo, no se produjo<br />
amplificación alguna, ni señal <strong>de</strong> hibridación, cuando dichos iniciadores se<br />
utilizaron en análisis PCR específica <strong>de</strong> ADN <strong>de</strong> aislados <strong>de</strong> F. oxysporum<br />
no patogénicos <strong>de</strong> garbanzo obtenidos <strong>de</strong> raíces <strong>de</strong> plantas sanas <strong>de</strong> éste,<br />
<strong>de</strong> aislados <strong>de</strong> otras formae speciales <strong>de</strong> F. oxysporum (i. e., melonis, niveum,<br />
lycopersici, phaseoli), ni <strong>de</strong> aislados pertenecientes a otras 47 especies <strong>de</strong><br />
<strong>Fusarium</strong> ( i. e., F. proliferatum, F. solani) (Tabla 6, Fig. 7).<br />
74
A M 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19<br />
2,6<br />
1,<br />
1,<br />
0,<br />
B<br />
1,<br />
Capítulo II<br />
Figura 7. Amplificación PCR específica utilizando ADN <strong>de</strong> aislados <strong>de</strong> <strong>Fusarium</strong><br />
oxysporum f. sp. ciceris, <strong>de</strong> F. oxysporum no patogénicos <strong>de</strong> garbanzo, <strong>de</strong> otras formae<br />
speciales, y <strong>de</strong> diferentes <strong>Fusarium</strong> spp., y los iniciadores Foc0-12fwd y rev (A); e<br />
hibridación <strong>de</strong> los productos <strong>de</strong> amplificación con el fragmento RAPD clonado<br />
<strong>de</strong> 1,5 kb, marcador <strong>de</strong> la forma specielis ciceris (clon Foc0-12) (B). Los números a la<br />
izquierda correspon<strong>de</strong>n a los pesos moleculares (kb) <strong>de</strong>l ‘0,1 kb lad<strong>de</strong>r XIV’<br />
(Roche Diagnostics) (Carril M). Carriles: 1-8, aislados <strong>de</strong> cada una <strong>de</strong> las raza 0,<br />
1B/C, 1A, 2 a 6, Foc–7802, –USA 3-1JG62, –7989, –8605, –8606, –8607, –8012,<br />
–9023; 9-12, aislados <strong>de</strong> F. oxysporum no patogénicos <strong>de</strong> garbanzo, Fo–8250, –<br />
9081, –90105, –506. 13, 14, aislados <strong>de</strong> F. oxysporum f. sp. melonis 9016, y F.<br />
oxysporum f. sp. niveum 8805; 15-19, aislados <strong>de</strong> F. acuminatum, F. avenaceum, F. lactis,<br />
F. proliferatum, y F. solani, respectivamente.<br />
75
Capítulo II<br />
Con objeto <strong>de</strong> <strong>de</strong>terminar la cantidad mínima <strong>de</strong> ADN <strong>de</strong>tectable en el<br />
análisis PCR con cada par <strong>de</strong> iniciadores, se realizó la amplificación PCR<br />
específica utilizando un intervalo <strong>de</strong> concentraciones <strong>de</strong> ADN y las<br />
condiciones <strong>de</strong>scritas con anterioridad. Todos los pares <strong>de</strong> iniciadores<br />
utilizados <strong>de</strong>tectaron hasta 0,1 ng <strong>de</strong> ADN fúngico (Fig. 8). También se<br />
exploró la posibilidad <strong>de</strong> utilizar PCR ‘multiplex’, en la que los diferentes<br />
pares <strong>de</strong> iniciadores se combinan en la misma mezcla <strong>de</strong> reacción para<br />
realizar la amplificación conjunta <strong>de</strong> varias bandas marcadoras. Sin<br />
embargo, <strong>de</strong>bido a la similitud en cuanto a los pesos moleculares <strong>de</strong> las<br />
bandas amplificadas, este proceso no produjo una amplificación que<br />
distinguiese con facilidad a las distintas razas <strong>de</strong> F. oxysporum f. sp. ciceris,<br />
por lo que fue <strong>de</strong>sestimado.<br />
También se exploró la posibilidad <strong>de</strong> que la combinación <strong>de</strong> distintos<br />
iniciadores pudiese generar bandas marcadoras para otras razas. Así, la<br />
utilización conjunta <strong>de</strong> los iniciadores FocR6-P18fwd y FocR0-M15rev,<br />
que fueron diseñados para i<strong>de</strong>ntificar a los aislados <strong>de</strong> las razas 6 y 0<br />
respectivamente, amplificó una única banda <strong>de</strong> 1,4 kb en las razas 1A y 6,<br />
sin que se apreciase amplificación alguna en el resto <strong>de</strong> las razas <strong>de</strong> F.<br />
oxysporum f. sp. ciceris o en aislados <strong>de</strong> F. oxysporum no patogénicos <strong>de</strong><br />
garbanzo (Fig. 6D). Esta pareja <strong>de</strong> iniciadores permitió la i<strong>de</strong>ntificación<br />
indirecta <strong>de</strong> los aislados <strong>de</strong> la raza 1A <strong>de</strong>bido a la posibilidad <strong>de</strong><br />
discriminar a éstos <strong>de</strong> los aislados <strong>de</strong> la raza 6 mediante PCR específica<br />
utilizando los iniciadores FocR6-O2fwd y rev.<br />
La utilización ‘ad hoc’ <strong>de</strong> los iniciadores <strong>de</strong>sarrollados en el análisis<br />
PCR específica permitió analizar 76 aislados <strong>de</strong> F. oxysporum f. sp. ciceris<br />
representativos <strong>de</strong> cada una <strong>de</strong> las ocho razas patogénicas <strong>de</strong>scritas, y <strong>de</strong><br />
un amplio origen geográfico <strong>de</strong>l patógeno. Los resultados indicaron una<br />
correlación casi absoluta en la i<strong>de</strong>ntificación racial <strong>de</strong> los aislados mediante<br />
PCR específica, y la previa caracterización mediante pruebas biológicas.<br />
Los análisis PCR específica utilizando los iniciadores FocR0-M15fwd<br />
y rev i<strong>de</strong>ntificaron correctamente a todos los aislados <strong>de</strong> la raza 0<br />
proce<strong>de</strong>ntes <strong>de</strong> España, Israel, Líbano, Túnez, Turquía, y Siria. Sin<br />
76
2,64<br />
1,5<br />
1,0<br />
0,5<br />
Foc-7802<br />
Fo-90101<br />
200<br />
150 150<br />
100<br />
50<br />
25<br />
10<br />
5<br />
1<br />
10-1 10-1 M 10-2<br />
10-3 10-4 10-5 10-3 10-4 10-5 C<br />
Capítulo II<br />
Figura 8. Cantidad <strong>de</strong> ADN <strong>de</strong>l aislado Foc–Tonini (raza 6) <strong>de</strong>tectada mediante<br />
la amplificación PCR utilizando los iniciadores Foc0-12fwd y Foc0-12rev,<br />
específicos <strong>de</strong> la forma specialis ciceris. Los números a la izquierda correspon<strong>de</strong>n a<br />
los pesos moleculares <strong>de</strong>l ‘0,1 kb lad<strong>de</strong>r XIV’ (Roche Diagnostics) (Carril M).<br />
Carriles 1 y 2, controles <strong>de</strong> amplificación <strong>de</strong> ADN <strong>de</strong>l aislado <strong>de</strong> la raza 0 <strong>de</strong><br />
<strong>Fusarium</strong> oxysporum f. sp. ciceris Foc-7802, y <strong>de</strong>l aislados <strong>de</strong> F. oxysporum no<br />
patogénico <strong>de</strong> garbanzo Fo-90101. Carriles 3-15, ng <strong>de</strong> ADN en la reacción PCR<br />
específica. Carril C, control sin ADN mol<strong>de</strong>.<br />
77
Capítulo II<br />
embargo, dichos análisis amplificaron el mismo fragmento <strong>de</strong> ADN en los<br />
aislados <strong>de</strong> la raza 1B/C Foc–9602 proce<strong>de</strong>nte <strong>de</strong> Túnez, y en los aislados<br />
Foc–T96 1-1, –T96 1-2, y –T96 2-1 proce<strong>de</strong>ntes <strong>de</strong> Turquía; pero ni en el<br />
resto <strong>de</strong> aislados <strong>de</strong> la raza 1B/C. El aislado Foc–9602 fue caracterizado<br />
como perteneciente a la raza 1B/C mediante pruebas <strong>de</strong> patogenicidad y<br />
mediante análisis RAPD; sin embargo, <strong>de</strong> las cinco bandas RAPD<br />
marcadoras <strong>de</strong> R1B/C, sólo se i<strong>de</strong>ntificaron dos <strong>de</strong> ellas en los perfiles<br />
RAPD generados por los distintos iniciadores. A<strong>de</strong>más, las interacciones<br />
cultivar-aislado i<strong>de</strong>ntificadas en las pruebas <strong>de</strong> biológicas sugirieron<br />
características patogénicas intermedias entre la raza 0 y la raza 1B/C.<br />
Igualmente, los análisis PCR específica utilizando los iniciadores<br />
FocR6-O2fwd y rev i<strong>de</strong>ntificaron correctamente a todos los aislados <strong>de</strong> la<br />
raza 6, pero a<strong>de</strong>más, amplificaron el mismo fragmento en el ADN <strong>de</strong><br />
algunos aislados <strong>de</strong> la raza 1A, como Foc–9027 PV1, y –9166. Estos<br />
aislados, aunque originalmente i<strong>de</strong>ntificados como pertenecientes a la raza<br />
1A, mostraron características patogénicas <strong>de</strong> la raza 6 en pruebas <strong>de</strong><br />
patogenicidad duplicadas (Alcalá Jiménez, 1995).<br />
78
Discusión<br />
Capítulo II<br />
En investigaciones anteriores (Capítulo I) i<strong>de</strong>ntificamos fragmentos RAPD<br />
marcadores <strong>de</strong> F. oxysporum f. sp. ciceris y sus razas 0, 1B/C, 5, y 6; sin<br />
embargo, la influencia <strong>de</strong> diversos factores (i. e., fuente <strong>de</strong>l ADN <strong>de</strong><br />
análisis, procedimiento <strong>de</strong> extracción <strong>de</strong> éste, presencia <strong>de</strong> contaminantes<br />
en la mezcla <strong>de</strong> reacción, etc.) sobre la reproducibilidad <strong>de</strong>l análisis RAPD,<br />
pue<strong>de</strong> cuestionar o disminuir su aplicabilidad diagnóstica. Por ello, resulta<br />
necesario <strong>de</strong>sarrollar metodologías mejoradas que obvien tales influencias<br />
y dificulta<strong>de</strong>s. Una <strong>de</strong> tales mejoras consiste en reemplazar la naturaleza<br />
aleatoria <strong>de</strong>l marcador <strong>de</strong> interés, propia <strong>de</strong> la tecnología RAPD, por<br />
secuencias contenidas en el genoma <strong>de</strong>l organismo diana. El objetivo <strong>de</strong> la<br />
investigación realizada en este Capítulo ha sido <strong>de</strong>sarrollar un método<br />
basado en la PCR específica que permita la i<strong>de</strong>ntificación consistente,<br />
rápida, y precisa <strong>de</strong> F. oxysporum f. sp. ciceris y diferenciar sus razas<br />
patogénicas.<br />
La estrategia que hemos seguido en nuestro estudio ha sido diseñar<br />
iniciadores específicos a partir <strong>de</strong> secuencias SCAR (‘Sequence<br />
Characterized Amplified Region’) proce<strong>de</strong>ntes <strong>de</strong> marcadores RAPD<br />
asociados a las razas <strong>de</strong>l patógeno. De esta manera, se han diseñado<br />
iniciadores para PCR específica que permiten distinguir inequívocamente a<br />
aislados <strong>de</strong> F. oxysporum f. sp. ciceris <strong>de</strong>: (a) aislados <strong>de</strong> F. oxysporum no<br />
patogénicos <strong>de</strong> garbanzo; (b) aislados pertenecientes a otras formae speciales<br />
<strong>de</strong> F. oxysporum; y (c) otras especies <strong>de</strong> <strong>Fusarium</strong>. A<strong>de</strong>más, mediante análisis<br />
PCR específica utilizando iniciadores ‘ad hoc’ ha sido posible diferenciar<br />
entre si a aislados <strong>de</strong> las razas 0, 1A, 5, y 6 <strong>de</strong> F. oxysporum f. sp. ciceris, así<br />
como a éstos <strong>de</strong> aislados pertenecientes a otras razas <strong>de</strong>l patógeno.<br />
En investigaciones anteriores i<strong>de</strong>ntificamos marcadores RAPD<br />
asociados a las razas 2, 3, y 4 <strong>de</strong> F. oxysporum f. sp. ciceris proce<strong>de</strong>ntes <strong>de</strong> la<br />
India; sin embargo, puesto que no disponíamos <strong>de</strong> un número<br />
suficientemente a<strong>de</strong>cuado <strong>de</strong> aislados representativos <strong>de</strong> estas razas,<br />
consi<strong>de</strong>ramos improce<strong>de</strong>nte continuar con el proceso experimental para<br />
<strong>de</strong>sarrollar iniciadores para PCR específica que permitan la i<strong>de</strong>ntificación<br />
<strong>de</strong> dichas razas.<br />
79
Capítulo II<br />
La estrategia que hemos seguido para el <strong>de</strong>sarrollo <strong>de</strong> iniciadores ha<br />
resultado muy útil, lo cual ha ayudado al éxito <strong>de</strong> nuestra investigación.<br />
Los avances metodológicos que se están produciendo en la última década,<br />
respecto <strong>de</strong> las técnicas relacionadas con el manejo <strong>de</strong>l ADN<br />
recombinante, pue<strong>de</strong>n hacer parecer a los no familiarizados con dicha<br />
tecnología que la clonación <strong>de</strong> ADN es un método meramente rutinario.<br />
En esta investigación, se han extremado las precauciones para asegurar la<br />
selección eficiente <strong>de</strong> clones que contuvieran los insertos correctos,<br />
mediante la realización <strong>de</strong> numerosos experimentos <strong>de</strong> hibridación. Así, en<br />
el curso <strong>de</strong> las hibridaciones realizadas se localizaron fragmentos RAPD <strong>de</strong><br />
igual peso molecular, pero cuyo posterior análisis con enzimas <strong>de</strong><br />
restricción mostró que contenían distintas secuencias <strong>de</strong> ADN. De no<br />
haber practicado las precauciones suficientes, la utilización <strong>de</strong> tales<br />
fragmentos podría haber concluido en el diseño <strong>de</strong> iniciadores ina<strong>de</strong>cuados<br />
para nuestros fines.<br />
Generalmente, se asume ina<strong>de</strong>cuadamente que fragmentos <strong>de</strong> ADN <strong>de</strong><br />
igual peso molecular que resultan amplificados en análisis RAPD <strong>de</strong><br />
aislados <strong>de</strong> una misma población/especie fúngica presentan homología en<br />
sus secuencias. Sin embargo, esta asunción no tiene por qué ser<br />
necesariamente cierta, a menos que se <strong>de</strong>muestre mediante análisis <strong>de</strong><br />
hibridación (Rieseberg, 1996; Thormann et al., 1994). Las precauciones<br />
adoptadas en nuestro trabajo, antes referidos, son concluyentes en cuanto<br />
a mostrar que los marcadores RAPD amplificados en todos los aislados <strong>de</strong><br />
una misma raza presentan una gran homología en sus secuencias.<br />
Las hibridaciones ‘Southern’ <strong>de</strong>l ADN genómico digerido con EcoRI,<br />
han puesto <strong>de</strong> manifiesto la naturaleza repetitiva <strong>de</strong> las secuencias <strong>de</strong> ADN<br />
contenidas en los fragmentos RAPD asociados a las razas <strong>de</strong> F. oxysporum<br />
f. sp. ciceris. La utilidad <strong>de</strong> las secuencias repetitivas para la generación <strong>de</strong><br />
SCARs ya ha sido puesta <strong>de</strong> manifiesto por otros autores (Wiglesworth et<br />
al., 1994), para la <strong>de</strong>tección específica <strong>de</strong> Peronospora tabacina D. B. Adams<br />
mediante análisis PCR. Aún así, en nuestra investigación, la capacidad <strong>de</strong>l<br />
análisis PCR específica <strong>de</strong> discriminar entre diferentes razas <strong>de</strong> F.<br />
oxysporum f. sp. ciceris, i. e., las razas 0 y 6, utilizando iniciadores que<br />
i<strong>de</strong>ntifican específicamente a los aislados <strong>de</strong> dichas razas, ha sido<br />
80
Capítulo II<br />
comprobada mediante la realización <strong>de</strong> hibridaciones <strong>de</strong> dicha<br />
amplificación con el fragmento amplificado. Esto <strong>de</strong>mostró que aunque<br />
una secuencia pue<strong>de</strong> encontrarse presente en el genoma <strong>de</strong> todas las razas<br />
<strong>de</strong> F. oxysporum f. sp. ciceris, dicha secuencia sólo es amplificada cuando se<br />
utiliza como mol<strong>de</strong> el ADN <strong>de</strong> los aislados <strong>de</strong> una raza en concreto.<br />
En cambio, el fragmento <strong>de</strong> ADN amplificado en los aislados <strong>de</strong> raza 5<br />
fue <strong>de</strong>tectado mediante hibridación también en el resto <strong>de</strong> las razas<br />
estudiadas, si bien la intensidad <strong>de</strong> la señal <strong>de</strong> hibridación fue mucho<br />
menor. Puesto que la amplificación en el análisis PCR visualizada en geles<br />
<strong>de</strong> agarosa fue <strong>de</strong>tectada únicamente cuando se utilizó ADN <strong>de</strong> los<br />
aislados <strong>de</strong> la raza 5, consi<strong>de</strong>ramos que el método <strong>de</strong>sarrollado es<br />
suficientemente preciso para su utilización en activida<strong>de</strong>s diagnosticas con<br />
vistas a la i<strong>de</strong>ntificación racial.<br />
La <strong>de</strong>tección <strong>de</strong> secuencias comunes en las distintas razas <strong>de</strong> F.<br />
oxysporum f. sp. ciceris no es necesariamente un hecho extraño. Puesto que<br />
F. oxysporum es <strong>de</strong> reproducción estrictamente asexual, las poblaciones <strong>de</strong>l<br />
hongo en la naturaleza están constituidas por individuos clonales (Leslie,<br />
1993; Kistler, 1997) que pue<strong>de</strong>n acumular información genética similar.<br />
F. oxysporum carece <strong>de</strong> un estado sexual conocido y la ocurrencia <strong>de</strong><br />
recombinación parasexual en condiciones naturales no ha sido<br />
documentada y pue<strong>de</strong> ser poco probable. Por tanto, la mutación parece ser<br />
la principal fuente <strong>de</strong> variación que proporcione y mantenga la variabilidad<br />
genética en este organismo. Para explicar gran parte <strong>de</strong> la variabilidad<br />
genética observada en F. oxysporum se ha postulado la actividad <strong>de</strong><br />
transposones (Daboussi y Langin, 1994; Daboussi et al., 1992). F. oxysporum<br />
contiene varias familias <strong>de</strong> este tipo <strong>de</strong> elementos, entre los que se<br />
encuentran los transposones ‘Fot1-like’. La mayor parte <strong>de</strong> las secuencias<br />
estudiadas en nuestra investigación han presentado similarida<strong>de</strong>s<br />
significativas con elementos transponibles, entre ellos el clon FocB10 (1,0<br />
kb–R2–OPF-12) cuya secuencia posee un 80% <strong>de</strong> similaridad con el<br />
transposón Fot1 i<strong>de</strong>ntificado en F. oxysporum f. sp. melonis (Daboussi et al.,<br />
1992). Los transposones pue<strong>de</strong>n constituir hasta el 5% <strong>de</strong>l genoma <strong>de</strong> F.<br />
oxysporum, y por tanto pue<strong>de</strong>n tener un gran impacto en la estructura y<br />
función <strong>de</strong> algunos genes, así como en la organización cromosómica<br />
81
Capítulo II<br />
general <strong>de</strong> esta especie (Daboussi y Langin, 1994). Por tanto, parece lógico<br />
pensar que los polimorfismos asociados a marcadores RAPD hayan tenido<br />
como origen el movimiento pasado o actual <strong>de</strong> este tipo <strong>de</strong> elementos en<br />
el genoma fúngico.<br />
Todos los pares <strong>de</strong> iniciadores para PCR específica diseñados en esta<br />
investigación han <strong>de</strong>tectado hasta 0,1 ng <strong>de</strong> ADN fúngico, en las<br />
condiciones <strong>de</strong> amplificación aquí <strong>de</strong>scritas. Esta cantidad <strong>de</strong> ADN<br />
fúngico pue<strong>de</strong> ser extraído con facilidad <strong>de</strong> diversos substratos naturales<br />
que pudieran contener al patógeno. Ello, junto con la robustez <strong>de</strong>l análisis<br />
PCR específica anima a estudiar la posible aplicación <strong>de</strong> la metodología<br />
que hemos <strong>de</strong>sarrollado en la <strong>de</strong>tección <strong>de</strong> F. oxysporum f. sp. ciceris y <strong>de</strong> sus<br />
razas patogénicas 0, 1A, 5, y 6, en suelo y en planta. Trabajos anteriores<br />
permitieron <strong>de</strong>tectar al patotipo <strong>de</strong> Marchitez <strong>de</strong> F. oxysporum f. sp. ciceris<br />
en el suelo (García Pedrajas et al., 1999) y en plantas <strong>de</strong> garbanzo<br />
infectadas (Kelly et al., 1998). Disponer <strong>de</strong> un método que permita la<br />
<strong>de</strong>tección rápida y temprana <strong>de</strong>l patógeno en semillas <strong>de</strong> garbanzo, pue<strong>de</strong><br />
ser <strong>de</strong> utilidad para evitar la dispersión <strong>de</strong> F. oxysporum f. sp. ciceris y <strong>de</strong> sus<br />
razas patogénicas a zonas <strong>de</strong> cultivo libres <strong>de</strong> ella. Similarmente, la<br />
aplicación <strong>de</strong> dicha metodología diagnóstica al suelo antes <strong>de</strong> la siembra<br />
permitiría tomar <strong>de</strong>cisiones respecto <strong>de</strong> la a<strong>de</strong>cuación <strong>de</strong> cultivares <strong>de</strong><br />
garbanzo <strong>de</strong> interés agronómico y comercial, en razón <strong>de</strong> la prevalencia <strong>de</strong><br />
razas <strong>de</strong>l patógeno virulentas sobre ellos. Finalmente, la utilización <strong>de</strong> la<br />
metodología diagnóstica en ambos escenarios pue<strong>de</strong> ayudar a <strong>de</strong>terminar<br />
los cambios en la estructura racial que puedan tener lugar en las<br />
poblaciones <strong>de</strong> F. oxysporum f. sp. ciceris, como consecuencia <strong>de</strong>l uso <strong>de</strong><br />
cultivares resistentes o <strong>de</strong> modificaciones ambientales en el cultivo.<br />
82
CAPÍTULO III<br />
Capítulo III<br />
Genealogías génicas <strong>de</strong>finen a <strong>Fusarium</strong><br />
oxysporum f. sp. ciceris como un grupo<br />
monofilético<br />
Introducción<br />
En los hongos que carecen <strong>de</strong> estado reproductivo sexual, y que por tanto<br />
se reproducen obligada y estrictamente por mecanismos asexuales, no se<br />
produce una recombinación regular y la variación genética que se pueda<br />
generar en ellos resulta principalmente <strong>de</strong> la acumulación <strong>de</strong> mutaciones.<br />
En consecuencia, en este tipo <strong>de</strong> hongos todo el genoma se encuentra<br />
ligado, es transmitido como una sola unidad <strong>de</strong> una generación a la<br />
siguiente, y las diferentes regiones <strong>de</strong>l genoma <strong>de</strong>ben poseer la misma<br />
historia evolutiva (Taylor et al., 1999b). Por lo tanto, la evolución <strong>de</strong><br />
caracteres fenotípicos en los hongos fitopatógenos <strong>de</strong> reproducción<br />
asexual, tales como la especificidad <strong>de</strong> huésped o cultivar, o la relación<br />
entre razas patogénicas, pue<strong>de</strong> ser estudiada mediante el análisis <strong>de</strong><br />
genealogías <strong>de</strong> genes que no tienen que mantener necesariamente una<br />
relación funcional directa con el fenotipo <strong>de</strong> interés, i. e., neutrales (ej.,<br />
O’Donnell et al., 1998; Steenkamp et al., 2000; Taylor et al., 1999a).<br />
<strong>Fusarium</strong> oxysporum Schlechtend: Fr. es una especie fúngica <strong>de</strong><br />
reproducción estrictamente asexual ubicua en los ambientes edáficos en el<br />
mundo. Los aislados <strong>de</strong> F. oxysporum pue<strong>de</strong>n causar Marchitez vascular o<br />
83
Capítulo III<br />
Podredumbre radical en numerosos cultivos agrícolas, y han sido<br />
clasificados en formae speciales en base a la patogenicidad huésped- específica<br />
que manifiestan (Nelson et al., 1981). Generalmente, los aislados <strong>de</strong> F.<br />
oxysporum pertenecientes a una forma specialis son genéticamente más<br />
similares entre sí que a los aislados <strong>de</strong> la especie patogénicamente<br />
especializados sobre otros huéspe<strong>de</strong>s, y por lo tanto, se suele asumir que<br />
los aislados <strong>de</strong>ntro <strong>de</strong> una forma specialis son evolutivamente <strong>de</strong> origen<br />
monofilético (Kistler, 1997; Tantaoui et al., 1996).<br />
A<strong>de</strong>más, en F. oxysporum la clonalidad ha sido asociada a la<br />
compatibilidad vegetativa (Gordon y Martyn, 1997; Kistler, 1997), ya que<br />
aislados <strong>de</strong>l hongo que pertenecen a un mismo grupo <strong>de</strong> compatibilidad<br />
vegetativa (VCG) muestran una alta similaridad genética <strong>de</strong>terminada<br />
mediante haplotipos <strong>de</strong> ADN mitocondrial (ADNmt) o <strong>de</strong> IGS<br />
(‘Intergenic Spacers’) (Gordon y Martyn, 1997).<br />
Sin embargo, algunos estudios basados en genealogías génicas han<br />
<strong>de</strong>mostrado que ciertas formae speciales <strong>de</strong> F. oxysporum pue<strong>de</strong>n tener<br />
orígenes múltiples e in<strong>de</strong>pendientes (i. e., son <strong>de</strong> origen polifilético),<br />
indicando que patogenicidad y virulencia han evolucionado más <strong>de</strong> una<br />
vez en el curso <strong>de</strong> su historia adaptativa (Baayen et al., 2000; O’Donnell et<br />
al., 1998).<br />
De forma similar, también se han encontrado excepciones notables en<br />
la correlación entre VCG y los haplotipos <strong>de</strong> ADNmt y <strong>de</strong> IGS. Así, por<br />
ejemplo, un aislado <strong>de</strong> F. oxysporum f. sp. melonis (Leach & Currence) W. C.<br />
Sny<strong>de</strong>r & H. N. Hansen perteneciente al VCG 0131 compartió los<br />
haplotipos <strong>de</strong> ADNmt e IGS <strong>de</strong> aislados patogénicos <strong>de</strong>l VCG 0134, en<br />
lugar <strong>de</strong> compartirlos con aislados representativos <strong>de</strong>l VCG 0131 (Appel y<br />
Gordon, 1995). A<strong>de</strong>más, aislados <strong>de</strong> F. oxysporum no patogénicos <strong>de</strong><br />
melón, pero vegetativamente compatibles con aislados <strong>de</strong> F. oxysporum f.<br />
sp. melonis <strong>de</strong> los VCGs 0131 y 0134, presentaron secuencias <strong>de</strong><br />
nucleótidos <strong>de</strong> la región IGS <strong>de</strong>l ADN ribosómico que fueron diferentes a<br />
las <strong>de</strong> los aislados patogénicos (Appel y Gordon, 1996). Este último<br />
ejemplo <strong>de</strong>muestra que, en algunos casos, la compatibilidad vegetativa<br />
pue<strong>de</strong> ser una coinci<strong>de</strong>ncia que haya resultado como consecuencia <strong>de</strong> la<br />
convergencia <strong>de</strong> caracteres en lugar <strong>de</strong> poseer un antepasado común.<br />
84
Capítulo III<br />
<strong>Fusarium</strong> oxysporum f. sp. ciceris (Padwick) Matuo & K. Sato, el agente<br />
causante <strong>de</strong> la Fusariosis Vascular <strong>de</strong>l garbanzo (Cicer arietinum L.), es<br />
patogénico <strong>de</strong> especies <strong>de</strong> Cicer (Kaiser et al., 1994), y ha sido <strong>de</strong>scrita en<br />
todas las zonas geográficas <strong>de</strong>l mundo cultivadoras <strong>de</strong> esta leguminosa<br />
(Jalali y Chand, 1992). La variación en los tipos <strong>de</strong> síndromes que el<br />
patógeno induce en la planta (patotipos) y en razas patogénicas según la<br />
reacción <strong>de</strong> cultivares <strong>de</strong> garbanzo a la infección, ha sido correlacionada<br />
con las distintas regiones geográficas don<strong>de</strong> existe el patógeno, y con<br />
polimorfismos en marcadores moleculares.<br />
En F. oxysporum f. sp. ciceris se han <strong>de</strong>scrito dos patotipos, <strong>de</strong>signados<br />
Amarillez y Marchitez, que se i<strong>de</strong>ntifican mediante pruebas <strong>de</strong><br />
patogenicidad en cultivares susceptibles <strong>de</strong> garbanzo (Trapero Casas y<br />
Jiménez Díaz, 1985). Estos dos patotipos también pue<strong>de</strong>n ser<br />
diferenciados mediante marcadores RAPD (‘Random Amplified<br />
Polymorphic DNA’), y se distribuyen en dos grupos diferentes mediante el<br />
análisis <strong>de</strong> ‘cluster’ <strong>de</strong> los productos <strong>de</strong> amplificación <strong>de</strong> su ADN con<br />
iniciadores arbitrarios (Kelly et al., 1994; Jiménez Gasco et al., 2001;<br />
Capítulo I); lo que sugiere que podrían no encontrarse muy relacionados<br />
genéticamente.<br />
A<strong>de</strong>más <strong>de</strong> la variación en patotipos, en F. oxysporum f. sp. ciceris existen<br />
ocho razas patogénicas (razas 0, 1A, 1B/C, 2, 3, 4, 5, y 6) que son<br />
i<strong>de</strong>ntificadas mediante la reacción diferencial <strong>de</strong> una serie <strong>de</strong> líneas o<br />
cultivares <strong>de</strong> garbanzo (Haware y Nene, 1982b; Jiménez Díaz et al., 1989b;<br />
1993a). Al igual que ocurre con los patotipos, algunas <strong>de</strong> estas razas<br />
pue<strong>de</strong>n ser diferenciadas mediante marcadores RAPD (Jiménez Gasco et<br />
al., 2001; Capítulo I).<br />
En cultivares <strong>de</strong> garbanzo susceptibles, las razas 0 y 1B/C inducen el<br />
síndrome <strong>de</strong> Amarillez, mientras que las razas 1A, 2, 3, 4, 5 y 6 inducen el<br />
síndrome <strong>de</strong> Marchitez (Jiménez Díaz et al., 1989b; 1993a; Trapero Casas y<br />
Jiménez Díaz, 1985). A<strong>de</strong>más, estas razas también presentan distintas<br />
distribuciones geográficas: las razas 2, 3, y 4 han sido <strong>de</strong>scritas únicamente<br />
en la India (Haware y Nene, 1982b), mientras que las razas 0, 1B/C, 5, y 6<br />
se encuentran principalmente en la región Mediterránea y en EEUU<br />
(California) (Halila y Strange, 1996; Jiménez Díaz et al., 1989b; 1993a). La<br />
85
Capítulo III<br />
raza 1A ha sido i<strong>de</strong>ntificada en la India (Haware y Nene, 1982b), California<br />
y la región Mediterránea (Jiménez Díaz et al., 1993a). Sin embargo, a pesar<br />
<strong>de</strong> existir una variación notable en relación a sintomatología, razas<br />
patogénicas, y distribución geográfica, todos los aislados <strong>de</strong> F. oxysporum f.<br />
sp. ciceris estudiados hasta el momento pertenecen a un mismo VCG<br />
(Nogales Monada, 1997).<br />
La diversidad fenotípica y geográfica que hemos observado en F.<br />
oxysporum f. sp. ciceris plantea la posibilidad <strong>de</strong> que este taxón sea <strong>de</strong> origen<br />
polifilético, al igual que se ha <strong>de</strong>mostrado en F. oxysporum f. sp. cubense (E.<br />
F. Smith) W. C. Sny<strong>de</strong>r & H. N. Hans. y F. oxysporum f. sp. melonis. Por<br />
tanto, el objetivo <strong>de</strong> las investigaciones contenidas en este Capítulo fue<br />
<strong>de</strong>mostrar la hipótesis nula <strong>de</strong> un origen monofilético para F. oxysporum f.<br />
sp. ciceris. Para alcanzar dicho objetivo, hemos estudiado la extensión y<br />
naturaleza <strong>de</strong> la variación existente en las secuencias <strong>de</strong> intrones <strong>de</strong> varios<br />
genes conservados. Estas secuencias fueron comparadas a las <strong>de</strong> otras<br />
formae speciales <strong>de</strong> F. oxysporum con el objeto <strong>de</strong> comprobar si la forma specialis<br />
ciceris es monofilética <strong>de</strong>ntro <strong>de</strong> este complejo <strong>de</strong> especies. Un objetivo<br />
adicional en nuestro estudio fue comparar la variación en dichas<br />
secuencias en relación con la variación <strong>de</strong> patotipos, razas patogénicas y<br />
distribución geográfica existente en el patógeno.<br />
Materiales y Métodos<br />
Aislados fúngicos<br />
En este estudio se han utilizado un total <strong>de</strong> 17 aislados <strong>de</strong> F. oxysporum f.<br />
sp. ciceris, representativos <strong>de</strong> las ocho razas patogénicas <strong>de</strong>scritas, y <strong>de</strong> un<br />
amplio rango geográfico <strong>de</strong>l patógeno (Tabla 9). A<strong>de</strong>más, se han incluido<br />
tres aislados <strong>de</strong> F. oxysporum no patogénicos <strong>de</strong> garbanzo, obtenidos <strong>de</strong><br />
raíces sanas <strong>de</strong> garbanzo (Tabla 9). Todos los aislados se habían<br />
caracterizados en cuanto a patotipo y a raza patogénica en trabajos<br />
anteriores (Jiménez Díaz et al., 1993a; Jiménez Gasco et al., 2001; Kelly et<br />
86
Capítulo III<br />
Tabla 9. Información sobre la raza y origen geográfico <strong>de</strong> los aislados <strong>de</strong><br />
<strong>Fusarium</strong> oxysporum f. sp. ciceris (Foc) y <strong>Fusarium</strong> oxysporum (Fo) no<br />
patogénicos <strong>de</strong> garbanzo utilizados en el análisis <strong>de</strong> genealogías génicas<br />
Aislado a Origen geográfico b Raza patogénica c<br />
GRUPO A<br />
Foc-7802 España 0<br />
Foc-1987-W17 EEUU 1B/C<br />
Foc-7989 India 1A<br />
Foc-1992R4N India 4<br />
Foc-9035 España 5<br />
Foc-Tonini EEUU 6<br />
GRUPO B<br />
Foc-9601 Túnez 0<br />
Foc-9602 Túnez 1B/C<br />
Foc-USA 3-1 JG62 EEUU 1B/C<br />
Foc-9168 Marruecos 1A<br />
Foc-8605 India 2<br />
Foc-1992R2N India 2<br />
Foc-8606 India 3<br />
Foc-1992R3N India 3<br />
Foc-8607 India 4<br />
Foc-9094 JG62 España 5<br />
Foc-9093PV1 España 6<br />
Fo-90101 España NP<br />
Fo-90105 España NP<br />
Fo-9169 Marruecos NP<br />
a Los genes analizados en los aislados <strong>de</strong>l Grupo A fueron factor <strong>de</strong> traducción y<br />
elongación 1α (EF1α), β-tubulina, histona 3, actina, y calmodulina. El gen EF1α<br />
fue el único analizado en los aislados <strong>de</strong>l Grupo B.<br />
b Los aislados <strong>de</strong> EEUU (California), España, y Marruecos proce<strong>de</strong>n <strong>de</strong> la micoteca<br />
<strong>de</strong>l Departamento <strong>de</strong> Protección <strong>de</strong> Cultivos, Instituto <strong>de</strong> Agricultura Sostenible,<br />
CSIC, Córdoba, España. Los aislados <strong>de</strong> la India fueron proporcionados por el Dr.<br />
M. P. Haware, ICRISAT, Hy<strong>de</strong>rabad, India. Los aislados proce<strong>de</strong>ntes <strong>de</strong> Túnez<br />
fueron cedidos por el Dr. M. H. Halila, Institute Nationale <strong>de</strong> la Recherche<br />
Agronomique, Ariana, Túnez.<br />
c Raza <strong>de</strong>terminada mediante pruebas <strong>de</strong> patogenicidad sobre líneas <strong>de</strong> garbanzo<br />
diferenciadoras (Alcalá Jiménez, 1995; Jiménez Gasco et al., 2001; Capítulo I). NP =<br />
no patogénicos <strong>de</strong> garbanzo.<br />
87
Capítulo III<br />
al., 1994; Capítulo I). En el grupo <strong>de</strong> 17 aislados se incluyeron al menos<br />
dos aislados representativos <strong>de</strong> cada raza, que fueron seleccionados para<br />
maximizar la variación geográfica entre aislados <strong>de</strong> una misma raza,<br />
siempre que fue posible (algunas <strong>de</strong> las razas presentan una distribución<br />
geográfica limitada). Todos los aislados se conservaron como cultivos<br />
monoconídicos, en suelo estéril a 4ºC, en oscuridad, y pertenecen a la<br />
colección <strong>de</strong> cultivos <strong>de</strong>l Departamento <strong>de</strong> Protección <strong>de</strong> Cultivos,<br />
Instituto <strong>de</strong> Agricultura Sostenible, CSIC, Córdoba. Los procedimientos<br />
para la obtención <strong>de</strong> cultivos activos <strong>de</strong> los aislados, el manejo <strong>de</strong> éstos, y<br />
la obtención <strong>de</strong> su micelio para extracciones <strong>de</strong> ADN, han sido <strong>de</strong>scritos<br />
con anterioridad (Pág. 22, 26, Capítulo I). El ADN genómico fue<br />
purificado a partir <strong>de</strong> micelio liofilizado y molido, mediante el método <strong>de</strong><br />
Rae<strong>de</strong>r y Broda (1985) (Pág. 22-28, Capítulo I).<br />
Amplificación <strong>de</strong> ADN y secuenciación<br />
Para este estudio se seleccionaron genes que contienen secuencias<br />
conservadas entre ascomicetos, y que presentan al menos un intrón. Los<br />
genes seleccionados fueron: factor <strong>de</strong> traducción y elongación 1α (EF1α),<br />
β-tubulina (β-tub), histona 3 (H3), actina (Act), y calmodulina (Cal).<br />
Los iniciadores y las condiciones <strong>de</strong> reacción para la amplificación PCR<br />
(‘Polymerase Chain Reaction’) <strong>de</strong> estos genes fueron <strong>de</strong>scritos<br />
anteriormente (Carbone y Kohn, 1999; Glass y Donaldson, 1995;<br />
O’Donnell et al., 1998) (Tabla 10). Los productos <strong>de</strong> PCR fueron<br />
purificados <strong>de</strong>l gel mediante los ‘kits’ ‘QIAquick PCR purification’ and<br />
‘QIAquick gel extraction’ (Qiagen Inc., Chatsworth, CA, EEUU).<br />
Todos los iniciadores fueron sintetizados por el Servicio <strong>de</strong> Síntesis <strong>de</strong><br />
Oligonucleótidos <strong>de</strong> la Universidad <strong>de</strong> Cornell, Ithaca, NY, EEUU. La<br />
secuenciación <strong>de</strong> los productos <strong>de</strong> PCR fue realizada por Servicio <strong>de</strong><br />
Secuenciación <strong>de</strong> ADN <strong>de</strong> la Universidad <strong>de</strong> Cornell.<br />
88
Capítulo III<br />
Tabla 10. Secuencia <strong>de</strong> los iniciadores que amplifican intrones <strong>de</strong> los<br />
genes altamente conservados: factor <strong>de</strong> traducción y elongación 1α<br />
(EF1α), β-tubulina, histona 3, actina, y calmodulina, utilizados en el<br />
análisis <strong>de</strong> genealogías génicas en <strong>Fusarium</strong> oxysporum f. sp. ciceris<br />
Gen amplificado Iniciadors Secuencia <strong>de</strong>l iniciador (5’→3’)<br />
EF1α a EF1 ATGGGTAAGGA(A/G)GACAAGAC<br />
EF2 GGA(G/A)GTACCAGT(G/C)ATCATGTT<br />
β-Tubulina b Bt1a TTCCCCCGTCTCCACTTCTTCATG<br />
Bt1b GACGAGATCGTTCATGTTGAACTC<br />
Histona 3 b H3-1a ACTAAGCAGACCGCCCGCAGG<br />
H3-1b GCGGGCGAGCTGGATGTCCTT<br />
Actina c ACT-512F ATGTGCAAGGCCGGTTTCGC<br />
ACT-783R TACGAGTCCTTCTGGCCCAT<br />
Calmodulina c CAL-228F GAGTTCAAGGAGGCCTTCTCCC<br />
CAL-737R CATCTTTCTGGCCATCATGG<br />
Iniciadores <strong>de</strong>scritos en: a O’Donnell et al., 1998; b Glass & Donaldson, 1995; y c Carbone<br />
& Kohn, 1999.<br />
Análisis filogenético<br />
Las secuencias génicas fueron editadas y alineadas con secuencias<br />
homólogas <strong>de</strong> aislados <strong>de</strong> otras formae speciales <strong>de</strong> F. oxysporum obtenidas <strong>de</strong><br />
GenBank, mediante Sequencher 3.1.1 (Gen Co<strong>de</strong>s, Ann Arbor, MI,<br />
EEUU). El análisis filogenético se realizó mediante PAUP* 4.0b4a<br />
(‘Phylogenetic Analysis Using Parsimony and Other Methods’) (Swofford,<br />
2000). Las inserciones se consi<strong>de</strong>raron como caracteres únicos. El análisis<br />
<strong>de</strong> parsimonia se realizó mediante la opción <strong>de</strong> búsqueda Heurística, con la<br />
adición aleatoria <strong>de</strong> 1.000 secuencias <strong>de</strong>l algoritmo ‘tree-bisection-<br />
89
Capítulo III<br />
reconnection branch swapping’, y la opción MULPARS. El análisis <strong>de</strong><br />
‘bootstrap’ se realizó con 1.000 repeticiones.<br />
Resultados<br />
Inicialmente en este estudio se secuenciaron y analizaron los genes EF1α,<br />
β-tub, H3, Act, y Cal. Estos genes se amplificaron a partir <strong>de</strong>l ADN<br />
genómico <strong>de</strong> un aislado <strong>de</strong> cada una <strong>de</strong> las razas 0, 1A, 1B/C, 4, 5, y 6 <strong>de</strong><br />
F. oxysporum f. sp. ciceris representativos <strong>de</strong> la mayor diversidad en patotipo,<br />
raza, y origen geográfico <strong>de</strong>l patógeno (Tabla 9, Grupo A), con objeto <strong>de</strong><br />
localizar polimorfismos en secuencias asociadas a dicho patotipos, razas<br />
patogénicas y distribución geográfica. Los resultados indicaron que las<br />
secuencias <strong>de</strong> los cinco genes son idénticas en los seis aislados <strong>de</strong> F.<br />
oxysporum f. sp. ciceris analizados (números <strong>de</strong> acceso <strong>de</strong> GenBank:<br />
AF346503, AF346505 a AF346508).<br />
Diversos autores (ej., Baayen et al., 2000; O’Donnell et al., 1998) han<br />
estudiado extensamente el gen EF1α en F. oxysporum para analizar la<br />
filogenia <strong>de</strong> varias <strong>de</strong> sus formae speciales. Por ello, en este trabajo se<br />
obtuvieron las secuencias EF1α <strong>de</strong> 11 aislados adicionales <strong>de</strong> F. oxysporum<br />
f. sp. ciceris y <strong>de</strong> tres aislados <strong>de</strong> F. oxysporum no patogénicos <strong>de</strong> garbanzo<br />
(Tabla 10, Grupo B). Todas las secuencias <strong>de</strong> EF1α <strong>de</strong> los aislados <strong>de</strong> F.<br />
oxysporum f. sp. ciceris fueron idénticas entre sí, in<strong>de</strong>pendientemente <strong>de</strong> la<br />
naturaleza racial o distribución geográfica <strong>de</strong> aquéllos. De forma similar,<br />
las secuencias EF1α <strong>de</strong> los tres aislados <strong>de</strong> F. oxysporum no patogénicos <strong>de</strong><br />
garbanzo (número <strong>de</strong> acceso <strong>de</strong> GenBank AF346504) resultaron idénticas<br />
entre sí, y diferentes <strong>de</strong> las <strong>de</strong> los aislados F. oxysporum f. sp. ciceris. Las<br />
diferencias entre las secuencias nucleotídicas fueron localizadas<br />
únicamente <strong>de</strong>ntro <strong>de</strong> los tres intrones existentes en el gen EF1α, tal<br />
90
Capítulo III<br />
como fue <strong>de</strong>scrito por O’Donnell et al. (1998) para otras formae speciales <strong>de</strong><br />
F. oxysporum.<br />
Para <strong>de</strong>mostrar si la genealogía génica inferida a partir <strong>de</strong> las secuencias<br />
<strong>de</strong> EF1α apoya la hipótesis <strong>de</strong> un origen monofilético para F. oxysporum f.<br />
sp. ciceris, las secuencias <strong>de</strong>l gen EF1α <strong>de</strong> 17 aislados <strong>de</strong>l patógeno y <strong>de</strong> tres<br />
aislados <strong>de</strong> F. oxysporum no patogénicos <strong>de</strong> garbanzo fueron comparadas<br />
con 24 secuencias EF1α obtenidas <strong>de</strong> GenBank. Estas 24 secuencias<br />
pertenecen a aislados <strong>de</strong> las siguientes 11 formae speciales <strong>de</strong> F. oxysporum:<br />
asparagi Cohen, canariensis Mercier & Louvet, cubense, dianthi (Prill. &<br />
Delacr.) W. C. Sny<strong>de</strong>r & H. N. Hans., gladioli (Massey) W. C. Sny<strong>de</strong>r & H.<br />
N. Hans., lini (Bolley) W. C. Sny<strong>de</strong>r & H. N. Hans., lycopersici (Sacc.) W. C.<br />
Sny<strong>de</strong>r & H. N. Hans., melonis, radicis-lycopersici W. R. Jarvis & R. A.<br />
Shoemaker, spinaciae (Sherb.) W. C. Sny<strong>de</strong>r & H. N. Hans., y tulipae Apt.,<br />
así como a aislados <strong>de</strong> F. oxysporum referidos como no patogénicos en la<br />
literatura fitopatológica. Las secuencias fueron seleccionadas como<br />
representativas <strong>de</strong> las agrupaciones principales <strong>de</strong> F. oxysporum (Baayen et<br />
al., 2000; O’Donnell et al., 1998).<br />
El análisis <strong>de</strong> parsimonia se realizó a partir <strong>de</strong> 701 caracteres, <strong>de</strong> los<br />
cuales 30 fueron filogenéticamente informativos. Se obtuvieron tres<br />
árboles filogenéticos <strong>de</strong> igual máxima parsimonia, que fueron comparados<br />
con la especie ‘fuera <strong>de</strong> grupo’ (especie ‘outgroup’) <strong>Fusarium</strong> spp. NRRL<br />
22903 (O’Donnell et al., 1998).<br />
Los aislados <strong>de</strong> F. oxysporum f. sp. ciceris formaron un grupo distintivo al<br />
ser comparados con aislados <strong>de</strong> otras formae speciales (Fig. 9). Esta<br />
agrupación, con un 62% <strong>de</strong> ‘bootstrap’, se resolvió <strong>de</strong>ntro <strong>de</strong>l grupo 1 sensu<br />
O’Donnell et al. (1998). Los tres aislados <strong>de</strong> F. oxysporum no patogénicos <strong>de</strong><br />
garbanzo, Fo–90101, –90105, y –9169, presentaron secuencias <strong>de</strong>l gen<br />
EF1α idénticas, in<strong>de</strong>pendientemente <strong>de</strong> su origen geográfico (Fo–9169,<br />
Marruecos; y Fo–90101 y –90105, España); a<strong>de</strong>más, estas secuencias<br />
fueron iguales a la <strong>de</strong> otro aislado <strong>de</strong> F. oxysporum (NRRL 25356) referido<br />
como no patogénico en la literatura fitopatológica, y a la <strong>de</strong> un aislado <strong>de</strong><br />
la forma specialis melonis (NRRL 26178). Las secuencias <strong>de</strong> estos aislados se<br />
91
Capítulo III<br />
resolvieron <strong>de</strong>ntro <strong>de</strong>l grupo 2 (O’Donnell et al., 1998), junto con la forma<br />
specialis lini y una secuencia <strong>de</strong> la forma specialis cubense (NRRL 25609).<br />
Discusión<br />
El principal objetivo <strong>de</strong> este trabajo fue <strong>de</strong>terminar si F. oxysporum f. sp.<br />
ciceris presenta un origen evolutivo monofilético. A pesar <strong>de</strong> la diversidad<br />
que presentan los aislados <strong>de</strong> este patógeno en cuanto a distribución<br />
geográfica, tipos <strong>de</strong> sintomatología que inducen en la planta susceptible, y<br />
la patogenicidad cultivar-específica, los aislados que hemos estudiado,<br />
representativos <strong>de</strong> seis <strong>de</strong> las ocho razas patogénicas <strong>de</strong> agente <strong>de</strong>scritas,<br />
poseen secuencias idénticas en los intrones <strong>de</strong> los cinco genes analizados<br />
(EF1α, β-tub, H3, Act, y Cal).<br />
De forma similar, los 11 aislados <strong>de</strong> F. oxysporum f. sp. ciceris adicionales<br />
estudiados no mostraron ningún polimorfismo en la secuencia <strong>de</strong> <strong>de</strong>l gen<br />
EF1α.<br />
Cuando se compararon las secuencias <strong>de</strong> EF1α <strong>de</strong> los 17 aislados <strong>de</strong> F.<br />
oxysporum f. sp. ciceris anteriores con las <strong>de</strong> tres aislados <strong>de</strong> F. oxysporum no<br />
patogénicos <strong>de</strong> garbanzo, y con las <strong>de</strong> 11 otras formae speciales <strong>de</strong> F.<br />
oxysporum, los aislados patogénicos <strong>de</strong> garbanzo formaron un grupo<br />
distintivo <strong>de</strong>l resto <strong>de</strong> los aislados (Fig. 9) <strong>de</strong>mostrando así su origen<br />
monofilético.<br />
Trabajos anteriores en nuestro laboratorio mostraron que los aislados<br />
<strong>de</strong> las diferentes razas <strong>de</strong> F. oxysporum f. sp. ciceris presentan el mismo<br />
patrón RFLP (‘Restriction Fragment Length Polymorphism’) <strong>de</strong>l ADNmt<br />
(Pérez-Artés et al., 1995), pertenecen a un único VCG (Nogales Moncada,<br />
92
86 F.o.lycopersici 26200<br />
F.o.melonis 26046<br />
57 F.o.lycopersici 26034<br />
F.o.tulipae 28974<br />
61 F.o.lycopersici 26383<br />
GRUPO 3<br />
58<br />
F.o.lycopersici 26203<br />
83 F.o.asparagi 28404<br />
65 F.o.radicis-lycopersici 26379<br />
F.o. cubense 26022<br />
F.o.spinaciae 26876<br />
F.oxysporum 25356<br />
74 58 F.o.melonis 26178<br />
F.oxysporum 90101 *<br />
GRUPO 2<br />
88 F.oxysporum 90105 *<br />
F.oxysporum 9169 *<br />
F.o.cubense 25609<br />
F.o.lini 29094<br />
57<br />
F.o.dianthi 28401<br />
76 F.o.gladioli 28918<br />
F.oxysporum 28366<br />
F.o.cubense 25607<br />
F.o.ciceris 1992R2N *<br />
F.o.ciceris 7802 *<br />
F.o.ciceris 7989 *<br />
F.o.ciceris 9035 *<br />
F.o.ciceris 1992R3N *<br />
F.o.ciceris 1992R4N *<br />
F.o.ciceris Tonini *<br />
62 F.o.ciceris USA-W17 *<br />
F.o.ciceris 9601 *<br />
F.o.ciceris 9602 *<br />
F.o.ciceris USA3-1JG62 *<br />
F.o.ciceris 8605 *<br />
GRUPO 1<br />
F.o.ciceris 8606 *<br />
F.o.ciceris 8607 *<br />
F.o.ciceris 9094 JG62 *<br />
F.o.ciceris 9093 PV1 *<br />
F.o.ciceris 9168 * 1 cambio<br />
100 F.oxysporum 25357<br />
F.o.cubense 25605<br />
65 F.o.canariensis 26035<br />
F.o.cubense 26029<br />
F.oxysporum 28371<br />
<strong>Fusarium</strong> sp. 22903<br />
Capítulo III<br />
Figura 9. Uno <strong>de</strong> tres árboles filogenéticos <strong>de</strong> igual máxima parsimonia inferidos<br />
a partir <strong>de</strong> la genealogía génica <strong>de</strong>l factor <strong>de</strong> traducción y elongación 1α (EF1α),<br />
que <strong>de</strong>muestra la monofilia en <strong>Fusarium</strong> oxysporum f. sp. ciceris. El asterisco indica<br />
aislados <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris y <strong>Fusarium</strong> oxysporum, en comparación<br />
con las secuencias <strong>de</strong> <strong>Fusarium</strong> oxysporum obtenidas <strong>de</strong> GenBank. Los números en<br />
las líneas representan valores <strong>de</strong> ‘bootstrap’ >50% basados en 1.000 repeticiones.<br />
93
Capítulo III<br />
1997), y poseen el mismo elemento <strong>de</strong> ADN repetitivo en sus genomas<br />
(Kelly, 1996). A<strong>de</strong>más, dichos aislados presentan una alta similaridad<br />
genética, indicada por el análisis <strong>de</strong> ‘cluster’ <strong>de</strong> datos RAPD (Jiménez<br />
Gasco et al., 2001; Kelly et al., 1994; Capítulo I). Aunque toda esta<br />
información sugiere un origen evolutivo monofilético para F. oxysporum f.<br />
sp. ciceris, la evi<strong>de</strong>ncia directa para su <strong>de</strong>mostración sólo pue<strong>de</strong> obtenerse<br />
mediante el análisis <strong>de</strong> genealogías génicas y su comparación con taxones<br />
relacionados.<br />
Las secuencias <strong>de</strong>l gen EF1α <strong>de</strong> tres aislados <strong>de</strong> F. oxysporum obtenidos<br />
<strong>de</strong> raíces <strong>de</strong> plantas sanas <strong>de</strong> garbanzo, pero no patogénicos <strong>de</strong> éste,<br />
fueron idénticas a la <strong>de</strong> un aislado <strong>de</strong> F. oxysporum f. sp. melonis. En cambio,<br />
otro aislado <strong>de</strong> F. oxysporum f. sp. melonis presentó una secuencia <strong>de</strong> EF1α<br />
igual a la <strong>de</strong> un aislado <strong>de</strong> F. oxysporum f. sp. lycopersici. El hecho <strong>de</strong> que los<br />
tres aislados <strong>de</strong> F. oxysporum estudiados no sean patogénicos <strong>de</strong> garbanzo<br />
pero puedan serlo <strong>de</strong> otras plantas, como melón, no ha sido abordado en<br />
nuestra investigación, por lo que esta posibilidad no pue<strong>de</strong> ser <strong>de</strong>scartada.<br />
La secuencia <strong>de</strong>l gen EF1α ha <strong>de</strong>mostrado ser un marcador<br />
filogenético excelente para estudiar la relación entre aislados <strong>de</strong> F.<br />
oxysporum (Baayen et al., 2000; O’Donnell et al., 1998). A<strong>de</strong>más, nuestro<br />
análisis filogenético <strong>de</strong> las secuencias EF1α <strong>de</strong> aislados patogénicos <strong>de</strong><br />
garbanzo junto con las otras obtenidas <strong>de</strong> GenBank, confirma la<br />
naturaleza polifilética <strong>de</strong> F. oxysporum f. sp. cubense y F. oxysporum f. sp.<br />
melonis (Fig. 9) (O’Donnell et al., 1998). Sin embargo, los aislados <strong>de</strong> las 11<br />
formae speciales a partir <strong>de</strong> los cuales se obtuvieron las secuencias <strong>de</strong> ADN<br />
<strong>de</strong> GenBank no han sido analizados en este trabajo, por lo que no<br />
po<strong>de</strong>mos <strong>de</strong>scartar la posibilidad <strong>de</strong> que estos aislados hayan sido<br />
caracterizados <strong>de</strong> forma errónea en cuanto a forma specialis.<br />
La interpretación más simple sobre el origen monofilético <strong>de</strong> F.<br />
oxysporum f. sp. ciceris, y <strong>de</strong> la falta <strong>de</strong> variación en las secuencias <strong>de</strong>l<br />
patógeno estudiadas en este trabajo, es que sus aislados <strong>de</strong>rivan <strong>de</strong> una<br />
población inicial fundadora pequeña (posiblemente un único individuo)<br />
que adquirió patogenicidad sobre Cicer spp. Como consecuencia <strong>de</strong> ello, la<br />
posterior diversificación en patotipos y razas patogénicas en dicho agente<br />
94
Capítulo III<br />
ha podido ser el resultado <strong>de</strong> la acumulación <strong>de</strong> pequeños cambios<br />
genéticos que hayan tenido lugar en fechas relativamente recientes<br />
(Gordon y Martyn, 1997). Aunque estos pequeños cambios genéticos no<br />
se han visto reflejados en las secuencias <strong>de</strong> los genes que hemos estudiado,<br />
si han podido ser <strong>de</strong>tectados mediante su asociación con marcadores<br />
RAPD. De hecho, los análisis <strong>de</strong> ‘cluster’ y <strong>de</strong> la varianza molecular<br />
(AMOVA, ‘Analysis of <strong>Molecular</strong> Variance’) <strong>de</strong> datos RAPD diferenciaron<br />
<strong>de</strong> forma consistente a los patotipos <strong>de</strong> Amarillez y Marchitez (Jiménez<br />
Gasco et al., 2001; Kelly et al., 1994; Capítulo I), y a las diferentes razas <strong>de</strong><br />
F. oxysporum f. sp. ciceris (Jiménez Gasco et al., 2001; Capítulo I). Ello<br />
sugiere que los marcadores RAPD están evolucionando más rápidamente<br />
que las secuencias <strong>de</strong> los genes utilizados para realizar análisis<br />
genealógicos.<br />
Basándonos en la conclusión <strong>de</strong> este trabajo sobre un origen evolutivo<br />
<strong>de</strong> monofilético <strong>de</strong> F. oxysporum f. sp. ciceris, nuestra hipótesis es que los<br />
polimorfismos tanto en fenotipos patogénicos como en marcadores<br />
moleculares <strong>de</strong>l patógeno, han <strong>de</strong>bido surgir <strong>de</strong>spués <strong>de</strong> que F. oxysporum f.<br />
sp. ciceris divergiese <strong>de</strong> otros taxones <strong>de</strong>ntro <strong>de</strong> F. oxysporum. En particular,<br />
nuestra hipótesis es que la variabilidad en patogenicidad que muestran los<br />
varios patotipos y razas <strong>de</strong>l agente sobre los diferentes tipos <strong>de</strong><br />
germoplasma <strong>de</strong> garbanzo, se <strong>de</strong>sarrolló en subpoblaciones <strong>de</strong>l hongo<br />
aisladas geográficamente que se produjeron <strong>de</strong>spués <strong>de</strong> la divergencia <strong>de</strong> la<br />
forma specialis ciceris <strong>de</strong>l resto <strong>de</strong> poblaciones <strong>de</strong> F. oxysporum.<br />
En Cicer spp. existen dos tipos <strong>de</strong> germoplasma, ‘kabuli’ y ‘<strong>de</strong>si’ (Singh,<br />
1987). Los garbanzos ‘<strong>de</strong>si’ (<strong>de</strong> semilla pequeña, angular, y coloreada) se<br />
cultivan principalmente en el subcontinente Indio, mientras que los<br />
garbanzos ‘kabuli’ (<strong>de</strong> semilla gran<strong>de</strong>, redon<strong>de</strong>ada y <strong>de</strong> color claro) se<br />
utilizan principalmente en la región Mediterránea y California.<br />
Curiosamente, el patotipo <strong>de</strong> Amarillez (que compren<strong>de</strong> a las razas 0 y<br />
1B/C) no ha sido <strong>de</strong>scrito en el subcontinente Indio, pero se encuentra <strong>de</strong><br />
forma generalizada en la región Mediterránea y en California (Halila y<br />
Strange, 1996; Jiménez Díaz et al., 1989b; 1993a; R. M. Jiménez Díaz, no<br />
publicado). La raza 0 no es patogénica sobre ‘JG-62’, el cultivar <strong>de</strong> garbanzo<br />
susceptible universal <strong>de</strong>l patotipo <strong>de</strong> Marchitez (que compren<strong>de</strong> a las razas<br />
95
Capítulo III<br />
1A, 2, 3, 4, 5, y 6), y es principalmente patogénica sobre garbanzos ‘kabuli’<br />
(Jiménez Díaz et al., 1989b; 1993a).<br />
En cambio, las fuentes <strong>de</strong> resistencia a F. oxysporum f. sp. ciceris se<br />
encuentran principalmente en el germoplasma tipo ‘<strong>de</strong>si’ (Haware et al.,<br />
1980), y la mayor parte <strong>de</strong> los genotipos ‘<strong>de</strong>si’ son resistentes a la raza 0<br />
(Jiménez Díaz et al., 1989b; 1993a). A<strong>de</strong>más, las razas 2, 3, y 4, <strong>de</strong>scritas<br />
hasta ahora únicamente en la India, son las razas más virulentas <strong>de</strong> las<br />
ocho razas patogénicas i<strong>de</strong>ntificadas hasta el momento (Halila y Strange,<br />
1996; Haware y Nene, 1982b; Jiménez Díaz et al., 1989b; 1993a) y son las<br />
razas que más distan genéticamente <strong>de</strong>l resto <strong>de</strong> las razas <strong>de</strong> F. oxysporum f.<br />
sp. ciceris (Jiménez Gasco et al., 2001; Kelly et al., 1994; Capítulo I).<br />
Esta correlación aparente entre patotipos y razas <strong>de</strong>l patógeno <strong>de</strong> una<br />
parte y a los tipos <strong>de</strong> germoplasma <strong>de</strong> garbanzo <strong>de</strong> otra, podría ser<br />
explicada si el patógeno está compuesto por poblaciones aisladas<br />
geográficamente y adaptadas al germoplasma local <strong>de</strong> garbanzo, incluso<br />
aunque todas parezcan haber evolucionado a partir <strong>de</strong>l mismo antepasado<br />
común. En cualquier caso, la <strong>de</strong>mostración <strong>de</strong> un origen monofilético para<br />
F. oxysporum f. sp. ciceris <strong>de</strong>sestima claramente la posibilidad <strong>de</strong> que las<br />
diferentes subpoblaciones hayan evolucionado a partir <strong>de</strong> orígenes<br />
diferentes, sobre distintos tipos <strong>de</strong> huéspe<strong>de</strong>s.<br />
La utilización <strong>de</strong> genealogías génicas en este Capítulo <strong>de</strong> la investigación<br />
ha permitido estudiar la evolución <strong>de</strong> F. oxysporum f. sp. ciceris como una<br />
grupo <strong>de</strong>rivado <strong>de</strong> una población fundadora pequeña o un individuo<br />
único. Aunque existe la posibilidad <strong>de</strong> que puedan <strong>de</strong>sarrollarse nuevas<br />
variantes <strong>de</strong>l patógeno con una mayor adaptación, que reemplacen a las<br />
poblaciones existentes, i. e., un ‘barrido’ selectivo; no existe evi<strong>de</strong>ncia <strong>de</strong><br />
que esto haya ocurrido en F. oxysporum f. sp. ciceris, a menos que los nuevos<br />
genotipos hayan tenido éxito en reemplazar a poblaciones existentes<br />
anteriormente en las principales zonas cultivadoras <strong>de</strong> garbanzo <strong>de</strong>l<br />
mundo. La uniformidad en las secuencias genéticas estudiadas a lo largo <strong>de</strong><br />
todo el rango geográfico es una notable evi<strong>de</strong>ncia <strong>de</strong> un origen único.<br />
Los resultados que hemos alcanzado en este estudio también resaltan la<br />
necesidad <strong>de</strong> seleccionar marcadores genéticos apropiados para respon<strong>de</strong>r<br />
96
Capítulo III<br />
a las cuestiones que le han dado origen, esto es, las secuencias <strong>de</strong> genes<br />
pue<strong>de</strong>n no revelar suficientes polimorfismos para distinguir entre los<br />
fenotipos <strong>de</strong> interés fitopatológico. Para esta aplicación, los marcadores<br />
RAPD han <strong>de</strong>mostrado ser <strong>de</strong> mayor utilidad (Assigbetse et al., 1994;<br />
Grajal Martin et al., 1993; Jiménez Gasco et al., 2001; Kelly et al., 1994;<br />
Capítulo I).<br />
97
CAPÍTULO IV<br />
Capítulo IV<br />
<strong>Caracterización</strong> <strong>de</strong> aislados <strong>de</strong> <strong>Fusarium</strong><br />
oxysporum f. sp. ciceris mediante ‘fingerprinting’<br />
genómico con secuencias <strong>de</strong> ADN repetitivo<br />
Introducción<br />
Las poblaciones <strong>de</strong> <strong>Fusarium</strong> oxysporum Schlechtend.:Fr. f. sp. ciceris<br />
(Padwick) Matuo & K. Sato, el agente causante <strong>de</strong> la Fusariosis Vascular<br />
<strong>de</strong>l garbanzo (Cicer arietinum L.), presentan una gran variabilidad<br />
patogénica. En F. oxysporum f. sp. ciceris se han i<strong>de</strong>ntificado dos patotipos<br />
(<strong>de</strong>nominados Amarillez y Marchitez) en función <strong>de</strong> la sintomatología que<br />
originan sus aislados en cultivares <strong>de</strong> garbanzo susceptibles; y ocho razas<br />
patogénicas (<strong>de</strong>nominadas 0, 1A, 1B/C, 2, 3, 4, 5, y 6), según la reacción<br />
que se <strong>de</strong>sarrolla al inocular una serie <strong>de</strong> líneas <strong>de</strong> garbanzo diferenciadoras<br />
(Haware y Nene, 1982b; Jiménez Díaz et al., 1989b; 1993a; Trapero Casas y<br />
Jiménez Díaz, 1985). Los mecanismos que han dado lugar a la aparición <strong>de</strong><br />
razas patogénicas en F. oxysporum f. sp. ciceris son <strong>de</strong>sconocidos. Puesto que<br />
F. oxysporum es un hongo anamórfico, la ausencia <strong>de</strong> un ciclo sexual<br />
conocido imposibilita el estudio genético <strong>de</strong>l patógeno mediante métodos<br />
convencionales.<br />
El conocimiento acerca <strong>de</strong> la extensión y distribución <strong>de</strong> la variabilidad<br />
genética existente tanto en las razas patogénicas <strong>de</strong> F. oxysporum f. sp. ciceris,<br />
como entre ellas como componentes poblacionales <strong>de</strong>l patógeno, es un<br />
99
Capítulo IV<br />
requisito importante a la hora <strong>de</strong> estudiar la relación genética entre dichas<br />
razas así como su posible origen evolutivo. Este conocimiento tiene<br />
importantes implicaciones no sólo en el manejo a<strong>de</strong>cuado <strong>de</strong> los cultivares<br />
<strong>de</strong> garbanzo resistentes a F. oxysporum f. sp. ciceris ya disponibles, sino<br />
también en el <strong>de</strong>sarrollo <strong>de</strong> nuevos cultivares con resistencia más efectiva y<br />
dura<strong>de</strong>ra a las diferentes razas <strong>de</strong>l patógeno.<br />
Los elementos <strong>de</strong> ADN repetitivo han mostrado ser herramientas muy<br />
útiles para estudios taxonómicos y epi<strong>de</strong>miológicos <strong>de</strong> hongos<br />
fitopatógenos (Goodwin et al., 1995; Levy et al., 1991; Roumen et al., 1997;<br />
Shull y Hamer, 1996; Zeigler et al., 1995), incluido F. oxysporum (Kistler et<br />
al., 1991; Mouyna et al., 1996; Namiki et al., 1994; 1998; Plyler et al., 2000;<br />
Tantaoui et al., 1996). Las hibridaciones <strong>de</strong> ADN genómico con sondas <strong>de</strong><br />
ADN repetitivo generan patrones <strong>de</strong> fragmentos (‘fingerprints’) <strong>de</strong> ADN<br />
que proporcionan información relativa a un gran número <strong>de</strong> loci <strong>de</strong> forma<br />
simultánea.<br />
Puesto que la probabilidad <strong>de</strong> que organismos no relacionados puedan<br />
compartir un mismo genotipo ‘multilocus’ es muy baja, el análisis <strong>de</strong><br />
‘fingerprints’ <strong>de</strong> ADN permite estructurar las poblaciones fúngicas en<br />
‘linajes clonales’; ésto es, grupos <strong>de</strong> aislados fúngicos que comparten una<br />
alta similaridad en patrones <strong>de</strong> ‘fingerprints’ <strong>de</strong> ADN (Zeigler et al., 1995).<br />
La interpretación evolutiva <strong>de</strong> esta aproximación fenética es que los<br />
aislados que componen un linaje clonal están relacionados genéticamente y<br />
<strong>de</strong>scien<strong>de</strong>n <strong>de</strong> un antepasado común. Una población fúngica con<br />
estructura <strong>de</strong> linajes es consistente con el modo <strong>de</strong> reproducción asexual<br />
<strong>de</strong>l patógeno, y generalmente, los distintos linajes clónicos presentan<br />
patrones <strong>de</strong> virulencia in<strong>de</strong>pendientes.<br />
Los estudios genéticos <strong>de</strong> poblaciones fúngicas mediante marcadores<br />
moleculares indican que existen diferentes patrones <strong>de</strong> diversidad entre las<br />
formae speciales <strong>de</strong> F. oxysporum (Kistler, 1997). Algunas formae speciales<br />
presentan escasa diversidad en haplotipos ‘multilocus’ basados en datos <strong>de</strong><br />
RFLP (‘Restriction Fragment Length Polymorphism’) <strong>de</strong> copia única;<br />
a<strong>de</strong>más, poseen similares haplotipos <strong>de</strong> ADN mitocondrial generados<br />
mediante RFLPs, y/o pertenecen a un único grupo <strong>de</strong> compatibilidad<br />
vegetativa (VCG). Este patrón <strong>de</strong> diversidad, tan simple, ha sido<br />
100
Capítulo IV<br />
i<strong>de</strong>ntificado en F. oxysporum f. sp. albedinis (Killian & Maire) Gordon<br />
(Fernán<strong>de</strong>z et al., 1997; Tantaoui et al., 1996), F. oxysporum f. sp. canariensis<br />
Mercier & Louvet (Plyler et al., 2000), y F. oxysporum f. sp. conglutinans<br />
(Wollenw.) W. C. Sny<strong>de</strong>r & H. N. Hansen (Bosland y Williams, 1987;<br />
Kistler et al., 1987). Dicho patrón <strong>de</strong> escasa similaridad es consistente con<br />
la estructura <strong>de</strong> una población resultante <strong>de</strong> la expansión <strong>de</strong> un único<br />
genotipo que ha tenido éxito en la adquisición <strong>de</strong> patogenicidad sobre un<br />
huésped <strong>de</strong>terminado (Kistler, 1997).<br />
Otras formae speciales <strong>de</strong> F. oxysporum poseen patrones <strong>de</strong> mayor<br />
diversidad que, aún siendo consistentes con la clonalidad consecuencia <strong>de</strong><br />
su sistema reproductivo asexual, se caracterizan por compren<strong>de</strong>r dos o<br />
más linajes <strong>de</strong>ntro <strong>de</strong> una forma specialis, que se refleja mediante la existencia<br />
<strong>de</strong> múltiples VCGs, y una diversidad alta <strong>de</strong> haplotipos mitocondriales y<br />
haplotipos ‘multilocus’ i<strong>de</strong>ntificados mediante RFLPs o RAPDs (‘Random<br />
Amplified Polymorphic DNA’). Estos patrones <strong>de</strong> mayor diversidad han<br />
sido i<strong>de</strong>ntificados en F. oxysporum f. sp. cubense (E. F. Smith) W. C. Sny<strong>de</strong>r<br />
& H. N. Hansen (Bentley et al., 1995; Koenig et al., 1997; O’Donnell et al.,<br />
1998), F. oxysporum f. sp. melonis (Leach & Currence) W. C. Sny<strong>de</strong>r & H. N.<br />
Hansen (Jacobson y Gordon, 1988; 1990), y F. oxysporum f. sp. lycopersici<br />
(Sacc.) W. C. Sny<strong>de</strong>r & H. N. Hansen (Elias y Schnei<strong>de</strong>r, 1991; Elias et al.,<br />
1993).<br />
Para explicar la relación entre formae speciales, razas, y VCGs en F.<br />
oxysporum se han propuesto varios mo<strong>de</strong>los evolutivos y se han i<strong>de</strong>ntificado<br />
numerosos ejemplos que reflejan cada uno <strong>de</strong> dichos mo<strong>de</strong>los (Correll,<br />
1991). En ciertas formae speciales (i. e., cubense, melonis) existe una compleja<br />
relación entre razas patogénicas y VCGs, <strong>de</strong> manera que distintos aislados<br />
<strong>de</strong>l patógeno pertenecientes a una misma raza pue<strong>de</strong>n ser adscritos a<br />
diferentes VCGs, y un mismo VCG pue<strong>de</strong> compren<strong>de</strong>r aislados <strong>de</strong><br />
diferentes razas (Appel y Gordon, 1994; Bentley et al., 1998; Jacobson y<br />
Gordon, 1990). Aunque numerosas investigaciones han <strong>de</strong>mostrado que<br />
pue<strong>de</strong> existir correlación entre VCGs y linajes clonales (ej., Elias et al.,<br />
1993; Koenig et al., 1997), existen muy pocos trabajos que hayan<br />
profundizado en la relación evolutiva entre razas que pertenezcan a un<br />
mismo VCG. Gordon y Martyn (1997) propusieron que una alta<br />
101
Capítulo IV<br />
similaridad genética entre razas patogénicas es indicativa <strong>de</strong> una posible<br />
evolución <strong>de</strong> unas razas a partir <strong>de</strong> otras mediante pequeños cambios<br />
genéticos (Gordon y Martyn, 1997). Aunque esta hipótesis es plausible,<br />
por el momento, en la literatura fitopatológica no se encuentran datos que<br />
la apoyen.<br />
En las investigaciones que han dado lugar a este Capítulo hemos<br />
utilizado sondas <strong>de</strong> ADN repetitivo para generar ‘fingerprints’ <strong>de</strong> ADN <strong>de</strong><br />
razas <strong>de</strong> F. oxysporum f. sp. ciceris, con el fin <strong>de</strong>: (1) examinar la variabilidad<br />
genética entre aislados <strong>de</strong> una misma raza, y entre distintas razas <strong>de</strong> este<br />
patógeno; (2) <strong>de</strong>terminar si las diferentes razas <strong>de</strong> F. oxysporum f. sp. ciceris<br />
están constituidas por linajes clonales genéticamente diferenciados, o en<br />
cambio, se encuentran agrupadas <strong>de</strong>ntro <strong>de</strong> un único linaje; y (3) estudiar la<br />
posible relación evolutiva entre dichas razas patogénicas.<br />
Materiales y Métodos<br />
Aislados fúngicos<br />
En este estudio se han utilizado 36 aislados <strong>de</strong> F. oxysporum f. sp. ciceris<br />
representativos <strong>de</strong> las ocho razas patogénicas <strong>de</strong>scritas (i. e., razas 0, 1A,<br />
1B/C, 2, 3, 4, 5, y 6) y <strong>de</strong> un amplio origen geográfico <strong>de</strong>l patógeno, así<br />
como tres aislados <strong>de</strong> F. oxysporum no patogénicos <strong>de</strong> garbanzo obtenidos<br />
<strong>de</strong> raíces <strong>de</strong> plantas sanas <strong>de</strong> garbanzo (Tabla 11). Todos los aislados <strong>de</strong> F.<br />
oxysporum f. sp. ciceris fueron caracterizados racialmente mediante pruebas<br />
<strong>de</strong> patogenicidad en trabajos anteriores (Jiménez Díaz et al., 1989b; 1993a;<br />
Jiménez Gasco et al., 2001; Kelly et al., 1994; Capítulo I), y proce<strong>de</strong>n <strong>de</strong> la<br />
colección <strong>de</strong> cultivos fúngicos <strong>de</strong>l Departamento <strong>de</strong> Protección <strong>de</strong><br />
Cultivos, Instituto <strong>de</strong> Agricultura Sostenible, CSIC, Córdoba.<br />
Los aislados se han mantenido como cultivos monoconídicos, en suelo<br />
estéril a 4ºC, en oscuridad. Los procedimientos para la obtención <strong>de</strong><br />
cultivos activos <strong>de</strong> los aislados fúngicos, el manejo <strong>de</strong> éstos, y la obtención<br />
102
Capítulo IV<br />
<strong>de</strong> su micelio para extracciones <strong>de</strong> ADN, han sido <strong>de</strong>scritos con<br />
anterioridad (Pág. 22, 26, Capítulo I; Pág. 52, Capítulo II).<br />
Extracción <strong>de</strong> ADN<br />
El ADN genómico <strong>de</strong> cada aislado se purificó a partir <strong>de</strong> micelio<br />
liofilizado y molido, según el método <strong>de</strong> Rae<strong>de</strong>r y Broda (1985) <strong>de</strong>scrito<br />
con anterioridad (Pág. 27-28, Capítulo I); la única excepción al método<br />
<strong>de</strong>scrito fue la utilización <strong>de</strong> 100 mg <strong>de</strong> micelio liofilizado y molido, con<br />
objeto <strong>de</strong> obtener ADN <strong>de</strong> mayor concentración, y en mayor cantidad<br />
para la realización <strong>de</strong> las digestiones con la enzima EcoRI.<br />
Hibridaciones ‘Southern’<br />
Se digirieron entre 3 y 5 µg <strong>de</strong> ADN <strong>de</strong> cada uno <strong>de</strong> los aislados <strong>de</strong> F.<br />
oxysporum f. sp. ciceris, y <strong>de</strong> los aislados <strong>de</strong> F. oxysporum no patogénicos <strong>de</strong><br />
garbanzo, con 15 a 25 U <strong>de</strong> la enzima EcoRI. Los fragmentos generados en<br />
la digestión enzimática se separaron mediante electroforesis en geles <strong>de</strong><br />
agarosa al 0,8 % <strong>de</strong> 15 x 25 cm, en tampón TAE (40 mM Tris-acetato; 1<br />
mM EDTA, pH 8,0) a 1 V·cm -1 durante 12-14 h. Posteriormente, los<br />
fragmentos separados electroforéticamente se transfirieron mediante vacío<br />
103
Capítulo IV<br />
Tabla 11. Información geográfica y racial sobre los aislados <strong>de</strong> <strong>Fusarium</strong><br />
oxysporum f. sp. ciceris (Foc), y <strong>de</strong> F. oxysporum (Fo) no patogénicos <strong>de</strong><br />
garbanzo, utilizados en el análisis <strong>de</strong> ‘fingerprints’ <strong>de</strong> ADN<br />
104<br />
Aislado Origen geográfico a<br />
Raza<br />
patogénica b<br />
Foc-7802 España 0<br />
Foc-7952 España 0<br />
Foc-8207 España 0<br />
Foc-82108 España 0<br />
Foc-82113 España 0<br />
Foc-9018 PV1 España 0<br />
Foc-9018 JG62 España 0<br />
Foc-9032 España 0<br />
Foc-91108 España 0<br />
Foc-91114 España 0<br />
Foc-9605 Túnez 0<br />
Foc-T3 Túnez 0<br />
Foc-USA 3-1 JG62 EEUU 1B/C<br />
Foc-1987-W17 EEUU 1B/C<br />
Foc-9602 Túnez 1B/C<br />
Foc-7989 India 1A<br />
Foc-9027 PV1 c España 1A<br />
Foc-9168 Marruecos 1A<br />
Foc-9165 Marruecos 1A<br />
Foc-9166 c Marruecos 1A<br />
Foc-8272 España 6<br />
Foc-9023 España 6<br />
Foc-9164 Marruecos 6<br />
Foc-9170 Marruecos 6<br />
Foc-Tonini EEUU 6
Capítulo IV<br />
Tabla 11. (Continuación) Información geográfica y racial sobre los<br />
aislados <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris (Foc), y <strong>de</strong> F. oxysporum (Fo) no<br />
patogénicos <strong>de</strong> garbanzo, utilizados en el análisis <strong>de</strong> ‘fingerprints’ <strong>de</strong> ADN<br />
Aislado Origen geográfico a<br />
Raza<br />
patogénica b<br />
Foc-8012 España 5<br />
Foc-9035 España 5<br />
Foc-9094 JG62 España 5<br />
Foc-USA 1-1 JG62 EEUU 5<br />
Foc-USA W6-1 EEUU 5<br />
Foc-8605 India 2<br />
Foc-1992R2N India 2<br />
Foc-8606 India 3<br />
Foc-1992R3N India 3<br />
Foc-8607 India 4<br />
Foc-1992R4N India 4<br />
Fo-8250 España NP<br />
Fo-9009 España NP<br />
Fo-90105 España NP<br />
a Los aislados <strong>de</strong> EEUU (California), España, y Marruecos proce<strong>de</strong>n <strong>de</strong> la<br />
micoteca <strong>de</strong>l Departamento <strong>de</strong> Protección <strong>de</strong> Cultivos, Instituto <strong>de</strong> Agricultura<br />
Sostenible, CSIC, Córdoba, España. Los aislados <strong>de</strong> la India fueron<br />
proporcionados por el Dr. M. P. Haware, ICRISAT, Hy<strong>de</strong>rabad, India. Los<br />
aislados proce<strong>de</strong>ntes <strong>de</strong> Túnez fueron cedidos por el Dr. M. H. Halila, Institute<br />
Nationale <strong>de</strong> la Recherche Agronomique, Ariana, Túnez.<br />
b Raza <strong>de</strong>terminada mediante pruebas <strong>de</strong> patogenicidad sobre líneas <strong>de</strong> garbanzo<br />
diferenciadoras (Alcalá Jiménez, 1995; Jiménez Gasco et al., 2001; Capítulo I).<br />
NP = no patogénicos <strong>de</strong> garbanzo.<br />
c Aislados caracterizados como pertenecientes a la raza 6 <strong>de</strong> F. oxysporum f. sp.<br />
ciceris en pruebas <strong>de</strong> patogenicidad duplicadas (Alcalá Jiménez, 1995).<br />
105
Capítulo IV<br />
(VacuGene XL y VacuGene Pump, Pharmacia LKB, Upsala, Suecia) a una<br />
membrana <strong>de</strong> nylon (Roche Diagnostics, Mannheim, Alemania).<br />
Como sondas <strong>de</strong> hibridación para generar ‘fingerprints’ <strong>de</strong> ADN se<br />
utilizaron fragmentos <strong>de</strong> ADN <strong>de</strong> naturaleza repetitiva i<strong>de</strong>ntificados con<br />
anterioridad a este estudio. Estos fragmentos <strong>de</strong> ADN fueron<br />
i<strong>de</strong>ntificados en el transcurso <strong>de</strong> los procesos <strong>de</strong> clonación y secuenciación<br />
<strong>de</strong> fragmentos RAPD marcadores <strong>de</strong> las razas 2 y 6 <strong>de</strong> F. oxysporum f. sp.<br />
ciceris, que se llevaron a cabo con objeto <strong>de</strong> diseñar iniciadores para<br />
reacciones <strong>de</strong> PCR específica (Ver Capítulos I y II). Los clones bacterianos<br />
que albergaron dichos fragmentos RAPD clonados fueron los siguientes:<br />
FocB10 (1,0 kb–raza 2–OPF12), FocO2 (1,0 kb–raza 6–OPI09), y FocP18<br />
(1,4 kb–raza 6–OPI09).<br />
La purificación <strong>de</strong> ADN plasmídico, así como la obtención <strong>de</strong> la sonda<br />
<strong>de</strong> ADN y su marcaje con DIG-11-dUTP(digoxigenina-3-O-metilcarbonilamino-caproil-5-(3-aminoalil)-uridina-5’-tri-fosfato),<br />
se realizaron por los<br />
procedimientos <strong>de</strong>scritos con anterioridad (Pág. 59-62 Capítulo II). Los<br />
tratamientos <strong>de</strong> prehibridación e hibridación se realizaron en condiciones<br />
<strong>de</strong> alta astringencia (68 ºC) (Sambrook et al., 1989). El revelado <strong>de</strong> las<br />
membranas <strong>de</strong> hibridación se realizó mediante métodos<br />
quimioluminiscentes <strong>de</strong>scritos en el Capítulo II (Pág. 62-63). Las<br />
hibridaciones se repitieron al menos dos veces.<br />
Análisis <strong>de</strong> datos<br />
Los datos obtenidos a partir <strong>de</strong> los ‘fingerprints’ <strong>de</strong> ADN generados<br />
mediante la hibridación <strong>de</strong> las digestiones <strong>de</strong> ADN genómico con sondas<br />
<strong>de</strong> ADN repetitivo, se han analizado mediante dos estrategias: (a) <strong>de</strong> tipo<br />
fenético, en el que el criterio utilizado es la totalidad <strong>de</strong> similitud <strong>de</strong><br />
caracteres entre organismos; y (b) <strong>de</strong> aproximación cladística, que analiza la<br />
relación entre los organismos teniendo en cuenta su historia evolutiva.<br />
Se construyó una matriz binaria resultante <strong>de</strong> combinar los datos<br />
generados por el análisis <strong>de</strong> hibridación <strong>de</strong> 36 aislados <strong>de</strong> F. oxysporum f. sp.<br />
106
Capítulo IV<br />
ciceris y tres aislados <strong>de</strong> F. oxysporum no patogénicos <strong>de</strong> garbanzo, con las<br />
tres sondas <strong>de</strong> ADN repetitivo FocB10, FocO2, y FocP18. En la matriz, la<br />
presencia o ausencia <strong>de</strong> los fragmentos i<strong>de</strong>ntificados se codificó como ‘1’ y<br />
‘0’, respectivamente.<br />
El análisis <strong>de</strong> similaridad fenético entre los 39 aislados <strong>de</strong> estudio se<br />
realizó mediante el programa NTSYSpc2.0 (Rohlf, 1988), utilizando el<br />
‘unweighted paired group method with arithmetic averages’ (UPGMA),<br />
que se basó en el coeficiente <strong>de</strong> similaridad <strong>de</strong> Jaccard (Sneath y Sokal,<br />
1973).<br />
El análisis cladístico se basó en un análisis <strong>de</strong> máxima parsimonia<br />
mediante el programa PAUP* 4.0b4a (‘Phylogenetic Analysis Using<br />
Parsimony and Other Methods’) (Swofford, 2000). El análisis <strong>de</strong><br />
parsimonia se realizó utilizando la búsqueda Heurística, y la significación<br />
<strong>de</strong> cada una <strong>de</strong> las agrupaciones generadas se confirmó mediante el análisis<br />
<strong>de</strong> ‘bootstrap’ con 1.000 repeticiones.<br />
Resultados<br />
En este estudio hemos utilizado sondas <strong>de</strong> ADN repetitivo <strong>de</strong> F. oxysporum<br />
f. sp. ciceris para generar ‘fingerprints’ <strong>de</strong> ADN genómico <strong>de</strong> razas <strong>de</strong> dicho<br />
agente. Este ADN repetitivo fue i<strong>de</strong>ntificado durante los procesos <strong>de</strong><br />
clonación y secuenciación <strong>de</strong> marcadores RAPD <strong>de</strong> las razas 2 (FocB10) y<br />
6 (FocO2 y FocP18) <strong>de</strong> F. oxysporum f. sp. ciceris, realizados para diseñar<br />
iniciadores <strong>de</strong> PCR específica. El análisis <strong>de</strong> las secuencias <strong>de</strong> dichos<br />
fragmentos <strong>de</strong> ADN indicó que éstos presentan similaridad con elementos<br />
transponibles <strong>de</strong> otros hongos citados en la literatura fitopatológica. En<br />
particular, la secuencia <strong>de</strong> FocB10 presentó un 80% <strong>de</strong> similaridad con el<br />
transposón Fot1 i<strong>de</strong>ntificado en F. oxysporum f. sp. melonis (Daboussi et al.,<br />
1992). Similarmente, la secuencia <strong>de</strong> FocO2 presentó un 52% <strong>de</strong><br />
similaridad con la codificante <strong>de</strong> una proteína similar a una transposasa<br />
i<strong>de</strong>ntificada en aislados <strong>de</strong> F. oxysporum (Okuda et al., 1998); y la secuencia<br />
107
Capítulo IV<br />
<strong>de</strong> FocP18 presentó cierta homología con transposasas putativas <strong>de</strong>scritas<br />
en numerosos organismos, aunque con niveles <strong>de</strong> similitud poco<br />
significativos.<br />
Los perfiles <strong>de</strong> hibridación generados con las tres sondas <strong>de</strong> ADN y el<br />
ADN genómico <strong>de</strong> los aislados <strong>de</strong> F. oxysporum f. sp. ciceris manifestaron<br />
una notable diferencia con respecto a los generados con ADN <strong>de</strong> aislados<br />
<strong>de</strong> F. oxysporum no patogénicos <strong>de</strong> garbanzo. Así, el clon FocB10 presentó<br />
homología con 20 a 30 <strong>de</strong> los fragmentos generados en la digestión <strong>de</strong>l<br />
ADN <strong>de</strong> F. oxysporum f. sp. ciceris con EcoRI; mientras que el patrón <strong>de</strong><br />
hibridación que se generó utilizando ADN <strong>de</strong> los aislados <strong>de</strong> F. oxysporum<br />
no patogénicos <strong>de</strong> garbanzo contuvo nueve ó 15 bandas <strong>de</strong> ADN (aislados<br />
Fo–8250 y –90105, respectivamente), o ninguna (aislado Fo–9009). El<br />
tamaño las bandas <strong>de</strong> hibridación observadas varió entre 1,5 y 12 kb,<br />
aproximadamente.<br />
Similarmente, la sonda <strong>de</strong> ADN FocO2 generó patrones <strong>de</strong> hibridación<br />
que contenían entre 20 y 30 bandas homólogas en aislados <strong>de</strong> F. oxysporum<br />
f. sp. ciceris, y sólo entre tres y 10 bandas en los aislados <strong>de</strong> F. oxysporum<br />
Fo–8250 y –90105 no patogénicos <strong>de</strong> garbanzo, respectivamente (Fig. 10).<br />
FocP18 fue probablemente la secuencia contenida más repetitivamente en<br />
el ADN genómico <strong>de</strong> F. oxysporum f. sp. ciceris. Así, el patrón <strong>de</strong> hibridación<br />
con la citada sonda fue tan profuso, que en el intervalo <strong>de</strong> peso molecular<br />
entre 5 y 10 kb no hubo resolución suficiente para i<strong>de</strong>ntificar bandas<br />
individuales <strong>de</strong>bido al ‘smear’ <strong>de</strong> fondo (rastro <strong>de</strong> hibridación continuo en<br />
el que no se i<strong>de</strong>ntifican bandas discretas). Aún así, en las hibridaciones se<br />
i<strong>de</strong>ntificaron ‘fingerprints’ <strong>de</strong> ADN formados por hasta 16 bandas<br />
claramente distinguibles. Al igual que con las otras sondas utilizadas, en las<br />
hibridaciones con esta secuencia <strong>de</strong> ADN repetitivo sólo se i<strong>de</strong>ntificaron<br />
tres o seis bandas <strong>de</strong> ADN homólogas en los aislados <strong>de</strong> F. oxysporum<br />
Fo–8250 y –90105 no patogénicos <strong>de</strong> garbanzo, respectivamente. Por el<br />
contrario, ninguna <strong>de</strong> las tres sondas <strong>de</strong> ADN repetitivo produjo señal <strong>de</strong><br />
hibridación alguna con el ADN genómico <strong>de</strong>l aislado <strong>de</strong> F. oxysporum Fo–<br />
9009.<br />
108
1<br />
5,<br />
2,<br />
1,<br />
1,0<br />
0,<br />
0,<br />
C 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18<br />
Capítulo IV<br />
Figura 10. ‘Fingerprint’ <strong>de</strong> ADN genómico <strong>de</strong> aislados <strong>de</strong> <strong>Fusarium</strong> oxysporum<br />
f. sp. ciceris representativos <strong>de</strong> todas las razas patogénicas <strong>de</strong>scritas (razas 0,<br />
1A, 1B/C, 2, 3, 4, 5, y 6), y <strong>de</strong> un amplio origen geográfico <strong>de</strong>l patógeno. La<br />
sonda utilizada fue FocO2, que contiene un elemento <strong>de</strong> ADN repetitivo<br />
i<strong>de</strong>ntificado en el genoma <strong>de</strong> F. oxysporum f. sp. ciceris. Los números a la<br />
izquierda indican el peso molecular aproximado (kb) <strong>de</strong> las bandas <strong>de</strong><br />
hibridación. Carriles: C, control <strong>de</strong> hibridación; 1-2, aislados <strong>de</strong> la raza 0 Foc–<br />
9018 JG62, –7802; 3-4, aislados <strong>de</strong> la raza 1B/C Foc–USA 3-1 JG62, –1987<br />
W17; 5, 6, aislados <strong>de</strong> cada una <strong>de</strong> las razas 2 y 4 Foc–8605, –1992R4N; 7-8,<br />
aislados <strong>de</strong> la raza 5 Foc–8012, – 9035; 9-13, aislados <strong>de</strong> la raza 1A Foc–7989,<br />
–9166, –9165, –9168, –9027 PV1; 14, 16, 17, aislados <strong>de</strong> la raza 6 Foc–Tonini,<br />
–8272, –9170; 15, 18, 19, aislados <strong>de</strong> F. oxysporum no patogénicos <strong>de</strong> garbanzo<br />
Fo–8250, –9009, y –90105, respectivamente.<br />
109
Capítulo IV<br />
Variabilidad genética entre razas <strong>de</strong> F. oxysporum f. sp. ciceris<br />
Las hibridaciones realizadas utilizando ADN genómico <strong>de</strong> los 36 aislados<br />
<strong>de</strong> F. oxysporum f. sp. ciceris y <strong>de</strong> tres aislados <strong>de</strong> F. oxysporum no patogénicos<br />
<strong>de</strong> garbanzo, y las tres sondas <strong>de</strong> ADN repetitivo FocB10, FocO2, y<br />
FocP18, produjeron 88 bandas claramente distinguibles y con la suficiente<br />
resolución para ser contabilizadas. Los patrones <strong>de</strong> ‘fingerprints’ <strong>de</strong> ADN<br />
fueron muy similares entre aislados <strong>de</strong> F. oxysporum f. sp. ciceris, aunque la<br />
presencia <strong>de</strong> ciertas bandas <strong>de</strong> hibridación permitió diferenciar visualmente<br />
a cada una <strong>de</strong> las razas mediante su perfil <strong>de</strong> hibridación.<br />
La similaridad entre los aislados fúngicos se <strong>de</strong>terminó mediante análisis<br />
<strong>de</strong> agregación (‘cluster’) <strong>de</strong> los datos <strong>de</strong> ‘fingerprinting’, en el que se<br />
consi<strong>de</strong>raron <strong>de</strong> forma conjunta las hibridaciones producidas por las tres<br />
sondas <strong>de</strong> ADN repetitivo. El <strong>de</strong>ndrograma así generado mostró que los<br />
aislados pertenecientes a una raza <strong>de</strong> F. oxysporum f. sp. ciceris son<br />
genéticamente más similares entre sí que lo son a los aislados<br />
pertenecientes a otras razas <strong>de</strong>l patógeno (Fig. 11).<br />
Los aislados <strong>de</strong> F. oxysporum f. sp. ciceris compartieron aproximadamente un<br />
32% <strong>de</strong> similaridad en ‘fingerprints’ <strong>de</strong> ADN genómico, y fueron<br />
claramente distinguibles <strong>de</strong> los aislados <strong>de</strong> F. oxysporum no patogénicos <strong>de</strong><br />
garbanzo incluidos en el análisis (Fig. 11). Dentro <strong>de</strong>l grupo <strong>de</strong> aislados <strong>de</strong><br />
F. oxysporum f. sp. ciceris se distinguieron dos grupos claramente<br />
correlacionados con la capacidad <strong>de</strong> los aislados <strong>de</strong> inducir el síndrome <strong>de</strong><br />
Amarillez (patotipo <strong>de</strong> Amarillez) o <strong>de</strong> Marchitez (patotipo <strong>de</strong> Marchitez),<br />
que compartieron aproximadamente un 50% <strong>de</strong> similaridad cada uno. A su<br />
vez, <strong>de</strong>ntro <strong>de</strong> los grupos correspondientes a cada uno <strong>de</strong> dichos<br />
patotipos, los aislados <strong>de</strong> F. oxysporum f. sp. ciceris se distribuyeron en<br />
agrupaciones correlacionadas con la naturaleza racial <strong>de</strong> cada aislado.<br />
110
0.00 0.25 0.50 0.75 1.00<br />
Capítulo IV<br />
Raza<br />
patogénica<br />
9018PV1 0<br />
9018JG62 0<br />
T3 0<br />
82108 0<br />
82113 0<br />
91108 0<br />
91114 0<br />
7802 0<br />
8207 0<br />
7952 0<br />
9032 0<br />
9605 0<br />
1987W17 1B/C<br />
USA3-1JG62 1B/C<br />
9602 1B/C<br />
7989 1A<br />
9168 1A<br />
8272 6<br />
9166 1A<br />
9170 6<br />
Tonini 6<br />
9165 1A<br />
9164 6<br />
9023 6<br />
9027PV1 1A<br />
8605 2<br />
1992R2N 2<br />
8606 3<br />
1992R3N 3<br />
1992R4N 4<br />
8607 4<br />
8012 5<br />
USA1-1JG62 5<br />
9035 5<br />
USAW6-1 5<br />
9094JG62 5<br />
8250 NP<br />
90105 NP<br />
9009 NP<br />
Figura 11. Dendrograma UPGMA generado a partir <strong>de</strong> la información obtenida<br />
mediante ‘fingerprints’ <strong>de</strong> ADN genómico <strong>de</strong> aislados <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp.<br />
ciceris (razas 0, 1A, 1B/C, 2 a 6) y F. oxysporum no patogénicos <strong>de</strong> garbanzo (NP)<br />
con las sondas <strong>de</strong> ADN repetitivo FocB10, FocO2, y FocP18. La escala inferior<br />
correspon<strong>de</strong> a la similaridad entre aislados basada en el coeficiente <strong>de</strong> Jaccard.<br />
111<br />
Amarillez Marchitez Marchitez
Capítulo IV<br />
El ‘cluster’ que agrupó a los aislados <strong>de</strong>l patotipo <strong>de</strong> Amarillez estuvo<br />
constituido por dos ‘subclusters’ (63-65%), correspondientes<br />
respectivamente a las razas 0 y 1B/C. De igual manera, <strong>de</strong>ntro <strong>de</strong>l ‘cluster’<br />
correspondiente al patotipo <strong>de</strong> Marchitez se distinguieron tres<br />
agrupaciones <strong>de</strong> aislados. De una parte, los aislados <strong>de</strong> las razas 1A y 6<br />
constituyeron un grupo (64% <strong>de</strong> similaridad) claramente diferenciado <strong>de</strong>l<br />
resto <strong>de</strong> aislados causantes <strong>de</strong>l síndrome <strong>de</strong> Marchitez. Este resto <strong>de</strong><br />
aislados estuvo comprendido por las razas 2, 3, 4, proce<strong>de</strong>ntes <strong>de</strong> la India,<br />
que formaron un grupo distinguible <strong>de</strong>l constituido por la raza 5, ambos<br />
comprendidos en el ‘cluster’ <strong>de</strong> aislados causantes <strong>de</strong> Marchitez (Fig. 11).<br />
Los aislados <strong>de</strong> la raza 5 se agruparon <strong>de</strong> forma distintiva al resto <strong>de</strong><br />
aislados <strong>de</strong> Marchitez, compartiendo un 90% <strong>de</strong> similaridad en los<br />
‘fingerprints’ <strong>de</strong> ADN genómico. No existió asociación alguna entre la<br />
distribución <strong>de</strong> los aislados en el <strong>de</strong>ndrograma y el origen geográfico <strong>de</strong> los<br />
aislados, a excepción <strong>de</strong> la indicada para los aislados <strong>de</strong> la India, que<br />
constituyeron un ‘cluster’ distintivo.<br />
Variabilidad genética entre aislados <strong>de</strong> F. oxysporum f. sp. ciceris<br />
pertenecientes a la misma raza<br />
En los ‘fingerprints’ <strong>de</strong> ADN genómico con sondas <strong>de</strong> ADN repetitivo, el<br />
nivel <strong>de</strong> similaridad <strong>de</strong> los polimorfismos i<strong>de</strong>ntificados entre aislados<br />
pertenecientes a una misma raza <strong>de</strong> F. oxysporum f. sp. ciceris varió<br />
notablemente según ésta. En particular, los aislados pertenecientes a la raza<br />
0 <strong>de</strong> F. oxysporum f. sp. ciceris compartieron aproximadamente un 57% <strong>de</strong><br />
similaridad (Fig. 11). En cambio, los aislados <strong>de</strong> la raza 5, proce<strong>de</strong>ntes <strong>de</strong><br />
España y California (EEUU), fueron genéticamente los más similares entre<br />
sí, con aproximadamente un 90% <strong>de</strong> similaridad en ‘fingerprints’ <strong>de</strong> ADN<br />
genómico. A<strong>de</strong>más, los aislados <strong>de</strong> la raza 0 se distribuyeron a su vez en<br />
dos agrupaciones con un 63 y 65% <strong>de</strong> similaridad cada uno, que no<br />
mostraron correlación geográfica alguna, ni asociación con los subgrupos<br />
112
Capítulo IV<br />
<strong>de</strong> virulencia <strong>de</strong>scritos <strong>de</strong>ntro <strong>de</strong> la raza 0 con anterioridad (Alcalá<br />
Jiménez, 1995; Jiménez-Díaz et al., 1993a).<br />
Los aislados pertenecientes a las razas 1A y 6 <strong>de</strong> F. oxysporum f. sp. ciceris<br />
se agruparon conjuntamente compartiendo aproximadamente un 64% <strong>de</strong><br />
similaridad en patrones <strong>de</strong> ‘fingerprints’ <strong>de</strong> ADN genómico. En la<br />
agrupación <strong>de</strong> los aislados <strong>de</strong> dichas razas se observó que los aislados Foc<br />
–9027 PV1, –9165, –9166, –9168, pertenecientes a la raza 1A, mostraron<br />
ser genéticamente más similares a los aislados <strong>de</strong> la raza 6 que a otros<br />
aislados <strong>de</strong> la raza 1A (i. e., Foc–7989). De igual manera, el aislado Foc–<br />
8272 caracterizado como raza 6, se agrupó junto a los aislados <strong>de</strong> la raza<br />
1A <strong>de</strong> F. oxysporum f. sp. ciceris (Fig. 11).<br />
Análisis filogenético <strong>de</strong> aislados <strong>de</strong> F. oxysporum f. sp. ciceris<br />
Con objeto <strong>de</strong> estudiar la posible relación evolutiva entre las razas <strong>de</strong> F.<br />
oxysporum f. sp. ciceris, se realizó un análisis filogenético basado en un<br />
método <strong>de</strong> máxima parsimonia a partir <strong>de</strong> los datos obtenidos mediante<br />
‘fingerprints’ <strong>de</strong> ADN genómico con las tres sondas <strong>de</strong> ADN repetitivo<br />
<strong>de</strong>scritos en el apartado anterior.<br />
Esta aproximación cladística produjo dos árboles igualmente<br />
parsimoniosos a partir <strong>de</strong> 67 caracteres filogenéticamente informativos<br />
[longitud <strong>de</strong>l árbol=202 pasos; índice <strong>de</strong> consistencia (CI)= 0,4158; índice<br />
<strong>de</strong> consistencia recalculado (RC)= 0,3271; índice <strong>de</strong> homoplasia (HI)=<br />
0,5842; índice <strong>de</strong> retención (RI)= 0,7866] (Fig. 12). La topografía general<br />
<strong>de</strong>l cladograma fue muy similar a la producida mediante el análisis fenético<br />
UPGMA.<br />
En dicho cladograma, el grupo <strong>de</strong> aislados <strong>de</strong> F. oxysporum f. sp. ciceris se<br />
diferenció claramente <strong>de</strong> los aislados <strong>de</strong> F. oxysporum no patogénicos <strong>de</strong><br />
garbanzo. A<strong>de</strong>más, los aislados <strong>de</strong> F. oxysporum f. sp. ciceris se distribuyeron<br />
en dos grupos correlacionados con el patotipo al que pertenecen; i. e., su<br />
capacidad <strong>de</strong> inducir el síndrome <strong>de</strong> Amarillez o <strong>de</strong> Marchitez.<br />
113
Capítulo IV<br />
A pesar <strong>de</strong> la similitud en la topografía <strong>de</strong> los análisis cladístico y<br />
fenético, en el análisis <strong>de</strong> máxima parsimonia se i<strong>de</strong>ntificaron varias<br />
diferencias con respecto al análisis UPGMA. En el análisis cladístico, el<br />
aislado Foc–9602, proce<strong>de</strong>nte <strong>de</strong> Túnez y caracterizado patogénicamente<br />
como raza 1B/C, se agrupó junto con los aislados <strong>de</strong> la raza 0 analizados<br />
(Fig. 12); mientras que el análisis UPGMA agrupó a dicho aislado junto a<br />
los aislados <strong>de</strong> la raza 1B/C proce<strong>de</strong>ntes <strong>de</strong> California (Fig. 11). En este<br />
contexto, es <strong>de</strong> resaltar que el análisis fenético analiza estrictamente la<br />
similaridad total en el patrón <strong>de</strong> homologías producido en las<br />
hibridaciones <strong>de</strong> ADN, mientras que la reconstrucción cladística refleja la<br />
relación entre los aislados teniendo en cuenta sólo los caracteres <strong>de</strong>rivados<br />
compartidos (i. e., un antepasado común). El aislado Foc–9602 fue<br />
caracterizado como perteneciente a la raza 1B/C mediante pruebas <strong>de</strong><br />
patogenicidad y mediante análisis RAPD; sin embargo, <strong>de</strong> las cinco bandas<br />
RAPD marcadoras <strong>de</strong> R1B/C i<strong>de</strong>ntificadas en los aislados <strong>de</strong> esta raza<br />
proce<strong>de</strong>ntes <strong>de</strong> California (EEUU), sólo tres <strong>de</strong> ellas se i<strong>de</strong>ntificaron en los<br />
perfiles <strong>de</strong> amplificación RAPD <strong>de</strong>l ADN genómico <strong>de</strong> Foc–9602<br />
utilizando iniciadores aleatorios (Capítulo I). A<strong>de</strong>más, el análisis <strong>de</strong>l ADN<br />
<strong>de</strong> este aislado mediante PCR (‘Polymerase Chain Reaction’) específica<br />
generó la banda marcadora <strong>de</strong> la raza 0 (Capítulo II).<br />
De igual forma, el análisis <strong>de</strong> máxima parsimonia mostró que los aislados<br />
<strong>de</strong> la raza 1A son más cercanos evolutivamente a las razas 2, 3, 4, y 5, que<br />
a los aislados <strong>de</strong> la raza 6. Similarmente, dicho análisis indicó que la raza 5<br />
es la más distante genéticamente, con un mayor número <strong>de</strong> pasos<br />
evolutivos (Fig. 12), y más cercana a las razas 2, 3, y 4, i<strong>de</strong>ntificadas<br />
únicamente en la India hasta ahora, que al resto <strong>de</strong> las razas causantes<br />
<strong>de</strong>l síndrome <strong>de</strong> Marchitez presentes en la Cuenca Mediterránea (razas 1A<br />
y 6).<br />
114
Capítulo IV<br />
71<br />
9018PV1 (R0)<br />
9018JG62 (R0)<br />
82108 (R0)<br />
T3 (R0)<br />
82113 (R0)<br />
91108 (R0)<br />
91114 (R0)<br />
59<br />
9602 (R1B/C)<br />
55<br />
7802 (R0)<br />
8207 (R0)<br />
53<br />
9032 (R0)<br />
7952 (R0)<br />
89<br />
9605 (R0)<br />
USAW17 (R1B/C)<br />
69<br />
USA 3-1 JG62 (R1B/C)<br />
83<br />
7989 (R1A)<br />
9168 (R1A)<br />
8272 (R6)<br />
90 8605 (R2)<br />
86 1992R2N (R2)<br />
8606 (R3)<br />
55 1992R3N (R3)<br />
1992R4N (R4)<br />
8607 (R4)<br />
87<br />
100<br />
95<br />
9166 (R1A)<br />
9165 (R1A)<br />
9170 (R6)<br />
59 8012 (R5)<br />
USA1-1JG62 (R5)<br />
9035 (R5)<br />
9094 JG62 (R5)<br />
70 USAW6-1 (R5)<br />
52<br />
Tonini (R6)<br />
9164 (R6)<br />
9023 (R6)<br />
9027PV1 (R1A)<br />
1 cambio<br />
8250 (NP)<br />
90105 (NP)<br />
9009 (NP)<br />
Figura 12. Uno <strong>de</strong> dos árboles filogenéticos <strong>de</strong> igual máxima parsimonia generados<br />
mediante PAUP en base a la información generada mediante ‘fingerprints’ <strong>de</strong> ADN<br />
genómico <strong>de</strong> aislados <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris (razas 0, 1A, 1B/C, 2 a 6),<br />
y <strong>de</strong> F. oxysporum no patogénicos <strong>de</strong> garbanzo (NP), generados con las sondas <strong>de</strong><br />
ADN repetitivo FocB10, FocO2, y FocP18. Los números en las líneas representan<br />
valores <strong>de</strong> ‘bootstrap’ >50% basados en 1.000 repeticiones.<br />
115
Capítulo IV<br />
Discusión<br />
Los objetivos <strong>de</strong> la investigación incluida en este Capítulo han sido<br />
<strong>de</strong>terminar la variabilidad genética existente entre los aislados <strong>de</strong> una<br />
misma raza, y entre las distintas razas patogénicas <strong>de</strong> F. oxysporum f. sp.<br />
ciceris; así como estudiar la posible relación evolutiva entre dichas razas.<br />
Para ello, hemos analizado los ‘fingerprints’ <strong>de</strong> ADN genómico <strong>de</strong> los<br />
aislados generados mediante hibridaciones con tres sondas <strong>de</strong> ADN<br />
repetitivo <strong>de</strong> F. oxysporum f. sp. ciceris, compuestas por secuencias que<br />
presentan similitud con elementos transponibles <strong>de</strong> otros hongos <strong>de</strong>scritos<br />
en la literatura fitopatológica.; y la similaridad existente entre los<br />
‘fingerprints’ <strong>de</strong> ADN <strong>de</strong> los distintos aislados <strong>de</strong> F. oxysporum f. sp. ciceris<br />
se analizó mediante dos aproximaciones, una <strong>de</strong> tipo fenético y otra <strong>de</strong><br />
tipo cladístico.<br />
El análisis <strong>de</strong> ‘fingerprints’ <strong>de</strong> ADN genómico ha puesto <strong>de</strong> manifiesto<br />
que los aislados <strong>de</strong> F. oxysporum f. sp. ciceris presentan patrones <strong>de</strong><br />
hibridación altamente correlacionados tanto con su capacidad <strong>de</strong> inducir<br />
un síndrome <strong>de</strong>terminado en la planta huésped (i. e., patotipos <strong>de</strong><br />
Amarillez y <strong>de</strong> Marchitez), como con su patogenicidad diferencial sobre<br />
líneas diferenciadoras <strong>de</strong> garbanzo (i. e., razas patogénicas),<br />
in<strong>de</strong>pendientemente <strong>de</strong>l tipo <strong>de</strong> análisis realizado. El análisis fenético <strong>de</strong> la<br />
similaridad entre ‘fingerprints’ <strong>de</strong> ADN mostró que, <strong>de</strong>ntro <strong>de</strong>l grupo <strong>de</strong>l<br />
patotipo <strong>de</strong> Amarillez, los aislados <strong>de</strong> F. oxysporum f. sp. ciceris se dividieron<br />
en dos grupos que correspon<strong>de</strong>n a la raza 0 y a la 1B/C. De igual manera,<br />
los aislados pertenecientes al patotipos <strong>de</strong> Marchitez se distribuyeron en<br />
varios grupos correspondientes a las razas 1A/6, raza 5, y al grupo <strong>de</strong> razas<br />
2, 3, y 4, <strong>de</strong>scritas únicamente en la India.<br />
Esta agrupación por similaridad <strong>de</strong> ‘fingerprints’ <strong>de</strong> los aislados <strong>de</strong> F.<br />
oxysporum f. sp. ciceris es consistente con la obtenida por Kelly et al. (1994)<br />
mediante el análisis RAPD con tres iniciadores aleatorios, y por Jiménez<br />
Gasco et al. (2001) (Capítulo I) mediante RAPDs utilizando siete<br />
iniciadores aleatorios diferentes a los anteriores. Es <strong>de</strong> resaltar que en los<br />
dos estudios referidos se utilizó una parte <strong>de</strong>l mismo grupo <strong>de</strong> aislados <strong>de</strong>l<br />
116
Capítulo IV<br />
patógeno que se ha utilizado en el análisis <strong>de</strong> ‘fingerprints’. Ésto sugiere<br />
que F. oxysporum f. sp. ciceris es un grupo clonal <strong>de</strong> aislados patógenos con<br />
una fuerte estructura racial, en el cual la correlación entre los marcadores<br />
genéticos neutrales, tales como los ‘fingerprints’ <strong>de</strong> ADN, y la<br />
patogenicidad, pue<strong>de</strong> ser utilizada para inferir conclusiones sobre la<br />
relación genética entre razas.<br />
En los últimos años se han realizado numerosos estudios <strong>de</strong> análisis <strong>de</strong><br />
‘fingerprints’ con ADN repetitivo en varias formae speciales <strong>de</strong> F. oxysporum,<br />
con objeto <strong>de</strong> inferir los mo<strong>de</strong>los <strong>de</strong> evolución entre razas patogénicas. La<br />
mayor parte <strong>de</strong> dichos estudios no mostró una correlación entre razas y<br />
evolución genética (Kim et al., 1992; Kistler et al., 1987; 1991; Manicom y<br />
Baayen, 1993; Manicom et al., 1990), aunque si ha sido muy clara la<br />
correlación entre ‘fingerprints’ <strong>de</strong> ADN y VCGs (Elias et al., 1993;<br />
Whitehead et al., 1992). Así, por ejemplo, en F. oxysporum f. sp. vasinfectum<br />
(Atk.) W. C. Sny<strong>de</strong>r & H. N. Hansen, el análisis RFLP <strong>de</strong>l ADN<br />
ribosómico (ADNr) y mitocondrial (ADNmt) no estableció una clara<br />
correlación entre razas pertenecientes a distintos VCGs y la variabilidad<br />
genética entre ellas, aunque los aislados <strong>de</strong> cada raza pudieron ser<br />
diferenciados al combinar la información <strong>de</strong>l análisis RFLP <strong>de</strong>l ADNr y<br />
<strong>de</strong>l ADNmt (Fernán<strong>de</strong>z et al., 1994). En la literatura fitopatológica no<br />
existen trabajos que analicen a fondo la evolución <strong>de</strong> razas en formae<br />
speciales monofiléticas, tal como F. oxysporum f. sp. ciceris.<br />
El análisis <strong>de</strong> ‘fingerprints’ <strong>de</strong> ADN reveló que algunos aislados <strong>de</strong> la<br />
raza 1A <strong>de</strong> F. oxysporum f. sp. ciceris (i. e., Foc–9027 PV1, –9166) son más<br />
similares genéticamente a los aislados <strong>de</strong> la raza 6 que al resto <strong>de</strong> los<br />
aislados <strong>de</strong> la raza 1A o al resto <strong>de</strong> las razas que inducen el síndrome <strong>de</strong><br />
Marchitez (Fig. 11). Esta agrupación ha sido i<strong>de</strong>ntificada con anterioridad<br />
(Jiménez Gasco et al., 2001; Capítulo I) y podría ser explicada bien por la<br />
caracterización errónea <strong>de</strong> algunos <strong>de</strong> los aislados, o por una alta<br />
similaridad genética entre dichas razas.<br />
De hecho, existe documentación que apoya las dos posibilida<strong>de</strong>s<br />
señaladas. Así, cuando la caracterización racial <strong>de</strong> los aislados <strong>de</strong> la raza 1A<br />
Foc–9027 JG62 y –9166 que realizó Alcalá Jiménez (1995) mediante<br />
pruebas <strong>de</strong> patogenicidad fue repetida para confirmar la reproducibilidad<br />
117
Capítulo IV<br />
<strong>de</strong> asignación a una raza, dichos aislados fueron caracterizados como<br />
pertenecientes a la raza 6 (Alcalá Jiménez, 1995). A<strong>de</strong>más, las reacciones<br />
<strong>de</strong> virulencia diferencial generadas por las razas 1A y 6 al inocular líneas<br />
diferenciadoras <strong>de</strong> garbanzo en pruebas <strong>de</strong> patogenicidad son muy<br />
similares entre sí, ya que las razas 1A y 6 se diferencian principalmente por<br />
la reacción <strong>de</strong> los cvs. ‘ICCV-2’ e ‘ICCV-4’ (resistentes a la raza 1A,<br />
susceptibles a la raza 6) (Halila y Strange, 1996; Jiménez Díaz et al., 1993a).<br />
Tales diferencias en la asignación <strong>de</strong> un aislado a una u otra raza por el<br />
fenotipo <strong>de</strong> la reacción <strong>de</strong> ‘ICCV-2’ a la inoculación, pue<strong>de</strong> estar asociada<br />
con el ciclo corto que caracteriza a este cultivar. En condiciones <strong>de</strong> cultivo<br />
en maceta en ambiente controlado óptimo para el crecimiento <strong>de</strong> la planta<br />
y el <strong>de</strong>sarrollo <strong>de</strong> la infección, tiene lugar una senescencia precoz <strong>de</strong> la<br />
planta que pue<strong>de</strong> ser confundida con una reacción patogénica si no se<br />
confirma la asociación entre sintomatología e infección mediante al<br />
aislamiento positivo <strong>de</strong>l agente en cultivo puro. Por todo ello, no es <strong>de</strong><br />
<strong>de</strong>scartar la posibilidad <strong>de</strong> un error en la caracterización racial <strong>de</strong> los<br />
aislados referidos, <strong>de</strong>bida a algún fallo durante la realización <strong>de</strong> las pruebas<br />
<strong>de</strong> patogenicidad. Asimismo, también es posible que estas dos razas<br />
patogénicas aún no se encuentren suficientemente separadas entre sí en<br />
términos evolutivos, para haber <strong>de</strong>sarrollado patrones <strong>de</strong> ‘fingerprinting’<br />
<strong>de</strong> ADN y patogénicos in<strong>de</strong>pendientes. Finalmente, también existe la<br />
posibilidad <strong>de</strong> que se haya producido una evolución convergente <strong>de</strong><br />
patogenicidad (i. e., raza 1A) a partir <strong>de</strong> antepasados genéticamente<br />
diferentes, dando lugar a dos grupos <strong>de</strong> ‘fingerprints’ <strong>de</strong> ADN <strong>de</strong>ntro <strong>de</strong> la<br />
raza 1A, uno <strong>de</strong> ellos genéticamente más similar al que presenta la raza 6.<br />
In<strong>de</strong>pendientemente <strong>de</strong> lo <strong>de</strong>scrito anteriormente, el análisis fenético<br />
(UPGMA) <strong>de</strong> los ‘fingerprints’ <strong>de</strong> ADN mostró que todos los aislados <strong>de</strong><br />
la raza 1A fueron más similares genéticamente a los aislados <strong>de</strong> la raza 6<br />
que al resto <strong>de</strong> las razas <strong>de</strong> F. oxysporum f. sp. ciceris (Fig. 11). En cambio, el<br />
análisis filogenético indicó que la raza 1A está más cercana evolutivamente<br />
a las razas 2, 3, 4, y 5, que a la raza 6 (Fig. 12). Por lo tanto, es necesario un<br />
análisis más profundo, tanto genético como patogénico, <strong>de</strong> las razas 1A y 6<br />
<strong>de</strong> F. oxysporum f. sp. ciceris para aclarar su relación genética.<br />
118
Capítulo IV<br />
El aislado Foc–9602, proce<strong>de</strong>nte <strong>de</strong> Túnez, fue caracterizado como<br />
perteneciente a la raza 1B/C <strong>de</strong> F. oxysporum f. sp. ciceris mediante pruebas<br />
<strong>de</strong> patogenicidad (Capítulo I). En este estudio, el análisis <strong>de</strong> similaridad<br />
fenética situó a este aislado junto a los aislados <strong>de</strong> la raza 1B/C<br />
proce<strong>de</strong>ntes <strong>de</strong> California (EEUU) (Fig. 11); mientras que mediante el<br />
análisis cladístico dicho aislado fue agrupado junto a los aislados <strong>de</strong> la raza<br />
0 (Fig. 12). En pruebas <strong>de</strong> patogenicidad (Capítulo I), el aislado Foc–9602<br />
mostró menor virulencia sobre el cultivar diferenciador C-104 que el resto<br />
<strong>de</strong> los aislados <strong>de</strong> F. oxysporum f. sp. ciceris analizados y que el aislado tipo<br />
<strong>de</strong> la raza 0 (Foc–7802); pero resultó más virulento que los aislados <strong>de</strong> raza<br />
0 sobre ‘ICCV-4’ (Capítulo I). Estas interacciones cultivar-aislado sugieren<br />
características patogénicas intermedias entre la raza 0 y la raza 1B/C.<br />
El análisis filogenético <strong>de</strong> los ‘fingerprints’ <strong>de</strong> ADN mostró que los<br />
aislados <strong>de</strong> la raza 5 (no <strong>de</strong>scrito en la India) se encuentran más cercanos<br />
evolutivamente a las razas 2, 3, y 4 (sólo <strong>de</strong>scritas en la India) que al resto<br />
<strong>de</strong> las razas presentes en la Cuenca Mediterránea (razas 1A y 6). Aunque la<br />
raza 5 y las razas 2, 3, y 4 se encuentran geográficamente separadas entre<br />
sí, dichas razas son las más similares en relación a la virulencia que<br />
manifiestan sobre líneas y cultivares diferenciadores. Esta agrupación<br />
pue<strong>de</strong> ser explicada si dichas razas constituyen grupos relacionados<br />
evolutivamente pero aislados geográficamente, y adaptados al<br />
germoplasma local <strong>de</strong> garbanzo, incluso aunque todas parezcan haber<br />
evolucionado a partir <strong>de</strong>l mismo antepasado.<br />
En Cicer spp. existen dos tipos <strong>de</strong> germoplasma, <strong>de</strong>nominados ‘kabuli’ y<br />
‘<strong>de</strong>si’ (Singh, 1987). Los garbanzos ‘<strong>de</strong>si’ (<strong>de</strong> semilla pequeña, angular, y<br />
coloreada) se cultivan principalmente en el subcontinente Indio, mientras<br />
que los garbanzos ‘kabuli’ (<strong>de</strong> semilla gran<strong>de</strong>, redon<strong>de</strong>ada y <strong>de</strong> color claro)<br />
se utilizan principalmente en la región Mediterránea y California.<br />
Si la patogenicidad sobre un huésped en particular constituyó un evento<br />
único y aislado en el proceso evolutivo <strong>de</strong>l patógeno, y el genotipo en<br />
cuestión se distribuyó geográficamente <strong>de</strong> forma generalizada, cabe esperar<br />
que el patógeno diferenciado sea genéticamente distinto a los aislados no<br />
patogénicos con los que coexiste. En cambio, la relación genética cercana<br />
entre aislados patogénicos y no patogénicos <strong>de</strong> una zona en particular<br />
119
Capítulo IV<br />
indica una <strong>de</strong>rivación o selección reciente <strong>de</strong> dicho patógeno a partir <strong>de</strong> la<br />
población <strong>de</strong> aislados no patogénicos (Gordon y Okamoto, 1992). La<br />
notable diferencia entre los ‘fingerprints’ <strong>de</strong> ADN genómico observadas<br />
entre los aislados <strong>de</strong> F. oxysporum f. sp. ciceris y los aislados <strong>de</strong> F. oxysporum<br />
no patogénicos <strong>de</strong> garbanzo, es consistente con la hipótesis <strong>de</strong> que la<br />
población actual <strong>de</strong> este patógeno <strong>de</strong>bió estar constituida por un único<br />
genotipo o un número limitado <strong>de</strong> ellos que adquirió la capacidad <strong>de</strong><br />
causar enfermedad en Cicer spp. Esta hipótesis está a<strong>de</strong>más apoyada por la<br />
existencia <strong>de</strong> un único VCG en F. oxysporum f. sp. ciceris (Nogales Moncada,<br />
1997), y <strong>de</strong> un único perfil <strong>de</strong> restricción <strong>de</strong>l ADN mitocondrial (Pérez<br />
Artés et al., 1995), así como por la ausencia <strong>de</strong> polimorfismos en las<br />
secuencias <strong>de</strong> cinco genes conservados (Capítulo III).<br />
Mientras que otras formae speciales compuestas por un único VCG han<br />
mostrado una diversidad genética muy limitada (i. e., formae speciales<br />
albedinis, y canariensis) (Plyler et al., 2000; Tantaoui et al., 1996), nuestros<br />
resultados indican que F. oxysporum f. sp. ciceris es una forma specialis<br />
altamente diversificada, patogénica y genéticamente. Esta diversidad pue<strong>de</strong><br />
ser reflejo <strong>de</strong> una larga historia <strong>de</strong> asociación con su planta huésped, el<br />
garbanzo, como especie cultivada. Aunque la presencia constatada más<br />
antigua <strong>de</strong>l garbanzo data <strong>de</strong>l año 5.450 a <strong>de</strong> C., en Turquía; información<br />
arqueológica reciente data el origen <strong>de</strong> esta planta hace 8.600 a 8.900 años<br />
a <strong>de</strong> C. A<strong>de</strong>más, dicha información indica que el garbanzo constituyó,<br />
junto a otras seis especies <strong>de</strong> plantas, la cuna <strong>de</strong> la agricultura localizada en<br />
el suroeste <strong>de</strong> la actual Turquía limitante con Siria (Lev-Yadun et al., 2000).<br />
Este ha sido el único lugar don<strong>de</strong> se han i<strong>de</strong>ntificado especímenes<br />
silvestres <strong>de</strong> garbanzo (Singh, 1997).<br />
Hipotéticamente, las razas patogénicas más antiguas han <strong>de</strong>bido<br />
acumular más diversidad genética que aquéllas originadas más<br />
recientemente (Goodwin et al., 1995), especialmente si asumimos el origen<br />
<strong>de</strong> una raza a partir <strong>de</strong> un único genotipo, seguido <strong>de</strong> su dispersión más o<br />
menos generalizada. A<strong>de</strong>más, se asume que la base primitiva <strong>de</strong> las formae<br />
speciales fue una población parasítica, no patogénica, que evolucionó hacia<br />
la adquisición <strong>de</strong> patogenicidad sobre un huésped <strong>de</strong>terminado (Correll,<br />
1991; Leslie, 1993). Por lo tanto, en un mo<strong>de</strong>lo <strong>de</strong> evolución en las que<br />
120
Capítulo IV<br />
unas razas patogénicas se originan a partir <strong>de</strong> otras a través <strong>de</strong> cambios<br />
genéticos más o menos sencillos (Gordon y Martyn, 1997), las razas más<br />
antiguas habrán <strong>de</strong> ser menos virulentas que las más recientemente<br />
evolucionadas.<br />
Nuestros resultados apoyan esta hipótesis evolutiva para F. oxysporum f.<br />
sp. ciceris, en tanto las que la evolución más reciente <strong>de</strong> las razas<br />
patogénicas va unida a una mayor virulencia <strong>de</strong> estas. Así, la raza 0 es la<br />
menos virulenta <strong>de</strong> todas las razas, y es la que más diversidad genética<br />
presenta (Fig. 11 y 12), por lo que correspon<strong>de</strong> a la más ancestral. Por el<br />
contrario, la raza 5 es la más virulenta <strong>de</strong> las i<strong>de</strong>ntificadas hasta ahora en la<br />
Cuenca Mediterránea, la que menor diversidad genética posee, y a la que<br />
por tanto correspon<strong>de</strong>ría un origen más reciente. A<strong>de</strong>más, <strong>de</strong>s<strong>de</strong> un punto<br />
<strong>de</strong> vista evolutivo, a la raza 0 <strong>de</strong>bería seguir la raza 1B/C, no sólo en<br />
cuanto a su nivel <strong>de</strong> virulencia, sino también en cuanto a su capacidad <strong>de</strong><br />
causar el síndrome <strong>de</strong> Amarillez en cultivares <strong>de</strong> garbanzo susceptibles.<br />
Esta hipótesis parece ser coherente con nuestros resultados, ya que estas<br />
dos razas están genéticamente más relacionadas entre sí que con el resto <strong>de</strong><br />
las razas <strong>de</strong> F. oxysporum f. sp. ciceris.<br />
El tipo <strong>de</strong> elementos <strong>de</strong> ADN repetitivo que hemos utilizado para<br />
generar ‘fingerprints’ <strong>de</strong> ADN genómico presentan secuencias<br />
características <strong>de</strong> transposones. Ello indica que la ganancia o pérdida<br />
paralela <strong>de</strong> fragmentos <strong>de</strong> ADN vía transposición o escisión genera un alto<br />
nivel <strong>de</strong> homoplasia, que pue<strong>de</strong> resultar en una falsa interpretación<br />
filogenética y hacer que este tipo <strong>de</strong> datos no sean convenientes como<br />
indicadores <strong>de</strong> <strong>de</strong>scen<strong>de</strong>ncia común (O’Hanlon y Peakall, 2000), aunque<br />
sean muy valiosos para el estudio <strong>de</strong> la similaridad entre aislados, en<br />
especial entre organismos clonales (Carbone et al., 1999). Sin embargo,<br />
tanto el análisis <strong>de</strong> similaridad fenética entre ‘fingerprints’ <strong>de</strong> ADN, como<br />
el análisis filogenético <strong>de</strong> éstos, han mostrado ser congruentes entre sí, y se<br />
correlacionan con las características fitopatógenas <strong>de</strong> los aislados <strong>de</strong> F.<br />
oxysporum f. sp. ciceris analizados.<br />
Numerosos estudios señalan la posibilidad <strong>de</strong> que el movimiento <strong>de</strong><br />
transposones en el genoma <strong>de</strong> un organismo sea uno <strong>de</strong> los mecanismos<br />
generadores <strong>de</strong> la gran variabilidad genética observada en F. oxysporum<br />
121
Capítulo IV<br />
(Daboussi, 1997; Daboussi y Langin, 1994), dando lugar a mutación <strong>de</strong><br />
genes o reorganizaciones genómicas mediante inserciones o escisiones<br />
(Kistler y Miao, 1992). Nuestro trabajo ha <strong>de</strong>mostrado que el genoma <strong>de</strong> F.<br />
oxysporum f. sp. ciceris contiene numerosas secuencias homólogas a<br />
secuencias similares a transposones <strong>de</strong>scritos en otros hongos, entre ellos<br />
Fot1 (Daboussi et al., 1992), cuya actividad en el sentido arriba indicado ha<br />
sido <strong>de</strong>mostrada. Este tipo <strong>de</strong> elementos constituyen una fuente potencial<br />
<strong>de</strong> variación que pue<strong>de</strong> dar lugar a la aparición eventual <strong>de</strong> nuevas razas a<br />
partir <strong>de</strong> las ya existentes. El seguimiento <strong>de</strong> genotipos mediante la<br />
generación <strong>de</strong> ‘fingerprints’ <strong>de</strong> ADN como los <strong>de</strong>scritos en este trabajo,<br />
será sumamente útil para <strong>de</strong>tectar los cambios genéticos que se puedan<br />
producir en las poblaciones <strong>de</strong> F. oxysporum f. sp. ciceris.<br />
122
CAPÍTULO V<br />
Capítulo V<br />
Mejora <strong>de</strong> la metodología <strong>de</strong> análisis <strong>de</strong><br />
poblaciones <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris<br />
mediante PCR entre elementos <strong>de</strong> ADN repetitivo<br />
(rep-PCR)<br />
Introducción<br />
La amplificación entre elementos repetitivos <strong>de</strong> ADN mediante análisis <strong>de</strong><br />
la reacción en ca<strong>de</strong>na <strong>de</strong> la polimerasa (PCR, ‘Polymerase Chain<br />
Reaction’), o rep-PCR (‘repetitive element-based PCR’), genera<br />
‘fingerprints’ (patrones <strong>de</strong> fragmentos) <strong>de</strong> ADN mediante la amplificación<br />
entre las múltiples copias <strong>de</strong> los elementos repetitivos dispersos en el<br />
genoma mol<strong>de</strong>. Esta metodología combina la simplicidad <strong>de</strong> la técnica <strong>de</strong>l<br />
análisis PCR, con el nivel <strong>de</strong> polimorfismos que pue<strong>de</strong> ser <strong>de</strong>tectado<br />
mediante la hibridación <strong>de</strong>l ADN genómico utilizando sondas basadas en<br />
secuencias repetitivas, y ha sido utilizada para el análisis <strong>de</strong> hongos,<br />
bacterias <strong>de</strong>l suelo, y bacterias fitopatógenas (E<strong>de</strong>l et al., 1995; George et<br />
al., 1997).<br />
En los análisis rep-PCR referidos, los iniciadores utilizados<br />
correspon<strong>de</strong>n a secuencias <strong>de</strong> regiones <strong>de</strong> ADN conservadas en bacterias,<br />
como la secuencia REP (‘Repetitive Extragenic Palindromic Sequence’)<br />
(Stern et al., 1984), las secuencias ERIC (‘Enterobacterial Repetitive<br />
123
Capítulo V<br />
Intergenic Consensus Sequences’) (Hulton et al., 1991), y el elemento BOX<br />
(Martin et al., 1992). La naturaleza altamente conservada <strong>de</strong> estas<br />
secuencias ha permitido su utilización para el análisis <strong>de</strong> numerosos<br />
microorganismos, incluidos hongos fitopatógenos. Sin embargo,<br />
posteriores estudios han <strong>de</strong>mostrado que se pue<strong>de</strong> alcanzar un mayor nivel<br />
<strong>de</strong> polimorfismos y especificidad si la rep-PCR se basa en elementos<br />
repetitivos endógenos, o propios <strong>de</strong>l organismo <strong>de</strong> estudio (George et al.,<br />
1997; 1998).<br />
En trabajos anteriores en esta Tesis (Capítulos II y IV) hemos<br />
i<strong>de</strong>ntificado secuencias <strong>de</strong> ADN repetitivo en el genoma <strong>de</strong> <strong>Fusarium</strong><br />
oxysporum Schlechtend.:Fr. f. sp. ciceris (Padwick) Matuo & K. Sato, el<br />
agente causante <strong>de</strong> la Fusariosis Vascular <strong>de</strong>l garbanzo (Cicer arietinum L.).<br />
Las poblaciones naturales <strong>de</strong> este patógeno presentan una gran variabilidad<br />
patogénica y genética. Respecto <strong>de</strong> la diversidad patogénica, experimentos<br />
<strong>de</strong> patogenicidad en condiciones controladas han llevado a i<strong>de</strong>ntificar dos<br />
patotipos, <strong>de</strong>nominados Amarillez y Marchitez y ocho razas patogénicas,<br />
<strong>de</strong>nominadas razas 0, 1A, 1B/C, 2, 3, 4, 5, y 6 (Halila y Strange, 1996;<br />
Haware y Nene, 1982b; Jiménez Díaz et al., 1989b; 1993a). Mientras que<br />
los patotipos <strong>de</strong> Amarillez y Marchitez son <strong>de</strong>terminados por las<br />
características <strong>de</strong>l síndrome patológico que inducen en cultivares <strong>de</strong><br />
garbanzo susceptibles (Trapero Casas, 1983; Trapero Casas y Jiménez<br />
Díaz, 1985), la asignación <strong>de</strong> los aislados <strong>de</strong> F. oxysporum f. sp. ciceris a las<br />
ocho razas patogénicas se basa en la reacción <strong>de</strong> líneas <strong>de</strong> garbanzo<br />
diferenciadoras en inoculaciones estandarizadas (Haware y Nene, 1982b;<br />
Jiménez Díaz et al., 1989b; 1993a).<br />
Las secuencias i<strong>de</strong>ntificadas en el genoma <strong>de</strong> F. oxysporum f. sp. ciceris<br />
antes referidas (Capítulos II y IV) han mostrado similaridad con<br />
transposones <strong>de</strong> genomas fúngicos <strong>de</strong>scritos en la literatura (Daboussi et<br />
al., 1992; Okuda et al., 1998). Cuando dichas secuencias <strong>de</strong> F. oxysporum f.<br />
sp. ciceris se utilizan como sondas en hibridaciones <strong>de</strong>l ADN genómico,<br />
estas secuencias repetitivas presentan homología con un alto número <strong>de</strong><br />
fragmentos (20 a 30) <strong>de</strong> ADN, generando ‘fingerprints’ cuya utilidad para<br />
<strong>de</strong>terminar la variabilidad genética existente entre las razas patogénicas <strong>de</strong><br />
F. oxysporum f. sp. ciceris ha sido <strong>de</strong>mostrada (Capítulo IV). De esta forma,<br />
124
Capítulo V<br />
hemos i<strong>de</strong>ntificado numerosos polimorfismos asociados a la naturaleza<br />
patotípica y racial <strong>de</strong> los aislados <strong>de</strong>l patógeno analizados. Los<br />
polimorfismos así i<strong>de</strong>ntificados pue<strong>de</strong>n ser <strong>de</strong>bidos a diferencias en el<br />
número <strong>de</strong> copias <strong>de</strong> estos elementos presentes en el genoma fúngico, o a<br />
diferencias en los lugares <strong>de</strong> inserción <strong>de</strong> ellos en dicho genoma. A<strong>de</strong>más,<br />
la información generada por dichos ‘fingerprints’ <strong>de</strong> ADN genómico ha<br />
permitido profundizar en la relación evolutiva entre las distintas razas<br />
patogénicas, con lo cual, las secuencias <strong>de</strong> ADN repetitivo <strong>de</strong>l patógeno<br />
constituyen herramientas <strong>de</strong> gran utilidad para el estudio <strong>de</strong> la estructura<br />
<strong>de</strong> las poblaciones <strong>de</strong> F. oxysporum f. sp. ciceris.<br />
La obtención <strong>de</strong> ‘fingerprints’ <strong>de</strong> ADN genómico para las<br />
investigaciones que constituyen el Capítulo IV <strong>de</strong> esta Tesis, se ha basado<br />
en la realización <strong>de</strong> hibridaciones tipo ‘Southern’ con sondas <strong>de</strong> ADN<br />
repetitivo. Esta metodología requiere <strong>de</strong> la utilización <strong>de</strong> una gran cantidad<br />
<strong>de</strong> ADN <strong>de</strong> alta calidad, así como <strong>de</strong> tiempo <strong>de</strong> análisis prolongado y<br />
amplios recursos, lo cual dificulta significativamente el análisis <strong>de</strong> un<br />
número elevado <strong>de</strong> aislados fúngicos. Por tanto, se necesita una<br />
metodología <strong>de</strong> estudio que sea tan informativa como el ‘fingerprinting’ <strong>de</strong><br />
ADN genómico <strong>de</strong>scrito anteriormente, pero cuya realización sea lo<br />
suficientemente sencilla y no necesitada <strong>de</strong> recursos excesivos como para<br />
po<strong>de</strong>r ser utilizada <strong>de</strong> forma rutinaria en el laboratorio.<br />
El objetivo <strong>de</strong> la investigación recogida en este Capítulo ha sido<br />
<strong>de</strong>mostrar la hipótesis <strong>de</strong> que los polimorfismos generados mediante la<br />
hibridación <strong>de</strong>l ADN genómico <strong>de</strong> F. oxysporum f. sp. ciceris con una sonda<br />
constituida por un elemento repetitivo endógeno en dicho genoma, en<br />
particular un transposón, pue<strong>de</strong>n ser i<strong>de</strong>ntificados mediante análisis rep-<br />
PCR basado en la amplificación <strong>de</strong>l ADN contenido entre las diferentes<br />
copias <strong>de</strong> dicho elemento. Si dicha hipótesis se prueba cierta, la<br />
información generada mediante hibridaciones ‘Southern’ será equivalente a<br />
la generada mediante el análisis rep-PCR, y este último procedimiento<br />
proporcionará un método alternativo más practicable para el análisis <strong>de</strong><br />
extensas poblaciones <strong>de</strong> F. oxysporum f. sp. ciceris.<br />
125
Capítulo V<br />
Materiales y Métodos<br />
Aislados fúngicos<br />
En este estudio se han utilizado 32 aislados <strong>de</strong> F. oxysporum f. sp. ciceris<br />
representativos <strong>de</strong> las ocho razas patogénicas <strong>de</strong>scritas (0, 1A, 1B/C, 2, 3,<br />
4, 5, y 6), y <strong>de</strong> un amplio origen geográfico <strong>de</strong>l patógeno (Tabla 12).<br />
Dichos aislados <strong>de</strong> F. oxysporum f. sp. ciceris fueron caracterizados<br />
racialmente en trabajos anteriores (Jiménez Díaz et al., 1989b; Jiménez<br />
Díaz et al., 1993a; Kelly et al., 1994; Capítulo I). Asimismo, se han incluido<br />
en el estudio cinco aislados <strong>de</strong> F. oxysporum obtenidos <strong>de</strong> raíces <strong>de</strong> plantas<br />
sanas <strong>de</strong> garbanzo y no patogénicos <strong>de</strong> éste (Tabla 12). Todos los aislados<br />
fúngicos utilizados proce<strong>de</strong>n <strong>de</strong> la colección <strong>de</strong>l Departamento <strong>de</strong><br />
Protección <strong>de</strong> Cultivos, Instituto <strong>de</strong> Agricultura Sostenible, CSIC,<br />
Córdoba, y se han mantenido almacenados como cultivos monoconídicos,<br />
en suelo estéril en tubos <strong>de</strong> ensayo, a 4ºC, en oscuridad. Los<br />
procedimientos para la obtención <strong>de</strong> cultivos activos <strong>de</strong> los aislados<br />
fúngicos, el manejo <strong>de</strong> éstos, y la obtención <strong>de</strong> su micelio para la<br />
extracción <strong>de</strong> ADN, han sido <strong>de</strong>scritos con anterioridad (Pág. 22, 26,<br />
Capítulo I; Pág. 52, Capítulo II).<br />
Extracción <strong>de</strong> ADN<br />
El ADN genómico <strong>de</strong> cada aislado se purificó a partir <strong>de</strong> micelio<br />
liofilizado y molido, según el método <strong>de</strong> Rae<strong>de</strong>r y Broda (1985) (Pág. 27-<br />
28, Capítulo I). Las muestras <strong>de</strong> ADN se diluyeron hasta una<br />
concentración <strong>de</strong> 25-50 ng/µl para la realización <strong>de</strong> las amplificaciones<br />
rep-PCR.<br />
126
Diseño <strong>de</strong> iniciadores<br />
Capítulo V<br />
Los iniciadores utilizados en la investigación se basaron en la secuencia <strong>de</strong>l<br />
clon bacteriano FocB10, que alberga el fragmento <strong>de</strong> 1,0 kb generado por<br />
el iniciador aleatorio OPF12 en análisis RAPD. Esta banda RAPD<br />
(‘Random Amplified Polymorphic DNA’) fue i<strong>de</strong>ntificada como asociada a<br />
la raza 2 <strong>de</strong> F. oxysporum f. sp. ciceris en trabajos anteriores en esta Tesis<br />
(Capítulos I y II). El análisis <strong>de</strong> la secuencia correspondiente a este<br />
fragmento mostró una alta similaridad con el transposón Fot1 i<strong>de</strong>ntificado<br />
en F. oxysporum f. sp. melonis (Leach & Currence) W. C. Sny<strong>de</strong>r & H. N.<br />
Hansen (Daboussi et al., 1992).<br />
Los dos iniciadores: Foc-TnB10fwd 5’-GGCGTTGGATAAGG-<br />
TTGCGATGGAGGTT-3’, y Foc-TnB10rev 5’-TCCGGGCATTTA-<br />
TCGTTTTGACTGGTA-3’, se diseñaron a partir <strong>de</strong> cada uno <strong>de</strong> los<br />
extremos <strong>de</strong> la secuencia <strong>de</strong>l fragmento RAPD mencionado, en<br />
orientación opuesta, <strong>de</strong> forma que los extremos 3’ estuvieran dirigidos<br />
hacia el exterior <strong>de</strong> la secuencia repetitiva. Los iniciadores se diseñaron<br />
mediante el programa PrimerSelect 3.11 <strong>de</strong>l paquete <strong>de</strong> ‘software DNA<br />
Star’ (Madison, WI, EEUU), y fueron sintetizados comercialmente por<br />
GENSET S. A. (Paris, Francia).<br />
Condiciones <strong>de</strong> amplificación rep-PCR<br />
Para los análisis rep-PCR se utilizó el sistema ‘Expand Long Template<br />
PCR System’ (Roche Diagnostics, Mannheim, Alemania), optimizado para<br />
realizar amplificaciones PCR <strong>de</strong> regiones genómicas largas. Las reacciones<br />
<strong>de</strong> amplificación se realizaron en un volumen total <strong>de</strong> 25 µl, que contenía<br />
0,4 µM <strong>de</strong> cada iniciador, 300 µM <strong>de</strong> cada dNTP, 2,5 µl <strong>de</strong> 10 x tampón S1<br />
suministrado por el distribuidor, 2U <strong>de</strong> Taq ADN Polimerasa (Roche<br />
Diagnostics, Mannheim, Alemania), 2,25 mM MgCl 2, y 50-100 ng <strong>de</strong> ADN<br />
fúngico.<br />
127
Capítulo V<br />
Tabla 12. Información geográfica y racial sobre los aislados <strong>de</strong> <strong>Fusarium</strong><br />
oxysporum f. sp. ciceris (Foc), <strong>de</strong> F. oxysporum (Fo) no patogénicos <strong>de</strong><br />
garbanzo, utilizados en el análisis rep-PCR<br />
128<br />
Aislado Origen geográfico a<br />
Raza<br />
patogénica b<br />
Foc-7802 España 0<br />
Foc-7952 España 0<br />
Foc-82108 España 0<br />
Foc-82113 España 0<br />
Foc-9018 PV1 España 0<br />
Foc-9032 España 0<br />
Foc-91108 I España 0<br />
Foc-T3 Túnez 0<br />
Foc-USA 3-1 JG62 EEUU 1B/C<br />
Foc-1987-W17 EEUU 1B/C<br />
Foc-9602 Túnez 1B/C<br />
Foc-7989 India 1A<br />
Foc-9027 PV1 c España 1A<br />
Foc-9168 Marruecos 1A<br />
Foc-9166 c Marruecos 1A<br />
Foc-8272 España 6<br />
Foc-9023 España 6<br />
Foc-9093 PV1 España 6<br />
Foc-9164 Marruecos 6<br />
Foc-9170 Marruecos 6<br />
Foc-Tonini EEUU 6
Capítulo V<br />
Tabla 12. (Continuación) Información geográfica y racial sobre los<br />
aislados <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris (Foc), <strong>de</strong> F. oxysporum (Fo) no<br />
patogénicos <strong>de</strong> garbanzo, utilizados en el análisis rep-PCR<br />
Aislado Origen geográfico a<br />
Raza<br />
patogénica b<br />
Foc-8012 España 5<br />
Foc-9035 España 5<br />
Foc-9094 JG62 España 5<br />
Foc-USA 1-1 JG62 EEUU 5<br />
Foc-USA W6-1 EEUU 5<br />
Foc-8605 India 2<br />
Foc-1992R2N India 2<br />
Foc-8606 India 3<br />
Foc-1992R3N India 3<br />
Foc-8607 India 4<br />
Foc-1992R4N India 4<br />
Fo-8250 España NP<br />
Fo-9081 España NP<br />
Fo-90101 España NP<br />
Fo-9169 Marruecos NP<br />
Fo-90105 España NP<br />
a Los aislados <strong>de</strong> EEUU (California), España, y Marruecos proce<strong>de</strong>n <strong>de</strong> la<br />
micoteca <strong>de</strong>l Departamento <strong>de</strong> Protección <strong>de</strong> Cultivos, Instituto <strong>de</strong> Agricultura<br />
Sostenible, CSIC, Córdoba, España. Los aislados <strong>de</strong> la India fueron<br />
proporcionados por el Dr. M. P. Haware, ICRISAT, Hy<strong>de</strong>rabad, India. Los<br />
aislados proce<strong>de</strong>ntes <strong>de</strong> Túnez fueron cedidos por el Dr. M. H. Halila, Institute<br />
Nationale <strong>de</strong> la Recherche Agronomique, Ariana, Túnez.<br />
b Raza <strong>de</strong>terminada mediante pruebas <strong>de</strong> patogenicidad en líneas <strong>de</strong> garbanzo<br />
diferenciadoras (Alcalá-Jiménez, 1995; Jiménez-Gasco et al., 2001; Capítulo I).<br />
NP=no patogénicos <strong>de</strong> garbanzo.<br />
c Aislados caracterizados como pertenecientes a la raza 6 <strong>de</strong> F. oxysporum f. sp.<br />
ciceris en pruebas <strong>de</strong> patogenicidad duplicadas (Alcalá Jiménez, 1995).<br />
129
Capítulo V<br />
Las amplificaciones se realizaron en el termociclador Perkin-Elmer<br />
2400 (Perkin-Elmer, Norwalk, CT). El ciclo <strong>de</strong> temperaturas consistió en<br />
2,5 min <strong>de</strong> <strong>de</strong>snaturalización inicial a 95ºC; cinco ciclos <strong>de</strong> 1 min <strong>de</strong><br />
<strong>de</strong>snaturalización a 94ºC, 1 min <strong>de</strong> complementación entre el ADN mol<strong>de</strong><br />
y el iniciador a 65ºC, y 10 min <strong>de</strong> polimerización a 68ºC; 30 ciclos <strong>de</strong> 30 s a<br />
94ºC, 1 min a 65ºC, y 10 min a 68ºC; y finalmente, 15 min <strong>de</strong> extensión a<br />
72ºC.<br />
Los productos <strong>de</strong> amplificación fueron separados mediante<br />
electroforesis en geles <strong>de</strong> agarosa al 1,5 % y 1,5 V·cm -1 , teñidos con<br />
bromuro <strong>de</strong> etidio, y visualizados bajo luz ultravioleta. Como marcador <strong>de</strong><br />
peso molecular se utilizó el ‘0,1-kb DNA lad<strong>de</strong>r XIV’ (Roche Diagnostics,<br />
Mannheim, Alemania) y ADN <strong>de</strong> fago λ digerido con la enzima PstI.<br />
Todas las reacciones <strong>de</strong> amplificación fueron repetidas al menos dos veces<br />
y siempre incluyeron controles negativos (sin ADN mol<strong>de</strong>).<br />
Análisis <strong>de</strong> datos<br />
Para <strong>de</strong>terminar la variabilidad genética entre los aislados <strong>de</strong> F. oxysporum f.<br />
sp. ciceris en el estudio se realizó una análisis <strong>de</strong> similaridad mediante el<br />
programa NTSYSpc2.0 (Rohlf, 1988), utilizando el ‘unweighted paired<br />
group method with arithmetic averages’ (UPGMA) que se basó en el<br />
coeficiente <strong>de</strong> similaridad <strong>de</strong> Jaccard (Sneath y Sokal, 1973). Para ello, se<br />
construyó una matriz binaria con la información obtenida <strong>de</strong> la<br />
amplificación rep-PCR <strong>de</strong> ADN <strong>de</strong> 32 aislados F. oxysporum f. sp. ciceris,<br />
representativos <strong>de</strong> las distintas razas patogénicas i<strong>de</strong>ntificadas y <strong>de</strong> un<br />
amplio origen geográfico <strong>de</strong>l patógeno; así como <strong>de</strong> cinco aislados <strong>de</strong> F.<br />
oxysporum no patogénicos <strong>de</strong> garbanzo. La presencia o ausencia <strong>de</strong> los<br />
fragmentos <strong>de</strong> ADN amplificados se i<strong>de</strong>ntificaron como ‘1’ y ‘0’,<br />
respectivamente.<br />
A<strong>de</strong>más <strong>de</strong> lo anterior, también se analizó mediante UPGMA la<br />
información generada por la hibridación <strong>de</strong> ADN genómico <strong>de</strong> F.<br />
oxysporum f. sp. ciceris con la sonda <strong>de</strong> ADN FocB10 (a partir <strong>de</strong> cuya<br />
130
Capítulo V<br />
secuencia se diseñaron los iniciadores utilizados para realizar los análisis <strong>de</strong><br />
rep-PCR). Para ello, se contabilizó la presencia o ausencia <strong>de</strong> los<br />
fragmentos homólogos en una matriz binaria, tal y como se ha <strong>de</strong>scrito<br />
para el análisis rep-PCR.<br />
Resultados<br />
Variabilidad en F. oxysporum f. sp. ciceris <strong>de</strong>terminada mediante<br />
análisis rep-PCR<br />
En este trabajo hemos utilizado un método basado en la PCR, para la<br />
generar patrones <strong>de</strong> amplificación tan informativos como los ‘fingerprints’<br />
<strong>de</strong> ADN genómico que se producen mediante su hibridación con sondas<br />
<strong>de</strong> ADN repetitivo. Para ello, nos hemos basado en un elemento <strong>de</strong> ADN<br />
repetitivo (transposón) obtenido <strong>de</strong>l propio genoma <strong>de</strong> F. oxysporum f. sp.<br />
ciceris, que presentó gran similaridad con el transposón Fot1 (Daboussi et<br />
al., 1992). Para la investigación se diseñaron dos iniciadores a partir <strong>de</strong> los<br />
extremos <strong>de</strong> la secuencia <strong>de</strong> dicho elemento repetitivo dirigidos hacia el<br />
exterior <strong>de</strong>l elemento, <strong>de</strong> forma que permitieran la amplificación entre<br />
copias <strong>de</strong>l transposón mediante PCR. Para el análisis se utilizaron<br />
condiciones <strong>de</strong> PCR que favorecieron la amplificación <strong>de</strong> fragmentos<br />
largos <strong>de</strong> ADN.<br />
El tamaño <strong>de</strong> los fragmentos <strong>de</strong> ADN amplificados varió entre 0,5 y<br />
más <strong>de</strong> 12 kb, aunque las bandas más reproducibles fueron las <strong>de</strong> peso<br />
molecular inferior a 12 kb. En los análisis, los perfiles <strong>de</strong> amplificación<br />
fueron reproducibles utilizando diferentes enzimas Taq Polimerasa,<br />
diferentes termocicladores, y cuando se realizaron en diferentes<br />
laboratorios (IAS, Córdoba; ‘Department of Plant Pathology’, Cornell<br />
University, Ithaca, NY, EEUU). Sin embargo, la amplificación <strong>de</strong><br />
fragmentos largos <strong>de</strong> ADN requiere condiciones <strong>de</strong> PCR muy eficientes, y<br />
el método hubo <strong>de</strong> ser optimizado antes <strong>de</strong> contabilizar las bandas <strong>de</strong><br />
ADN para proce<strong>de</strong>r a su análisis.<br />
131
Capítulo V<br />
Los análisis <strong>de</strong> ADN <strong>de</strong> aislados <strong>de</strong> F. oxysporum f. sp. ciceris generaron<br />
perfiles <strong>de</strong> amplificación que los diferenciaron <strong>de</strong> los <strong>de</strong> F. oxysporum no<br />
patogénicos <strong>de</strong> garbanzo. A<strong>de</strong>más, mediante el patrón <strong>de</strong> amplificación<br />
rep-PCR también se pudo diferenciar a los aislados <strong>de</strong> F. oxysporum f. sp.<br />
ciceris pertenecientes a una raza <strong>de</strong> los aislados pertenecientes a otras razas<br />
(Fig. 13). Mientras que el número <strong>de</strong> bandas amplificadas en aislados <strong>de</strong> F.<br />
oxysporum f. sp. ciceris varió entre 10 y 20, los aislados <strong>de</strong> F. oxysporum no<br />
patogénicos <strong>de</strong> garbanzo estudiados produjeron un número <strong>de</strong> bandas<br />
significativamente inferior (entre 2 y 5).<br />
Análisis <strong>de</strong> los datos rep-PCR<br />
La información generada mediante amplificaciones rep-PCR se utilizó para<br />
realizar un análisis <strong>de</strong> agrupación (‘cluster’) <strong>de</strong> los aislados <strong>de</strong> F. oxysporum<br />
f. sp. ciceris según la similaridad que presentaron en los perfiles producidos.<br />
Se contabilizaron un total <strong>de</strong> 33 fragmentos <strong>de</strong> ADN. El análisis UPGMA<br />
<strong>de</strong> las bandas amplificadas mediante rep-PCR distinguió a los aislados <strong>de</strong><br />
F. oxysporum f. sp. ciceris <strong>de</strong> los <strong>de</strong> F. oxysporum no patogénicos <strong>de</strong> garbanzo<br />
(Fig. 14). A<strong>de</strong>más, los aislados F. oxysporum f. sp. ciceris se distribuyeron en<br />
dos grupos principales, que estuvieron altamente correlacionados con la<br />
capacidad <strong>de</strong> los aislados <strong>de</strong> inducir el síndrome <strong>de</strong> Amarillez (patotipo <strong>de</strong><br />
Amarillez) o <strong>de</strong> Marchitez (patotipo <strong>de</strong> Marchitez). Estos ‘clusters’<br />
compartieron aproximadamente un 60 y un 50% <strong>de</strong> similaridad en sus<br />
perfiles <strong>de</strong> amplificación rep-PCR, respectivamente.<br />
A su vez, <strong>de</strong>ntro <strong>de</strong>l grupo correspondiente a cada patotipo <strong>de</strong> F.<br />
oxysporum f. sp. ciceris se distinguieron varias agrupaciones <strong>de</strong> aislados que<br />
se correlacionaron con su naturaleza racial. Así, <strong>de</strong>ntro <strong>de</strong>l grupo<br />
correspondiente al patotipo <strong>de</strong> Amarillez se i<strong>de</strong>ntificaron dos ‘subclusters’<br />
<strong>de</strong> aislados que correspondieron respectivamente a las razas 0 y 1B/C.<br />
132
1<br />
5,<br />
2,<br />
1,<br />
1,<br />
1,<br />
0,<br />
M 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18<br />
Capítulo V<br />
Figura 13. Amplificación rep-PCR <strong>de</strong> aislados <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris.<br />
La amplificación <strong>de</strong>l ADN existente entre copias <strong>de</strong>l elemento repetitivo FocB10<br />
genera patrones como el mostrado, que están altamente correlacionados con la<br />
naturaleza patotípica y racial <strong>de</strong> los aislados <strong>de</strong>l patógeno. Los números a la<br />
izquierda indican el peso molecular aproximado (kb) <strong>de</strong> las bandas <strong>de</strong><br />
amplificación. Carriles: M, marcador <strong>de</strong> peso molecular; 1-3, 9, 11, aislados <strong>de</strong><br />
la raza 0 Foc–9032, –82113, –7952, –T3, –9018 PV1; 4, 6, aislados <strong>de</strong> la raza<br />
1A Foc–9168, –9027 PV1; 5, 8, aislados <strong>de</strong> la raza 6 Foc–9170, –8272; 10,<br />
aislado <strong>de</strong> la raza 1B/C Foc–9602; 12, 13, aislados <strong>de</strong> la raza 5 Foc–9035, –<br />
USA W6-1; 14-16, aislados <strong>de</strong> cada una <strong>de</strong> las razas 2, 3 y 4 Foc–1992R2N, –<br />
1992R3N, –1992R4N; 7, 17, 18, aislados <strong>de</strong> F. oxysporum no patogénicos <strong>de</strong><br />
garbanzo Fo–8250, –9081, y –9169, respectivamente.<br />
133
Capítulo V<br />
De igual manera, <strong>de</strong>ntro <strong>de</strong>l ‘cluster’ correspondiente al patotipo <strong>de</strong><br />
Marchitez se distinguieron tres agrupaciones <strong>de</strong> aislados. Los aislados <strong>de</strong><br />
las razas 1A y 6 constituyeron un grupo (65% <strong>de</strong> similaridad) claramente<br />
diferenciado <strong>de</strong>l resto <strong>de</strong> aislados causantes <strong>de</strong> dicho síndrome. Los<br />
aislados pertenecientes a la raza 5 formaron un grupo (90% <strong>de</strong> similaridad)<br />
distinguible <strong>de</strong>l resto, <strong>de</strong>ntro <strong>de</strong> los aislados <strong>de</strong>l patotipo <strong>de</strong> Marchitez (Fig.<br />
14). Finalmente, los aislados <strong>de</strong> las razas 2, 3, 4, proce<strong>de</strong>ntes <strong>de</strong> la India, se<br />
agruparon en un tercer ‘cluster’.<br />
Análisis comparativo entre los resultados <strong>de</strong> las hibridaciones ‘Southern’ y<br />
el análisis rep-PCR<br />
Para confirmar que la información generada mediante la amplificación rep-<br />
PCR entre copias <strong>de</strong> elementos repetitivos, es equivalente a la generada<br />
cuando utilizamos dicho elemento como sonda para hibridación <strong>de</strong> ADN<br />
genómico, se compararon los análisis <strong>de</strong> similaridad UPGMA <strong>de</strong> los<br />
resultados obtenidos mediante ambos métodos.<br />
La distribución <strong>de</strong> los aislados fúngicos según su similaridad en el<br />
análisis <strong>de</strong> los datos generados mediante rep-PCR, fue muy similar a la<br />
obtenida <strong>de</strong> ‘fingerprints’ <strong>de</strong> ADN generados mediante la hibridación <strong>de</strong><br />
ADN genómico <strong>de</strong> F. oxysporum f. sp. ciceris con el elemento repetitivo<br />
FocB10. Así, los ‘clusters’ formados en dichos análisis estuvieron<br />
altamente correlacionados con las características patogénicas <strong>de</strong> los<br />
aislados.<br />
Los aislados <strong>de</strong> F. oxysporum f. sp. ciceris, que compartieron un 25% <strong>de</strong><br />
similaridad, se diferenciaron claramente <strong>de</strong> los aislados <strong>de</strong> F. oxysporum no<br />
patogénicos <strong>de</strong> garbanzo. Dentro <strong>de</strong>l grupo <strong>de</strong> aislados <strong>de</strong> F. oxysporum f.<br />
sp. ciceris, los aislados se distribuyeron en dos ‘clusters’ principales según su<br />
134
0.00 0.25 0.50 0.75 1.00<br />
Capítulo V<br />
Raza<br />
patogénica<br />
7802 0<br />
91108I 0<br />
82108 0<br />
7952 0<br />
82113 0<br />
9032 0<br />
T3<br />
0<br />
9018PV1 0<br />
USA3-1JG62 1B/C<br />
USAW17 1B/C<br />
9602 1B/C<br />
8605 2<br />
1992R2N 2<br />
8606 3<br />
1992R3N 3<br />
1992R4N 4<br />
8607 4<br />
8012 5<br />
USA14201JG62 5<br />
9094JG62 5<br />
9035 5<br />
USAW6-1 5<br />
7989 1A<br />
9168 1A<br />
8272 6<br />
9023 6<br />
9093PV1 6<br />
9027PV1 1A<br />
9166 1A<br />
Tonini 6<br />
9164 6<br />
9170 6<br />
90101 NP<br />
90105 NP<br />
9081 NP<br />
9169<br />
NP<br />
8250 NP<br />
Figura 14. Dendrograma UPGMA generado a partir <strong>de</strong> la información obtenida<br />
mediante la amplificación rep-PCR <strong>de</strong> ADN <strong>de</strong> aislados <strong>de</strong> <strong>Fusarium</strong> oxysporum f.<br />
sp. ciceris (razas 0, 1A, 1B/C, 2 a 6) y <strong>de</strong> F. oxysporum no patogénicos <strong>de</strong> garbanzo<br />
(NP). La amplificación rep-PCR se produce entre copias <strong>de</strong>l elemento repetitivo<br />
FocB10, lo cual genera ‘fingerprints’ <strong>de</strong> ADN. La escala inferior correspon<strong>de</strong> a la<br />
similaridad entre aislados basada en el coeficiente <strong>de</strong> Jaccard.<br />
135<br />
Amarillez Marchitez
Capítulo V<br />
asignación a patotipo (Amarillez y Marchitez). A su vez, los aislados <strong>de</strong><br />
cada patotipo se distribuyeron en grupos que correspondieron a las razas<br />
patogénicas i<strong>de</strong>ntificadas en F. oxysporum f. sp. ciceris (Fig. 15)<br />
Dentro <strong>de</strong>l ‘cluster’ que incluyó los aislados <strong>de</strong> Amarillez (45% se<br />
similaridad) se distinguieron dos subgrupos que correspondieron a los<br />
aislados <strong>de</strong> las razas 0 y 1B/C; mientras el grupo <strong>de</strong> aislados <strong>de</strong>l patotipo<br />
<strong>de</strong> Marchitez (50% <strong>de</strong> similaridad) se subdividió en tres agrupaciones que<br />
correspondieron a los aislados <strong>de</strong> las razas 1A/6, la raza 5, y las razas 2, 3,<br />
y 4.<br />
Esta agrupación <strong>de</strong> aislados es notablemente similar a obtenida<br />
mediante el análisis <strong>de</strong> resultados obtenidos en amplificaciones rep-PCR<br />
(Fig. 14, 15). De igual manera, en ambos análisis se han <strong>de</strong>tectado<br />
aproximadamente los mismos porcentajes <strong>de</strong> similaridad entre grupos o<br />
subgrupos (Fig. 14, 15).<br />
La única diferencia resaltable entre los <strong>de</strong>ndrogramas UPGMA<br />
generados mediante los diferentes métodos <strong>de</strong> análisis concierne a la<br />
agrupación <strong>de</strong> los aislados <strong>de</strong> la raza 5 <strong>de</strong> F. oxysporum f. sp. ciceris. Así,<br />
mientras que el análisis <strong>de</strong> los datos generados mediante rep-PCR mostró<br />
que los aislados <strong>de</strong> la raza 5 fueron más similares a las razas 2, 3, y 4,<br />
<strong>de</strong>scritas únicamente en la India (Fig. 14); el análisis <strong>de</strong> los datos generados<br />
mediante la hibridación con el elemento FocB10 situó a los aislados <strong>de</strong> la<br />
raza 5 igualmente distantes <strong>de</strong> los <strong>de</strong> las razas 2, 3, y 4, que <strong>de</strong> los aislados<br />
<strong>de</strong> las razas 1A y 6 que junto con aquéllos constituyen el patotipo <strong>de</strong><br />
Marchitez (Fig. 15).<br />
136
0.00 0.25 0.50 0.75 1.00<br />
Capítulo V<br />
Raza<br />
patogénica<br />
9018PV1 0<br />
9018JG62 0<br />
T3 0<br />
82108 0<br />
7802 0<br />
8207 0<br />
9032 0<br />
7952 0<br />
9605 0<br />
82113 0<br />
91108 0<br />
91114 0<br />
1978W17 1B/C<br />
USA3-1JG62 1B/C<br />
9602 1B/C<br />
7989 1A<br />
9168 1A<br />
9166 1A<br />
9165 1A<br />
9170 6<br />
Tonini 6<br />
9164 6<br />
9023 6<br />
9027PV1 1A<br />
8272 6<br />
8605 2<br />
1992R2N 2<br />
8606 3<br />
1992R3N 3<br />
1992R4N 4<br />
8607 4<br />
8012 5<br />
USA1-1JG62 5<br />
9035 5<br />
USAW6-1 5<br />
9094JG62 5<br />
90101 NP<br />
8250 NP<br />
9009 NP<br />
Figura 15. Dendrograma UPGMA generado a partir <strong>de</strong> la información <strong>de</strong><br />
‘fingerprints’ obtenidos mediante hibridaciones ‘Southern’ <strong>de</strong> ADN genómico <strong>de</strong><br />
aislados <strong>de</strong> <strong>Fusarium</strong> oxysporum f. sp. ciceris (razas 0, 1A, 1B/C, 2 a 6) y <strong>de</strong> F.<br />
oxysporum no patogénicos <strong>de</strong> garbanzo (NP), con el elemento repetitivo FocB10.<br />
La escala inferior correspon<strong>de</strong> a la similaridad entre aislados basada en el<br />
coeficiente <strong>de</strong> Jaccard.<br />
137<br />
Amarillez Marchitez
Capítulo V<br />
Discusión<br />
El estudio <strong>de</strong> ‘fingerprints’ <strong>de</strong> ADN genómico generados mediante<br />
hibridaciones con elementos <strong>de</strong> ADN repetitivo, ha mostrado ser una<br />
herramienta importante para analizar la estructura genética <strong>de</strong> las razas<br />
patogénicas <strong>de</strong> F. oxysporum f. sp. ciceris, así como para compren<strong>de</strong>r la<br />
relación genética entre ellas (Capítulo IV). Sin embargo, el prolongado<br />
tiempo <strong>de</strong> análisis y amplitud <strong>de</strong> recursos que lleva implícita la utilización<br />
<strong>de</strong> dicha metodología <strong>de</strong> análisis, hacen que sea difícil su aplicación<br />
rutinaria al estudio <strong>de</strong> un elevado número <strong>de</strong> aislados componentes <strong>de</strong><br />
poblaciones <strong>de</strong> este patógeno, que es necesario para realizar inferencias<br />
sobre su diversidad genética.<br />
Nuestro objetivo en este trabajo ha sido <strong>de</strong>sarrollar un método que,<br />
basado en el análisis PCR, posibilite la i<strong>de</strong>ntificación <strong>de</strong>l mismo nivel <strong>de</strong><br />
polimorfismos que proporciona la hibridación <strong>de</strong> ADN genómico con<br />
elementos repetitivos contenidos en él. Para ello, hemos diseñado una<br />
pareja <strong>de</strong> iniciadores que amplifican las regiones <strong>de</strong> ADN existentes entre<br />
las diferentes copias <strong>de</strong> un elemento repetitivo, que se encuentran<br />
dispersas en el genoma <strong>de</strong> F. oxysporum f. sp. ciceris. Este elemento<br />
repetitivo fue i<strong>de</strong>ntificado en dicho genoma, y presenta aproximadamente<br />
un 80% <strong>de</strong> similaridad al transposón Fot1 i<strong>de</strong>ntificado en F. oxysporum f. sp.<br />
melonis (Daboussi et al., 1992).<br />
Mediante el análisis PCR en condiciones que favorecen la amplificación<br />
<strong>de</strong> regiones largas <strong>de</strong> ADN, utilizando los referidos iniciadores orientados<br />
hacia el exterior <strong>de</strong>l elemento repetitivo, se generaron patrones <strong>de</strong><br />
amplificación con 10 a 20 bandas, que resultaron válidos para discriminar<br />
entre razas <strong>de</strong> F. oxysporum f. sp. ciceris. Aunque el potencial <strong>de</strong><br />
discriminación entre aislados <strong>de</strong>l patógeno que ofrece la hibridación <strong>de</strong><br />
ADN genómico con el elemento repetitivo fue mayor que el que<br />
proporciona el análisis rep-PCR, es conveniente consi<strong>de</strong>rar algunas<br />
limitaciones técnicas en la valoración comparativa <strong>de</strong> las dos metodologías.<br />
Así, mientras el primer método requiere mucho tiempo y gran cantidad <strong>de</strong><br />
ADN puro, la preparación tediosa <strong>de</strong> la sonda <strong>de</strong> hibridación, y la<br />
138
Capítulo V<br />
realización <strong>de</strong> las hibridaciones ‘Southern’; el método rep-PCR sólo<br />
requiere minipreparaciones <strong>de</strong> ADN y la realización <strong>de</strong> los análisis PCR.<br />
El análisis <strong>de</strong> similaridad <strong>de</strong> los patrones <strong>de</strong> amplificación<br />
proporcionados por el análisis rep-PCR, y su comparación con los<br />
obtenidos mediante hibridaciones ‘Southern’, ha puesto <strong>de</strong> manifiesto que<br />
la información generada por ambos métodos es equivalente. Así, el análisis<br />
UPGMA <strong>de</strong> los datos generados mediante el análisis rep-PCR distribuye a<br />
los aislados <strong>de</strong> F. oxysporum f. sp. ciceris en agrupaciones altamente<br />
correlacionadas tanto con el tipo <strong>de</strong> síndrome patogénico que inducen en<br />
la planta huésped (i. e., patotipos <strong>de</strong> Amarillez y <strong>de</strong> Marchitez), como con<br />
su patogenicidad diferencial sobre líneas diferenciadoras <strong>de</strong> garbanzo (i. e.,<br />
razas patogénicas). Esta distribución <strong>de</strong> aislados en grupos <strong>de</strong> similaridad,<br />
así como los valores <strong>de</strong> similaridad entre dichos grupos, son comparables a<br />
los que se obtuvieron en el análisis UPGMA <strong>de</strong> los patrones generados<br />
mediante las hibridaciones con la sonda FocB10.<br />
La única diferencia digna <strong>de</strong> ser <strong>de</strong>stacada entre el resultado <strong>de</strong>l análisis<br />
<strong>de</strong> ‘clusters’ <strong>de</strong> la información generada por el análisis rep-PCR respecto<br />
<strong>de</strong>l correspondiente al <strong>de</strong> la generada por la hibridaciones <strong>de</strong> ADN, fue la<br />
agrupación <strong>de</strong> los aislados pertenecientes a la raza 5 <strong>de</strong> F. oxysporum f. sp.<br />
ciceris. Mientras que el análisis UPGMA <strong>de</strong> los perfiles <strong>de</strong> amplificación<br />
rep-PCR indicó que los aislados <strong>de</strong> la raza 5 son genéticamente más<br />
similares a las razas 2, 3, y 4 <strong>de</strong>scritas solamente en la India, que a las razas<br />
1A y 6 <strong>de</strong>scritas en varias áreas <strong>de</strong> cultivo; el análisis <strong>de</strong> los datos <strong>de</strong><br />
hibridación con FocB10 indicó que los aislados <strong>de</strong> dicha raza 5 presentan<br />
la misma similaridad a todas las razas <strong>de</strong> F. oxysporum f. sp. ciceris que<br />
pertenecen al patotipo <strong>de</strong> Marchitez. Sin embargo, el análisis conjunto <strong>de</strong><br />
los resultados generados por la hibridación con la sonda FocB10 y con<br />
otras dos secuencias <strong>de</strong> naturaleza repetitiva, mostró la misma agrupación<br />
<strong>de</strong> razas que la obtenida mediante el análisis <strong>de</strong> los perfiles <strong>de</strong><br />
amplificación rep-PCR <strong>de</strong>sarrollado en este Capítulo. De igual manera, el<br />
análisis <strong>de</strong> máxima parsimonia <strong>de</strong> los mismos datos indicó un estrecha<br />
relación filogenética entre el grupo <strong>de</strong> razas 2, 3, y 4, proce<strong>de</strong>ntes <strong>de</strong> la<br />
India, y la raza 5 <strong>de</strong> F. oxysporum f. sp. ciceris.<br />
139
Capítulo V<br />
La metodología <strong>de</strong> análisis rep-PCR basada en la utilización como<br />
iniciadores <strong>de</strong> elementos repetitivos endógenos al organismo en estudio,<br />
ha sido aplicada por otros autores anteriormente con éxito. Los elementos<br />
repetitivos, y en particular el elemento MGR586, han sido utilizados<br />
extensamente para la caracterización <strong>de</strong> las poblaciones <strong>de</strong> Magnaporthe<br />
grisea (T. T. Hebert) Yaegashi & U<strong>de</strong>gawa; <strong>de</strong> hecho, George et al. (1998)<br />
utilizaron la amplificación entre copias <strong>de</strong> estos elementos para inferir la<br />
relación genética entre los aislados <strong>de</strong> dicho patógeno.<br />
En nuestra investigación, a<strong>de</strong>más <strong>de</strong> FocB10 hemos i<strong>de</strong>ntificado en el<br />
genoma <strong>de</strong> F. oxysporum f. sp. ciceris otras dos secuencias <strong>de</strong> ADN que<br />
poseen homología con un alto número <strong>de</strong> fragmentos generados mediante<br />
su digestión con EcoRI, y cuyas secuencias han mostrado similaridad con<br />
elementos transponibles ya <strong>de</strong>scritos en otros hongos. Es presumible que<br />
la utilización <strong>de</strong> estas secuencias para diseñar nuevos iniciadores podrá<br />
generar una mayor cantidad <strong>de</strong> ‘fingerprints’ <strong>de</strong> ADN mediante análisis<br />
rep-PCR; y a<strong>de</strong>más, que los nuevos iniciadores así generados podrán ser<br />
utilizados combinadamente para realizar amplificaciones entre los<br />
diferentes elementos repetitivos.<br />
En consecuencia, los resultados <strong>de</strong> las investigaciones incluidas en este<br />
Capítulo han puesto <strong>de</strong> manifiesto la utilidad <strong>de</strong> la metodología rep-PCR<br />
como herramienta <strong>de</strong> análisis para generar ‘fingerprints’ <strong>de</strong> ADN válidos<br />
para la caracterización y estudio filogenético <strong>de</strong> razas <strong>de</strong> F. oxysporum f. sp.<br />
ciceris, que a<strong>de</strong>más supone la disponibilidad <strong>de</strong> una estrategia <strong>de</strong> estudio<br />
alternativa a la convencional basada en la realización <strong>de</strong> hibridaciones <strong>de</strong><br />
ADN genómico con sondas <strong>de</strong> interés. El método <strong>de</strong>scrito en este trabajo<br />
parece especialmente a<strong>de</strong>cuado para el estudio <strong>de</strong> diversidad en las<br />
poblaciones <strong>de</strong> F. oxysporum f. sp. ciceris, para lo cual es necesaria la<br />
caracterización <strong>de</strong> un elevado número <strong>de</strong> aislados <strong>de</strong>l patógeno<br />
representativos <strong>de</strong> la variabilidad <strong>de</strong> dichas poblaciones.<br />
140
DISCUSIÓN GENERAL<br />
Discusión General<br />
F. oxysporum f. sp. ciceris (Padwick) Matuo & K. Sato es paradigmática entre<br />
las formae speciales <strong>de</strong> F. oxysporum en términos <strong>de</strong> diversidad patogénica. Así,<br />
los componentes <strong>de</strong> las poblaciones naturales <strong>de</strong> este hongo anamórfico<br />
<strong>de</strong> suelo pue<strong>de</strong>n ser diferenciados en los patotipos <strong>de</strong> Amarillez y<br />
Marchitez, por el síndrome que inducen en cultivares <strong>de</strong> garbanzo<br />
susceptibles (Trapero Casas, 1983; Trapero Casas y Jiménez Díaz, 1985), y<br />
en razas patogénicas (0, 1A, 1B/C, 2, 3, 4, 5, y 6) según la reacción a la<br />
infección <strong>de</strong> líneas <strong>de</strong> garbanzo diferenciadoras (Haware y Nene, 1982b;<br />
Jiménez Díaz et al., 1989b; 1993a). Esta diversidad patogénica tiene<br />
relevancia respecto <strong>de</strong>l uso eficiente <strong>de</strong> cultivares <strong>de</strong> garbanzo resistentes a<br />
la enfermedad y su <strong>de</strong>sarrollo mediante mejora genética, que es la<br />
estrategia <strong>de</strong> control <strong>de</strong> la Fusariosis Vascular <strong>de</strong>l garbanzo más práctica y<br />
económicamente eficiente.<br />
Esta Tesis Doctoral se ha centrado en dos temas principales que<br />
guardan relación directa con dicho uso eficiente y <strong>de</strong>sarrollo <strong>de</strong> resistencia<br />
huésped: (1) la i<strong>de</strong>ntificación molecular específica <strong>de</strong> F. oxysporum f. sp.<br />
ciceris y <strong>de</strong> sus razas patogénicas; y (2) el estudio <strong>de</strong> su variabilidad genética,<br />
así como <strong>de</strong> la posible relación evolutiva entre dichas razas. La<br />
disponibilidad <strong>de</strong> una metodología <strong>de</strong> diagnóstico que permita la<br />
i<strong>de</strong>ntificación consistente, rápida y precisa <strong>de</strong> las razas patogénicas <strong>de</strong> F.<br />
oxysporum f. sp. ciceris, facilitaría la caracterización <strong>de</strong> la estructura <strong>de</strong><br />
virulencia <strong>de</strong>l patógeno en áreas extensas <strong>de</strong> cultivo, que es requisito<br />
imprescindible para la a<strong>de</strong>cuada elección y disposición en tiempo y espacio<br />
<strong>de</strong> la resistencia disponible.<br />
Para ello, hemos utilizado herramientas experimentales basadas en<br />
técnicas <strong>de</strong> biología molecular disponibles, que nos ha permitido<br />
<strong>de</strong>sarrollar una metodología eficiente para i<strong>de</strong>ntificar <strong>de</strong> forma rápida,<br />
141
Discusión General<br />
precisa y consistente a los aislados <strong>de</strong> F. oxysporum f. sp. ciceris y <strong>de</strong> las razas<br />
patogénicas 0, 1A, 5, y 6, que son prevalentes en la Cuenca Mediterránea.<br />
De igual manera, este tipo <strong>de</strong> herramientas experimentales nos ha<br />
permitido estudiar la diversidad genética existente en las poblaciones <strong>de</strong> F.<br />
oxysporum f. sp. ciceris, y el nivel <strong>de</strong> similaridad entre aislados <strong>de</strong> una misma<br />
raza; así como profundizar en el conocimiento <strong>de</strong> la posible evolución <strong>de</strong><br />
este patógeno.<br />
Nuestra estrategia experimental para <strong>de</strong>sarrollar un método <strong>de</strong><br />
i<strong>de</strong>ntificación <strong>de</strong> F. oxysporum f. sp. ciceris y discriminación entre sus razas<br />
patogénicas, se ha basado en la asociación inicial <strong>de</strong> polimorfismos <strong>de</strong><br />
ADN amplificados mediante RAPD-PCR (‘Random Amplified<br />
Polymorphic DNA-Polymerase Chain Reaction’) (Williams et al., 1990) a<br />
cada una <strong>de</strong> las razas <strong>de</strong> F. oxysporum f. sp. ciceris. En el <strong>de</strong>sarrollo <strong>de</strong> dicha<br />
estrategia hemos i<strong>de</strong>ntificado marcadores RAPD que distinguen a F.<br />
oxysporum f. sp. ciceris y a cada una <strong>de</strong> las razas 0, 1B/C, 5, y 6; cuya<br />
fiabilidad y reproducibilidad han sido contrastadas en ‘ensayos ciegos’ <strong>de</strong><br />
naturaleza biológica y molecular.<br />
Posteriormente, las secuencias <strong>de</strong> los fragmentos RAPD asociados a F.<br />
oxysporum f. sp. ciceris y a las razas patogénicas referidas sirvió para diseñar<br />
iniciadores basados en el genoma <strong>de</strong>l patógeno para el análisis PCR<br />
específica. Esta estrategia ha supuesto un gran éxito en nuestro trabajo, ya<br />
que hemos diseñado iniciadores para análisis PCR específica que permiten<br />
distinguir inequívocamente a aislados <strong>de</strong> F. oxysporum f. sp. ciceris <strong>de</strong>: (a)<br />
aislados <strong>de</strong> F. oxysporum no patogénicos <strong>de</strong> garbanzo; (b) aislados<br />
pertenecientes a otras formae speciales <strong>de</strong> F. oxysporum; y (c) otras especies <strong>de</strong><br />
<strong>Fusarium</strong>. A<strong>de</strong>más, mediante análisis PCR específica utilizando iniciadores<br />
‘ad hoc’ ha sido posible diferenciar entre sí a aislados <strong>de</strong> las razas 0, 1A, 5,<br />
y 6 <strong>de</strong> F. oxysporum f. sp. ciceris, así como a éstos <strong>de</strong> aislados pertenecientes<br />
a otras razas <strong>de</strong>l patógeno.<br />
En los últimos años, numerosos trabajos han <strong>de</strong>mostrado la utilidad <strong>de</strong><br />
la técnica RAPD para relacionar marcadores moleculares y razas<br />
patogénicas <strong>de</strong> formae speciales <strong>de</strong> F. oxysporum (Assigbetse et al., 1994;<br />
Grajal-Martín et al., 1993; Manulis et al., 1994; Migheli et al., 1998). Sin<br />
embargo, son limitados los trabajos en que los marcadores RAPD<br />
142
Discusión General<br />
i<strong>de</strong>ntificados han sido subsiguientemente <strong>de</strong>sarrollados en SCARs<br />
(‘Sequence Characterized Amplified Regions’), y utilizados para el<br />
posterior <strong>de</strong>sarrollo <strong>de</strong> iniciadores para análisis PCR específica. La<br />
robustez inherente <strong>de</strong>l análisis PCR específica comparado con la<br />
sensibilidad <strong>de</strong> la técnica RAPD a factores experimentales, hace que la<br />
metodología que hemos <strong>de</strong>sarrollado en esta investigación suponga una<br />
mejora para el análisis <strong>de</strong> la estructura <strong>de</strong> virulencia <strong>de</strong> F. oxysporum f. sp.<br />
ciceris; alternativa a la i<strong>de</strong>ntificación tradicional <strong>de</strong> razas <strong>de</strong>l patógeno<br />
mediante pruebas <strong>de</strong> patogenicidad, que es costosa en tiempo, espacio y<br />
recursos, y pue<strong>de</strong> ser influida por la variabilidad inherente al sistema<br />
experimental.<br />
A<strong>de</strong>más, ligeras modificaciones en la técnica <strong>de</strong> análisis pue<strong>de</strong> permitir<br />
la aplicación <strong>de</strong> la metodología que hemos <strong>de</strong>sarrollado, para la <strong>de</strong>tección<br />
<strong>de</strong>l patógeno y sus razas patogénicas utilizando ADN total extraído <strong>de</strong><br />
otros substratos distintos <strong>de</strong>l micelio puro <strong>de</strong>l hongo, como tejido vegetal<br />
infectado y suelo infestado. Anteriores trabajos en nuestro laboratorio<br />
<strong>de</strong>mostraron esta posibilidad, utilizando iniciadores para la <strong>de</strong>tección <strong>de</strong>l<br />
patotipo <strong>de</strong> Marchitez <strong>de</strong> F. oxysporum f. sp. ciceris (García Pedrajas et al.,<br />
1999; Kelly et al., 1998). Disponer <strong>de</strong> un método que permita la <strong>de</strong>tección<br />
rápida y temprana <strong>de</strong>l patógeno en semillas <strong>de</strong> garbanzo pue<strong>de</strong> ser <strong>de</strong><br />
utilidad para evitar la dispersión <strong>de</strong> F. oxysporum f. sp. ciceris y <strong>de</strong> sus razas<br />
patogénicas a zonas <strong>de</strong> cultivo libres <strong>de</strong> ella. Similarmente, la aplicación <strong>de</strong><br />
la nueva metodología al diagnóstico en suelo antes <strong>de</strong> la siembra permitirá<br />
tomar <strong>de</strong>cisiones respecto <strong>de</strong> la a<strong>de</strong>cuación <strong>de</strong> cultivares <strong>de</strong> garbanzo <strong>de</strong><br />
interés agronómico y comercial, en razón <strong>de</strong> las razas <strong>de</strong>l patógeno<br />
existentes ellos. Finalmente, la utilización <strong>de</strong> dicha metodología diagnóstica<br />
pue<strong>de</strong> ayudar a <strong>de</strong>tectar cambios en la estructura racial que puedan tener<br />
lugar en las poblaciones <strong>de</strong> F. oxysporum f. sp. ciceris, como consecuencia <strong>de</strong><br />
la utilización <strong>de</strong> cultivares resistentes o <strong>de</strong> modificaciones ambientales en<br />
el cultivo.<br />
La diversidad fenotípica que observamos en F. oxysporum f. sp. ciceris, y<br />
su amplia distribución geográfica, planteó la posibilidad <strong>de</strong> que este taxón<br />
fuese <strong>de</strong> origen polifilético. Estudios basados en genealogías génicas han<br />
<strong>de</strong>mostrado que ciertas formae speciales <strong>de</strong> F. oxysporum pue<strong>de</strong>n tener<br />
143
Discusión General<br />
orígenes múltiples e in<strong>de</strong>pendientes (i. e., son <strong>de</strong> origen polifilético),<br />
indicando que patogenicidad y virulencia han evolucionado más <strong>de</strong> una<br />
vez en el curso <strong>de</strong> su historia adaptativa (Baayen et al., 2000; O’Donnell et<br />
al., 1998). Para contrastar la hipótesis nula <strong>de</strong> monofilia en F. oxysporum f.<br />
sp. ciceris, hemos estudiado la extensión y naturaleza <strong>de</strong> la variación<br />
existente en las secuencias <strong>de</strong> intrones <strong>de</strong> varios genes conservados; y<br />
algunas <strong>de</strong> estas secuencias fueron comparadas a las <strong>de</strong> otras formae speciales<br />
<strong>de</strong> F. oxysporum.<br />
A pesar <strong>de</strong> la diversidad que presenta F. oxysporum f. sp. ciceris en lo<br />
referente a su distribución geográfica, los tipos <strong>de</strong> sintomatología que<br />
induce en la planta susceptible, y la patogenicidad cultivar-específica,<br />
aislados <strong>de</strong> F. oxysporum f. sp. ciceris representativos <strong>de</strong> diversas razas y<br />
variada proce<strong>de</strong>ncia geográfica poseen idénticas secuencias en los intrones<br />
<strong>de</strong> cinco genes altamente conservados en genomas fúngicos: factor <strong>de</strong><br />
traducción y elongación 1α (EF1α), β-tubulina, histona 3, actina, y<br />
calmodulina. A<strong>de</strong>más, el análisis <strong>de</strong> máxima parsimonia <strong>de</strong> las secuencias<br />
<strong>de</strong>l gen EF1α <strong>de</strong> aislados <strong>de</strong> F. oxysporum f. sp. ciceris representativos <strong>de</strong><br />
todas las razas <strong>de</strong>l patógeno, y <strong>de</strong> secuencias representativas <strong>de</strong> 11 formae<br />
speciales <strong>de</strong> F. oxysporum, agrupó a los aislados <strong>de</strong> F. oxysporum f. sp. ciceris <strong>de</strong><br />
forma distintiva respecto <strong>de</strong>l resto <strong>de</strong> los aislados, todo ello indicando el<br />
origen monofilético <strong>de</strong> esta forma specialis.<br />
La interpretación más simple y lógica sobre el origen monofilético <strong>de</strong> F.<br />
oxysporum f. sp. ciceris, y la falta <strong>de</strong> variación en las secuencias <strong>de</strong>l patógeno<br />
estudiadas en este trabajo, es que sus aislados <strong>de</strong>rivan <strong>de</strong> una población<br />
inicial fundadora pequeña (posiblemente un único individuo) que adquirió<br />
patogenicidad sobre Cicer spp. Como consecuencia <strong>de</strong> ello, la posterior<br />
diversificación <strong>de</strong> dicho agente en patotipos y razas patogénicas ha podido<br />
ser el resultado <strong>de</strong> la acumulación <strong>de</strong> pequeños cambios genéticos que<br />
hayan tenido lugar en fechas posteriores (Gordon y Martyn, 1997). Esta<br />
hipótesis está a<strong>de</strong>más apoyada por la existencia <strong>de</strong> un único grupo <strong>de</strong><br />
compatibilidad vegetativa (VCG) en F. oxysporum f. sp. ciceris (Nogales<br />
Moncada, 1997), y <strong>de</strong> un único perfil <strong>de</strong> restricción <strong>de</strong>l ADN mitocondrial<br />
(Pérez Artés et al., 1995).<br />
144
Discusión General<br />
Aunque estos pequeños cambios genéticos no se han visto reflejados en<br />
las secuencias <strong>de</strong> los genes que hemos estudiado, si han podido ser<br />
<strong>de</strong>tectados mediante su asociación con marcadores RAPD. De hecho, los<br />
análisis <strong>de</strong> ‘cluster’ <strong>de</strong> datos RAPD diferenciaron <strong>de</strong> forma consistente a<br />
los patotipos <strong>de</strong> Amarillez y Marchitez (Jiménez Gasco et al., 2001; Kelly et<br />
al., 1994), y a las diferentes razas <strong>de</strong> F. oxysporum f. sp. ciceris (Jiménez<br />
Gasco et al., 2001). Ello sugiere que los marcadores RAPD están<br />
evolucionando más rápidamente que las secuencias <strong>de</strong> los genes utilizados<br />
para realizar análisis genealógicos.<br />
Una vez <strong>de</strong>mostrado el origen monofilético <strong>de</strong> F. oxysporum f. sp. ciceris,<br />
nos propusimos profundizar en el estudio <strong>de</strong> la variabilidad genética<br />
existente en las razas <strong>de</strong> F. oxysporum f. sp. ciceris, bajo la asunción <strong>de</strong> que en<br />
este hongo carente <strong>de</strong> reproducción sexual todo el genoma se encuentra<br />
ligado, es transmitido como una sola unidad <strong>de</strong> una generación a la<br />
siguiente, y las diferentes regiones <strong>de</strong>l genoma <strong>de</strong>ben poseer la misma<br />
historia evolutiva (Taylor et al., 1999b). Por lo tanto, la evolución <strong>de</strong><br />
caracteres fenotípicos en este tipo <strong>de</strong> hongos fitopatógenos mitospóricos,<br />
tales como la especificidad <strong>de</strong> huésped o cultivar, o la relación entre razas<br />
patogénicas, pue<strong>de</strong> ser estudiada mediante el análisis <strong>de</strong> regiones <strong>de</strong>l ADN<br />
que no tienen que mantener necesariamente una relación funcional directa<br />
con el fenotipo <strong>de</strong> interés, i. e., neutrales.<br />
Por ello, hemos analizado ‘fingerprints’ <strong>de</strong> ADN genómico <strong>de</strong> los<br />
aislados <strong>de</strong> las diferentes razas <strong>de</strong> F. oxysporum f. sp. ciceris, generados<br />
mediante hibridaciones <strong>de</strong> dicho ADN con tres sondas <strong>de</strong> ADN repetitivo<br />
i<strong>de</strong>ntificadas en el propio genoma <strong>de</strong>l patógeno, y compuestas por<br />
secuencias que presentan similitud con elementos transponibles <strong>de</strong> otros<br />
hongos <strong>de</strong>scritos en la literatura fitopatológica. La similaridad existente<br />
entre los ‘fingerprints’ <strong>de</strong> ADN <strong>de</strong> los distintos aislados <strong>de</strong> F. oxysporum f.<br />
sp. ciceris se analizó mediante dos aproximaciones, una <strong>de</strong> tipo fenético y<br />
otra <strong>de</strong> tipo cladístico.<br />
El análisis <strong>de</strong> ‘fingerprints’ <strong>de</strong> ADN genómico puso <strong>de</strong> manifiesto que<br />
los aislados <strong>de</strong> F. oxysporum f. sp. ciceris presentan patrones <strong>de</strong> hibridación<br />
altamente correlacionados tanto con su capacidad <strong>de</strong> inducir un síndrome<br />
<strong>de</strong>terminado en la planta huésped (i. e., patotipos <strong>de</strong> Amarillez y <strong>de</strong><br />
145
Discusión General<br />
Marchitez), como con su patogenicidad diferencial sobre líneas<br />
diferenciadoras <strong>de</strong> garbanzo (i. e., razas patogénicas), in<strong>de</strong>pendientemente<br />
<strong>de</strong>l tipo <strong>de</strong> análisis fenético o cladístico realizado.<br />
Mientras que otras formae speciales compuestas por un único VCG han<br />
mostrado una diversidad genética muy limitada (Plyler et al., 2000;<br />
Tantaoui et al., 1996), nuestros resultados indican que F. oxysporum f. sp.<br />
ciceris es una forma specialis altamente diversificada, patogénica y<br />
genéticamente. Esta diversidad pue<strong>de</strong> ser reflejo <strong>de</strong> una larga historia <strong>de</strong><br />
asociación con su planta huésped, el garbanzo, una <strong>de</strong> las especies<br />
cultivadas que constituyeron la cuna <strong>de</strong> la agricultura, hecho documentado<br />
por información arqueológica reciente (Lev-Yadun et al., 2000; Singh,<br />
1997).<br />
Hipotéticamente, las razas patogénicas más antiguas han <strong>de</strong>bido<br />
acumular más diversidad genética que aquéllas originadas más<br />
recientemente (Goodwin et al., 1995), especialmente si asumimos el origen<br />
<strong>de</strong> una raza a partir <strong>de</strong> un único genotipo, seguido <strong>de</strong> su dispersión más o<br />
menos generalizada. Por lo tanto, en un mo<strong>de</strong>lo <strong>de</strong> evolución en las que<br />
unas razas patogénicas se originan a partir <strong>de</strong> otras a través <strong>de</strong> cambios<br />
genéticos más o menos sencillos y su persistencia en la población <strong>de</strong>l<br />
agente es consecuencia <strong>de</strong> la selección impuesta por la resistencia en el<br />
huésped (Gordon y Martyn, 1997), las razas más antiguas habrán <strong>de</strong> ser<br />
más diversas genéticamente y menos virulentas, que las más recientemente<br />
evolucionadas.<br />
Nuestros resultados apoyan dicho mo<strong>de</strong>lo evolutivo para F. oxysporum f.<br />
sp. ciceris, e indica que el avance en la evolución <strong>de</strong> las razas patogénicas va<br />
unida a mayor virulencia <strong>de</strong> éstas. Así, la raza 0 es la menos virulenta <strong>de</strong><br />
todas las razas, y es la que más diversidad genética presenta (indicado por<br />
el análisis <strong>de</strong> datos RAPD, y por el análisis <strong>de</strong> ‘fingerprints’ <strong>de</strong> ADN) por<br />
lo que correspon<strong>de</strong> a la más ancestral. Por el contrario, la raza 5 es la más<br />
virulenta <strong>de</strong> las i<strong>de</strong>ntificadas hasta ahora en la Cuenca Mediterránea, la que<br />
menor diversidad genética posee, y a la que por tanto correspon<strong>de</strong>ría un<br />
origen más reciente. A<strong>de</strong>más, <strong>de</strong>s<strong>de</strong> un punto <strong>de</strong> vista evolutivo, a la raza 0<br />
<strong>de</strong>bería seguir la raza 1B/C, no sólo en cuanto a su nivel <strong>de</strong> virulencia, sino<br />
también en cuanto a su capacidad <strong>de</strong> causar el síndrome <strong>de</strong> Amarillez en<br />
146
Discusión General<br />
cultivares <strong>de</strong> garbanzo susceptibles. Esta hipótesis parece ser coherente<br />
con nuestros resultados, ya que estas dos razas están genéticamente más<br />
relacionadas entre sí que con el resto <strong>de</strong> las razas <strong>de</strong> F. oxysporum f. sp.<br />
ciceris. A<strong>de</strong>más, los aislados <strong>de</strong> la raza 5 (no <strong>de</strong>scrito en la India) se<br />
encuentran más cercanos evolutivamente a las razas 2, 3, y 4 (sólo <strong>de</strong>scritas<br />
en la India) que al resto <strong>de</strong> las razas presentes en la Cuenca Mediterránea<br />
(razas 1A y 6). Aunque la raza 5 y las razas 2, 3, y 4 se encuentran<br />
geográficamente separadas entre sí, dichas razas son las más similares en<br />
relación a la virulencia que manifiestan sobre líneas y cultivares<br />
diferenciadores.<br />
Toda la información generada en esta Tesis Doctoral anima a<br />
profundizar en el conocimiento sobre la relación evolutiva entre razas <strong>de</strong><br />
F. oxysporum f. sp. ciceris. Un mejor entendimiento <strong>de</strong> dicha evolución<br />
podría ayudar a pre<strong>de</strong>cir los cambios que puedan ocurrir en la estructura<br />
racial existente en un suelo, al someterlo al uso continuado <strong>de</strong> cultivares <strong>de</strong><br />
garbanzo que presenten resistencia a una o alguna <strong>de</strong> las razas <strong>de</strong> F.<br />
oxysporum f. sp. ciceris.<br />
Finalmente, aunque existen numerosas investigaciones que han<br />
<strong>de</strong>mostrado la correlación entre VCGs y linajes clonales en F. oxysporum<br />
(ej., Elias et al., 1993; Koenig et al., 1997), existen muy pocos trabajos que<br />
hayan profundizado en la relación evolutiva entre razas que pertenezcan a<br />
un mismo VCG. Gordon y Martyn (1997) propusieron que una alta<br />
similaridad genética entre razas patogénicas es indicativa <strong>de</strong> una posible<br />
evolución <strong>de</strong> unas razas a partir <strong>de</strong> otras mediante pequeños cambios<br />
genéticos (Gordon y Martyn, 1997). Aunque esta hipótesis es plausible,<br />
por el momento no se encuentran en la literatura fitopatológica datos que<br />
la apoyen. Este es el primer trabajo en que se realiza un análisis tan<br />
profundo <strong>de</strong> una forma specialis <strong>de</strong> F. oxysporum <strong>de</strong> origen claramente<br />
monofilético, en el que la información patogénica y genética indica la<br />
posible evolución <strong>de</strong> unas razas a partir <strong>de</strong> otras.<br />
147
CONCLUSIONES<br />
Las conclusiones <strong>de</strong> esta Tesis Doctoral son las siguientes:<br />
Conclusiones<br />
1. El análisis RAPD ha permitido i<strong>de</strong>ntificar fragmentos <strong>de</strong> ADN que<br />
son diagnósticos <strong>de</strong> F. oxysporum f. sp. ciceris, así como <strong>de</strong> las razas 0,<br />
1B/C, 5, y 6 <strong>de</strong>l patógeno. La aplicabilidad, reproducibilidad, y<br />
fiabilidad <strong>de</strong> este método diagnóstico han sido confirmadas mediante<br />
‘ensayos ciegos’ <strong>de</strong> aislados <strong>de</strong> F. oxysporum f. sp. ciceris cuya i<strong>de</strong>ntidad<br />
racial era <strong>de</strong>sconocida para el operador.<br />
2. El análisis UPGMA <strong>de</strong> los perfiles <strong>de</strong> amplificación RAPD indicó una<br />
alta similaridad genética entre los aislados <strong>de</strong> F. oxysporum f. sp. ciceris, y<br />
agrupó a éstos en ‘clusters’ que estuvieron correlacionados con el<br />
patotipo a que pertenecen. Los aislados <strong>de</strong>l patotipo <strong>de</strong> Amarillez<br />
constituyeron un grupo distintivo que incluye a los aislados <strong>de</strong> las<br />
razas 0 y 1B/C; mientras que los aislados <strong>de</strong>l patotipo <strong>de</strong> Marchitez<br />
constituyeron otro ‘cluster’ conteniendo a los aislados <strong>de</strong> las razas 1A,<br />
2, 3, 4, 5, y 6. El análisis AMOVA <strong>de</strong> los mismos datos fue<br />
consistente con estos resultados.<br />
3. Se han diseñado iniciadores para PCR específica, basados en las<br />
secuencias <strong>de</strong> los marcadores RAPD asociados a las razas <strong>de</strong> F.<br />
oxysporum f. sp. ciceris i<strong>de</strong>ntificados, que permiten distinguir<br />
inequívocamente a aislados <strong>de</strong> F. oxysporum f. sp. ciceris <strong>de</strong> aislados <strong>de</strong><br />
F. oxysporum no patogénicos <strong>de</strong> garbanzo; <strong>de</strong> aislados pertenecientes a<br />
otras formae speciales <strong>de</strong> F. oxysporum; y <strong>de</strong> otras especies <strong>de</strong> <strong>Fusarium</strong>.<br />
Asimismo, se han diseñado iniciadores para PCR específica que han<br />
hecho posible diferenciar entre sí a aislados <strong>de</strong> las razas 0, 1A, 5, y 6<br />
149
Conclusiones<br />
150<br />
<strong>de</strong> F. oxysporum f. sp. ciceris, así como a éstos <strong>de</strong> aislados pertenecientes<br />
a otras razas <strong>de</strong>l patógeno.<br />
4. El análisis <strong>de</strong> ‘fingerprints’ <strong>de</strong> ADN genómico generados mediante<br />
hibridación con sondas <strong>de</strong> ADN repetitivo, ha puesto <strong>de</strong> manifiesto<br />
que los aislados <strong>de</strong> F. oxysporum f. sp. ciceris presentan perfiles <strong>de</strong><br />
hibridación altamente correlacionados tanto con su capacidad <strong>de</strong><br />
inducir un síndrome <strong>de</strong>terminado en la planta huésped (i. e., patotipos<br />
<strong>de</strong> Amarillez y <strong>de</strong> Marchitez), como con su patogenicidad diferencial<br />
sobre líneas diferenciadoras <strong>de</strong> garbanzo (i. e., razas patogénicas).<br />
5. Hemos <strong>de</strong>sarrollado un método <strong>de</strong> análisis <strong>de</strong> ADN genómico <strong>de</strong> F.<br />
oxysporum f. sp. ciceris, basado en la amplificación entre copias <strong>de</strong> un<br />
elemento repetitivo i<strong>de</strong>ntificado en el genoma <strong>de</strong>l patógeno (rep-<br />
PCR). La información generada por ‘fingerprints’ <strong>de</strong> ADN obtenidos<br />
con este método es equivalente a la obtenida mediante hibridaciones<br />
con el elemento repetitivo, lo cual supone un método alternativo, pero<br />
más simple, para investigar la diversidad en las poblaciones <strong>de</strong> F.<br />
oxysporum f. sp. ciceris basado en el estudio <strong>de</strong> un elevado número <strong>de</strong><br />
aislados <strong>de</strong>l patógeno.<br />
6. A pesar <strong>de</strong> la diversidad <strong>de</strong> este patógeno en su distribución<br />
geográfica, los tipos <strong>de</strong> sintomatología que induce en la planta<br />
susceptible, y la patogenicidad cultivar-específica, los aislados <strong>de</strong> F.<br />
oxysporum f. sp. ciceris poseen idénticas secuencias <strong>de</strong> los intrones <strong>de</strong><br />
cinco genes altamente conservados en genomas fúngicos: EF1α, βtub,<br />
H3, Act, y Cal.<br />
7. El análisis <strong>de</strong> máxima parsimonia <strong>de</strong> las secuencias <strong>de</strong>l gen EF1α <strong>de</strong><br />
17 aislados <strong>de</strong> F. oxysporum f. sp. ciceris y <strong>de</strong> otras 24 secuencias<br />
representativas <strong>de</strong> 11 formae speciales <strong>de</strong> F. oxysporum, agrupó a los<br />
aislados <strong>de</strong> F. oxysporum f. sp. ciceris <strong>de</strong> forma distintiva respecto <strong>de</strong>l<br />
resto <strong>de</strong> los aislados, que <strong>de</strong>muestra el origen monofilético <strong>de</strong> esta<br />
forma specialis.<br />
8. La interpretación lógica <strong>de</strong> la monofilia <strong>de</strong> F. oxysporum f. sp. ciceris<br />
sugiere que éste <strong>de</strong>riva <strong>de</strong> una población inicial fundadora pequeña
Conclusiones<br />
(posiblemente un único individuo) que adquirió patogenicidad sobre<br />
Cicer spp., y cuya subsiguiente diversificación en patotipos y razas<br />
patogénicas ha podido resultar <strong>de</strong> la acumulación <strong>de</strong> cambios<br />
genéticos relativamente simples.<br />
9. F. oxysporum f. sp. ciceris es un grupo clonal <strong>de</strong> aislados patogénicos<br />
con una fuerte estructura racial, en el cual la correlación entre los<br />
marcadores genéticos neutrales, tales como RAPDs y ‘fingerprints’ <strong>de</strong><br />
ADN, y la patogenicidad, pue<strong>de</strong> ser utilizada para inferir conclusiones<br />
sobre la relación genética entre razas.<br />
10. Nuestra hipótesis evolutiva es que, en F. oxysporum f. sp. ciceris, unas<br />
razas patogénicas se han originado a partir <strong>de</strong> otras, y que el avance<br />
evolutivo <strong>de</strong> dichas razas patogénicas va unido a un incremento <strong>de</strong><br />
virulencia <strong>de</strong> éstas (i. e., número <strong>de</strong> líneas diferenciadoras raciales<br />
susceptibles). Así, la raza 0 es la menos virulenta <strong>de</strong> todas las razas, y<br />
la más antigua <strong>de</strong> ellas en razón <strong>de</strong> su mayor diversidad genética. Por<br />
el contrario, la raza 5 es la más virulenta <strong>de</strong> las i<strong>de</strong>ntificadas hasta<br />
ahora en la Cuenca Mediterránea, la que menor diversidad genética<br />
posee, y a la que por tanto correspon<strong>de</strong>ría un origen evolutivo más<br />
reciente.<br />
151
BIBLIOGRAFÍA CITADA<br />
Bibliografía<br />
Alcalá Jiménez, A. R. 1995. Aspectos epi<strong>de</strong>miológicos en el patosistema<br />
<strong>Fusarium</strong> oxysporum Schlechtend.:Fr. f. sp. ciceris (Padwick) Matuo & K.<br />
Sato / Cicer arietinum L. Tesis Doctoral. Universidad <strong>de</strong> Córdoba,<br />
Córdoba, 284 pp.<br />
Allen, D. J. 1983. The Pathology of Tropical Food Legumes. John Wiley<br />
and Sons, New York. 413 pp.<br />
Altschul, S. F., Mad<strong>de</strong>n, T. L., Schäffer, A. A., Zhang, J., Zhang, Z., Miller,<br />
W., y Lipman, D. J. 1997. Gapped BLAST and PSI-BLAST: a new<br />
generation of protein database search programs. Nucleic Acids<br />
Research 25: 3389-3402.<br />
Appel, D., y Gordon, T. R. 1994. Local and regional variation in<br />
populations of <strong>Fusarium</strong> oxysporum from agricultural fields.<br />
Phytopathology 84: 786-791.<br />
Appel, D., y Gordon, T. R. 1995. Intraspecific variation within populations<br />
of <strong>Fusarium</strong> oxysporum based on RFLP analysis of the intergenic spacer<br />
region of the rDNA. Experimental Mycology 19: 120-128.<br />
Appel, D., y Gordon, T. R. 1996. Relationships among pathogenic and<br />
nonpathogenic isolates of <strong>Fusarium</strong> oxysporum based on the partial<br />
sequence of the intergenic spacer region of the ribosomal DNA.<br />
<strong>Molecular</strong> Plant-Microbe Interactions 9: 125-138.<br />
Assigbetse, K. B., Fernán<strong>de</strong>z, D., Dubois, M. P., y Geiger, J. P. 1994.<br />
Differentiation of <strong>Fusarium</strong> oxysporum f. sp. vasinfectum races on cotton by<br />
153
Bibliografía<br />
154<br />
Random Amplified Polymorphic DNA (RAPD) analysis.<br />
Phytopathology 84: 622-626.<br />
Ayyar, V. R., e Iyer, R. B. 1936. A preliminary note on the mo<strong>de</strong> of<br />
inheritance of reaction to wilt in Cicer arietinum L. Proceedings of the<br />
Indian Aca<strong>de</strong>my of Sciences 3: 438-443.<br />
Baayen, R. P., O’Donnell, K., Bonants, P. J. M., Cigelnik, E., Kroon, L. P.<br />
N. M., Roebroeck, E. J. A., y Waalwijk, C. 2000. Gene genealogies and<br />
AFLP analyses in the <strong>Fusarium</strong> oxysporum complex i<strong>de</strong>ntify monophyletic<br />
and nonmonophyletic formae speciales causing wilt and rot diseases.<br />
Phytopathology 90: 891-900.<br />
Bentley, S., Pegg, K. G., y Dale, J. L. 1994. Optimization of RAPDfingerprinting<br />
to analyze genetic variation within populations of<br />
<strong>Fusarium</strong> oxysporum f. sp. cubense. Journal of Phytopathology 142: 64-78.<br />
Bentley, S., Pegg, K. G., y Dale, J. L. 1995. Genetic variation among a<br />
world-wi<strong>de</strong> collection of isolates of <strong>Fusarium</strong> oxysporum f. sp. cubense<br />
analysed by RAPD-PCR fingerprinting. Mycological Research 99: 1378-<br />
1384.<br />
Bentley, S., Pegg, K. G., Moore, N. Y., y Bud<strong>de</strong>nhagen, I. W. 1998.<br />
Genetic variation among vegetative compatibility groups of <strong>Fusarium</strong><br />
oxysporum f. sp. cubense analyzed by DNA fingerprinting. Phytopathology<br />
88: 1283-1293.<br />
Bosland, P. W., y Williams, P. H. 1987. An evaluation of <strong>Fusarium</strong> oxysporum<br />
from crucifers, based on pathogenicity, isozyme polymorphism,<br />
vegetative compatibility and geographic origin. Canadian Journal of<br />
Botany 65: 2067-2073.<br />
Bouhot, D., y Rouxel, F. 1971. Technique sèlective et quantitative<br />
d´analyse <strong>de</strong>s <strong>Fusarium</strong> oxysporum et <strong>Fusarium</strong> solani dans le sol. Mo<strong>de</strong><br />
d´emploi. Annals <strong>de</strong> Phytopathologie 3: 251-254.<br />
Bouslama, M. 1980. Chickpea improvement in Tunisia. Proc. Int.<br />
Workshop Improvement: 277-280: ICRISAT, Hy<strong>de</strong>rabad, India.
Bibliografía<br />
Bridge, P. D., Arora, D. K., Reddy, C. A., y Elan<strong>de</strong>r, R. P. (eds.) 1998a.<br />
Applications of PCR in Mycology. CAB International, Wallingford,<br />
Oxon, UK. 357 pp.<br />
Bridge, P. D., Couteaudier, Y., y Clarckson, J. M. (eds.) 1998b. <strong>Molecular</strong><br />
Variability of Fungal Plant Pathogens. CAB International, Wallingford,<br />
Oxon, UK. 319 pp.<br />
Brown, J. K. M. 1998. Surveys of variation in pathogen populations and<br />
their application to disease control. En: The Epi<strong>de</strong>miology of Plant<br />
Diseases: 73-102. D. G. Jones (ed.).<br />
Bud<strong>de</strong>nhagen, I. W., y Workneh, F. 1988. <strong>Fusarium</strong> wilt of chickpea in<br />
California. (Abstract) Phytopathology 78: 1563.<br />
Bud<strong>de</strong>nhagen, I. W., Workneh, F., y Bosque Pérez, N. 1988. Chickpea<br />
improvement and chickpea diseases in California. International<br />
Chickpea Newsletter 19: 9-10.<br />
Bunting, T. E., Plumley, K. A., Clarke, B. B., y Hillman, B. I. 1996.<br />
I<strong>de</strong>ntification of Magnaporthe poae by PCR and examination of its<br />
relationship to other fungi by analysis of their nuclear rDNA ITS-1<br />
regions. Phytopathology 86: 398-404.<br />
Cabrera <strong>de</strong> la Colina, J. 1986. Variación patogénica en <strong>Fusarium</strong> oxysporum<br />
Schlechtend. emend. Sny<strong>de</strong>r & Hansen y F. solani (Mart.) Sacc. emend.<br />
Sny<strong>de</strong>r & Hansen agentes <strong>de</strong> la Marchitez y Podredumbre <strong>de</strong> Raíz <strong>de</strong>l<br />
garbanzo en Andalucía. Tesis Doctoral. Universidad <strong>de</strong> Córdoba.<br />
Córdoba, 237 pp.<br />
Cabrera <strong>de</strong> la Colina, J., Trapero Casas, A., y Jiménez Díaz, R. M. 1985.<br />
Races of <strong>Fusarium</strong> oxysporum f. sp. ciceri in Andalucía. International<br />
Chickpea Newsletter 13: 24-26.<br />
Cabrera <strong>de</strong> la Colina, J., Trapero Casas, A., y Jiménez Díaz, R. M. 1987.<br />
Host range and symptomless carriers of <strong>Fusarium</strong> spp. Infecting<br />
chickpeas in Andalucía. En: Proceedings of the 7th Congress of the<br />
Mediterranean Phytopathological Union (SEF/MPU, eds.), pp. 20-26.<br />
155
Bibliografía<br />
Carbone, I., y Kohn, L. M. 1999. A method for <strong>de</strong>signing primer sets for<br />
speciation studies in filamentous ascomycetes. Mycologia 91: 553-556.<br />
Carbone, I., An<strong>de</strong>rson, J. B., y Kohn, L. M. 1999. Patterns of <strong>de</strong>scent in<br />
clonal lineages and their multilocus fingerprints are resolved with<br />
combined gene genealogies. Evolution 53: 11-21.<br />
Car<strong>de</strong>r, J. H., y Barbara, D. I. 1994. <strong>Molecular</strong> variation within some<br />
Japanese isolates of Verticillium dahliae. Plant Pathology 43: 947-950.<br />
Coddington, A., y Gould, D. S. 1992. Use of RFLPs to i<strong>de</strong>ntify races of<br />
fungal pathogens. En: Techniques for the rapid <strong>de</strong>tection of Plant<br />
Pathogens: 162-176. J. M. Duncan y L. Torrance (eds.). Brithish Society<br />
for Plant Pathology. Blackwell Science Publications, Oxford, UK.<br />
Correll, J. C. 1991. The relationship between formae specialis, races and<br />
vegetative compatibility groups in <strong>Fusarium</strong> oxysporum. Phytopathology<br />
81: 1061-1063.<br />
Daboussi, M. J. 1997. Fungal transposable elements and genome<br />
evolution. Genetica 100: 253-260.<br />
Daboussi, M. J., y Langin, T. 1994. Transposable elements in the fungal<br />
plant pathogen <strong>Fusarium</strong> oxysporum. Genetica 93: 49-59.<br />
Daboussi, M. J., Langin, T., y Brygoo, Y. 1992. Fot1, a new family of<br />
fungal transposable elements. <strong>Molecular</strong> & General Genetics 232: 12-<br />
16.<br />
Don, R. H., Cox, P. T., Wainwright, B. J., Baker, K., y Mattick, J. S. 1991.<br />
‘Touch-down’ PCR to circumvent spurious priming during gene<br />
amplification. Nucleic Acids Research 19: 4008.<br />
Echandi, E. 1970. Wilt of chickpeas or garbanzo beans (Cicer arietinum)<br />
incited by <strong>Fusarium</strong> oxysporum. Phytopathology 60: 1539.<br />
E<strong>de</strong>l, V., Steinberg, C., Avelage, I., Laguerre, G., y Alabouvette, C. 1995.<br />
Comparison of three molecular methods for the characterization of<br />
<strong>Fusarium</strong> oxysporum strains. Phytopathology 85: 579-585.<br />
156
Bibliografía<br />
Elias, K. S., y Schnei<strong>de</strong>r, R. W. 1991. Vegetative compatibility groups in<br />
<strong>Fusarium</strong> oxysporum f. sp. lycopersici. Phytopathology 81: 159-162.<br />
Elias, K. S., Zamir, D., Lichtman-Pleban, T., y Katan, T. 1993. Population<br />
structure of <strong>Fusarium</strong> oxysporum f. sp. lycopersici: Restriction fragment<br />
length polymorphisms provi<strong>de</strong> genetic evi<strong>de</strong>nce that vegetative<br />
compatibility group is an indicator of evolutionary origin. <strong>Molecular</strong><br />
Plant-Microbe Interactions 6: 565-572.<br />
Excoffier, L., Smouse, P. E., y Quattro, J. M. 1992. Analysis of molecular<br />
variance inferred from metric distances among DNA haplotypes:<br />
Application to human mitochondrial DNA restriction data. Genetics<br />
131: 479-491.<br />
Fernán<strong>de</strong>z, D., Ouinten, M., Tantaoui, A., y Geiger, J.-P. 1997. <strong>Molecular</strong><br />
records of micro-evolution within the Algerian population of <strong>Fusarium</strong><br />
oxysporum f. sp. albedinis during its spread to new oases. European<br />
Journal of Plant Pathology 103: 485-490.<br />
Frisullo, S., Ciccarese F., Amenduni, M., y Zamani, H. R. 1989. Wilt of<br />
chickpea (Cicer arietinum L.) by <strong>Fusarium</strong> oxysporum f. sp. ciceris Schlech. in<br />
southern Italy. Difesa <strong>de</strong>lle Piante 12: 181-182.<br />
García Pedrajas, M. D., Bainbridge, B. W., Heale, J. B., Pérez Artés, E., y<br />
Jiménez Díaz, R. M. 1999. A simple PCR-based method for the<br />
<strong>de</strong>tection of the chickpea wilt pathogen <strong>Fusarium</strong> oxysporum f. sp. ciceris<br />
in artificial and natural soils. European Journal of Plant Pathology 105:<br />
251-259.<br />
George, M. L., Nelson, R. J., Zeigler, R. S., y Leung, H. 1998. Rapid<br />
population analysis of Magnaporthe grisea by using rep-PCR and<br />
endogenous repetitive DNA sequences. Phytopathology 88: 223-229.<br />
George, M. L. C., Bustamam, M., Cruz, W. T., Leach, J. E., y Nelson, R. J.<br />
1997. Movement of Xanthomonas oryzae pv. oryzae in Southern Asia<br />
<strong>de</strong>tected using PCR-based PCR fingerprinting. Phytopathology 87: 302-<br />
309.<br />
157
Bibliografía<br />
Ghanekar, A. M., Sethi, S. C., Jalali, B. L., y Nene, Y. L. 1990. ICCV6<br />
(ICCC32) a kabuli chickpea resistant to <strong>Fusarium</strong> wilt and Root rots.<br />
International Chickpea Newsletter 23: 22-23.<br />
Glass, N. L., y Donaldson, G. C. 1995. Development of primers sets<br />
<strong>de</strong>sign for use with the PCR to amplify conserved genes from<br />
filamentous ascomycetes. Applied and Environmental Microbiology 61:<br />
1323-1330.<br />
Goodwin, S. B., Drenth, A., y Fry, W. E. 1992. Cloning and genetic<br />
analyses of two highly polymorphic, mo<strong>de</strong>rately repetitive nuclear<br />
DNAs from Phytophthora infestans. Curr. Genet. 22: 107-115.<br />
Goodwin, S. B., Sujkowski, L. S., y Fry, W. E. 1995. Rapid evolution of<br />
pathogenicity within clonal lineages of the potato blight disease fungus.<br />
Phytopathology 85: 669-676.<br />
Gordon, T. R., y Martyn, R. D. 1997. The evolutionary biology of <strong>Fusarium</strong><br />
oxysporum. Annual Review of Phytopathology 35: 111-128.<br />
Gordon, T. R., y Okamoto, D. 1992. Population structure and the<br />
relationship between pathogenic and nonpathogenic strains of <strong>Fusarium</strong><br />
oxysporum. Phytopathology 82: 73-77.<br />
Grajal Martin, M. J., Simon, C. J., y Muehlbauer, F. J. 1993. Use of<br />
Random Amplified Polymorphic DNA (RAPD) to characterize race 2<br />
of <strong>Fusarium</strong> oxysporum f. sp. pisi. Phytopathology 83: 612-614.<br />
Gumber, R. K., Kumar, J., y Haware, M. P. 1995. Inheritance of resistance<br />
to <strong>Fusarium</strong> wilt in chickpea. Plant Breeding 114: 277-279.<br />
Halila, H. M., y Harrabi, M. M. 1990. Breeding for dual resistance to<br />
Ascochyta and wilt diseases in chickpea. Options Mediterr. Ser. Semin.<br />
9: 163-166.<br />
Halila, M. H. y Strange, R. N. 1996. I<strong>de</strong>ntification of the causal agent of<br />
wilt of chickpea in Tunisia as <strong>Fusarium</strong> oxysporum f. sp. ciceri race 0.<br />
Phytopathologia Mediterranea 35: 67-74.<br />
158
Bibliografía<br />
Haware, M. P. 1990. <strong>Fusarium</strong> wilt and other important diseases of<br />
chickpea in the Mediterranean area. Options Mediterr. Ser. Semin. 9:<br />
61-64.<br />
Haware, M. P., y Nene, Y. L. 1982a. Symptomless carriers of the chickpea<br />
wilt <strong>Fusarium</strong>. Plant Disease 66: 250-251.<br />
Haware, M. P., y Nene, Y. L. 1982b. Races of <strong>Fusarium</strong> oxysporum f. sp.<br />
ciceris. Plant Disease 66: 809-810.<br />
Haware, M. P., Kumar, J., y Reddy, M. V. 1980. Disease resistance in<br />
kabuli-<strong>de</strong>si chickpea introgression. Proceedings of the International<br />
Workshop on Chickpea Improvement. ICRISAT, Hy<strong>de</strong>rabad, India,<br />
67-69.<br />
Haware, M. P., Nene, Y. L., y Mathur, S. B. 1986a. Seed-borne diseases of<br />
chickpea. En: Technical Bulletin (Danish Government Institute of Seed<br />
Technology for Developing Countries, ed.) Vol. 1, 32 pp.<br />
Haware, M. P., Nene, Y. L., y Mathur, S. B. 1986b. Survival of <strong>Fusarium</strong><br />
oxysporum f. sp. ciceris in soil in absence of chickpea. En: Management of<br />
Soilborne Diseases of Crop Plants: Proceedings of National Seminar,<br />
pp. 8-10.<br />
Haware, M. P., Nene, Y. L., y Rajeshwari, R. 1978. Erradication of<br />
<strong>Fusarium</strong> oxysporum f. sp. ciceris transmitted in chickpea seed.<br />
Phytopathology 68: 1364-1367.<br />
Haware, M. P., Jiménez Díaz, R. M., Amin, K. S., Phillips, J. C., y Halila,<br />
M. H. 1989. Integrated management of wilt and root rots of chickpea.<br />
Proceedings of the International Workshop of Chickpea in the<br />
Nineties, ICARDA/ICRISAT. Patacheru, A. P., India.<br />
Helms, D., Panella, L., Bud<strong>de</strong>nhagen, I. W., Workneh, F., Tucker, C. L.,<br />
Foster, K. W., y Gepts, P. L. 1992. Registration of ‘UC 15’ chickpea.<br />
Crops Sci. 32: 500.<br />
Henson, J. M., y French, R. 1993. The polymerase chain reaction and plant<br />
disease diagnosis. Annual Review Phytopathology 31: 81-109.<br />
159
Bibliografía<br />
Hoagland, D. R., y Arnon, D. I. 1950. The water culture method for<br />
growing plants without soil. Calif Exp Stn Circ No 347.<br />
Hulton, C. S. J., Higgins, C. F., y Sharp., P. M. 1991. ERIC sequences: A<br />
novel family of repetitive elements in the genomes of Escherichia coli,<br />
Salmonella typhimurium and other enterobacteria. <strong>Molecular</strong> Microbiology<br />
5: 825-834.<br />
Jacobson, D. J., y Gordon, T. R. 1988. Vegetative compatibility and selfincompatibility<br />
within <strong>Fusarium</strong> oxysporum f. sp. melonis. Phytopathology<br />
78: 668-672.<br />
Jacobson, D. J., y Gordon, T. R. 1990. Variability of mitochondrial DNA<br />
as an indicator of relationships between populations of <strong>Fusarium</strong><br />
oxysporum f. sp. melonis. Mycological Research 94: 734-744.<br />
Jalali, B. L., y Chand, H. 1992. Chickpea wilt. In: Singh US,<br />
Mukhopadhayay AN, Kumar J, Chaube HS, eds. Plant Diseases of<br />
International Importance. Vol. I. Diseases of cereals and pulses.<br />
Englewood Cliffs, NY: Prentice Hall, 429-444.<br />
Jiménez Díaz, R. M. 1994. La Fusariosis Vascular <strong>de</strong>l garbanzo causada<br />
por <strong>Fusarium</strong> oxysporum f. sp. ciceris: 15 años <strong>de</strong> Investigación en España.<br />
Investigación Agraria: Producción y Protección Vegetales. Fuera <strong>de</strong><br />
Serie 2: 285-294.<br />
Jiménez Díaz, R. M., y Trapero Casas, A. 1985. Use of fungici<strong>de</strong><br />
treatments and host resistance to control the wilt and root rot complex<br />
of chickpeas. Plant Disease 69: 591-595.<br />
Jiménez Díaz, R. M., Basallote Ureba, M. J., y Rapoport, H. 1989a.<br />
Colonization and pathogenesis in chickpea infected by races of <strong>Fusarium</strong><br />
oxysporum f. sp. ciceri. Vascular Wilt Diseases of Plants. NATO ASI. Ser.<br />
Vol. H 28: 113-121. E. C. Tjamos y C. H. Beckman (eds.) Springer-<br />
Verlag, Berlin.<br />
Jiménez Díaz, R. M., Trapero Casas, A., y Cabrera <strong>de</strong> la Colina, J. 1989b.<br />
Races of <strong>Fusarium</strong> oxysporum f. sp. ciceri infecting chickpea in southern<br />
160
Bibliografía<br />
Spain. Vascular Wilt Diseases of Plants. NATO ASI. Ser. Vol. H 28:<br />
515-520. E. C. Tjamos y C. H. Beckman (eds.) Springer-Verlag, Berlin.<br />
Jiménez Díaz, R. M., Trapero Casas, A., y Cubero, I. 1989c. Importance of<br />
chickpea soilborne diseases in the Mediterranean basin. Proceedings of<br />
the Consultancy Meeting on Breeding for Disease Resistance in kabuli<br />
Chickpeas. ICARDA, Aleppo, Siria.<br />
Jiménez-Díaz, R. M., Alcalá-Jiménez, A. R., Hervás, A. y Trapero-Casas, J.<br />
L. 1993a. Pathogenic variability and hosts resistance in the <strong>Fusarium</strong><br />
oxysporum f. sp. ciceris / Cicer arietinum pathosystem. En: Proc. Eur.<br />
Semin. <strong>Fusarium</strong> Mycotoxins, Taxonomy, Pathogenicity and Host<br />
Resistance, 3rd (pp 87-94) Hodowsla Ròslin Aklimatyazacja i<br />
Nasiennictwo. Plant Breeding and Acclimatization Institute, Radzikóv,<br />
Polonia.<br />
Jiménez Díaz, R. M., Singh, K. B., Trapero Casas, A., y Trapero Casas, J.<br />
L. 1991. Resistance in kabuli chickpea to <strong>Fusarium</strong> wilt. Plant Disease<br />
75: 914-918.<br />
Jiménez Díaz, R. M., Crino, P., Halila, M. H., Mosconi, C., y Trapero<br />
Casas, A. 1993b. Screening for resistance to Ascochyta blight and<br />
<strong>Fusarium</strong> wilt in chickpea. Breeding for Stress Tolerance in Cool-<br />
Season Food Legumes: 77-95. K. B. Singh y M. C. Saxena (eds.), John<br />
Wiley & Sons, Chichester, Reino Unido.<br />
Jiménez Gasco, M. M., Pérez Artés, E., y Jiménez Díaz, R. M. 2001.<br />
I<strong>de</strong>ntification of pathogenic races 0, 1B/C, 5, and 6 of <strong>Fusarium</strong><br />
oxysporum f. sp. ciceris with random amplified polymorphic DNA<br />
(RAPD). European Journal of Plant Pathology 107: 237-248.<br />
Kaiser, W. J., Alcalá Jiménez, A. R., Hervás Vargas, A., Trapero Casas, J.<br />
L., y Jiménez Díaz, R. M. 1994. Screening of wild Cicer species for<br />
resistance to race 0 and 5 pf <strong>Fusarium</strong> oxysporum f. sp. ciceris. Plant<br />
Disease 78: 962-967.<br />
Kannaiyan, J. 1981. Diseases of chickpea in Malawi. International<br />
Chickpea Newsletter 4: 16.<br />
161
Bibliografía<br />
Keim, P., Shoemaker, R. C., y Palmer, R. G. 1989. Restriction fragment<br />
length polymorphism diversity in soybean. Theoretical and Applied<br />
Genetics 77: 786-792.<br />
Kelly, A. G. 1996. Genome analysis of races and pathotypes in the fungus<br />
<strong>Fusarium</strong> oxysporum f. sp. ciceris infecting chickpea. Tesis Doctoral. King’s<br />
College London, Universidad <strong>de</strong> Londres, Londres, Reino Unido. 206<br />
pp.<br />
Kelly, A. G., Bainbridge, B. W., Heale, J. B., Pérez Artés, E., y Jiménez<br />
Díaz, R. M. 1998. In planta-polymerase-chain-reaction <strong>de</strong>tection of the<br />
wilt-inducing pathotype of <strong>Fusarium</strong> oxysporum f. sp. ciceris in chickpea<br />
(Cicer arietinum L.). Physiological and <strong>Molecular</strong> Plant Pathology 52:<br />
397-409.<br />
Kelly, A. G., Alcalá-Jiménez, A. R., Bainbridge, B. W., Heale, J. B., Pérez-<br />
Artés, E., y Jiménez-Díaz, R. M. 1994. Use of genetic fingerprinting and<br />
Random Amplified Polymorphic DNA to characterize pathotypes of<br />
<strong>Fusarium</strong> oxysporum f. sp. ciceris infecting chickpea. Phytopathology 84:<br />
1293-1298.<br />
Kistler, H. C. 1997. Genetic diversity in the plant-pathogenic fungus<br />
<strong>Fusarium</strong> oxysporum. Phytopathology 87: 474-479.<br />
Kistler, H. C., y Miao, V. P. N. 1992. Mew mo<strong>de</strong>s of genetic change in<br />
filamentous fungi. Annual Review of Phytopathology 30: 131-152.<br />
Kistler, H. C., Momol, E. A., y Benny, U. 1991. Repetitive genomic<br />
sequences for <strong>de</strong>termining relatedness among strains of <strong>Fusarium</strong><br />
oxysporum. Phytopathology 81: 331-336.<br />
Kistler, H. C., Bosland, P. W., Benny, U. K., Leong, S., y Williams, P. H.<br />
1987. Relatedness of strains of <strong>Fusarium</strong> oxysporum from crucifers<br />
measured by examination of mitochondrial and ribosomal DNA.<br />
Phytopathology 77: 1289-1293.<br />
Koenig, R. L., Ploetz, R. C., y Kistler, H. C. 1997. <strong>Fusarium</strong> oxysporum f. sp.<br />
cubense consists of a small number of divergent and globally distributed<br />
clonal lineages. Phytopathology 87: 915-923.<br />
162
Bibliografía<br />
Kumar, J., y Haware, M. P. 1982. Inheritance to <strong>Fusarium</strong> wilt of chickpea.<br />
Phytopathology 72: 1035-1036.<br />
Kumar, J., Haware, M. P., y Smithson, J. B. 1985. Registration of four<br />
short duration <strong>Fusarium</strong> wilt-resistant kabuli (garbanzo) chickpea<br />
germplasm. Crop Science 25: 576-577.<br />
Lee, S. B., White, T. J., y Taylor, J. W. 1993. Detection of Phytophthora<br />
species by oligonucleoti<strong>de</strong> hibridization to amplified ribosomal DNA<br />
spacers. Phytopathology 83: 177-181.<br />
Leslie, J. F. 1993. Fungal vegetative compatibility. Annual Review of<br />
Phytopathology 31: 127-151.<br />
Lévesque, C. A., Vrain, T. C., y De Boer, S. H. 1994. Development of a<br />
species-specific probe for Pythium ultimum using amplified ribosomal<br />
DNA. Phytopathology 84: 474-478.<br />
Lev-Yadun, S., Gopher, A., y Abbo, S. 2000. The cradle of agriculture.<br />
Science 288: 1602-1603.<br />
Levy, M., Romao, J., Marchetti, M. A., y Hamer, J. E. 1991. DNA<br />
fingerprinting with a dispersed repeated sequence resolves pathotype<br />
diversity in the rice blast fungus. The Plant Cell 3: 95-102.<br />
López García, H. 1974. Inheritance of the character resistance to wilt<br />
(<strong>Fusarium</strong> sp.) in chickpea (Cicer arietinum) un<strong>de</strong>r field conditions.<br />
Agricultura Técnica en México 3: 286-289.<br />
Manulis, S., Kogan, N., Reuven, M., y Ben Yephet, Y. 1994. Use of RAPD<br />
technique for i<strong>de</strong>ntification of <strong>Fusarium</strong> oxysporum f. sp. dianthi from<br />
carnation. Phytopathology 84: 98-101.<br />
Martin, R. R., James, D., y Lévesque, C. A. 2000. Impacts of molecular<br />
diagnostic technologies on plant disease management. Annual Review<br />
of Phytopathology 38: 207-239.<br />
Martin, B., Humbert, O., Camara, M., Guenzi, E., Walker, J., Mitchell, T.,<br />
Andrew, P., Prudhomme, M., Alloing, G., Hakenbeck, R., Morrison, D.<br />
A., Boulnois, G. J., y Claverys, J. P. 1992. A highly conserved repeated<br />
163
Bibliografía<br />
164<br />
DNA element located in the chromosome of Streptococcus pneumoniae.<br />
Nucleic Acids Research 20: 3479-3483.<br />
McDonald, B. A. 1997. The populations genetics of fungi: tools and<br />
techniques. Phytopathology 87: 448-453.<br />
Michelmore, R. W., y Hulbert, S. H. 1987. <strong>Molecular</strong> markers for genetic<br />
analysis of phytopathogenic fungi. Annual Review of Phytopathology<br />
25: 383-404.<br />
Migheli, Q., Briatore, E., y Garibaldi, A. 1998. Use of random amplified<br />
polymorphic DNA (RAPD) to i<strong>de</strong>ntify races 1, 2, 3 and 8 of <strong>Fusarium</strong><br />
oxysporum f. sp. dianthi in Italy. European Journal of Plant Pathology<br />
104: 49-57.<br />
Milgroom, M. G., y Fry, W. E. 1997. Contributions of population genetics<br />
to plant disease epi<strong>de</strong>miology and management. Advances in Botanical<br />
Research 24: 2-30<br />
Miller, S. A. 1996. Detecting propagules of plant pathogenic fungi.<br />
Advances in Botanical Research 23:73-102.<br />
Morales, G. J. A. 1986. Chickpea breeding program in Sonora.<br />
International Chickpea Newsletter 15: 11-12.<br />
Mouyna, I., Renard, J. L., y Brygoo, Y. 1996. DNA polymorphism among<br />
<strong>Fusarium</strong> oxysporum f. sp. elaeidis populations from oil palm, using a<br />
repeated and dispersed sequence ‘Palm’. Current Genetics 30: 174-180.<br />
Mullis, K. B., y Faloona, F. A. 1987. Specific synthesis of DNA in vitro via<br />
a polymerase-catalysed chain reaction. Methods in Enzymology 155:<br />
335-350.<br />
Namiki, F., Shiomi, T., Kayamura, Y., y Tsuge, T. 1994. Characterization<br />
of the formae speciales of <strong>Fusarium</strong> oxysporum causing wilts of cucurbits<br />
by DNA fingerprinting with nuclear repetitive DNA sequences.<br />
Applied and Environmental Microbiology 60: 2684-2691.
Bibliografía<br />
Namiki, F., Shiomi, T., Kayamura, Y., y Tsuge, T. 1998. Pathogenic and<br />
genetic variation in the Japanese strains of <strong>Fusarium</strong> oxysporum f. sp.<br />
melonis. Phytopathology 88: 804-810.<br />
Narasimhan, R. 1929. A preliminary note on a <strong>Fusarium</strong> parasitic on Bengal<br />
gram (Cicer arietinum). Madras Agricultural Department Year Book: 5-<br />
11.<br />
Navas Cortés, J. A., Trapero Casas, A., y Jiménez Díaz, R. M. 1998.<br />
Influence on relative humidity and temperature on <strong>de</strong>velopment of<br />
Didymella rabiei on chickpea <strong>de</strong>bris. Plant Pathology 47: 57-66.<br />
Navas Cortés, J. A.., Alcalá Jiménez, A. R., Hau, B., y Jiménez Díaz, R. M.<br />
2000. Influence of inoculum <strong>de</strong>nsity of races 0 and 5 of <strong>Fusarium</strong><br />
oxysporum f. sp. ciceris on <strong>de</strong>velopment of <strong>Fusarium</strong> wilt in chickpea<br />
cultivars. European Journal of Plant Pathology 106: 135-146.<br />
Navas Cortés, J. A., Rodríguez Jurado, D., Trapero Casas, J. L., y Jiménez<br />
Díaz, R. M. 1994. Resistencia combinada a la raza 5 <strong>de</strong> <strong>Fusarium</strong><br />
oxysporum f. sp. ciceris y a Rabia en garbanzo. En: Resúmenes, Congreso<br />
<strong>de</strong> la Sociedad Española <strong>de</strong> Fitopatología VII, Sitges: 52.<br />
Nazar, R. N., Hu, X., Schmidt, J., Culham, D. y Robb, J. 1991. Potential<br />
use of PCR-amplified ribosomal intergenic sequences in the <strong>de</strong>tection<br />
and differentiation of Verticillium wilt pathogens. Physiol. Mol. Plant<br />
Pathol. 39: 1-11.<br />
Nelson, P. E., Toussoun, T. A., y Cook, R. J. (eds.) 1981. <strong>Fusarium</strong>:<br />
Diseases, Biology and Taxonomy. University Park, PA, USA: The<br />
Pennsylvania University Press.<br />
Nelson, P. E., Toussoun, T. A., y Marasas, W. F. O. (eds.) 1983. <strong>Fusarium</strong><br />
species: An Illustrated Manual for I<strong>de</strong>ntification. University Park, PA,<br />
USA: The Pennsylvania University Press.<br />
Nene, Y. L. 1980. Diseases of chickpea. ICRISAT. Proc. Int. Workshop<br />
Improvement: 171-187. ICRISAT, Hy<strong>de</strong>rabad, India.<br />
165
Bibliografía<br />
Nene, Y. L., y Haware, M. P. 1980. Screening for chickpea resistance to<br />
wilt. Plant Disease 64: 379-380.<br />
Nene, Y. L., y Reddy, M. V. 1987. Chickpea diseases and their control. En:<br />
The Chickpea: 233-270. M. C. Saxena, y K. B. Singh, eds. CAB<br />
International. Wallingford, Oxon, U. K.<br />
Nene, Y. L., Sheila, V. K., y Sharma, S. B. 1984. A world list of chickpea<br />
(Cicer arietinum L.) and pigeonpea (Cajanus cajan L.) pathogens. ICRISAT<br />
Pulse Pathology Progress Report 8: 19.<br />
Nogales Moncada, A. M. 1997. Compatibilidad vegetativa en <strong>Fusarium</strong><br />
oxysporum f. sp. ciceris and <strong>Fusarium</strong> oxysporum f. sp. melonis agentes,<br />
respectivamente, <strong>de</strong> las Fusariosis vasculares <strong>de</strong>l garbanzo y melón.<br />
Tesis Doctoral. Universidad <strong>de</strong> Córdoba, Córdoba, 216 pp.<br />
O’Donnell, K. O., Kistler, H. C., Cigelnik, E., y Ploetz, R. C. 1998.<br />
Multiple evolutionary origins of the fungus causing Panama disease of<br />
banana: Concordant evi<strong>de</strong>nce from nuclear and mitochondrial gene<br />
genealogies. Proceedings of the National Aca<strong>de</strong>my of Sciences 95:<br />
2044-2049.<br />
Okuda, M., Ikeda, K., Namiki, F., Nishi, K., y Tsuge, T. 1998. Tfo1: An<br />
Ac-like transposon from the plant pathogenic fungus <strong>Fusarium</strong><br />
oxysporum. <strong>Molecular</strong> and General Genetics 258: 599-607.<br />
Paran, I., y Michelmore, R. W. 1993. Development of reliable PCR-based<br />
markers linked to downy mil<strong>de</strong>w resistance genes in lettuce. Theoretical<br />
and Applied Genetics 85: 985-993.<br />
Pérez Artés, E., González Roncero, M. I., y Jiménez Díaz, R. M. 1995.<br />
Restriction fragment length polymorphism analysis of the<br />
mitochondrial DNA of <strong>Fusarium</strong> oxysporum f. sp. ciceris. Journal of<br />
Phytopathology 143: 105-109.<br />
Pérez Artés, E., Rodríguez Jurado, D., y Jiménez Díaz, R. M. 1996.<br />
<strong>Caracterización</strong> <strong>de</strong> aislados <strong>de</strong> <strong>Fusarium</strong> oxysporum no patogénicos <strong>de</strong><br />
garbanzo mediante análisis <strong>de</strong> polimorfismos <strong>de</strong> ADN. En: Resúmenes,<br />
166
Bibliografía<br />
Congreso <strong>de</strong> la Sociedad Española <strong>de</strong> Fitopatología, VIII, Córdoba,<br />
España: 78.<br />
Phillips, J. C. 1988. A distinct race of chickpea wilt in California.<br />
International Chickpea Newsletter 18: 19-21.<br />
Plyler, T. R., Simone, G. W., Fernan<strong>de</strong>z, D., y Kistler, H. C. 2000. Genetic<br />
diversity among isolates of <strong>Fusarium</strong> oxysporum f.sp. canariensis. Plant<br />
Pathology 49 155-164.<br />
Rae<strong>de</strong>r, U., y Broda, P. 1985. Rapid DNA preparation from filamentous<br />
fungi. Letters of Applied Microbiology 1: 17-20.<br />
Raheja, P. C., y Das, G. P. 1957. Development studies in crop plants II.<br />
Effect of cultural treatments on the inci<strong>de</strong>nce of gram wilt. Indian<br />
Journal of Agricultural Science 27: 237-250.<br />
Reddy, M. V., Raju, T. N., y Nene, N. Y. 1990. Evaluation of chickpea<br />
lines in the All Indian Coordinated Varietal Trials for wilt and root rots<br />
resistance. International Chickpea Newsletter 23:22.<br />
Rieseberg, L. H. 1996. Homology among RAPD fragments in<br />
interespecific comparisons. <strong>Molecular</strong> Ecology 5: 99-105.<br />
Rohlf, F. J. 1988. NTSYS-pc: Numerical Taxonomy and Multivariate<br />
Analysis System. Version 1.80. Exeter Software, Setauket, NY, EEUU.<br />
Roumen, E., Levy, M., y Notteghem, J. L. 1997. Characterisation of the<br />
European pathogen population of Magnaporthe grisea by DNA<br />
fingerprinting and pathotype analysis. European Journal of Plant<br />
Pathology 103: 363-371.<br />
Sambrook, J., Fritsch, E. F., y Maniatis, T. (eds.) 1989. <strong>Molecular</strong> Cloning:<br />
A Laboratory Manual, 2ª ed. CSHL Press. Cold Spring Harbor, Nueva<br />
York, EEUU.<br />
Schilling, A. G., Möller, E. M., y Geiger H. H. 1996. Polymerase chain<br />
reaction-based assays for species-specific <strong>de</strong>tection of <strong>Fusarium</strong><br />
culmorum, F. graminearum, y F. avenaceum. Phytopathology 86: 515-522.<br />
167
Bibliografía<br />
Shull, V., y Hamer, J. E. 1996. Rearrangements at a DNA-fingeprint locus<br />
in the rice blast fungus. Current Genetics 30: 263-271.<br />
Sidhu, J. S., Singh, K. B., y Slinkard, A. E. 1983. Inheritance of resistance<br />
to <strong>Fusarium</strong> wilt in chickpea. Journal of Heredity 74: 68.<br />
Singh, K. B. 1987. Chickpea breeding. En: The Chickpea: 127-162. M. C.<br />
Saxena, y K. B. Singh, eds. CAB International. Wallingford, Oxon, UK.<br />
Singh, K. B. 1988. Prospects of <strong>de</strong>veloping new genetic material and<br />
breeding methodologies for chickpea improvement. Proceedings of the<br />
International Workshop on Present Status and Future Prospects of<br />
Chickpea Crop Production and Improvement in the Mediterranean<br />
Countries. CIHEAM/EEU AGRIMED/ICARDA.<br />
Singh, K. B. 1997. Chickpea (Cicer arietinum L.). Field Crops Research 53:<br />
161- 170.<br />
Singh, K. B., y Dahiya, K. B. 1973. Breeding for wilt resistance in<br />
chickpea. Symposium on wilt problems and breeding for wilt resistance<br />
in Bengal gram (Abstract): 13-14. Indian Agricultural Research<br />
Institute, Nueva Delhi, India.<br />
Singh, K. B., y Reddy, M. V. 1991. Advances in disease resistance breeding<br />
in chickpea. Advances in Agronomy 45: 191-222.<br />
Singh, K. B., Kumar, J., Haware, M. P., y Smithson, J. B. 1987a. Genetics<br />
of resistance to <strong>Fusarium</strong> wilt in chickpea. En: Genetics and Plant<br />
Pathogenesis: 339-344. P. R. Day y G. J. Jellis (eds.). Blackwell Scientific<br />
Publications, Oxford, UK.<br />
Singh, K. B., Kumar, J., Smithson, J. B., y Haware, M. P. 1987b.<br />
Complementation between genes for resistance to race 1 of <strong>Fusarium</strong><br />
oxysporum f. sp.ciceri in chickpea. Plant Pathology 36: 539-543.<br />
Sneath, P., y Sokal, R. 1973. Numerical Taxonomy: the Principles and<br />
Practice of Numerical Classification. W.H. Freeman and Co., San<br />
Francisco, CA, EEUU.<br />
168
Bibliografía<br />
Sny<strong>de</strong>r, W. C., y Hansen, H. N. 1940. The species concept in <strong>Fusarium</strong>.<br />
American Journal of Botany 27: 64-67.<br />
Steenkamp, E., Britz, H., Coutinho, T., Wingfield, B., Marasas, W., y<br />
Wingfield, M. 2000. <strong>Molecular</strong> characterization of <strong>Fusarium</strong> subglutinans<br />
associated with mango malformation. <strong>Molecular</strong> Plant Pathology 1:<br />
187-193.<br />
Stepanova, M. Y. 1971. Spread of <strong>Fusarium</strong> oxysporum in legumes. Trudy.<br />
Ves. Inst. Zashch. 29: 100.<br />
Stern, M. J., Ames, G. F. L., Smith, N. H., Robinson, E. C., y Higgins, C.<br />
F. 1984. Repetitive extragenic palindromic sequences, a major<br />
component of the bacterial genome. Cell 37: 1015-1026.<br />
Swofford, D. L. 2000. PAUP*. Phylogenetic Analysis Using Parsimony<br />
(*and other methods). Version 4. Sinauer Associates, Sun<strong>de</strong>rland, MA,<br />
EEUU.<br />
Tantaoui, A., Ouiten, M., Geiger, J. P., y Fernan<strong>de</strong>z, D. 1996.<br />
Characterization of a single clonal lineage of <strong>Fusarium</strong> oxysporum f. sp.<br />
albedinis causing bayoud disease of date palm in Morocco.<br />
Phytopathology 86: 787-792.<br />
Taylor, J. W., Geiser, D. M., Burt, A., y Koufopanou, V. 1999a. The<br />
evolutionary biology and population genetics un<strong>de</strong>rlying fungal strain<br />
typing. Clinical Microbiology Reviews 12: 126-146.<br />
Taylor, J. W., Jacobson, D. J., y Fischer, M. C. 1999b. The evolution of<br />
asexual fungi: reproduction, speciation and classification. Annual<br />
Review Phytopathology 37: 197-246.<br />
Thormann, C. E., Ferreira, M. E., Camargo, L. E. A., Tivang, J. G., y<br />
Osborn, T. C. 1994. Comparison of RFLP and RAPD markers to<br />
estimate genetic realtionships withing and among cruciferous species.<br />
Theoretical and Applied Genetics 88: 973-980.<br />
Trapero Casas, A. 1983. La Marchitez y Podredumbre <strong>de</strong> la Raíz <strong>de</strong>l<br />
garbanzo en el Valle <strong>de</strong>l Guadalquivir: Importancia, distribución,<br />
169
Bibliografía<br />
170<br />
etiología, epi<strong>de</strong>miología y medios <strong>de</strong> lucha. Tesis Doctoral, Universidad<br />
<strong>de</strong> Córdoba, Córdoba, 295 pp.<br />
Trapero Casas, A., y Jiménez Díaz, R. M. 1985. Fungal wilt and root rot<br />
diseases of chickpea in Southern Spain. Phytopathology 75: 1146-1151.<br />
Trapero Casas, A., y Jiménez Díaz, R. M. 1986 a. La ‘Seca’ <strong>de</strong>l garbanzo en<br />
Andalucía. Comunicaciones Agrarias. Serie Protección Vegetal, nº 4: 64.<br />
Trapero Casas, A., y Jiménez Díaz, R. M. 1986 b. Influence of sowing date<br />
on <strong>Fusarium</strong> wilt and Ascochyta blight of chickpea in southern Spain.<br />
Abstracts, International Food Legume Research Conference on Pea,<br />
Lentil, Faba bean and Chickpea. Spokane, Washington, USA. College<br />
of Agriculture, University of Idaho, USA.<br />
Upadhyaya, H. D., Haware, M. P., Kumar, J., y Smithson, J. B. 1983a.<br />
Resistance to wilt in chickpea. I. Inheritance of late-wilting in response<br />
to race 1. Euphytica 32: 447-452.<br />
Upadhyaya, H. D., Smithson, J. B., Haware, M. P., y Kumar, J. 1983b.<br />
Resistance to wilt in chickpea. II. Further evi<strong>de</strong>nce for two genes for<br />
resistance to race 1. Euphytica 32: 749-755.<br />
van Rheenen, H. A., Reddy, M. V., Kumar, J., y Haware, M. P. 1992.<br />
Breeding for resistance to soil-borne diseases in chickpea. En: Disease<br />
Resistance Breeding in Chickpea: 57-60. K. B. Singh y M. C. Saxena,<br />
eds. ICARDA, Aleppo, Siria.<br />
Ward, E. 1994. Use of polymerase chain reaction for i<strong>de</strong>ntifying plant<br />
pathogens. En: Ecology of Plant Pathogens: 143-160. J. P. Blakeman y<br />
B. Williamson, eds. CAB International, Oxon, UK.<br />
White, T. J., Bruns, T., Lee, S., y Taylor, J. 1990. Amplification and direct<br />
sequencing of fungal ribosomal RNA genes for phylogenetics. Páginas<br />
315-322 en: PCR Protocols. A Gui<strong>de</strong> to Methods and Applications. M.<br />
A. Innis, D. H. Gelfand, J. J. Sninsky, y T. J. White, eds. Aca<strong>de</strong>mic<br />
Press, Inc., San Diego, CA, EEUU.
Bibliografía<br />
Whitehead, D. S., Coddington, A., y Lewis, B. G. 1992. Classification of<br />
races by DNA polymorphism analysis and vegetative compatibility<br />
grouping in <strong>Fusarium</strong> oxysporum f. sp. pisi. Physiological and <strong>Molecular</strong><br />
Plant Pathology 41: 295-305.<br />
Wiglesworth, M. D., Nesmith, W. C., Schardl, C. L., Li, D. y Siegel, M. R.<br />
1994. Use of specific repetitive sequences in Peronospora tabacina for early<br />
<strong>de</strong>tection of the tobacco blue mold pathogen. Phytopathology 84: 425-<br />
430.<br />
Williams, J. G. K., Kubelik, A. R., Livak, K. J., Rafolski, J. A. y Tingey, S.<br />
V. 1990. DNA polymorphisms amplified by arbitrary primers are useful<br />
as genetic markers. Nucleic Acids Research 18: 6531-6535<br />
Woo, S. L., Zoina, A., <strong>de</strong>l Sorbo, G., Lorito, M., Nanni, B., Scala, F., y<br />
Noviello, C. 1996. Characterization of <strong>Fusarium</strong> oxysporum f. sp. phaseoli<br />
by pathogenic races VCGs, RFLPs, and RAPDs. Phytopathology 86:<br />
966-973.<br />
Zeigler, R. S., Cuoc, L. X., Scott, R. P., Bernardo, M. A., Chen, D. H.,<br />
Valent, B., y Nelson, R. J. 1995. The relationship between lineage and<br />
virulence in Pyricularia grisea in the Philippines. Phytopathology 85: 443-<br />
451.<br />
171