Help es V e IV Genetica Laura 2012 - web matic
Help es V e IV Genetica Laura 2012 - web matic
Help es V e IV Genetica Laura 2012 - web matic
You also want an ePaper? Increase the reach of your titles
YUMPU automatically turns print PDFs into web optimized ePapers that Google loves.
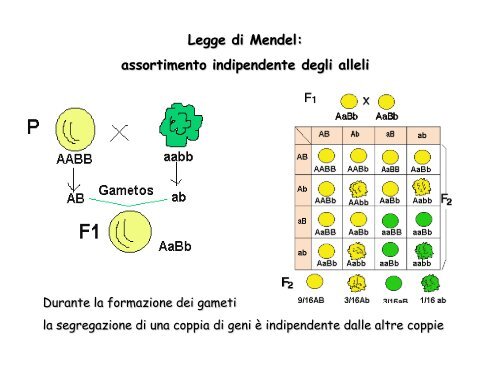
Legge di Mendel:<br />
assortimento indipendente degli alleli<br />
Durante la formazione dei gameti<br />
la segregazione di una coppia di geni è indipendente dalle altre coppie
Da cosa dipende il chi-quadro?<br />
Χ 2 = Σ<br />
(foss – fatt) 2<br />
fatt<br />
1. Dagli scarti fra valori osservati e att<strong>es</strong>i<br />
2. Dal numero di addendi<br />
Gradi di libertà: n-1
RICOMBINAZIONE:<br />
qualunque proc<strong>es</strong>so che generi<br />
un prodotto diverso dai genotipi parentali<br />
•RICOMBINAZIONE GENERALE O OMOLOGA:<br />
Avviene di solito fra <strong>es</strong>t<strong>es</strong>e regioni di sequenze nucleotidiche<br />
omologhe<br />
•RICOMBINAZIONE SITO SPECIFICA:<br />
I siti di rottura e riunione tra due molecole di DNA (o tra due<br />
segmenti della st<strong>es</strong>sa molecola) sono compr<strong>es</strong>i all’interno di<br />
specifiche regioni molto brevi (< 25 nucleotidi)<br />
•RICOMBINAZIONE NON OMOLOGA:<br />
Evento piuttosto raro che coinvolge sequenze nucleotidiche non<br />
omologhe.
NO CROSSING OVER<br />
A Cm<br />
A Cm Meioisi I<br />
a cm<br />
a cm<br />
CROISSNG OVER<br />
A Cm<br />
A cm<br />
a Cm<br />
a cm<br />
LEZIONE 4 GENI CONCATENATI<br />
A Cm<br />
A Cm<br />
a cm<br />
a cm<br />
A Cm<br />
A cm<br />
Meioisi II<br />
Meioisi I Meioisi II<br />
a Cm<br />
a cm<br />
A Cm<br />
A Cm<br />
a cm<br />
a cm<br />
gameti<br />
A Cm<br />
A cm<br />
a cm<br />
a cm<br />
gameti<br />
L’analisi di linkage si basa sulla co-segregazione alla meiosi di 2 o più loci più<br />
frequentemente di quanto ci si aspetta per caso. Se 2 loci vengono ereditati insieme<br />
frequentemente, è probabile che siano localizzati vicini fra di loro sul cromosoma<br />
NR<br />
R<br />
R<br />
NR
Il mezzo ideale per Studiare associazione e<br />
ricombinazione è il reincrocio<br />
Rapporti fenotipici att<strong>es</strong>i (diibrido): 1: 1: 1: 1<br />
F(P) = F(NP) = 0,5<br />
Se c’è associazione: F(P) > 0,5, F(NP)
A B<br />
a b<br />
RAPPRESENTAZIONE GRAFICA DEI CROMOSOMI<br />
DI UN IND<strong>IV</strong>IDUO DIIBRIDO AaBb<br />
A B<br />
a b<br />
Forma CIS<br />
Entrambe gli alleli dominanti<br />
su un cromosoma<br />
e i rec<strong>es</strong>sivi sull’omologo<br />
A B<br />
a b<br />
a b<br />
A B<br />
A b<br />
a B<br />
Forma TRAS<br />
Su ogni cromosoma sono concatenati<br />
un allele dominante e uno rec<strong>es</strong>sivo<br />
VARI MODI DI SCR<strong>IV</strong>ERE LA STESSA COSA: PRENDIAMO AD ESEMPIO LA FORMA CIS<br />
A a<br />
B b<br />
B A<br />
b a<br />
ERRATO<br />
PERCHÉ<br />
I DUE ALLELI DELLO STESSO GENE<br />
SONO SULLO STESSO CROMOSOMA<br />
A a<br />
B b
Reincrocio<br />
Aa Bb X aa bb<br />
Frequenze fenotipiche: AB Ab aB ab TOT<br />
Osservate 78 122 118 82 400<br />
Att<strong>es</strong>e 100 100 100 100 400<br />
I fenotipi Ab e aB sono pr<strong>es</strong>enti in frequenze superiori all’att<strong>es</strong>a<br />
Perciò Ab e aB sono i fenotipi parentali:<br />
A b<br />
a B<br />
La distanza fra locus A e locus B è<br />
A B<br />
Trans cis<br />
a b<br />
d = NR / NT = (78+82)/400 = 0,4 frequenza-cioè 40 cM
Se tutti i cromosomi subiscono crossing-over alla meiosi, la frequenza di<br />
ricombinazione è del 50%<br />
NR non può <strong>es</strong>sere > ½ NT<br />
(in qu<strong>es</strong>to caso, si considerano i geni indipendenti)<br />
Per cui d MAX = 0,5.<br />
La frequenza di ricombinazione è metà della frequenza<br />
di crossing over
ANALISI DI LINKAGE DI CARATTERI MENDELIANI<br />
� Determinare la frequenza con cui due loci ricombinano fra loro alla<br />
meiosi = TETHA (��<br />
TETHA (�� =<br />
Num Ricombinanti<br />
Num Totale di individui<br />
• Se due loci sono su cromosomi diversi segregano indipendentemente. La probabilità<br />
che vengano ereditati insieme è del 1/2 -> � = 50%<br />
• e due loci sono vicini fra loro sullo st<strong>es</strong>so cromosoma saranno ereditati insieme più<br />
frequentemente -> � < 50%<br />
Tanto più sono vicini, tanto più piccola è la probabilità che avvenga un crossing-over<br />
LA FREQUENZA DI RICOMBINAZIONE (�)<br />
E’ UNA MISURA DELLA DISTANZA GENETICA
Problema. 1.<br />
Due geni che influenzano le caratteristiche del baccello nel fagiolo<br />
risiedono sul cromosoma 5. Il baccello stretto è rec<strong>es</strong>sivo rispetto al<br />
baccello normale; il baccello bianco è rec<strong>es</strong>sivo rispetto al baccello<br />
chiazzato. Una pianta di linea pura con baccelli stretti e chiazzati viene<br />
incrociata con una pianta di linea pura, a baccelli normali e bianchi. Gli<br />
individui di F1 sono poi incrociati con individui a baccello stretto e bianco,<br />
con qu<strong>es</strong>ti risultati:<br />
144 baccelli stretti e chiazzati<br />
150 baccelli normali e bianchi<br />
11 baccelli normali e chiazzati<br />
9 baccelli stretti e bianchi.<br />
(a) Stimare la distanza fra i due loci<br />
(b) Disegnare i cromosomi 5 in ognuna delle 4 classi fenotipiche della F2.
Parentali<br />
S b<br />
s B<br />
S B<br />
s b<br />
Ricombinanti<br />
stretto (s) < (S) NORMALE<br />
bianco (b) < (B) CHIAZZATO<br />
stretto<br />
CHIAZZATO<br />
0,063 = 6,3 cM<br />
(ssBB) (SSbb)<br />
100% (SsBb)<br />
s B<br />
Parentali<br />
NORMALE<br />
NORMALE<br />
bianco<br />
CHIAZZATO<br />
(SsBb) stretto bianco<br />
(ssbb)<br />
s b<br />
s b<br />
stretto NORMALE stretto NORMALE<br />
CHIAZZATO BIANCO<br />
bianco<br />
Chiazzato<br />
144 150 11 9<br />
s B<br />
s b<br />
S b<br />
s b<br />
S B<br />
s b s b<br />
Ricombinanti
Procmlema 2.<br />
Nei gatti due geni mutanti dominanti determinano i caratteri coda mozza (Cm) e pelo<br />
ispido (I). Quando gatti eterozigoti per entrambe i geni con coda mozza e pelo ispido<br />
sono stati incrociati con gatti normali, la loro progenie è risultata divisa nei seguenti<br />
quattro fenotipi:<br />
79 con pelo ispido,<br />
103 normali,<br />
95 con pelo ispido e coda mozza,<br />
75 con coda mozza.<br />
(a) Si determinino i genotipi dei genitori;<br />
(b)Calcolare la frequenza di ricombinazione fra Cm e I.
Parentali<br />
I Cm<br />
i cm<br />
I cm<br />
i Cm<br />
Ricombinanti<br />
liscio<br />
Coda mozza<br />
(IiCmcm)<br />
ispido<br />
Coda mozza<br />
i cm i cm<br />
Parentali<br />
(iicmcm)<br />
liscio Coda normale<br />
(iiCmcm) (IiCmcm) (iicmcm) (Iicmcm)<br />
i cm<br />
Pelo ISPIDO (I) > (i) liscio<br />
Coda MOZZA (Cm) > (cm) normale<br />
ispido<br />
Coda mozza<br />
I Cm<br />
liscio<br />
Coda normale<br />
i Cm<br />
i cm<br />
i cm<br />
ispido<br />
Coda normale<br />
103 95 75 79<br />
Freq di ric=<br />
75+79<br />
75+79+103+95<br />
I cm<br />
i cm i cm<br />
Ricombinanti<br />
= 0,44
Problema 3.<br />
Consideriamo i dati di un reincrocio tra piantine di fragole. Nelle fragole, v è una<br />
mutazione autosomica rec<strong>es</strong>siva che determina la forma del allungata del frutto, e g è<br />
una mutazione autosomica rec<strong>es</strong>siva che ne determina il colore giallo della polpa. Le<br />
fragole selvatiche hanno la polpa rossa e forma arrotondata. Linee pure di fragole a<br />
frutto allungato e polpa rossa (v/v e g+/g+) furono incrociate con fragole a corpo<br />
arrotondato e polpa gialla ((v+/v+ e g/g). Piantine della F1 con frutto arrotondato a<br />
polpa rossa furono incrociate con piantine a polpa gialla e frutto allungato. I risultato<br />
su un totale di 3236 piantine pr<strong>es</strong>enta:<br />
283 con frutto rosso, arrotondato<br />
1294 con frutto rosso, allungato<br />
1418 con frutto giallo, arrotondato<br />
241 con frutto giallo, allungato<br />
(a) Verificare se i geni segregano indipendentemente.<br />
(cm) Calcolare l’unità di mappa che distanzia i due geni.
a) Per verificare se i due geni segregano indipendentemente bisogna<br />
o calcolare il Χ 2<br />
Freq att<strong>es</strong>e = 241+283+1294+1418 = 809 per fenotipo<br />
4<br />
(809-241) 2 (809-283) 2 (809-1294) 2 (809-1418) 2<br />
Χ<br />
809 809 809 809<br />
2 = + + + = 1400<br />
df = 4-1 = 3<br />
Devo rifiutare l’ipot<strong>es</strong>i nulla quindi non segregano indipendentemente<br />
O calcolare la distanza di mappa e verificare che non sia uguale a 50 cM<br />
b) Distanza fra i due loci<br />
283 frutto rosso, arrotondato 1294 frutto rosso, allungato<br />
241 frutto giallo, allungato 1418 frutto giallo, arrotondato<br />
Ricombinanti<br />
Parentali<br />
241+283<br />
Dist di mappa = x 100 = 16,2 cM<br />
241+283+1294+1418
Problema 4.<br />
Negli studenti del primo anno di Biologia sono stati identificati due geni<br />
mutanti dominanti che determinano i caratteri repulsione allo studio della<br />
genetica (Rg) e voglia di andare al mare (M). La classe è risultata divisa nei<br />
seguenti quattro fenotipi:<br />
74 repulsione allo studio, voglia di andare al mare<br />
14 repulsione allo studio, n<strong>es</strong>suna voglia di andare al mare<br />
13 propensione allo studio, voglia di andare al mare<br />
79 propensione allo studio, n<strong>es</strong>suna voglia di andare al mare<br />
Considerato che per ogni ragazzo uno dei due genitori pr<strong>es</strong>enta genotipo<br />
eterozigote per entrambi i geni e l’altro è omozigote rec<strong>es</strong>sivo per entrambe<br />
i geni:<br />
(a) si determinino i parentali;<br />
(b) si calcoli la distanza di mappa fra Rg e M.
Parentali<br />
Rg M<br />
rg m<br />
Rg m<br />
rg M<br />
Ricombinanti<br />
Repulsione studio (Rg) > (rg) propensione<br />
Voglia di Mare (M) > (m) no mare<br />
(Rgrg Mm) (rgrg mm)<br />
(RgrgMm) (rgrgmm) (Rgrg mm) (rgrg Mm)<br />
Repulsione<br />
Voglia mare<br />
Rg M<br />
rg m<br />
Repulsione<br />
Voglia mare<br />
Parentali<br />
propensione<br />
no mare<br />
rg m<br />
rg m<br />
propensione<br />
no mare<br />
Repulsione<br />
no mare<br />
Rg m<br />
rg m<br />
Ricombinanti<br />
rg m<br />
rg m<br />
propensione<br />
voglia mare<br />
74 79 14 13<br />
rg M<br />
rg m<br />
Dist di mappa = x 100 = 15 cM<br />
14+13<br />
74+79+14+13
LEZIONE 7<br />
Incrocio a tre punti
Problema 1. Nella pianta di cacao tre geni rec<strong>es</strong>sivi, associati sul med<strong>es</strong>imo<br />
cromosoma, codificano per il colore dei semi bianchi (w) o violetti (W), fiori<br />
bianco-rosei (sc) o scarlatti (Sc) e per il colore de frutto giallo (v) o verde<br />
(V). Una pianta di cacao omozigote per tutti e tre gli alleli rec<strong>es</strong>sivi è<br />
incrociata con una pianta omozigote per tutti gli alleli dominanti. La F1<br />
risultante è in seguito reincrociata con una pianta omozigote per gli alleli<br />
rec<strong>es</strong>sivi in un incrocio di controllo a tre punti, da cui si ottiene la seguente<br />
progenie:<br />
w sc V 87<br />
W Sc v 94<br />
W Sc V 3479<br />
w sc v 3478<br />
W sc V 1515<br />
w Sc v 1531<br />
w Sc V 292<br />
W sc v 280<br />
_____________________________<br />
totale 10756<br />
a) Determinare l’ordine dei geni sui cromosomi.<br />
b) Calcolate le distanze di mappa tra i diversi geni.<br />
c) Calcolate se <strong>es</strong>iste interferenza.
Incrocio a tre punti<br />
Prima di tutto identificare quali sono i parentali e quali i doppi ricombinanti<br />
Qu<strong>es</strong>to serve a capire l’ordine dei geni!<br />
Doppi ricombinanti<br />
Meno frequenti<br />
Parentali<br />
Più frequenti<br />
w sc V 87<br />
W Sc v 94<br />
W Sc V 3479<br />
w sc v 3478<br />
W sc V 1515<br />
w Sc v 1531<br />
w Sc V 292<br />
W sc v 280<br />
Gene centrale<br />
W V Sc<br />
w v sc
Stabilito qual è l’ordine dei geni si può calcolare la distanza di mappa:<br />
W V Sc<br />
w v sc<br />
Tra W e V<br />
Dove la distanza di mappa si <strong>es</strong>prime con la frequenza di ricombinazione<br />
dei ricombinanti W v sc 280<br />
w V Sc 292<br />
dei doppi ricombinanti W v Sc 94<br />
w V sc 87<br />
753<br />
Distanza di mappa<br />
ric 1°reg + d ric<br />
= F (ric) x100 = x100 = 753<br />
Prima regione<br />
x 100 = 7 %<br />
totale<br />
10756
Calcolata la distanza tra W e v si procede a calcolare la distanza:<br />
W<br />
7 cM<br />
V Sc<br />
w v sc<br />
Tra V e Sc<br />
Dove la distanza di mappa si <strong>es</strong>prime con la frequenza di ricombinazione<br />
dei ricombinanti W V sc 1515<br />
w v Sc 1531<br />
dei doppi ricombinanti W v Sc 94<br />
w V sc 87<br />
3227<br />
Distanza di mappa<br />
= F (ric) x100 =<br />
ric 2°reg + d ric<br />
x100 =<br />
3227<br />
Seconda regione<br />
x 100 = 30 %<br />
totale<br />
10756
Stabilito qual è la distanza di mappa fra i tre geni<br />
Ora si può valutare quale sia l’INTERFERENZA DI CROSSING-OVER.<br />
Prima si calcola il<br />
W<br />
7 cM<br />
V<br />
30 cM<br />
Sc<br />
w v sc<br />
Coefficiente di coincidenza:<br />
Frequenza att<strong>es</strong>a<br />
del doppio<br />
ricombinante<br />
Probabilità che possa<br />
avvenire un Crossing over<br />
in qu<strong>es</strong>ta prima regione<br />
Frequenza dei doppi ricombinanti<br />
Distanza di mappa<br />
Prima regione<br />
100<br />
Distanza di mappa<br />
seconda regione<br />
100<br />
Probabilità che possa<br />
avvenire un Crossing over<br />
in qu<strong>es</strong>ta seconda regione
Interferenza<br />
F (ric. oss)=<br />
94+87<br />
= 0,017<br />
10756<br />
Coeff. di coinc = = 0,8<br />
F (ric. att)= 0,07 X 0.30 = 0,021<br />
INTERFERENZA = 1 - Coeff. di coinc. = 1- 0,8 = 0,2<br />
CHE SIGNIFICA CHE C’E’ UN 20% DI INTERFERENZA<br />
Viene da sé che se il Coeff di coincidenza è uguale a 1<br />
Allora non c’è interferenza<br />
Per cui meno coincidono i valori osservato rispetto agli att<strong>es</strong>i<br />
Più importante sarà l’interferenza tra i crossing-over
Problema 2.<br />
Nei pinguini il carattere guancia e gola neri (B) è dominante sul colore bianco (b), sullo<br />
st<strong>es</strong>so cromosoma sono pr<strong>es</strong>enti i l carattere doppia banda pettorale (A) dominante<br />
sulla colorazione omogenea (a) e il carattere piumaggio blu-grigio (c) rec<strong>es</strong>sivo rispetto<br />
al colore nero (C).<br />
Dall’incrocio di individui a piumaggio blu-grigio, pettorale di colore omogeneo e guancia<br />
e gola bianchi con individui eterozigoti a piumaggio nero, pettorale con doppia banda<br />
bianco nera e guancia e gola neri è risultata la seguente progenie:<br />
fenotipo progenie N. Individui<br />
ABC 70<br />
abc 70<br />
abC 310<br />
ABc 310<br />
AbC 100<br />
aBc 100<br />
Abc 20<br />
aBC 20<br />
_______________________________________<br />
Totale 1000<br />
Determinate il genotipo degli individui incrociati;l’ordine dei geni, le rispettive distanze, e<br />
il coefficiente di interferenza.
Abc 310<br />
abC 310<br />
AB =<br />
BC =<br />
Abc 20<br />
aBC 20<br />
parentali Doppi ricomb<br />
20+20+100+100<br />
1000<br />
20+20+70+70<br />
1000<br />
x100 = 24 cM<br />
x100 = 18 cM<br />
AbC 100<br />
aBc 100<br />
ABC 70<br />
abc 70<br />
Ricomb 1° R Ricomb 2° R<br />
24 cM<br />
18 cM<br />
A B C<br />
F (ric. oss)= 20+20 = 0,04<br />
1000<br />
Interferenza = 1- = 0,074<br />
F (ric. att)= 24 X 18 = 0,0432<br />
100 100
Problema 3.<br />
Reincrocio di un quadruplo eterozigote con il parentale rec<strong>es</strong>sivo. E’ noto l’ordine dei geni.<br />
Si osservano:<br />
Non ricombinanti 486 Stimare le distanze di mappa<br />
Ricombinanti in I regione 61<br />
Ricombinanti in II regione 143<br />
Ricombinanti in III regione 226<br />
Ricombinanti in I e II regione 10<br />
Ricombinanti in I e III regione 45<br />
Ricombinanti in II e III regione 27<br />
Ricombinanti in I, II e III regione 2<br />
Totale 1000<br />
Stimare l’interferenza.<br />
AB =<br />
BC =<br />
CD =<br />
61+10+45+2<br />
1000<br />
143+10+27+2<br />
1000<br />
226+45+27+2<br />
1000<br />
11,8 cM<br />
18,2 cM 30 cM<br />
A B C<br />
F (3 ric. oss)= 2 = 0,002<br />
1000<br />
Interferenza = 1- = 0,7<br />
F (ric. att)= 11,8 x 18,2 x 30 = 0,0432<br />
100 100 100<br />
x100 = 11,8 cM<br />
x100 = 18,2 cM<br />
x100 = 30 cM<br />
D
Problema. 4.<br />
Data una distanza di mappa A-B =10 e B-C = 20:<br />
A B C<br />
10 cM 20 cM<br />
a) Data una percentuale di doppi ricombinanti pari a 1.6% che valore ha l’interferenza?<br />
F (ric. oss)= 1,6 = 0,016<br />
Interferenza = 1- 100<br />
= 0,2<br />
10 20<br />
F (ric. att)= X = 0,02<br />
100 100<br />
b) Dato un valore di 40% di interferenza quanti sono i doppi ricombinanti?<br />
E quanto è la percentuale dei parentali?<br />
F (ric. oss) = ?<br />
Interferenza = 1- = 40 = 0,4<br />
10 x 20 100<br />
100 100<br />
x<br />
- = -1 + 0,04 x = 0,06 x 0,02 x = 1,2<br />
0,02<br />
% Parentali = 100 - ric 1° - ric 2° - D ric<br />
1 – = 0,04<br />
x<br />
0,02<br />
Ric Oss = 1,2 %<br />
100 - ric 1°reg + 1,2<br />
x100 = 10 -<br />
ric 2°reg + 1,2 x100 = 20 - 1,2 = 71,2 %<br />
100<br />
100
E<br />
Problema. 5.<br />
Dato il seguente schema di distanze di mappa. Disegnare l’ordine dei geni.<br />
B C D E F<br />
A 25 1 19 7 20<br />
B 26 6 32 5<br />
C 20 6 21<br />
D 26 1<br />
E 27<br />
C<br />
1 cM<br />
A 25 cM<br />
B<br />
A 26 cM B<br />
C 20 cM A 19 cM D 6 cM B<br />
6 cM<br />
C 7 cM A 26 cM D<br />
B<br />
32 cM<br />
1 cM 5 cM<br />
E C A 20 cM D F B<br />
27 cM 21 cM