Genomica Lez.2.pdf - Dipartimento di Biologia
Genomica Lez.2.pdf - Dipartimento di Biologia
Genomica Lez.2.pdf - Dipartimento di Biologia
You also want an ePaper? Increase the reach of your titles
YUMPU automatically turns print PDFs into web optimized ePapers that Google loves.
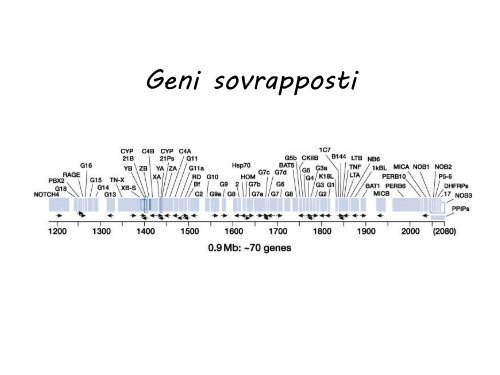
Geni sovrapposti
Lo splicing alternativo aumenta in modo considerevole la complessità del<br />
trascrittoma (e quin<strong>di</strong> del proteoma).
Splicing Alternativo<br />
Oltre il 90% dei geni umani è in grado <strong>di</strong> esprimere più <strong>di</strong> un trascritto (ed è<br />
quin<strong>di</strong> soggetto a splicing alternativo). Le <strong>di</strong>verse isoforme <strong>di</strong> splicing possono<br />
avere specificità a livello <strong>di</strong> tessuto, <strong>di</strong> con<strong>di</strong>zione fisiologica, o patologica.<br />
%<br />
35<br />
30<br />
25<br />
20<br />
15<br />
10<br />
5<br />
0<br />
17,635 Human genes<br />
1 2 -5 6 -1 0 1 1 -2 0 2 1 -3 0 3 1 -5 0 >5 0<br />
Number of Transcripts/ Gene
Uno stesso gene può esprimere proteine con funzioni<br />
opposte: l’esempio dell’attività della Caspasi 9 (CASP9)<br />
La forma costitutiva della proteina (CASP9, 9 esoni, 416 aa) induce<br />
apoptosi. Essa contiene un Caspase recruitment domain (CARD) e un dominio<br />
caspasi Peptidase_C14.<br />
L’isoforma più corta della proteina (CASP9S, 5 esoni, 266 aa) contiene un<br />
dominio Caspase recruitment domain (CARD) e un dominio tronco della<br />
Peptidase_C14. Questa isoforma è priva dell’attività proteasica e agisce da<br />
inibitore dell’apoptosi.
Uno stesso gene può co<strong>di</strong>ficare per proteine in<strong>di</strong>rizzate a<br />
<strong>di</strong>versi compartimenti cellulari: l’esempio del gene NFS1<br />
La proteina co<strong>di</strong>ficata dal gene NFS1 fornisce zolfo inorganico ai cluster ferro-zolfo<br />
rimuovendo lo zolfo dalla cisteina, e formando alanina nel processo. Questo gene<br />
utilizza siti <strong>di</strong> inizio alternativi della traduzione per generare una isoforma<br />
mitocondriale ed una isoforma citoplasmatica. La selezione del sito <strong>di</strong> inizio della<br />
trascrizione è regolata dal pH citosolico.<br />
L’isoforma che co<strong>di</strong>fica per la proteina mitocondriale (457 aa) contiene un peptide<br />
segnale e un dominio aminotrasnferasico.<br />
L’altra isoforma, che deriva sa un sito <strong>di</strong> inizio alternativo della trascrizione co<strong>di</strong>fica<br />
per una proteina più corta (397 aa) priva del peptide segnale ma contenente il<br />
dominio aminotransferasico.
Uno stesso gene può co<strong>di</strong>ficare trascritti soggetti ad un <strong>di</strong>verso<br />
meccanismo <strong>di</strong> regolazione post-trascrizionale: l’esempio <strong>di</strong> SLC11A2<br />
Il gene SLC11A2 (<strong>di</strong>valent cation transporter) co<strong>di</strong>fica per (almeno) due <strong>di</strong>verse<br />
isoforme, solo una delle quali risponde alla concentrazione del ferro (i.e. i livelli della<br />
proteina aumentano sensibilmente in seguito alla carenza <strong>di</strong> ferro). Responsabile del<br />
meccanismo <strong>di</strong> regolazione è un “Iron Responsive Element (IRE)” nella regione 3’UTR<br />
presente solo in una delle due isoforme.<br />
IRE<br />
Nell’uomo il trascritto contenente l’IRE (16 exons) co<strong>di</strong>fica per una proteina <strong>di</strong> 561 aa<br />
(NM_000617). Il trascritto privo <strong>di</strong> IRE (17 exons) co<strong>di</strong>fica per una proteina <strong>di</strong> 568<br />
aa.<br />
La stessa situazione si verifica nel topo, per cui l’unica entry RefSeq<br />
corrisponde alla isoforma priva <strong>di</strong> IRE (NM_008732). Il meccanismo <strong>di</strong><br />
risposta al ferro appare specifico del tipo cellulare.<br />
IRE
Famiglie geniche<br />
• Famiglie geniche classiche (istoni, globine)<br />
• Geni co<strong>di</strong>ficanti prodotti con domini altamente<br />
conservati (Homeobox, Paired box, Forkhead,<br />
ecc.)<br />
• Geni co<strong>di</strong>ficanti proteine contenenti corti motivi<br />
conservati, correlati ad una comune funzione<br />
(DEAD box, WD domain, ecc.).<br />
• Superfamiglie (immunoglobuline, recettori G<br />
protein coupled, ecc.) .
DEAD (Asp-Glu-Ala-Asp)<br />
WD (Trp-Asp)
Superfamiglia delle Ig<br />
09_10.jpg
Famiglie geniche<br />
Le famiglie geniche possono essere generate attraverso <strong>di</strong>versi meccanismi:<br />
• poliploi<strong>di</strong>zzazione del genoma (famiglia dei geni omeotici)<br />
• duplicazione <strong>di</strong> segmenti genomici<br />
•duplicazione <strong>di</strong> un singolo gene (geni per a e b globine)<br />
• retrotrascrizione
Duplicazioni geniche: poliploi<strong>di</strong>zzazione (2R)<br />
R1<br />
R2<br />
esaploi<strong>di</strong> Duplicazioni genomiche dedotte<br />
Due “Round” <strong>di</strong> duplicazioni<br />
genomiche nei progenitori dei<br />
vertebrati, probabilmente una<br />
subito prima e una subito dopo<br />
la <strong>di</strong>versificazione degli Agnatha<br />
(lampreda e affini).<br />
One to four<br />
Invertebrati<br />
Vertebrati<br />
Sintenie; cluster <strong>di</strong> geni Hox
Duplicazioni geniche: poliploi<strong>di</strong>zzazione (2R)<br />
Successivamente alla duplicazione genomica possono intervenire eventi <strong>di</strong><br />
acquisto e per<strong>di</strong>ta <strong>di</strong> geni che mo<strong>di</strong>ficano la struttura dei cluster.
Famiglia dei geni omeotici<br />
Il Cluster <strong>di</strong> geni Hox è quadruplicato nei mammiferi rispetto a Drosophila<br />
Drosophila<br />
Vertebrati<br />
Evoluzione della famiglia dei geni omeotici attraverso un processo a due sta<strong>di</strong>:<br />
1) nel primo sta<strong>di</strong>o, eventi <strong>di</strong> duplicazione in cis del gene primor<strong>di</strong>ale hanno<br />
prodotto i <strong>di</strong>versi componenti del cluster negli invertebrati<br />
2) nel secondo sta<strong>di</strong>o, eventi <strong>di</strong> duplicazione in trans dell’intero cluster hanno<br />
prodotti cluster multipli<br />
Nei vertebrati, la duplicazione genica è stata accompagnata da per<strong>di</strong>ta <strong>di</strong> geni
Cromosoma 22<br />
Famiglia dei geni per le globine<br />
Progenitore<br />
proto-a<br />
Progenitore<br />
proto-b<br />
Cromosoma 16 Cromosoma 11
Duplicazione genica<br />
Produzione <strong>di</strong> due copie identiche <strong>di</strong> un gene<br />
Delle due copie, una continua a svolgere la propria funzione,<br />
l’altra può andare incontro a <strong>di</strong>versi destini<br />
Il gene duplicato mantiene la stessa<br />
funzione del gene ancestrale (istoni)<br />
Gene redundancy<br />
L’accumulo <strong>di</strong> mutazioni porta<br />
all’inattivazione del gene duplicato,<br />
trasformandolo in pseudogene<br />
(pseudogeni delle a e b globine)<br />
Destino geni duplicati<br />
Il gene duplicato, non essendo sottoposto<br />
alla stessa pressione selettiva del gene<br />
ancestrale, può accumulare mutazioni<br />
casuali<br />
L’accumulo <strong>di</strong> mutazioni fa sì che il gene<br />
duplicato possa acquisire una nuova<br />
funzione utile per l’organismo<br />
(le nuove funzioni acquisite possono<br />
<strong>di</strong>ventare specie-specifiche)
Una terza alternativa è che tutte le copie possono essere mantenute nella loro<br />
forma originale; l’effetto omogeneizzante dell’evoluzione concertata favorisce<br />
quest’ultima possibilità che agirebbe rallentando la <strong>di</strong>versità genetica e quin<strong>di</strong> il<br />
<strong>di</strong>vergere della funzionalità. Per esempio dall’analisi dei geni delle globine in varie<br />
specie è emerso che questi geni inizialmente sono andati incontro a questo<br />
fenomeno <strong>di</strong> omogeneizzazione me<strong>di</strong>ante conversione genica, ma quando le sequenze<br />
<strong>di</strong>versero sufficientemente per rendere inefficace questo meccanismo, i geni<br />
iniziarono ad evolvere in<strong>di</strong>pendentemente.<br />
Tutti i primati hanno 2 α globine. Assumiamo quin<strong>di</strong> che l’antenato comune dei<br />
primati avesse due geni α-globinici.<br />
• Se α1 e α2 della stessa specie si sono separati circa 300 milioni <strong>di</strong> anni fa<br />
dovrebbero aver accumulato molti cambiamenti AA.<br />
• Si osserva un’alta omogeneità intraspecifica:<br />
la conclusione e che i geni α1 e α2 non si sono evoluti in modo in<strong>di</strong>pendente, ma in<br />
maniera concertata.
La maggior parte delle sequenze ripetute, sia co<strong>di</strong>ficanti che non co<strong>di</strong>ficanti,<br />
presentano la capacità <strong>di</strong> evolvere in maniera concertata. Quando vengono<br />
comparati i membri <strong>di</strong> una famiglia ripetuta, la maggiore similarità <strong>di</strong><br />
sequenza si trova all’interno della specie (Paralogia) piuttosto che tra le<br />
specie (Ortologia), suggerendo che i membri della famiglia non evolvono<br />
in<strong>di</strong>pendentemente gli uni dagli altri.<br />
Il processo molecolare che conduce alla omogenizzazione delle sequenze <strong>di</strong><br />
DNA appartenenti ad una data famiglia ripetuta è chiamata evoluzione<br />
concertata. L’evoluzione concertata avviene a causa <strong>di</strong> vari meccanismi<br />
genetici che producono scambio tra le sequenze <strong>di</strong> DNA non alleliche<br />
all’interno <strong>di</strong> un genoma. Questi meccanismi comprendono il crossing-over<br />
ineguale, lo scambio ineguale tra cromati<strong>di</strong> fratelli e meccanismi simili alla<br />
conversione genica ed amplificazione che sono particolarmente attivi nel caso<br />
<strong>di</strong> sequenze <strong>di</strong> DNA ripetuto in tandem.
Con il termine <strong>di</strong> conversione genica si intende invece un trasferimento non<br />
reciproco <strong>di</strong> sequenze tra una coppia <strong>di</strong> sequenze <strong>di</strong> DNA non alleliche<br />
(conversione genica interlocus) o alleliche (conversione genica interallelica).<br />
Delle due sequenze che interagiscono una, la sequenza donatrice, rimane<br />
immutata, mentre l’altra, la ricevente, viene mo<strong>di</strong>ficata.<br />
Un possibile meccanismo ipotizza la formazione <strong>di</strong> un eteroduplex tra un<br />
filamento della sequenza donatrice e quello complementare della ricevente,<br />
con successiva conversione del ricevente grazie al meccanismo della riparazione<br />
degli accoppiamenti errati: gli enzimi <strong>di</strong> riparazione del DNA riconoscono che<br />
i due filamenti dell’eteroduplex non sono perfettamente appaiati e<br />
correggono la sequenza del ricevente per renderla perfettamente<br />
complementare alla sequenza del filamento donatore.
EVOLUZIONE CONCERTATA<br />
Negli eucarioti alcuni geni sono presenti in copie multiple. Negli organismi complessi,<br />
per esempio, i geni per l’RNA ribosomale sono tipicamente presenti in centinaia o<br />
anche migliaia <strong>di</strong> copie. Senza dubbio alcuno, queste copie si sono originate per<br />
duplicazione. In seguito alla duplicazione ci si potrebbe attendere che ogni singola copia<br />
<strong>di</strong> un gene acquisisca delle mutazioni e <strong>di</strong>verga. La selezione potrebbe limitare la<br />
mutazione nelle regioni co<strong>di</strong>ficanti, ma, se ne esistono molte copie, ci aspetteremmo,<br />
specie a livello delle sequenze non co<strong>di</strong>ficanti, che sussista un certo grado <strong>di</strong><br />
<strong>di</strong>vergenza. Al contrario <strong>di</strong> quanto atteso, numerosi stu<strong>di</strong> hanno rivelato che spesso le<br />
sequenze nucleoti<strong>di</strong>che risultano alquanto omogenee in seno alle <strong>di</strong>verse copie <strong>di</strong> un<br />
gene.<br />
Inoltre, anche le sequenze non co<strong>di</strong>ficanti risultano omogenee, il che suggerisce che la<br />
selezione non sia responsabile <strong>di</strong> questa purificazione. Quando gli stessi geni vengono<br />
esaminati in una seconda specie strettamente correlata, si trova che anche le<br />
sequenze <strong>di</strong> quest’ultima sono omogenee, ma spesso <strong>di</strong>verse dalle sequenze omogenee<br />
trovate nella prima specie.<br />
Queste osservazioni hanno fatto concludere che un qualche processo a livello molecolare<br />
mantenga <strong>di</strong> continuo l’uniformità tra copie multiple della stessa sequenza all’interno<br />
<strong>di</strong> una specie. Allo stesso tempo, il processo permette una rapida <strong>di</strong>fferenziazione tra<br />
specie: il meccanismo dell’evoluzione concertata non è chiaramente compreso, ma<br />
l’evoluzione concertata ha conseguenze importanti su come evolvano i geni e<br />
rappresenta una forza evolutiva <strong>di</strong> cui si ignorava l’esistenza prima che alla genetica <strong>di</strong><br />
popolazioni fossero applicate le moderne tecniche molecolari.
Duplicazioni intra-geniche<br />
Nuove funzioni geniche possono essere acquisite me<strong>di</strong>ante riarrangiamento <strong>di</strong> segmenti<br />
genici co<strong>di</strong>ficanti per domini proteici strutturali<br />
2 meccanismi:<br />
- duplicazione dei domini<br />
- rimescolamento dei<br />
domini
Duplicazioni intra-geniche<br />
Esempio <strong>di</strong> duplicazione <strong>di</strong> domini strutturali: gene per il collagene a2 <strong>di</strong> tipo<br />
338 ripetizioni Gly-X-Y, presenti in 42<br />
dei 52 esoni del gene. Ogni esone<br />
co<strong>di</strong>fica per un numero completo <strong>di</strong><br />
ripetizioni.<br />
Evoluzione del gene me<strong>di</strong>ante<br />
duplicazione degli esoni che ha portato<br />
alla ripetizione <strong>di</strong> domini strutturali<br />
Esempio <strong>di</strong> rimescolamento dei domini strutturali: gene per l’attivatore<br />
del plasminogeno tissutale<br />
TPA<br />
4 esoni co<strong>di</strong>ficanti domini strutturali<br />
<strong>di</strong>versi: 1° esone simile a quelli della<br />
fibronectina, proteina che lega la<br />
fibrina, 2° esone co<strong>di</strong>fica per un<br />
dominio tipico dei fattori <strong>di</strong> crescita<br />
3° e 4° esone co<strong>di</strong>ficano per<br />
strutture kringle (legano i coaguli <strong>di</strong><br />
fibrina) presenti nel gene per<br />
plasminogeno
Pseudogeni<br />
• Copie <strong>di</strong>fettive dell’intera sequenza <strong>di</strong> un gene funzionale (o<br />
della sua porzione co<strong>di</strong>ficante), o copie troncate, mancanti<br />
<strong>di</strong> porzioni al 5’, al 3’, o frammenti interni.
Pseudogeni non processati<br />
• Contengono tutte le regioni funzionali del gene<br />
• Presentano codoni <strong>di</strong> stop inappropriati<br />
• Originati per duplicazione genica o crossing-over ineguale<br />
Pseudogeni processati (retropseudogeni)<br />
• Contengono solo le sequenze esoniche e una sequenza oligo dA/dT<br />
• Copiati dall’mRNA in cDNA e reintegrati nel genoma<br />
• Se sono espressi sono detti retrogeni
Pseudogeni<br />
La Trascrittasi Inversa co<strong>di</strong>ficata da elementi LINE può retrotrascrivere un<br />
mRNA in cDNA che successivamente può essere integrato a caso in un<br />
cromosoma. Se sul sito <strong>di</strong> inserimento è casualmente presente un promotore<br />
il retrogene può essere eventualmente espresso e <strong>di</strong>ventare funzionale.<br />
Normalmente, questo non accade e lo pseudogene comincia ad accumulare<br />
mutazioni casuali che <strong>di</strong>struggono la ORF (eventualmente) funzionale.