Structure et multiplication des virus
Structure et multiplication des virus
Structure et multiplication des virus
Create successful ePaper yourself
Turn your PDF publications into a flip-book with our unique Google optimized e-Paper software.
<strong>Structure</strong> Virion = particule virale (ou forme extracellulaire) 1 <strong>virus</strong> = génome + capside, +/- enveloppe lipidiqueGénome+Capside (core)=Nucléocapsi<strong>des</strong>piculesmatriceenveloppe(péplos)<strong>virus</strong> nu<strong>virus</strong> enveloppé5
Capside Une protéine ou un p<strong>et</strong>it nombre de protéines différentesAuto-assemblageSymétrie hélicoïdale ou icosaédriqueQuelques formes complexes6
Capsi<strong>des</strong> à symétrie hélicohlicoïdaleForme tubulaireSymétrie hélicoïdale7
Virus de la stomatite vésiculaireVirus de la rougeole8
Capsi<strong>des</strong> à symétrie icosaédriqueHexonPenton20 triangles équilatéraux12 somm<strong>et</strong>s30 arêtesSous-unité morphologique = Capsomère (penton ou hexon)Nombre total de capsomères = 12 pentons + X hexons9
Exemple : Adéno<strong>virus</strong> : 12 pentons + 240 hexonsAdéno<strong>virus</strong>252 capsomères10
Capsi<strong>des</strong> complexes<strong>virus</strong> de la variole<strong>virus</strong> du sida11
Enveloppe Virus enveloppés <strong>et</strong> <strong>virus</strong> non enveloppés (nus) Bicouche lipidique :– Dérivée <strong>des</strong> membranes cellulaires (bourgeonnement)– Glycoprotéines d’origines virales Conséquence : enveloppe = fragilité– Dégradation rapide dans le milieu extérieur (température, <strong>des</strong>siccation)– Dégradation dans le tube digestif (enzymes digestives, pH acide)– Dégradation par les solvants <strong>des</strong> lipi<strong>des</strong>12
Stabilité dansl’environnementVirus enveloppésNONVirus nusOUIElimination dans les selles NON (exceptions) OUIElimination dans la gorge OUI OUIContamination interhumainedirecte (respiratoire, salivaire,sexuelle, sanguine, verticale)Contamination interhumaineindirecte, fécale-oraleOUINONOUIOUITempérature de stockage delongue durée <strong>des</strong>prélèvements pour isolement-80 °C -20°CInactivation, désinfectionFacile(solvants <strong>des</strong> lipi<strong>des</strong>, alcool,dérivés iodés, …)Difficile(dérivés chlorés, aldéhy<strong>des</strong>)
Glycoprotéines d’enveloppe(spicules en ME)Domaine externeHémagglutinineNeuraminidaseDomain<strong>et</strong>ransmembranaireDomaine interneUn spicule viralgp = glycoprotéineVirus grippal14
Génomes virauxARNMonocaténaire (1 brin)Bicaténaire (2 brins)LinéaireSegmentéSegmentéADNMonocaténaire (1 brin)Bicaténaire (2 brins)LinéaireCirculairePartiellement bicaténaire15
Virus ADNgénome ex famille taille du génome protéines majeuresmonocaténairebicaténairepartielbicaténairecirculairebicaténairelinéaireParvoviridaeB19 (+ <strong>et</strong> -)HepadnaviridaeHBVPapovaviridaeVJC - HPVAdenoviridaeHerpesviridaePoxviridae4,5 - 6 kb 33,2 kb 74,5 - 7,5 kb 6 - 930 - 38 kpb120 - 225 kpb130 - 375 kpb> 10> 20> 100taux de mutation : 1 / 10 millions de nucléoti<strong>des</strong>16
Virus ARNgénome ex famille taille protéinesmajeuresmonocaténaire linéaire+ ou -monocaténaire linéaire2 brins +Picornaviridae +Virus polioRhabdoviridae –Virus de la rageR<strong>et</strong>roviridaeVirus du sida7 - 9 kb 410 - 14 kb 57 - 10 kb 7monocaténairesegmentéOrthomyxoviridaeVirus de la grippe10 - 15 kb6 – 8 segments6 – 8bicaténaire segmentéReoviridaeRota<strong>virus</strong>18 – 30 kpb10 – 12 segments6 - 10taux de mutation : 1 / 10 000 nucléoti<strong>des</strong>=> Variabilité génétique <strong>des</strong> <strong>virus</strong> à ARN17
Epissage alternatif ou différentiel1 gèneARNm précurseurépissage différentielARNmprotéine Aprotéine B18
Chevauchement <strong>des</strong> cadres de lecturegénomecadres de lectureprotéines A B19
Taille <strong>des</strong> <strong>virus</strong>•Moran U, Cell, 2010HIV = 100 nmPicornaviridae = 20-30 nmCMV = 230 nmNon visibles au microscope optique (+++)Notion d’agents filtrables ou ultrafiltrablesFiltre de Chamberland20
Classification <strong>et</strong> nomenclatureCritères de classification1 - acide nucléique : nature <strong>et</strong> structure2 - symétrie de la capside3 - présence ou absence d'enveloppeNomenclaturefamille : Nomviridae (en un mot)sous-famille : Nomvirinae (en un mot)genre : Nom<strong>virus</strong> (en un mot)espèce : Nom – <strong>virus</strong> ( en plusieurs mots)Nom courant : le <strong>virus</strong> de la rage ou le <strong>virus</strong> de l’herpès21
Ac. nucléique Symétrie Enveloppe Famille Genres EspècesADN Icosaédrique + Herpesviridae Simplex<strong>virus</strong>Varicello<strong>virus</strong>Lymphocrypto<strong>virus</strong>Cytomegalo<strong>virus</strong>Roseolo<strong>virus</strong>Herpes<strong>virus</strong> types 1 <strong>et</strong> 2 (HSV)Varicelle –Zona (VZV)Epstein Barr (EBV)Cytomégalo<strong>virus</strong> (CMV)Herpes<strong>virus</strong> types 6, 7 (HHV)ADN Icosaédrique + Hepadnaviridae Orthohepadna<strong>virus</strong> Virus de l'hépatite B (HBV)ADN Icosaédrique − Adenoviridae Mastadeno<strong>virus</strong> Adeno<strong>virus</strong> A-GARN Icosaédrique − Picornaviridae Entero<strong>virus</strong>Hepato<strong>virus</strong>ARN Icosaédrique − Reoviridae Rota<strong>virus</strong> Rota<strong>virus</strong> A-EEntero<strong>virus</strong> humain A, B, C, D(polio<strong>virus</strong>, écho<strong>virus</strong>, coxsackie<strong>virus</strong> A,B, entero<strong>virus</strong>)Rhino<strong>virus</strong> humain A, B, CVirus de l’hépatite A (VHA)ARN Icosaédrique + Flaviviridae Hepaci<strong>virus</strong> Virus de l'hépatite C (VHC)ARN Hélicoïdale + Orthomyxoviridae Influenza<strong>virus</strong> A, B, C Virus influenza A, B, CARN Hélicoïdale + Paramyxoviridae Respiro<strong>virus</strong>Rubula<strong>virus</strong>Morbilli<strong>virus</strong>Pneumo<strong>virus</strong>M<strong>et</strong>apneumo<strong>virus</strong>ARN Complexe + R<strong>et</strong>roviridae Lenti<strong>virus</strong> VIH 1-2Tableau : exemple de <strong>virus</strong> d’intérêt en pathologie humaineVirus parainfluenza 1 <strong>et</strong> 3Virus <strong>des</strong> oreillons , PIV 2-4Virus de la rougeoleVirus respiratoire syncytialM<strong>et</strong>apneumo<strong>virus</strong> humain
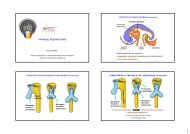
Cycle de <strong>multiplication</strong> virale1 fixation / 2 pénétration3 décapsidation4 réplication <strong>et</strong>expression dugénome5 assemblage6 libérationCycle replication = cycle de <strong>multiplication</strong>Une <strong>des</strong> étapes est la « réplication » du génome23
1. Fixation du <strong>virus</strong> sur la surface cellulaire• Ligand viral : protéine de capside (<strong>virus</strong> nu) ou glycoprotéine d’enveloppe(<strong>virus</strong> enveloppé)• Récepteur cellulaire sur la membrane cytoplasmique (+/- co-récepteur)• Le <strong>virus</strong> ne peut infecter que les cellules qui expriment les récepteursnécessaires à son attachement : spectre d’hôte, tropisme cellulaire24
2. Pénétration P: fusion (<strong>virus</strong> enveloppés)HerpesviridaeR<strong>et</strong>roviridaeParamyxoviridae25
2. Pénétration P: endocytose<strong>virus</strong> enveloppésOrthomyxoviridaeRhabdoviridaeArenaviridaeBunyaviridaeTogoviridaeFlaviviridae<strong>virus</strong> nusAdenoviridaePolyomaviridae26
2. Pénétration Pdirecte <strong>des</strong> Picornaviridae3. DécapsidationD• Dégradation de la capside pour libérer le génome viral27
4. RéplicationR La cellule passe sous le contrôle du <strong>virus</strong> Production massive <strong>des</strong> constituants viraux (aci<strong>des</strong> nucléiques, protéines) Baisse <strong>des</strong> synthèses <strong>des</strong> constituants cellulaires Réplication du génome ADN polymérase virale ou cellulaire : <strong>virus</strong> ADN Réplicase virale (ARN polymérase ARN dépendante) : <strong>virus</strong> ARN Rétro-transcriptase (ADN polymérase ARN dépendante) : VIH <strong>et</strong> VHB Expression du génome : Les génomes fils servent de matrice pour la synthèse <strong>des</strong> ARNm Les ARNm viraux sont traduits par les ribosomes cellulaires28
Le système de BaltimoreVIILes génomes viraux doivent synthétiser <strong>des</strong> ARNm « lisibles » par lesribosomes de l’hôte29
Exemple : Réplication d’un <strong>virus</strong> à ARN simple brin (+)+ARN-30
5. Assemblage <strong>des</strong> virions Auto-assemblage <strong>des</strong> protéines de capside Protéases virales ou cellulaires : maturation<strong>des</strong> protéines de structures Site d’assemblage Cytoplasme pour de nombreux <strong>virus</strong> Noyau pour les Herpesviridae, Adenoviridae <strong>et</strong>ParvoviridaeARNm (+)TraductionFormation de lanucléocapsideClivage de lapolyprotéine P1Assemblage <strong>des</strong>protéines encapsomère31
6. Libération <strong>des</strong> virions• Pour les <strong>virus</strong> nus : lyse de la cellule• Pour les <strong>virus</strong> enveloppés : Lyse de la cellule Bourgeonnement à travers les membranes cellulairesnucléocapsidematriceprotéinesd’enveloppe32
Cycle de <strong>multiplication</strong> virale1 fixation / 2 pénétration3 décapsidation4 réplication <strong>et</strong>expression dugénome5 assemblageDurée d'un cycle : 8 - 50 hVirions fils : 1.000 - 100.0006 libération33
Les infections viralesVirionTransformanteoncogéniqueLytiqueLatenteCellulePermissiveRéactivationChroniqueoupersistante productive34
Conclusion Parasites intracellulaires absolus Réplication nécessite l’infection d’une cellule Détournent la machinerie cellulaire de synthèse <strong>des</strong> macromolécules Forte interférence avec le métabolisme cellulaire Connaissance <strong>des</strong> mécanismes de réplication Compréhension de la pathogénie Développement <strong>des</strong> antiviraux35
AMPHIle TD portera sur les9 premiers coursFORUMLaboratoirede virologieTD VIRO36