Clonagem de expressão - Biologia Molecular e Genética
Clonagem de expressão - Biologia Molecular e Genética
Clonagem de expressão - Biologia Molecular e Genética
You also want an ePaper? Increase the reach of your titles
YUMPU automatically turns print PDFs into web optimized ePapers that Google loves.
<strong>Clonagem</strong> <strong>de</strong> <strong>expressão</strong>
Tópicos<br />
Significado da clonagem <strong>de</strong> <strong>expressão</strong><br />
Expressão heteróloga em E. coli<br />
Tipos <strong>de</strong> vectores <strong>de</strong> <strong>expressão</strong><br />
Expressão heteróloga em Saccharomyces cerevisiae<br />
Vectores <strong>de</strong> clonagem em leveduras<br />
Expressão heteróloga em células <strong>de</strong> mamífero<br />
Vectores <strong>de</strong> clonagem em células <strong>de</strong> mamífero
Significado da clonagem <strong>de</strong> <strong>expressão</strong><br />
Muitas das manipulações em clonagem molecular (clonagem geral) não<br />
requerem a <strong>expressão</strong> do DNA clonado.<br />
A clonagem <strong>de</strong> <strong>expressão</strong> permite obter o produto do gene<br />
clonado.<br />
O produto é utilizado para diferentes fins:<br />
• O transcrito da sequência clonada (transcrição in vitro) po<strong>de</strong> servir como<br />
sonda <strong>de</strong> hibridação.<br />
• A proteína expressa po<strong>de</strong> servir para i<strong>de</strong>ntificar o gene clonado, numa<br />
biblioteca <strong>de</strong> <strong>expressão</strong>, por hibridação com o anticorpo específico.<br />
• O DNA recombinante po<strong>de</strong> servir para produzir proteínas <strong>de</strong> valor<br />
comercial.
Expressão heteróloga em E. coli (1)<br />
• Objectivo – Produção <strong>de</strong> gran<strong>de</strong> quantida<strong>de</strong> <strong>de</strong> proteína recombinante<br />
• Factores <strong>de</strong> selecção do sistema <strong>de</strong> <strong>expressão</strong><br />
• Principais factores da <strong>expressão</strong> <strong>de</strong> genes clonados<br />
• Expressão em E. coli<br />
- Vantagens e <strong>de</strong>svantagens<br />
- Razões da <strong>expressão</strong> não eficiente<br />
- Configuração <strong>de</strong> vectores eficientes<br />
- Regulação transcricional<br />
Promotor<br />
Terminador<br />
- Regulação traducional<br />
Iniciação da tradução<br />
Estimuladores traducionais<br />
Terminação da tradução
Expressão heteróloga em E. coli (2)<br />
- Promotores<br />
- Frequência <strong>de</strong> utilização <strong>de</strong> codões<br />
- Proteólise<br />
- Expressão citoplasmática<br />
- Expressão periplasmática<br />
- Secreção extracelular<br />
- Fusões transcricionais<br />
- Fusões traducionais<br />
Adição <strong>de</strong> tags<br />
Adição <strong>de</strong> sinais <strong>de</strong> secreção (Protein targeting)
Factores <strong>de</strong> selecção do sistema <strong>de</strong> <strong>expressão</strong><br />
• Características do crescimento celular<br />
• Níveis <strong>de</strong> <strong>expressão</strong> (intracelular e/ou extracelular)<br />
• Modificações pós-traducionais<br />
• Activida<strong>de</strong> biológica da proteína relevante<br />
• Dificulda<strong>de</strong>s, custos
Principais factores da <strong>expressão</strong> <strong>de</strong> genes clonados
Vantagens e <strong>de</strong>svantagens da <strong>expressão</strong> em E. coli<br />
Vantagens Desvantagens<br />
• Vasto conhecimento da genética e<br />
fisiologia <strong>de</strong> E. coli<br />
• Diversida<strong>de</strong> <strong>de</strong> vectores <strong>de</strong> clonagem<br />
• Fácil controlo da <strong>expressão</strong> génica<br />
• Fácil crescimento com elevadas<br />
produções<br />
• Secreção do produto no meio <strong>de</strong> cultura<br />
• Não realiza modificações póstraducionais<br />
e tem capacida<strong>de</strong><br />
limitada <strong>de</strong> formar pontes<br />
dissulfídricas<br />
• Activida<strong>de</strong> biológica e<br />
imunogenicida<strong>de</strong> po<strong>de</strong>m diferir<br />
da proteína natural<br />
• Elevado conteúdo <strong>de</strong><br />
endotoxinas<br />
• Falta <strong>de</strong> um mecanismo <strong>de</strong><br />
secreção
Razões da <strong>expressão</strong> não eficiente em E. coli<br />
• Características estruturais da sequência <strong>de</strong> nucleótidos do gene<br />
• Instabilida<strong>de</strong> do mRNA<br />
• Reduzida eficiência traducional<br />
• Dificulda<strong>de</strong> <strong>de</strong> enrolamento da proteína<br />
• Diferenças <strong>de</strong> utilização <strong>de</strong> codões<br />
• Toxicida<strong>de</strong> da proteína para o hospe<strong>de</strong>iro
Configuração <strong>de</strong> vectores <strong>de</strong> <strong>expressão</strong> eficientes<br />
MCS<br />
Características do promotor (P)<br />
- Sequências -10, -35<br />
- Elemento UP (sequência a montante da box -35,<br />
estimulador da transcrição)<br />
- Forte (eficientemente reconhecido pela polimerase <strong>de</strong><br />
RNA)<br />
- Regulação forte (baixo nível <strong>de</strong> <strong>expressão</strong> basal<br />
<strong>de</strong>vido ao gene regulador, R, presente no<br />
vector ou integrado no cromossoma <strong>de</strong> E. coli)<br />
- Indução fácil (térmica ou química)<br />
- Posicionamento (10-100 pb da sequência <strong>de</strong> Shine-<br />
Dalgarno - SD)<br />
Elevado nº <strong>de</strong> cópias,<br />
elevado nº <strong>de</strong> mol<strong>de</strong>s<br />
<strong>de</strong> transcrição<br />
Funções do terminador da<br />
transcrição – TT<br />
- A jusante da sequência clonada<br />
impe<strong>de</strong> a transcrição através <strong>de</strong> outro<br />
promotor, o que po<strong>de</strong> inibir a sua<br />
função, fenómeno conhecido por<br />
oclusão do promotor<br />
- Aumenta a estabilida<strong>de</strong> do mRNA<br />
UAA - codão stop mais frequente<br />
UAAU - codão stop mais eficiente
Promotores utilizados em E. coli<br />
Promotor (origem) Regulação Indução<br />
P lac (E. coli) lacI, lacI q IPTG<br />
P tac , P híbrido (E. coli, box -35 do P do operão do lacI, lacI q IPTG<br />
triptofano, box -10 do P do operão da lactose)<br />
P L ( λ) λcIts857 Térmica<br />
P T7 (T7) λcIts857 Térmica<br />
lacI, lacI q IPTG
Representação esquemática da repressão do promotor P L <strong>de</strong> λ<br />
O repressor codificado<br />
pelo gene cI857 é sensível<br />
à temperatura.
Utilização do gene cI857 no sistema <strong>de</strong> <strong>expressão</strong> P T7<br />
Sistema <strong>de</strong> <strong>expressão</strong>: Plasmídio com P L a montante do gene 1<br />
Vector com P T7 a montante do gene em estudo<br />
Genoma do hospe<strong>de</strong>iro com λcI857<br />
O gene 1 codifica a polimerase <strong>de</strong> RNA T7 específica do promotor P T7<br />
A polimerase <strong>de</strong> RNA <strong>de</strong> E. coli reconhece P L<br />
A polimerase <strong>de</strong> RNA <strong>de</strong> E. coli é selectivamente inibida pela rifampicina<br />
1º Indução – Síntese da polimerase <strong>de</strong> RNA T7 por elevação da temperatura <strong>de</strong><br />
incubação <strong>de</strong> E. coli<br />
2º Adição <strong>de</strong> rifampicina – Inibição da polimerase <strong>de</strong> RNA <strong>de</strong> E. coli<br />
Nestas condições só ocorre síntese da proteína do gene em estudo.
Sistema <strong>de</strong> <strong>expressão</strong> P T7 sob controlo do P lac<br />
Regulação lacI, lacI q<br />
Indução por IPTG
Efeito da diferente utilização <strong>de</strong> codões na <strong>expressão</strong> génica<br />
com baixa frequência <strong>de</strong> utilização em E. coli<br />
Haverá baixa <strong>expressão</strong> proteica se o DNA clonado tiver codões pouco utilizados em E. coli.<br />
Soluções: Mutagénese sítio-específica (alteração <strong>de</strong> nucleótidos sem alteração <strong>de</strong><br />
aminoácidos)<br />
Co<strong>expressão</strong> do gene argU que codifica o tRNA<br />
Arg (AGG/AGA)
Tipos <strong>de</strong> vectores <strong>de</strong> <strong>expressão</strong><br />
Os vectores <strong>de</strong> <strong>expressão</strong> são <strong>de</strong> dois tipos:<br />
Vectores <strong>de</strong> fusão transcricional – fornecem apenas o promotor<br />
Vectores <strong>de</strong> fusão traducional – fornecem o promotor e os sinais<br />
<strong>de</strong> tradução (RBS e codão <strong>de</strong> iniciação da tradução)
Fusões transcricionais e traducionais (1)<br />
Em ambos os vectores o inserto <strong>de</strong>ve estar clonado na orientação correcta.
Fusões transcricionais e traducionais (2)<br />
• Na fusão transcricional, o promotor e o gene constituem uma unida<strong>de</strong><br />
transcricional, isto é, a transcrição iniciada a partir do promotor continua através<br />
do gene clonado.<br />
O vector fornece o promotor.<br />
O inserto contém os sinais <strong>de</strong> tradução – RBS e codão <strong>de</strong> iniciação da<br />
tradução.<br />
É produzida proteína nativa.<br />
• Na fusão traducional também se forma uma unida<strong>de</strong> transcricional.<br />
O vector fornece o promotor e os sinais <strong>de</strong> tradução.<br />
É produzida proteína <strong>de</strong> fusão ou proteína híbrida, em que parte do<br />
produto da tradução (proteína ou polipéptido) é <strong>de</strong>rivado do inserto e parte do<br />
vector.
Requisitos da fusão traducional<br />
• É preciso conhecer a sequência <strong>de</strong> nucleótidos do inserto e avaliar o resultado<br />
da clonagem num <strong>de</strong>terminado local.<br />
• O inserto <strong>de</strong>ve estar na mesma grelha <strong>de</strong> leitura do ATG do vector.<br />
• Po<strong>de</strong> ser necessário mudar <strong>de</strong> vector, ou modificar o vector ou o inserto<br />
(utilizando linkers ou adaptadores). Alternativamente, o inserto po<strong>de</strong> ser<br />
produzido por PCR, com primers que contêm um local <strong>de</strong> restrição que produz a<br />
grelha correcta.<br />
• É fundamental testar por sequenciação o produto recombinante para<br />
confirmar a correcta inserção do fragmento <strong>de</strong> DNA. A perda <strong>de</strong> uma base<br />
durante a ligação resulta numa grelha <strong>de</strong> leitura incorrecta.<br />
• As proteínas toleram uma variação consi<strong>de</strong>rável <strong>de</strong> aminoácidos na<br />
extremida<strong>de</strong> N, mas para fins terapêuticos em humanos <strong>de</strong>vem ser utilizados<br />
produtos iguais aos naturais.
1.<br />
2.<br />
Exemplo <strong>de</strong> fusão traducional em fase<br />
Em 1., fusão traducional em fase; em 2., fusão traducional incorrecta.
<strong>Clonagem</strong> num vector <strong>de</strong> <strong>expressão</strong> plasmídico<br />
lac<br />
A sequência <strong>de</strong> DNA que codifica a<br />
proteína relevante é clonada num<br />
vector <strong>de</strong> <strong>expressão</strong>.<br />
O vector <strong>de</strong> <strong>expressão</strong> tem um<br />
promotor forte, adjacente ao gene<br />
que codifica a proteína, que origina<br />
gran<strong>de</strong>s quantida<strong>de</strong>s <strong>de</strong> mRNA.<br />
O DNA recombinante é<br />
introduzido em bactérias,<br />
leveduras, células <strong>de</strong> insecto, ou<br />
células <strong>de</strong> mamífero, on<strong>de</strong> o gene<br />
clonado é transcrito e traduzido<br />
eficientemente.<br />
A clonagem <strong>de</strong> <strong>expressão</strong> permite a produção <strong>de</strong> gran<strong>de</strong>s quantida<strong>de</strong>s da proteína relevante.
Vectores <strong>de</strong> <strong>expressão</strong> em duas etapas<br />
Expressão <strong>de</strong> genes letais<br />
P T7 – promotor com um controlo muito apertado
Vector pET-3<br />
N<strong>de</strong>I<br />
BamHI<br />
O promotor do fago T7 permite elevado nível <strong>de</strong> <strong>expressão</strong>; a sequência lí<strong>de</strong>r do gene 10 (estimulador da<br />
tradução) do fago T7 assegura elevado nível <strong>de</strong> tradução; N<strong>de</strong>I e BamHI são locais <strong>de</strong> clonagem. A<br />
clonagem em BamHI origina uma proteína <strong>de</strong> fusão contendo 13 aminoácidos N-terminal do gene 10 do<br />
fago T7.
<strong>Clonagem</strong> num vector λ <strong>de</strong> <strong>expressão</strong> (1)<br />
Figure 7-21. [Adapted from J. D. Watson et al., 1992, Recombinant DNA,2d ed., Scientific American Books.]<br />
Vector λgt11<br />
A i<strong>de</strong>ntificação do DNA<br />
clonado é feita por hibridação<br />
com um anticorpo específico da<br />
proteína expressa.
Detecção <strong>de</strong> proteínas com anticorpos
Funções do tag<br />
O tag é uma curta sequência <strong>de</strong> nucleótidos que codifica um péptido com<br />
poucos aminoácidos.<br />
• Po<strong>de</strong> ser adicionado à extremida<strong>de</strong> N ou C do produto do gene clonado.<br />
• Permite a <strong>de</strong>tecção e purificação <strong>de</strong> proteínas.<br />
• Péptidos <strong>de</strong> fusão específicos conferem vantagens à proteína alvo durante a<br />
<strong>expressão</strong>: aumentam a solubilida<strong>de</strong>, protegem da proteólise, melhoram a<br />
conformação, aumentam a produção.<br />
• São vários os tags utilizados nos vectores <strong>de</strong> clonagem:<br />
His6<br />
Glutationa-S-transferase (GST)<br />
Proteína <strong>de</strong> ligação a maltose (MBP)<br />
Proteína ver<strong>de</strong> fluorescente (GFP)<br />
Epítopos
Tag adicionado ao inserto<br />
Por engenharia genética, um tag (epítopo) po<strong>de</strong> ser adicionado ao inserto.<br />
Figure 8-48. © 2000 by W. H. Freeman and Company
Vectores com tag
Detecção <strong>de</strong> proteínas com tag
Purificação <strong>de</strong> proteínas com tag<br />
As proteínas com tag são purificadas em colunas <strong>de</strong> cromatografia <strong>de</strong> afinida<strong>de</strong>.
A partir <strong>de</strong> EcoRI os locais<br />
múltiplos <strong>de</strong> clonagem <strong>de</strong><br />
pEGX-4T-1, -2 e –3<br />
configuram as três grelhas <strong>de</strong><br />
leitura possíveis dos codões.<br />
Vectores <strong>de</strong> fusão génica pGEX-4T<br />
A proteína <strong>de</strong> fusão é<br />
constituída pela componente<br />
N-terminal <strong>de</strong> GST (tag) e pela<br />
componente C-terminal da<br />
proteína pretendida.<br />
Po<strong>de</strong> ser purificada numa<br />
coluna <strong>de</strong> purificação <strong>de</strong><br />
afinida<strong>de</strong> <strong>de</strong> glutationa (ex.<br />
sefarose 4B glutationa) e<br />
clivada com trombina.
Purificação <strong>de</strong> complexos proteicos associados a proteínas <strong>de</strong> fusão com GST (1)<br />
Figure 8-50. © 2002 by Bruce Alberts, Alexan<strong>de</strong>r Johnson, Julian Lewis, Martin Raff, Keith Roberts, and Peter Walter.
Vectores <strong>de</strong> fusão génica pRSET<br />
tag<br />
EK – local <strong>de</strong> clivagem<br />
pela enterocinase para<br />
remoção do tag
Funções do péptido sinal<br />
• A secreção <strong>de</strong> proteínas por bactérias <strong>de</strong>pen<strong>de</strong> normalmente da presença<br />
<strong>de</strong> um péptido sinal na extremida<strong>de</strong> N. As proteínas são reconhecidas pela<br />
maquinaria <strong>de</strong> secreção e transportadas através da membrana<br />
citoplasmática.<br />
• A sequência <strong>de</strong> nucleótidos que codifica o péptido sinal existe no vector ou<br />
é incorporada no inserto. A secreção nem sempre acontece, <strong>de</strong>pen<strong>de</strong>ndo da<br />
estrutura da proteína.
Gene repórter<br />
Gene repórter é um gene que produz uma proteína que po<strong>de</strong> ser <strong>de</strong>tectada e<br />
quantificada utilizando um teste simples.<br />
Exemplos <strong>de</strong> genes repórter:<br />
GFP (Green fluorescent protein)<br />
lacZ (β-galactosidase)<br />
luc (luciferase)
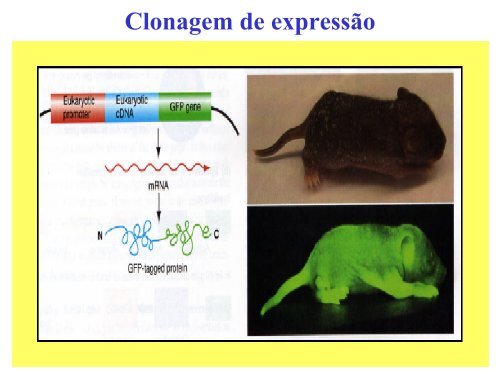
Utilização <strong>de</strong> genes repórter na localização <strong>de</strong> proteínas recombinantes<br />
A) Proteína com tag GFP<br />
A) O gene recombinante codifica uma proteína <strong>de</strong> fusão que contém GFP C-terminal.<br />
B) Ratinho transgénico com GFP em fusão<br />
com uma proteína epitelial<br />
B) O ratinho contém um transgene marcado com GFP expresso no corpo; o ratinho fluoresce quando iluminado com<br />
luz UV.<br />
Figure 19. 18 Genetics: From genes to Genomes, 2/e. (© McGraw-Hill Companies, 2004)
Vectores promoter-probe<br />
Os vectores promoter-probe permitem <strong>de</strong>tectar a existência <strong>de</strong> promotor no<br />
fragmento clonado, através da <strong>expressão</strong> do gene repórter (gene lacZ)
Avaliação do nível <strong>de</strong> <strong>expressão</strong> <strong>de</strong> promotores<br />
Teste <strong>de</strong> <strong>de</strong>tecção da β-galactosidase em que se utiliza o substrato ONPG<br />
A análise dos dados revela:<br />
• A eficiência <strong>de</strong> tradução do gene lacZ é maior do que a do gene em estudo<br />
(por comparação entre fusões transcricionais e traducionais).<br />
• Não há oclusão <strong>de</strong> promotor (por comparação entre resultados <strong>de</strong> diferentes<br />
fragmentos). A activação da transcrição do gene em estudo é maior quando os dois<br />
promotores estão presentes.
Expressão heteróloga em Saccharomyces cerevisiae<br />
Vantagens<br />
• Organismo unicelular (altemativa eucariótica <strong>de</strong> E. coli)<br />
• <strong>Genética</strong> e fisiologia conhecidas<br />
• Crescimento rápido<br />
• Vários promotores fortes (CYC1, ADH1, GAL1O)<br />
• Plasmídio natural <strong>de</strong>nominado 2-µm<br />
• Modificações pós-traducionais<br />
• Não patogénico<br />
• Elevada produção proteica<br />
• Produção industrial
ARS – Sequência <strong>de</strong><br />
replicação autónoma<br />
Vectores <strong>de</strong> clonagem em leveduras<br />
Nestes vectores, a função dos<br />
insertos é estudada por transfecção<br />
da estirpe <strong>de</strong> levedura <strong>de</strong> genótipo<br />
a<strong>de</strong>quado.<br />
São necessárias marcas <strong>de</strong><br />
selecção para a <strong>de</strong>tecção <strong>de</strong> clones<br />
recombinantes.<br />
As origens <strong>de</strong> replicação são os<br />
locais necessários para as enzimas<br />
<strong>de</strong> replicação bacterianas e <strong>de</strong><br />
levedura iniciarem o processo <strong>de</strong><br />
replicação.<br />
O DNA <strong>de</strong>rivado do plasmídio<br />
natural <strong>de</strong> levedura, 2-µm, tem a<br />
sua própria origem <strong>de</strong> replicação.<br />
Representações simplificadas <strong>de</strong> quatro tipos diferentes <strong>de</strong> vectores <strong>de</strong> clonagem em leveduras.<br />
Figure 13-11. © 2000 by W. H. Freeman and Company.
Especificida<strong>de</strong>s da clonagem em leveduras<br />
Métodos <strong>de</strong> transfecção em leveduras<br />
• Protoplastos<br />
• Acetato <strong>de</strong> lítio<br />
• Electroporação<br />
Leveduras utilizadas como hospe<strong>de</strong>iros<br />
• Saccharomyces cerevisiae<br />
• Kluyveromyces lactis<br />
• Schizosaccharomyces pombe<br />
• Yarrowia lipolytica<br />
• Hansenula polymorpha<br />
• Pichia pastoris (Os vectores <strong>de</strong> <strong>expressão</strong> com o promotor AOX1- gene que codifica a<br />
enzima álcool oxidase - geram níveis elevados do produto pretendido (gramas/litro), com menores<br />
custos do que os sistemas <strong>de</strong> insectos e <strong>de</strong> mamíferos. Ao contrário dos sistemas bacterianos, os<br />
vectores <strong>de</strong> P. pastoris não são mantidos epissomicamente, mas são construídos para serem<br />
integrados num cromossoma da levedura. São vectores shuttle com origens <strong>de</strong> replicação em<br />
levedura e em E. coli.)<br />
As leveduras utilizadas como hospe<strong>de</strong>iros são geralmente mutantes auxotróficos.<br />
Exemplos: TRP1, LEU2, URA3<br />
A selecção das células transfectadas é feita por complementação <strong>de</strong> <strong>de</strong>ficiências<br />
metabólicas.
<strong>Clonagem</strong> num plasmídio epissómico <strong>de</strong> levedura<br />
Há menor número <strong>de</strong> antibióticos<br />
disponíveis para os quais as leveduras são<br />
sensíveis (embora alguns fungicidas possam<br />
ser utilizados) e, por isso, a selecção baseiase,<br />
frequentemente, na complementação <strong>de</strong><br />
mutações auxotróficas da estirpe hospe<strong>de</strong>ira.<br />
Os vectores que replicam como<br />
plasmídios em S. cerevisiae são muitas vezes<br />
bastante instáveis, na medida em que ten<strong>de</strong>m<br />
a ser perdidos na cultura <strong>de</strong>vido à acumulação<br />
<strong>de</strong> células sem plasmídio. Isto é <strong>de</strong>vido a uma<br />
partição errada durante a mitose.<br />
Os vectores com ARS são muito instáveis,<br />
mas quando incluem o centrómero são mais<br />
estáveis.<br />
Os vectores são, em geral, vectores <strong>de</strong><br />
<strong>expressão</strong> porque expressar o gene clonado é<br />
uma das finalida<strong>de</strong>s da utilização <strong>de</strong><br />
leveduras como hospe<strong>de</strong>iro.
<strong>Clonagem</strong> num vector YAC
Características do vector YAC e do hospe<strong>de</strong>iro<br />
CEN4 - sequência centromérica<br />
TEL - sequências teloméricas<br />
As sequências do vector são:<br />
ARS1 - sequência <strong>de</strong> replicação autónoma em levedura<br />
Amp - gene que confere resistência à ampicilina para selecção em E. coli<br />
ori - origem <strong>de</strong> replicação para propagação em E. coli<br />
SUP4 - gene que codifica o tRNA supressor da mutação a<strong>de</strong>-2 do hospe<strong>de</strong>iro e restaura a<br />
activida<strong>de</strong> selvagem, originando colónias brancas<br />
TRP1 e URA3 - genes envolvidos no metabolismo do triptofano e uracilo, respectivamente<br />
Características do hospe<strong>de</strong>iro (estirpe AB1380 <strong>de</strong> levedura):<br />
a<strong>de</strong>-2 - mutação ocre no gene envolvido no metabolismo da a<strong>de</strong>nina que resulta na<br />
acumulação <strong>de</strong> pigmento vermelho (colónias vermelhas). A clonagem <strong>de</strong> um fragmento <strong>de</strong><br />
DNA exógeno no gene SUP4, inactiva a função supressora do gene, originando o fenótipo<br />
mutante.<br />
trp1 e ura3 - alelos complementados pelos alelos correspon<strong>de</strong>ntes do vector, constituindo<br />
um sistema <strong>de</strong> selecção para i<strong>de</strong>ntificar as células que contêm o vector YAC.
Expressão heteróloga em células <strong>de</strong> mamífero<br />
Realização <strong>de</strong> modificações pós-traducionais idênticas às naturais.<br />
Expressão transitória – <strong>expressão</strong> obtida sem integração do vector<br />
Expressão estável – <strong>expressão</strong> resultante da integração do vector <strong>de</strong> <strong>expressão</strong><br />
num dos cromossomas do hospe<strong>de</strong>iro<br />
Promotores utilizados na construção <strong>de</strong> vectores <strong>de</strong> células <strong>de</strong> mamífero:<br />
P SV40 (Simian Virus)<br />
P RSV-LTR (Rous Sarcoma Virus)<br />
P MSV-LTR (Murine Sarcoma Virus)<br />
P BPV-1 (Bovine Pappilomavirus Type 1)<br />
P CMV (Citomegalovirus)
Vector <strong>de</strong> <strong>expressão</strong> em células <strong>de</strong> mamífero, pcDNA3.1/myc-HIS
Características do vector pcDNA3.1/myc-HIS<br />
Vector shuttle que permite elevado nível <strong>de</strong> <strong>expressão</strong> constitutiva em células <strong>de</strong><br />
mamífero<br />
P CMV – promotor forte <strong>de</strong> citomegalovírus que assegura elevado nível <strong>de</strong> <strong>expressão</strong><br />
BGH pA – elemento da sequência <strong>de</strong> polia<strong>de</strong>nilação da hormona <strong>de</strong> crescimento bovino<br />
que permite a produção <strong>de</strong> uma extremida<strong>de</strong> 3’ <strong>de</strong>finida no mRNA obtido a partir do<br />
inserto.<br />
neo – gene marcador, regulado por um promotor/estimulador e sequência poli A SV40,<br />
que permite a selecção por crescimento em G418 (geneticina)<br />
Polylinker – contém locais múltiplos <strong>de</strong> clonagem<br />
6xHis – sequência que especifica seis resíduos <strong>de</strong> histidina consecutivos que facilita a<br />
purificação da proteína recombinante<br />
Epítopo myc – tag que permite a triagem com anticorpos da proteína recombinante<br />
utilizando um anticorpo específico para esta sequência<br />
Term – sinais <strong>de</strong> terminação da tradução<br />
SV40 ori – origem <strong>de</strong> replicação em células <strong>de</strong> mamífero<br />
Componentes <strong>de</strong> propagação em E. coli<br />
pUC ori – origem <strong>de</strong> replicação <strong>de</strong>rivada do plasmídio ColE1<br />
Amp – gene que confere resistência à ampicilina<br />
f1 ori – origem <strong>de</strong> replicação do fago filamentoso f1 que permite produzir recombinantes<br />
<strong>de</strong> ca<strong>de</strong>ia simples
Características das células COS<br />
As células COS permitem que qualquer DNA circular com uma origem <strong>de</strong><br />
replicação SV40 replique in<strong>de</strong>pen<strong>de</strong>ntemente do DNA celular.<br />
É uma linha estável <strong>de</strong> células <strong>de</strong> rim <strong>de</strong> macaco ver<strong>de</strong> Aficano <strong>de</strong>rivada da<br />
linha celular <strong>de</strong> macaco, CV1, permissiva <strong>de</strong> SV40. Quando as células CV1 são<br />
infectadas com SV40, ocorre o ciclo lítico normal do vírus.<br />
As células CV1 foram transformadas por integração nos cromossomas <strong>de</strong> um<br />
fragmento <strong>de</strong> DNA genómico <strong>de</strong> SV40 contendo uma origem <strong>de</strong> replicação<br />
mutante.<br />
As células COS resultantes (CV1 com origem <strong>de</strong>fectiva <strong>de</strong> SV40) expressam<br />
constitutivamente (estavelmente) o antigénio T codificado por SV40, a única<br />
proteína viral que é necessária para activar a origem <strong>de</strong> replicação <strong>de</strong> SV40.
Vector pBK-CMV
Características do vector pBK-CMV