V1-CAP 01
V1-CAP 01
V1-CAP 01
Create successful ePaper yourself
Turn your PDF publications into a flip-book with our unique Google optimized e-Paper software.
DOMÍNIO BACTERIA<br />
REINO MONERA<br />
REINO MONERA<br />
Bactérias<br />
(Germes e Micróbios)<br />
Número de espécies<br />
No mundo: cerca de 4.200<br />
No Brasil: ?<br />
Conhecidas no estado de São Paulo: ?<br />
Estimadas no estado de São Paulo: ?<br />
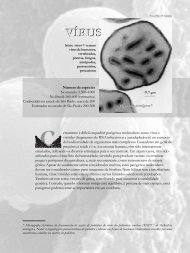
Microbispora sp.*<br />
Foto: Elisa Espósito<br />
actérias são organismos relativamente simples, microscópicos, cujo<br />
material genético não está envolvido por uma membrana nuclear (células<br />
procarióticas) Possuem importância indiscutível na sustentabilidade da<br />
biosfera e têm um papel preponderante nos ciclos biogeoquímicos. As<br />
espécies bacterianas são definidas filogeneticamente, empregando-se para tal a relação<br />
evolutiva entre seqüências de genes conservados, notadamente o RNA ribossômico 16S<br />
(rRNA 16S), e o grau de similaridade entre os genomas dos organismos (homologia<br />
DNA:DNA). A incrível versatilidade metabólica das bactérias justifica a sua ocorrência em<br />
diferentes ambientes e condições de sobrevivência. O número de espécies descritas na<br />
literatura, cerca de 4.200, a maioria representada por organismos cultivados, está muito<br />
aquém do estimado, representando menos de 1% do total de espécies existentes na natureza.<br />
Os estudos de levantamentos ecológicos e os registros sobre a diversidade de bactérias no<br />
estado de São Paulo são escassos, requerendo, portanto, um estímulo à execução de projetos<br />
temáticos e catalogação sistemática da diversidade de bactérias, complementada com esforços<br />
de análise e síntese do papel destes microrganismos no funcionamento de ecossistemas.<br />
* Micrografia eletrônica de varredura de actinomiceto em solo.
DIVERSIDADE NO DOMÍNIO BACTERIA<br />
VANDERLEI PEREZ CANHOS 1 , GILSON PAULO MANFIO 2 ,<br />
ROSANA FILOMENA VAZOLLER 2,3 & VIVIAN HELENA PELLIZARI 4<br />
1 Faculdade de Engenharia de Alimentos, Universidade Estadual de Campinas,<br />
Rua Professor Zeferino Vaz, s/nº, 13081-970 Campinas, SP, e-mail: vcanhos@bdt.org.br<br />
2 Coleção de Culturas Tropical, Fundação André Tosello,<br />
Rua Latino Coelho, 13<strong>01</strong>, 13087-<strong>01</strong>0 Campinas, SP, e-mail: gilson@bdt.org.br<br />
3 Escola de Engenharia de São Carlos, Universidade de São Paulo<br />
Av. Dr. Carlos Botelho, 1465, 13560-970 São Carlos, SP, e-mail: vazoller@uol.com.br<br />
4 Instituto de Ciências Biomédicas II, Universidade de São Paulo,<br />
Cidade Universitária, 05508-900 São Paulo, SP, e-mail: vivianp@usp.br<br />
1. Introdução<br />
1<br />
Bactérias constituem um grupo de microrganismos de ocorrência cosmopolita nos mais diversos hábitats.<br />
Apresentam uma enorme diversidade de vias metabólicas, reunindo organismos especializados na utilização de<br />
compostos orgânicos (heterotróficos e organotróficos) ou inorgânicos como fonte de energia (quimiorganotróficos<br />
e litotróficos), e aqueles capazes utilizar luz como fonte de energia no metabolismo (fototróficos).<br />
Estudos baseados na análise direta da diversidade bacteriana em amostras ambientais através de métodos<br />
moleculares, sem a etapa de isolamento e cultivo, têm revelado um novo cenário sobre a distribuição de<br />
microrganismos no meio ambiente. Os resultados indicam que alguns grupos do Domínio Bacteria apresentam<br />
distribuição cosmopolita, ao passo que outros parecem estar restritos a ambientes particulares (Schlegel &<br />
Jannasch, 1992). Alguns dos grupos filogenéticos de distribuição cosmopolita são bem conhecidos a partir de<br />
estudos de isolamento e cultivo, ao passo que outros são ainda pouco conhecidos/estudados ou não foram ainda<br />
detectados através de cultivo. Os representantes de Proteobacteria (bactérias púrpura fotossintéticas), Cytophagales<br />
(grupo dos Bacteroides/Cytophaga/Flexibacter) e as duas divisões de organismos Gram-positivos (Actinobacteria e<br />
Gram-positivos de baixo conteúdo de G+C), de distribuição cosmopolita, são exemplos de organismos bem<br />
conhecidos através de representantes cultivados. Em contraste, organismos das Divisões Acidobacterium, bactérias<br />
verdes não-sulfurosas e Verrucomicrobia, que também apresentam distribuição cosmopolita, evidenciada por estudos<br />
de seqüências de rRNA 16S clonadas do meio ambiente, são pouco representados por organismos cultivados e,<br />
conseqüentemente, pouco se sabe sobre a sua biologia e propriedades em geral (Hugenholtz et al., 1998a).<br />
Organismos litotróficos e as comunidades que eles suportam contribuem significativamente para a química<br />
da biosfera, atuando nos ciclos biogeoquímicos e processos de lixiviação de rochas e formação de solos. O<br />
metabolismo litotrófico, com produção de energia a partir de compostos inorgânicos, tais como hidrogênio e<br />
compostos reduzidos de ferro e enxofre, é mais amplamente distribuído entre os grupos filogenéticos de<br />
Bacteria e Archaea que as formas de metabolismo fototrófico e organotrófico (Kandler, 1994; Pace, 1997).<br />
Ambientes que permitem a ocorrência de organismos litotróficos, tais como habitats abaixo da superfície terrestre<br />
e os habitats geotermais, são mais prevalentes na Terra que ambientes ricos em matéria orgânica. Considerando a<br />
importância do metabolismo litotrófico nos processos ambientais, é notável a relativa falta de informação sobre<br />
a diversidade e biologia destes organismos e sobre as comunidades que eles suportam. Fatores que contribuem<br />
para a falta de conhecimento são as limitações para o isolamento e cultivo destes em laboratório. De modo geral,<br />
a descrição de comunidades microbianas requer o emprego de técnicas que não envolvam o cultivo em laboratório,<br />
uma vez que apenas uma pequena fração (
4<br />
V.P. Canhos et al.<br />
além das 12 divisões descritas por Woese (1987), baseadas principalmente em seqüências de rRNA 16S de<br />
organismos cultivados, 12 novas divisões putativas, descritas em estudo recente envolvendo a análise de seqüências<br />
de rDNA 16S isoladas diretamente do meio ambiente (Hugenholtz et al., 1998a), e 12 linhas de descendência<br />
descritas em outros estudos (Maidak et al., 1997). O termo “divisão” é definido como um grupo filogenético<br />
contendo duas ou mais seqüências de rDNA 16S reprodutivelmente monofiléticas e não afiliadas com os outro<br />
grupos filogenéticos que integram o Domínio Bacteria (Hugenholtz et al., 1998a,b).<br />
Durante a última década, a taxonomia de bactérias sofreu grandes mudanças. Novas categorias de informações<br />
de valor taxonômico potencial se tornaram disponíveis (e.g., quimiotaxonomia, composição de bases de DNA,<br />
hibridização DNA-DNA, ribotipagem etc.) e possibilitaram a diferenciação de organismos antes alocadas em<br />
grupos heterogêneos, assim como a revelação de dissimilaridades antes não detectadas. Outro fator importante<br />
foram os avanços recentes em técnicas de análises filogenéticas. Filogenia é atualmente uma base importante na<br />
classificação de bactérias. O uso integrado de características fenotípicas e genotípicas, denominado taxonomia<br />
polifásica, está tornando possíveis grandes avanços na classificação de bactérias.<br />
A nomenclatura de bactérias é controlada pelo “Código de Nomenclatura de Bactérias” (Lapage et al.,<br />
1975), sendo o nome do táxon bacteriano baseado na prioridade de publicação válida, legítima e efetiva. Desde<br />
<strong>01</strong> de janeiro de 1980, a prioridade de designar nomes a novas espécies de bactérias é baseada na “Approved List<br />
of Bacterial Names”, que compilou os nomes válidos até aquela data, e nas “Validation Lists”, publicadas<br />
trimestralmente no International Journal of Systematic Bacteriology (IJSB), que compilam os novos nomes<br />
descritos em trabalhos científicos publicados no próprio IJSB e em outros periódicos científicos.<br />
Nos quatro volumes do Bergey’s Manual of Systematic Bacteriology (Krieg & Holt, 1984; Sneath & Holt, 1986;<br />
Staley & Holt, 1989; Williams et al., 1989), são descritos 33 agrupamentos ou seções de microrganismos que<br />
compreendem os representantes dos Domínios Bacteria e Archaea, sendo uma seção dedicada exclusivamente<br />
às Archaea (Staley & Holt, 1989). Nos manuais de Bergey é descrito um total de 384 gêneros de bactérias nas<br />
outras 32 seções. Atualmente (dados de outubro/98), bactérias e archaeas compreendem um total de 4.167<br />
espécies com descrição taxonômica válida, distribuídas em 791 gêneros (http://www.dsmz.de/bactnom/<br />
bactname.htm). A classificação hierárquica dos táxons no Domínio Bacteria pode ser encontrada no site do NCBI<br />
Taxonomy Homepage (http://www.ncbi.nlm.nih.gov/Taxonomy/tax.html).<br />
2. Diversidade bacteriana<br />
A Figura 1 mostra as divisões alocadas no Domínio Bacteria, de acordo com a classificação filogenética<br />
construída com base na comparação de seqüências do rRNA 16S (Madigan et al., 1997), apresentando o estado da<br />
arte em 1987, em contraste com a classificação recentemente compilada por Hugenholtz et al. (1998a). Os<br />
estudos filogenéticos, até o momento, não foram capazes de resolver a ordem das ramificações das divisões no<br />
Domínio Bacteria. A diversidade bacteriana é vista como uma radiação “em leque” de diferentes grupos<br />
filogenéticos, representados pelas divisões; à exceção da divisão Aquificales, que ramifica-se mais profundamente<br />
nas árvores filogenéticas da maioria dos estudos (Hugenholtz et al., 1998a,b). Algumas das principais divisões do<br />
Domínio Bacteria, segundo uma compilação de vários autores (Woese, 1987; Logan, 1994; Madigan et al., 1997;<br />
Hugenholtz et al., 1998a; Tortora et al. 1998a, b), são descritas a seguir.<br />
Aquificales: Compreende a divisão com radiação mais profunda no Domínio Bacteria, cujos organismos<br />
são comumente associados a ambientes geotermais. Todas as seqüências de Aquificales recuperadas diretamente<br />
de amostras ambientais são provenientes de habitats com temperaturas elevadas. Os organismos isolados e<br />
cultivados em laboratório são termófilos e quimiotróficos, metabolizando hidrogênio.<br />
Thermotogales: Compreendem bacilos hipertermófilos anaeróbios, cuja temperatura ótima de crescimento<br />
é 80ºC. Uma espécie cultivada caracteriza esse grupo, Thermotoga maritima, cuja célula apresenta morfologia<br />
bastante peculiar, destacando-se a presença de um envoltório, além da parede celular.<br />
Thermus/Deinococcus: Os representantes desta Divisão são encontrados no solo e no ar. São organismos<br />
Gram-positivos, porém filogeneticamente distantes do grupo das bactérias Gram-positivas. Espécies de Deinococcus<br />
são altamente resistentes a radiação ionizante (até 30.000 Gy). Linhagens de Thermus spp. são termófilas extremas.
Bacteria<br />
Figura 1. Representação radial dos grupos filogenéticos de Bacteria conhecidos em 1987 (figura menor, baseada em<br />
Woese, 1987) e em 1997 (baseado em Hugenholtz et al., 1998a). Os setores em cunha indicam a ocorrência de duas ou<br />
mais seqüências representativas naquele nível de radiação. Doze novas divisões, indicadas como OP1 a OP12, são propostas<br />
com base em dados de estudos recentes (Hugenholtz et al., 1998a). Figura reproduzida com permissão da American<br />
Society for Microbiology.<br />
Espiroquetas: Em geral, são organismos de vida livre, encontrados em ambientes aquáticos, com<br />
metabolismo anaeróbio e aeróbio facultativo. O formato das células é típico, revelando uma estrutura delgada,<br />
flexível e helicoidal. São classificados em 6 gêneros, com base em seu habitat, patogenicidade e características<br />
morfo-fisiológicas, a saber: Borrelia, Cristispira, Leptonema, Leptospira, Spirochaeta e Treponema. Alguns organismos<br />
deste grupo são agentes de enfermidades graves, como, por exemplo, o Treponema pallidum, causador da sífilis.<br />
Bactérias verdes sulfurosas (green sulfur bacteria): A morfologia dessas bactérias é bastante diversa,<br />
incluindo bacilos sem motilidade, espirilos e cocos. São encontradas em sistemas aquáticos e locais que contêm<br />
enxofre elementar, como águas termais ou sedimentos de lagos e rios. São organismos estritamente anaeróbios e<br />
fototróficos obrigatórios. Os compostos reduzidos de enxofre sustentam o metabolismo energético nos organismos<br />
das espécies do gênero Chlorobium e Pelodictyon.<br />
Cytophagales: Grupo fisiologicamente diverso, incluindo bactérias anaeróbias estritas do gênero Bacteroides,<br />
aeróbias estritas do gênero Sporocytophaga, e uma grande variedade de bacilos com movimento deslizante, como<br />
as espécies do gênero Cytophaga. Compreende organismos quimiorganotróficos, encontrados na água e no solo,<br />
sendo algumas espécies celulolíticas. Organismos do gênero Flavobacterium são bastante conhecidos pela atividade<br />
de deterioração de alimentos.<br />
Planctomycetes: São bactérias aeróbias, encontradas em ambientes aquáticos, alocadas nos gêneros Pirella<br />
e Planctomyces. Estes organismos não apresentam peptidioglicano na parede celular. As células encontram-se,<br />
5
6<br />
V.P. Canhos et al.<br />
muitas vezes, aderidas à superfície de substratos pelo auxílio de estruturas celulares externas características, tais<br />
como hastes, hifas ou apêndices citoplasmáticos. Alguns organismos podem apresentar reprodução por brotamento.<br />
Chlamydia: São bactérias parasitas obrigatórias, patógenos de animais e humanos, alocados em três espécies:<br />
Chlamydia pneumoniae, C. psittaci e C. trachomatis. A morfologia celular é esférica regular, com cerca de 1 µm de<br />
diâmetro, e os organismos apresentam, sob condições ideais, um ciclo de vida de 48 h.<br />
Bactérias Gram-positivas com Baixo Teor de G+C: As bactérias Gram-positivas são separadas em dois<br />
grandes grupos, filogeneticamente distintos, cujos organismos podem ser, de maneira geral, discriminados de<br />
acordo com o teor de guanina e citosina (G+C) no DNA.<br />
O grupo dos organismos com baixo teor de G+C é bastante heterogêneo e compreende bacilos, cocos e<br />
organismos pleomórficos com reação de coloração celular Gram-positiva, com exceção das espécies de Mycoplasma,<br />
organismos que não apresentam parede celular. Congrega os organismos do gênero Clostridium, as bactérias<br />
formadoras de ácido lático (Lactobacillus spp., Lactococcus spp.) e vários cocos (Ruminococcus spp., Peptococcus spp.,<br />
Peptostreptococcus spp., Sarcina spp. e Staphylococcus spp.). O metabolismo inclui homo- e hetero-fermentação e<br />
respiração, sendo alguns organismos micro-aerófilos, anaeróbios facultativos ou anaeróbios estritos. Os Grampositivos<br />
formadores de esporos podem ser exemplificados pelos gêneros Bacillus, Clostridium e Sporosarcina.<br />
Bactérias Gram-positivas com Alto Teor de G+C ou Actinobacteria: A Divisão Actinobacteria<br />
(Stackebrandt et al., 1997) compreende o grupo dos organismos com alto teor de G+C da família Actinomycetales<br />
e gêneros relacionados.<br />
Sem dúvida, os actinomicetos possuem aspectos morfológicos e de ciclo celular diferenciados dos demais<br />
organismos Gram-positivos. Em geral, são aeróbios, não são móteis na fase vegetativa e apresentam pleomorfismo<br />
em alguma fase do ciclo celular, podendo formar filamentos. São divididos em 8 grupos. Exemplos comuns são<br />
os organismos do gêneros Arthrobacter, Corynebacterium, Nocardia, Rhodococcus, Streptomyces e Mycobacterium. Os<br />
actinomicetos incluem ainda os organismos microaerófilos/anaeróbios obrigatórios e formadores de ácido<br />
propiônico, alocados nos gêneros Actinomyces, Bifidobacterium e Butyrivibrio.<br />
Cyanobacteria (blue green algae): As cianobactérias são organismos fototróficos oxigênicos<br />
compreendendo várias espécies formadoras de filamentos. São organizadas em 5 subdivisões, que compreendem<br />
gêneros como Anabaena, Dermocarpa e Oscillatoria, encontradas em ambientes aquáticos e terrestres. Algumas<br />
espécies são fixadoras de nitrogênio. Um exemplo é o gênero Prochloron, um simbionte marinho.<br />
Proteobacteria (bactérias púrpuras): Constituem o maior e mais diverso grupo de bactérias cultivadas,<br />
alocadas em 5 subdivisões: alfa (a), beta (b), gama (g), delta (d) e epsilon (e). Os organismos alocados nesses<br />
grupos apresentam coloração Gram-negativa e uma enorme diversidade de morfologia e metabolismo, mesmo<br />
dentro de uma mesma radiação filogenética.<br />
O grupo alfa compreende a maioria das bactérias púrpura não-sulfurosas e organismos não-fotossintéticos<br />
com metabolismo diverso. As radiações dentro da subdivisão alfa são geralmente correlacionadas com propriedades<br />
fenotípicas, destacando-se os grupos das nitrificantes (Nitrobacter spp.) e denitrificantes (Rhodopseudomonas spp.),<br />
organismos com capacidade de fixação de nitrogênio (gêneros Bradyrhizobium, Rhizobium e outros), parasitas<br />
intracelulares de células eucarióticas (gêneros Bartonella e Rochalimaea). Organismos formadores de prosteca,<br />
como Caulobacter e Prosthecomicrobium, também são alocados nesta subdivisão.<br />
No grupo beta estão alocadas as espécies de Rhodocyclus, Spirillum, Thiobacillus e Zymomonas, entre outros 12<br />
gêneros definidos.<br />
O grupo gama compreende os gêneros Chromatium, Escherichia e outras bactérias entéricas, além de Legionella,<br />
Vibrio e outros 9 gêneros.<br />
O grupo delta reúne os gêneros Bdellovibrio, Myxococcus e bactérias redutoras do íon sulfato (sulphate-reducing<br />
bacteria).<br />
No grupo epsilon estão alocados os gêneros Campylobacter, Helicobacter, Thiovulum e Wolinella.<br />
As proteobactérias apresentam grande diversidade de morfologia celular e fisiologia. As estratégias para<br />
obtenção de energia são várias, incluindo organismos com metabolismo quimiolitotrófico, quimiorganotróficos e<br />
fototrófico, além de outras vias metabólicas especializadas em organismos adaptados a nichos ecológicos diversos<br />
(e.g., Thiobacillus spp.).
Bacteria<br />
Divisões Bacterianas Abundantes, Porém Pouco Conhecidas<br />
Acidobacterium: Divisão recentemente reconhecida, compreendendo três espécies de bactérias cultivadas:<br />
Acidobacterium capsulatum (Hiraishi et al., 1995), um organismo heterotrófico aeróbio moderadamente acidófilo,<br />
Holophaga foetida (Liesack et al., 1994), um aneróbio estrito fermentador de compostos aromáticos; e Geothrix<br />
fermentans (Lonergan et al., 1996), um anaeróbio estrito fermentador de acetato, além de diversas seqüências<br />
ambientais isoladas de aqüíferos contaminados, água do mar profunda, lago de água doce, mangue e, principalmente,<br />
de solos (Kuske et al., 1997; Ludwig et al., 1997). A ocorrência de seqüências de organismos da Divisão Acidobacterium<br />
em diversos estudos ambientais sugere que estes microrganismos são componentes ecológicos significativos em<br />
diversos ecossistemas, particularmente em comunidades de solo (Hugenholtz et al., 1998a).<br />
Bactérias Verdes Não-Sulfurosas: Grupo constituído por organismos com amplo espectro fenotípico,<br />
incluindo representantes cultivados com metabolismo fototrófico anóxico (Chloroflexus spp.) e organotrófico<br />
termófilo (Herpetosiphon aurantiacus, Sphaerobacter thermophilus e Thermomicrobium roseum). Espécies de Chloroflexus<br />
são metabolicamente versáteis, pois podem também se desenvolver no escuro, como organismos<br />
quimiorganotróficos, sob condições aeróbias. A diversidade metabólica destes organismos e a ocorrência de<br />
organismos não-cultivados em diversos habitats, incluindo aqüíferos, ambientes geotermais e marinhos, sedimentos<br />
e solo, sugerem que estes organismos possam ter um papel ecológico importante nestes ambientes.<br />
Verrucomicrobia: Divisão recentemente proposta (Hedlund et al., 1997), com alguns gêneros de organismos<br />
cultivados, incluindo Verrucomicrobium spinosum, Prosthecobacter spp., isolados de água doce, e linhagens de<br />
ultramicrobactérias (organismos com aproximadamente 0.1 µm 3 em volume), isoladas de solo (Janssen et al., 1997).<br />
Estudos de seqüências ambientais indicam que Verrucomicrobia, assim como os organismos da Divisão Acidobacterium,<br />
são abundantes e amplamente distribuídos na natureza, particularmente em solos. Estimativas para alguns<br />
representantes deste grupo (linhagem EA25 da subdivisão 2 de Verrucomicrobia), através de PCR, são da ordem de<br />
10 7 a 10 8 células por grama de solo, aproximadamente 1 a 10% da contagem microbiana total (Lee et al., 1996).<br />
Divisão OP11: A Divisão OP11 representa um grupo candidato de microrganismos recentemente proposta<br />
(Hugenholtz et al., 1997, 1998a,b), para a qual não existem ainda linhagens cultivadas. Seqüências de OP11 foram<br />
detectadas em vários estudos independentes conduzidos em habitats diversos, tais como diferentes tipos de<br />
solo, sedimentos de água doce, subsolo profundo e fontes termais, sugerindo que estes organismos possam<br />
desempenhar papéis ecológicos importantes (Hugenholtz et al., 1998a).<br />
3. Estado do conhecimento<br />
O desenvolvimento de novas técnicas de detecção e caracterização de microrganismos a partir do emprego<br />
da metodologia de PCR (polymerase chain reaction) - incluindo a detecção e classificação de microrganismos a partir<br />
de seqüências de rRNA 16S amplificadas a partir de DNA/RNA extraído de comunidades microbianas, e do<br />
emprego de técnicas de hibridização com sondas grupo-específicas diretamente em amostras de DNA/RNA<br />
das mesmas comunidades - permitiu grandes avanços na detecção e caracterização taxonômica dos<br />
microorganismos em ambientes naturais.<br />
Os avanços na utilização de técnicas moleculares para o estudo in situ estão abrindo novas fronteiras no<br />
estudo da ecologia microbiana (Amann et al., 1995). A primeira tentativa de caracterização da diversidade microbiana<br />
presente em uma amostra ambiental, através da análise do rRNA, ocorreu aproximadamente há uma década atrás.<br />
Nestes estudos, o rRNA 5S de amostras provenientes de ambientes diferenciados, como bactérias simbiontes de<br />
invertebrados quimioautotróficos e bactérias de uma fonte termal a 95 ºC, foi extraído e analisado por eletroforese.<br />
Mais tarde, moléculas de rRNA 16S e 23S passaram a ser empregadas, uma vez que oferecem maior informação,<br />
uma vez que as sequências têm aproximadamente 1.500 e 3.000 nucleotídeos, respectivamente, com grau de<br />
conservação suficiente para permitir a análise filogenética da informação molecular. Após o advento da metodologia<br />
de PCR, fragmentos de rRNA 16S passaram a ser amplificados seletivamente, a partir de amostras de DNA total<br />
extraído da amostra de interesse (Amann et al., 1995).<br />
O DNA e o RNA podem ser extraídos diretamente das populações microbianas e analisados através de<br />
diferentes técnicas moleculares para determinação da complexidade da comunidade e a identificação dos seus<br />
7
8<br />
V.P. Canhos et al.<br />
membros. Diferentes estratégias podem ser empregadas para a análise da diversidade genética microbiana em<br />
uma amostra ambiental, tais como a clonagem e o seqüenciamento de fragmentos de rDNA 16S amplificados por<br />
PCR (Amann et al., 1995; Pace et al., 1985), ou através da análise direta da diversidade de seqüências no pool de<br />
fragmentos, através de métodos eletroforéticos de alta resolução. Esta última abordagem representa uma nova<br />
estratégia para a análise da diversidade microbiana, baseada na eletroforese de fragmentos relativamente pequenos<br />
de rDNA 16S (~500 pares de bases), amplificados por PCR, e analisados em gel de poliacrilamida contendo um<br />
gradiente linear de agentes desnaturantes. Na análise de “DGGE” (denaturing gradient gel electrophoresis), o pool de<br />
produtos de PCR amplificados a partir de uma amostra ambiental, contendo fragmentos de rDNA 16S de mesmo<br />
comprimento, mas com seqüências de bases diferentes, pode ser separado de acordo com o comportamento de<br />
desnaturação particular de cada fragmento, relacionado ao conteúdo total de G+C e à distribuição destas ao<br />
longo do fragmento. O resultado final é um padrão de bandas de DNA, distribuído ao longo do gradiente de<br />
desnaturação do gel, de acordo com os respectivos pontos de desnaturação específicos de cada fragmento.<br />
A análise de DGGE tem sido usada no estudo da diversidade bacteriana em diferentes ambientes, tais como<br />
biorreatores experimentais, água e sedimentos de fontes termais e outros ambientes extremos, assim como nos<br />
estudos de estrutura e dinâmica de comunidades microbianas aquáticas, como bacterioplancton de água doce. A<br />
metodologia de DGGE proporciona ainda a possibilidade de isolamento e seqüenciamento de bandas distintas,<br />
permitindo a análise filogenética dos membros da comunidade (Muyzer et al., 1996; van Elsas et al., 1998).<br />
No Brasil, Rosado et al. (1998) empregaram a técnica de DGGE para estudar a diversidade do gene nifH de<br />
linhagens isoladas de Paenibacillus spp. e organismos presentes em amostras de solo. Amostras de rizosfera de<br />
solos de várzea e cerrado brasileiro de Minas Gerais foram estudadas, demonstrando a diversidade dos genes<br />
nifH nas diferentes populações de P. azotofixans. A diversidade de P. azotofixans associados a diferentes partes de<br />
plantas de milho cultivadas em solos brasileiros também foi caracterizada através de métodos de tipagem molecular<br />
(Seldin et al., 1998).<br />
Estudos envolvendo amplificação, clonagem e seqüenciamento de rRNA 16S, realizados em amostras de<br />
solos amazônicos, revelaram a ocorrência de microrganismos pouco comuns em amostras ambientais e<br />
demonstraram também o impacto do desmatamento sobre a diversidade microbiana. Os resultados demonstraram<br />
o predomínio de organismos do Domínio Bacteria (98) em relação a Archaea (2), sendo que algumas seqüências<br />
sugerem a existência de um novo grupo de Proteobacteria (Bornema & Triplett, 1997).<br />
Algumas propriedades de organismos ainda não-cultivados e/ou desconhecidos podem ser inferidas com<br />
base nas propriedades de organismos cultivados filogeneticamente relacionados. As seqüências de rRNA 16S<br />
podem também servir como modelo para o desenho de sondas moleculares para a detecção e estudo destes<br />
organismos na natureza. A aplicação de métodos moleculares na caracterização de comunidades microbianas na<br />
natureza vem permitindo uma expansão significativa da extensão do conhecimento sobre diversidade microbiana.<br />
Seqüências isoladas do meio ambiente são raramente idênticas a seqüências de organismos isolados e cultivados,<br />
sugerindo que o nosso conhecimento da estrutura filogenética da biosfera microbiana com base em estudos de<br />
isolamento e cultivo é seriamente limitado.<br />
A enorme diversidade e riqueza de organismos do Domínio Bacteria é amplamente reconhecida, porém o<br />
inventário é pobre. Como anteriormente observado, menos de 1% do total de espécies existentes é conhecido.<br />
Isto torna a diversidade dessas espécies um enigma a ser desvendado<br />
De acordo com Tiedje (1994), as seguintes questões devem ser consideradas nos estudos sobre diversidade<br />
microbiana:<br />
· O que avaliar nos estudos sobre diversidade de microrganismos?<br />
· Qual a extensão (ordem de grandeza) dessa diversidade em nosso planeta?<br />
· Qual a distribuição espacial de microrganismos? Uma amostra de solo no Brasil possui a mesma diversidade<br />
de organismos que uma amostra de solo da Austrália, por exemplo?<br />
· Qual o impacto destas diferenças sobre a sustentabilidade da biosfera e potencial de novas descobertas<br />
em biotecnologia?<br />
· Quais os métodos mais apropriados para desvendar a diversidade bacteriana?
Bacteria<br />
Frente a estas questões e suas implicações, é muito importante, sobretudo, a definição de “espécie” bacteriana,<br />
pois o termo é menos preciso para organismos procariontes do que para eucariontes. Para responder estas<br />
questões tornam-se necessários inventários em diferentes níveis de resolução taxonômica, abrangendo desde o<br />
nível macro-evolutivos (e.g., Divisões e Famílias) até a caracterização da diversidade infra-específica de populações<br />
(subespécies e genótipos).<br />
Outras questões a serem abordadas:<br />
· É possível isolar e/ou cultivar os organismos estudados nos diferentes ambientes? Qual a importância<br />
de associações interespecíficas?<br />
· Qual a importância ecológica de organismos “raros” em diferentes comunidades bacterianas?<br />
· Existe especialização em organismos simbiontes associados a animais e plantas?<br />
Maiores esforços para ampliar o conhecimento sobre a diversidade microbiana e filogenia de bactérias são<br />
fundamentais para a exploração de novos isolados de interesse ambiental e industrial (Istock et al 1996). É<br />
preciso também evidenciar a importância da taxonomia em estudos sobre a evolução das espécies. Segundo estes<br />
autores, há mais de três séculos os naturalistas procuram descrever as formas divergentes de vida na Terra para<br />
explicar a evolução, mas a descrição das células procarióticas, que parece primordial para os estudos evolutivos,<br />
está apenas iniciando.<br />
A ênfase dada à sistemática molecular microbiana nos últimos anos parece ser uma resposta mais rápida ao<br />
entendimento da diversidade bacteriana, somando-se, sem dúvida, às práticas tradicionais para a compreensão da<br />
estrutura e função fisiológica das células procarióticas. A taxonomia tradicional não deve prevalecer, pois a<br />
identificação de espécies não é meramente uma questão de denominar uma espécie bacteriana, mas de situá-la,<br />
sobretudo, em seu contexto ecológico. Análises fenotípicas e genotípicas combinadas (taxonomia polifásica), são<br />
fundamentais no esclarecimento da diversidade bacteriana.<br />
No estado de São Paulo, tomando-se como exemplo os estudos desenvolvidos em ecologia microbiana<br />
que resultaram no isolamento e taxonomia polifásica de bactérias, observa-se uma predominância de estudos de<br />
bactérias patogênicas presentes em sistemas aquáticos. São relatados estudos sobre víbrios patogênicos em<br />
amostras de Crassostrea gigas e mexilhões (Perna perna) do litoral de São Paulo (Matté, 1993), com a identificação de<br />
linhagens de Vibrio alginolyticus, V. cholerae não-O:1, V. fluvialis (somente detectado em ostras), V. furnissii, V.<br />
mimicus, V. parahemolyticus e V. vulnificus; detecção de V. cholerae O:1 e não-O:1 em amostras de diferentes localidades<br />
do estado de São Paulo, através da amplificação de genes codificadores para toxinas específicas por PCR (Rivera<br />
et al. 1995); detecção de Salmonella e Vibrio cholerae O:1 em análises de avaliação sanitária de águas em diversas<br />
localidades de São Paulo (Rivera & Martins, 1996); e o isolamento e identificação de Aeromonas spp. (A. caviae, A.<br />
hydrophila, A. jandaei e A. sobria) em amostras de água e sedimento da Represa de Guarapiranga (Matté, 1995).<br />
A Companhia de Tecnologia de Saneamento do Estado de São Paulo (CETESB) se destaca na condução de<br />
trabalhos para identificação de microrganismos de importância ambiental e sanitária, particularmente na avaliação<br />
de bactérias patogênicas em águas continentais e costeiras (Sanchez, 1986) e bactérias anaeróbias estritas (Desulfovibrio<br />
sp. e Desulfotomaculum sp.; Vazoller et al., 1988).<br />
Alguns grupos de pesquisas no estado de São Paulo vêm desenvolvendo estudos de microbiologia aplicada<br />
voltada para a ecologia microbiana e a exploração tecnológica da diversidade de microrganismos, incluindo a<br />
aplicação de bactérias em biorremediação ambiental de áreas poluídas (Pellizari et al, 1996) e resíduos de pesticidas<br />
(Esposito et al., 1998), biodigestão de compostos recalcitrantes em efluentes industriais (Souza et al., 1991;<br />
Vazoller, 1995, 1997), biotransformação (Cagnon et al., 1998), estudo sistemático de fitopatógenos de importância<br />
agrícola (Robbs, 1981; Jabuonsky et al., 1986; Malavolta-Júnior, 1996; Beretta et al., 1997; Machado et al., 1997;<br />
Rosato et al., 1998), e bactérias ambientais contaminantes de importantes processos de produção industrial<br />
(Pinhati et al., 1997).<br />
Dentre os grupos de pesquisa inter-institucionais no estado de São Paulo atuantes em sistemática molecular<br />
bacteriana com aplicações ambientais, destacam-se:<br />
· CCT/Fundação André Tosello e FEA/UNICAMP: Diversidade, bioprospecção, taxonomia polifásica e<br />
preservação de microrganismos de importância ambiental e agro-industrial (C. Y. Umino, S. Y. Eguchi, G.<br />
P. Manfio e V. P. Canhos);<br />
9
10<br />
V.P. Canhos et al.<br />
· CETESB, CBMEG/UNICAMP e CCT/Fundação André Tosello: Diversidade molecular de tiobacilos<br />
em ambientes antrópicos (L. M. M. Ottoboni, M. I. Z. Sato, G. S. Valent, N. C. Stoppe, C. M. Rech, C. A.<br />
Coimbrão e G. P. Manfio);<br />
· IBSBF, CBMEG/UNICAMP e CCT/Fundação André Tosello: Diversidade de bactérias fitopatogênicas<br />
(J. R. Neto, S. A. L. Destéfano, L. O. S. Beriam, V. A. Malavolta Júnior, I. Gatti de Almeida, Y. B. Rosato,<br />
F. Fantinatti e G P. Manfio);<br />
· ICB/USP e CCT/Fundação André Tosello: Caracterização de bactérias endofíticas fixadoras de nitrogênio<br />
e bactérias associadas a húmus (C. A. Moreira-Filho, C. Pavan e M.-A. van Sluys, H. R. Barbosa e G. P.<br />
Manfio);<br />
· ICB/USP e Escola de Engenharia São Carlos/USP: Diversidade de bactérias associadas à degradação de<br />
compostos recalcitrantes (V. H. Pellizari, R. Vazoller, E. Esposito, R. Montone, M. Bicego, I. N. G.<br />
Rivera e E. Foresti).<br />
4. Conservação ex-situ<br />
A caracterização taxonômica polifásica de bactérias, é fundamental para a identificação dos principais<br />
componentes do microbiota envolvido em processos ambientais e para o estudo da diversidade biológica (fenotípica<br />
e genômica) e funcional dos mesmos. O depósito destes recursos genéticos em coleções ex-situ, possibilita a<br />
realização de estudos complementares posteriores e o acúmulo de conhecimento científico sobre a diversidade<br />
bacteriana do estado de São Paulo e a potencial exploração tecnológica destes organismos em aplicações<br />
biotecnológicas e agro-industriais.<br />
Via de regra, o acesso à informações integradas sobre acervos/publicações científicas é difícil e boa parte<br />
dos microrganismos estudados em projetos desenvolvidos com recursos públicos são perdidos, devido ao<br />
emprego de métodos de preservação inadequados e à falta de política institucional de apoio à manutenção de<br />
acervos científicos. Salvo raras exceções, microrganismos isolados de ecossistemas do estado de São Paulo não<br />
estão disponíveis para estudos comparativos e aplicações biotecnológicas.<br />
Merecem destaque especial a Coleção de Culturas Tropical (CCT), da Fundação André Tosello, e a Coleção<br />
de Bactérias Fitopatogênicas do Instituto Biológico (IBSBF), da Seção de Bacteriologia Fitopatológica, Instituto<br />
Biológico, Campinas.<br />
A Coleção de Culturas Tropical (CCT) conta com infra-estrutura física adequada para a realização de<br />
trabalhos de taxonomia polifásica e preservação de microrganismos. Esta capacitação permite a realização de<br />
trabalhos de caracterização convencional (micromorfologia e caracterização bioquímica), quimiotaxonômica (análise<br />
de componentes de parece celular) e molecular (RAPD, ribotipagem, ARDRA e seqüenciamento de rDNA 16S),<br />
assim como a preservação de microrganismos por liofilização e criogenia. A CCT está ampliando as instalações<br />
para atuar como um centro depositário de material biológico associado a processos de patentes, sob credenciamento<br />
do Instituto Nacional de Propriedade Industrial (INPI), e também como centro de deposito de acervos<br />
especializados de diversidade microbiana de biomas brasileiros, visando a atender aos requerimentos da nova Lei<br />
de Acesso a Recursos Genéticos (em tramitação no Congresso Nacional) e aos compromissos assumidos na<br />
implementação da Convenção sobre a Diversidade Biológica (CDB). A CCT possui um acervo com mais de<br />
6.500 linhagens de interesse industrial e ambiental de microrganismos não-patogênicos ao homem (bactérias,<br />
fungos e leveduras; nível de contenção P2). O acervo CCT inclui linhagens-tipo relevantes para estudos<br />
taxonômicos, assim como linhagens-referência para testes de desinfecção, esterilização e testes de desafio ao<br />
ataque por microrganismos associados à biodeterioração e biocorrosão. Ao longo da última década, fruto do<br />
trabalho de pesquisa associado ao desenvolvimento de teses de Pós-graduação e pesquisas aplicadas, a CCT<br />
estruturou várias coleções especializadas de microrganismos associados a ecossistemas paulistas, incluindo um<br />
relevante acervo de microrganismos coletados na Reserva Ecológica Juréia-Itatins e um acervo de organismos<br />
associados à produção agrícola e processamento industrial da cana-de-açúcar no estado de São Paulo. Mais<br />
recentemente, a equipe da CCT, em colaboração com a Faculdade de Engenharia de Alimentos/Unicamp, vem<br />
realizando estudos ecológicos do microbiota associado a doenças de plantas cítricas e processos industriais de
Bacteria<br />
produção de sucos concentrado de laranja. As informações disponíveis sobre as linhagens depositadas na CCT<br />
estão estruturadas no sistema SDMS (Strain Database Management System), um sistema de banco de dados interativo<br />
que permite a realização de buscas sobre nomes, códigos da linhagem em outras coleções de cultura, origem,<br />
taxonomia, ecologia, propriedades e aplicações das linhagens do acervo, inclusive as referências bibliográficas.<br />
O sistema SDMS está disponível para consulta on-line na Internet no endereço http://www.bdt.org.br/cct/.<br />
A Coleção de Bactérias Fitopatogênicas do Instituto Biológico (IBSBF), da Seção de Bacteriologia<br />
Fitopatológica, Instituto Biológico, possui infra-estrutura para a caracterização convencional e preservação de<br />
bactérias associadas a doenças de cultivares de planta de interesse agrícola. A Seção atende a solicitações de<br />
interessados e processa cerca de 200 amostras por mês. Os dados do acervo IBSBF estão informatizados e<br />
disponíveis on-line em parceria estabelecida com a Base de Dados Tropical (BDT), no endereço http://<br />
www.bdt.org.br/bdt/ibsbf/. Ao longo dos últimos 20 anos, a coleção do IBSBF construiu o melhor arquivo de<br />
bactérias fitopatogênicas do país e hoje a coleção conta com mais de 1.000 linhagens identificadas e preservadas<br />
por liofilização. É um acervo único, de grande relevância para o mapeamento de patógenos de plantas e agrobiodiversidade.<br />
Os acervos da CCT e IBSBF encontram-se também listados no Catálogo Nacional de Linhagens<br />
– Bactérias (Canhos et al., 1989).<br />
5. Recomendações<br />
Visando o fortalecimento de grupos atuantes em taxonomia polifásica de bactérias no estado de São Paulo,<br />
recomenda-se:<br />
· Estruturação de uma rede cooperativa de laboratórios e especialistas em microbiologia sistemática e<br />
taxonomia polifásica de bactérias, voltada principalmente para a caracterização e identificação de isolados<br />
de importância ambiental, agrícola e industrial;<br />
· Estruturação de acervos representativos da diversidade microbiana associada aos ecossistemas do estado<br />
de São Paulo;<br />
· Estabelecimento de um programa de treinamento, com cursos de curta e média duração, destinados à<br />
formação de pessoal em nível técnico e/ou universitário, com foco em taxonomia polifásica, ecologia<br />
microbiana e exploração tecnológica da diversidade microbiana;<br />
· Disponibilização através da Internet de métodos e protocolos em taxonomia polifásica, bases de dados<br />
com características taxonômicas e tecnológicas de linhagens de interesse industrial e ambiental; e<br />
· Realização de estudos e publicação de resultados de trabalhos científicos nas áreas específicas de atuação<br />
dos participantes do projeto, enfocando o aspecto de aplicação prática dos conhecimentos gerados na<br />
exploração tecnológica da diversidade microbiana do estado de São Paulo.<br />
6. Bibliografia<br />
Amann, R.I.; Ludwig, W. & Schleifer, K.H. 1995. Phylogenetic identification and in situ detection of individual<br />
microbial cells without cultivation. Microbiological Reviews, 59: 143-169.<br />
Berreta, M.J.G.; Barthe, G.A.; Ceccardi, T.L.; Lee, R.F. & Derrick, K.S. 1997. A survey for strains of<br />
Xylella fastidiosa in Citrus affected by citrus variegated chlorosis and Citrus blight in Brazil. Plant Disease,<br />
81: 1196-1198.<br />
Bornema, J. & Triplett, E.W. 1997. Molecular microbial diversity in soils from eastern Amazonia: evidence<br />
for unususal microorganisms and microbial population shifts associated with deforastation. Applied and<br />
Environmental Microbiology, 63: 2647-2653.<br />
Cagnon, J.R.; Porto, A.L.M.; Manfio, G.P.; Eguchi, S.Y. & Marsaioli, A.J. 1998. First evaluation of the<br />
Brazilian microorganisms biocatalytic potential. Tetrahedron: Asymmetry (in press).<br />
11
12<br />
V.P. Canhos et al.<br />
Canhos, V.P.; Souza, S. & Canhos, D.A.L. (eds.). 1989. Catálogo nacional de linhagens, vol. I - Bactérias.<br />
Campinas: Fundação Tropical de Pesquisas e Tecnologia “André Tosello”.<br />
van Elsas, J.D.; Duarte, G.F.; Rosado, A.S. & Smalla, K. 1998. Microbiological and molecular biological<br />
methods for monitoring microbial inoculants and their effect in the soil environment Journal of<br />
Microbiological Methods, 32: 133-54.<br />
Esposito, E.; Paulillo, S.M. & Manfio, G.P. 1998. Biodegradation of the herbicide diuron in soil by indigenous<br />
actinomycetes. Chemosphere, 37: 541-548.<br />
Hedlund, B.P.; Gosink, J.J. & Staley, J.T. 1997. Verrucomicrobia div. nov., a new division of the Bacteria<br />
containing three new species of Prosthecobacter. Antonie van Leeuwenhoek, 72: 29-38.<br />
Hiraishi, A.; Kishimoto, N.; Kosako, Y.; Wakao, N. & Tano, T. 1995. Phylogenetic position of the<br />
menaquinone-containing acidophilic chemoorganotroph Acidobacterium capsulatum. FEMS Microbiology Letters,<br />
132: 91-94<br />
Hugenholtz, P.; Goebel, B.M. & Pace, N.R. 1998a. Impact of culture-independent studies on the emerging<br />
phylogenetic view of bacterial diversity. Journal of Bacteriology, 180: 4765-4774.<br />
Hugenholtz, P.; Pitulle, K.L. & Pace, N.R. 1998b. Novel division level bacterial diversity in a Yellowstone<br />
hot spring. Journal of Bacteriology, 180: 366-376.<br />
Istock, C.A.; Bell, J.A.; Fergusson, N. & Istock, N.L. 1996. Bacterial species and evolution: theoretical and<br />
practical perspectives. Journal of Industrial Microbiology, 17: 137-150.<br />
Jabuonsky, R.E.; Takatsu, A. & Retfschneider, F.J.B. 1986. Levantamento e identificação de espécies de<br />
Erwinia de diferentes plantas hospedeiras e regiões do Brasil. Fitopatologia Brasileira, 11: 185-195.<br />
Janssen, P.H.; Schuhmann, A.; Morschel, E. & Rainey, F.A. 1997. Novel anaerobic ultramicrobacteria<br />
belonging to the Verrucomicrobiales lineage of bacterial descent isolated by dilution culture from anoxic rice<br />
paddy soil. Applied and Environmental Microbiology, 63: 1382-1388.<br />
Kandler, O. 1994. The early diversification of life. Nobel Symposia, 84: 152-160.<br />
Krieg, N.R. & Holt, J.G. (eds.). 1984. Bergey’s manual of systematic bacteriology. Volume 1: Gram-negatives<br />
of general, medical, or industrial importance. Baltimore: Williams & Wilkins, Co.<br />
Kuske, C.R.; Barns, S.M. & Busch, J.D. 1997. Diverse uncultivated bacterial groups from soils of the arid<br />
southwestern United States that are present in many geographic regions. Applied and Environmental<br />
Microbiology, 63: 3614-362.<br />
Lapage, S.P.; Sneath, P.H.A.; Lessel, E.F.; Skerman, V.B.D.; Seeliger, H.P.R. & Clark, W.A. 1975.<br />
International code of nomenclature of bacteria. Revision 1975. Washington, D.C.: American Society for<br />
Microbiology.<br />
Lee, S.-Y.; Bollinger, J.; Bezdicek, D. & Ogram, A. 1996. Estimation of the abundance of an uncultured soil<br />
bacterial strain by a competitive quantitative PCR method. Applied and Environmental Microbiology, 62:<br />
3787-3793.<br />
Liesack, W., F. Bak, J.U. Kreft, and E. Stackebrandt. 1994. Holophaga foetida gen. nov., sp. nov., a new<br />
homoacetogenic bacterium degrading methoxylated aromatic compounds. Archives of Microbiology, 162:<br />
85-90.<br />
Logan, N.A. 1994. Bacterial systematics. Oxford: Backwell Scientific Publications.<br />
Lonergan, D.J.; Jenter, H.L.; Coates, J.D.; Phillips, E.J.P.; Schmidt, T. & Lovely, D.R. 1996. Phylogenetic<br />
analysis of dissimilatory Fe(III)-reducing bacteria. Journal of Bacteriology, 178: 2402-2408.<br />
Ludwig, W.; Bauer, S.H.; Bauer, M.; Held, I.; Kirchhof, G.; Schulze, R.; Huber, I.; Spring, S.; Hartmann,<br />
A. & Schleifer. K.-H. 1997. Detection and in situ identification of representatives of a widely distributed<br />
new bacterial phylum. FEMS Microbiology Letters, 153: 181-190.
Bacteria<br />
Machado, M.A.; Targon, M.L.P.N.; Beretta, M.J.G.; Laranjeira, F.F. & Carvalho, A.S. 1997. Detecção de<br />
Xylella fastidiosa em espécies e variedades de citros sobre-enxertadas em laranja Pera com clorose variegada<br />
dos citros (CVC). Fitopatologia Brasileira, 22: 30-33.<br />
Madigan, M.T.; Martinko, J.M. & Parker, J. 1997. Brock biology of microorgansims. 8 a<br />
Ed. Caps. 15, 16 e<br />
17. New Jersey: Prentice-Hall. p. 606-768.<br />
Maidak, B.L.; Olsen, G.J.; Larsen, N.; Overbeek, R.; McCaughey, M.J. & Woese, C.R. 1997. The RDP<br />
(Ribosomal Database Project). Nucleic Acids Res., 25: 109-110.<br />
Malavolta-Júnior, V.A. 1996. Contribuição ao conhecimento de bactérias do gênero Xanthomonas patogênicas a<br />
cereais de inverno. Tese de Doutorado. Piracicaba: Escola Superior de Agricultura “Luiz de Queiroz”<br />
(USP).<br />
Matté, G.R. 1993. Isolamento de víbrios potencialmente patogênicos em moluscos bivalves. Tese de Doutorado.<br />
São Paulo: Universidade de São Paulo (USP).<br />
Matté, M.H. 1995. Pesquisa de Aeromonas spp. potencialmente patogênicas em alguns pontos da Represa de<br />
Guarapiranga destinados à recreação e captação para abastecimento público. Dissertação de Mestrado. São<br />
Paulo: Universidade de São Paulo (USP).<br />
Muyzer G.; Hottentrager, S.; Teske, T. & Wawer C. 1996. Denaturing gradient gel electrophoresis of PCRamplified<br />
16S rDNA – a new molecular approach to analyse the genetic diversity of mixed microbial<br />
communities. In: Molecular methods ecology manual. Cap. 3.4.4. p 1-23.<br />
Pace, N.R. 1997. A molecular view of microbial diversity and the biosphere. Science, 276: 734-740.<br />
Pace, N.R.; Stahl, D.A.; Lane, D.J. & Olsen, G.J. 1985. Analyzing natural microbial populations by rRNA<br />
sequences. ASM News, 51: 4-12.<br />
Pellizari, V.H.; Bezborodnikov, S.; Quensen, J.F. 3rd & Tiedje, J.M. 1996. Evaluation of strains isolated by<br />
growth on naphthalene and biphenyl for hybridization of genes to dioxygenase probes and polychlorinated<br />
biphenyl-degrading ability. Applied and Environmental Microbiology, 62: 2053-2058.<br />
Pinhati, M.E.M.C.; Variane, S.; Eguchi, S.Y. & Manfio, G.P. 1997. Detection of acidothermophilic bacilli<br />
in industrialized fruit juices. Fruit Processing, 7: 350-353.<br />
Rivera, I.G. & Martins, M.T. 1996. Bactérias enteropatogênicas no ambiente aquático. Revista de Ciências<br />
Farmacêuticas, 17: 115-136.<br />
Rivera, I.G.; Chowdhury, M.A.R.; Sanchez, P.S.; Dato, M.I.; Huq, A.; Colwell, R.R. & Martins, M.T.<br />
1995. Detection of cholera (ctx) and zonula occludens (zot) toxin genes in Vibrio cholerae O1, O139 and non-<br />
O1 strains. World Journal of Microbiology and Biotechnology, 11: 572-577.<br />
Robbs, C.F. 1981. Taxonomia e bioecologia e principais representantes do gênero Erwinia no Brasil. Fitopatologia<br />
Brasileira, 6: 304-309.<br />
Rosado, A.S.; Duarte G.F.; Seldin, L. & van Elsas, J.D. 1998. Genetic diversity of nih genes sequences in<br />
Paenibacillus azotofixans strains and soil samples analysed by denaturing gradient gel electrophoresis of<br />
PCR-amplified gene fragments. Applied and Environmental Microbiology, 64: 2770-2779.<br />
Rosato, Y.B.; Neto, J.R.; Miranda, V.S.; Carlos, E.F. & Manfio, G.P. 1998. Diversity of a Xylella fastidiosa<br />
population isolated from Citrus sinensis affected by Citrus variegated chlorosis in Brazil. Systematic and<br />
Applied Microbiology, 21: 593-598.<br />
Sanchez, P. 1986. Evaluation of the sanitary quality of marine recreational waters and sands from beaches of<br />
the São Paulo State, Brazil. Water Science and Technology, 18: 61-72.<br />
Schlegel, H.G. & Jannasch, H.W. 1992. Prokaryotes and their habitats. In: Ballows, A., Truper, H.G.;<br />
Dworkin, M.; Harder, W. & Schleifer, K.-H. (eds.). The prokaryotes. Vol. I. New York: Springer<br />
Verlag. p. 75-125.<br />
13
14<br />
V.P. Canhos et al.<br />
Seldin, L.; Rosado, A.S.; William da Cruz, D.; Nobrega, A.; van Elsas, J.D. & Paiva, E. 1998. Comparison<br />
of Paenibacillus azotofixans strains isolated from rhizoplane, rhizosphere, and non-root-associated soil from<br />
maize planted in two different Brazilian soils. Applied and Environmental Microbiology, 64: 3860-3868.<br />
Sneath, P.H.A. & Holt, J.G. (eds.). 1986. Bergey’s manual of systematic bacteriology. Volume 2: Gram<br />
positives other than Actinomycetes. Baltimore: Williams & Wilkins, Co.<br />
Souza, M.E.; Fuzaro, G. & Polegato, A.R. 1991. Thermophilic anaerobic digestion of vinasse in pilot plant<br />
UASB reactor. In: Proceedings of Sixth International Symposium on Anaerobic Digestion, São Paulo,<br />
Brazil. p. 191-200.<br />
Stackebrandt, E.; Rainey, F.A. & Ward-Rainey, N.L. 1997. Proposal for a new hierarchic classification<br />
system, Actinobacteria classis nov. International Journal of Systematic Bacteriology, 47: 479-491.<br />
Staley, J.T. & Holt, J.G. (eds.). 1989. Bergey’s manual of systematic bacteriology. Volume 3: Archaeobacteria,<br />
Cyanobacteria, and the remaining Gram-negatives. Baltimore: Williams & Wilkins, Co.<br />
Tiedje, J.M. 1994. Microbial Diversity: of value to whom? ASM News, 60: 524-525.<br />
Tortora, G.J.; Funke, B.R. & Case, C.L. 1998a. The microbial world. Cap. 1. In: Microbiology – an introduction.<br />
Tortora, G.J.; Funke, B.R. & Case, C.L. 6 Ed. The Benjamim Cummings Publishing Company.<br />
Tortora, G.J.; Funke, B.R. & Case, C.L. 1998b. Bacteria. Cap. 11. In: Microbiology – an introduction. Tortora,<br />
G.J.; Funke, B.R. & Case, C.L. 6 Ed. The Benjamim Cummings Publishing Company, Inc.<br />
Vazoller, R.F. 1995. Avaliação do ecossistema microbiano de um biodigestor anaeróbio de fluxo ascendente e<br />
manta de lodo, operado com vinhaça sob condições termofílicas. Tese de Doutorado. São Carlos: Escola<br />
de Engenharia de São Carlos/USP.<br />
Vazoller, R.F. 1997. Microbial aspects of thermophilic anaerobic biodigestion of vinasse. In: Proceedings of<br />
ISME 7 – Progress in Microbial Ecology. Santos. p. 527-532.<br />
Vazoller, R.F.; Rech, C.M.; Figueiredo, M.G. & Giaj-Levra, L.A. 1988. Bacterial identification of granular<br />
sludge from domestic sewage UASB-reactor. In: Proceedings of the 5th International Symposium on<br />
Anaerobic Digestion, Bologna, Itália. p. 61-64.<br />
Williams, S.T.; Sharpe, M.E. & Holt, J.G. (eds.). 1989. Bergey’s manual of systematic bacteriology. Volume<br />
4: Actinomycetes. Baltimore: Williams & Wilkins, Co.<br />
Woese, C.R. 1987. Bacterial evolution. Microbiological Reviews, 51: 221-271.