Genetische Diagnostik bei Entwicklungsstörungen und seltenen ...
Genetische Diagnostik bei Entwicklungsstörungen und seltenen ...
Genetische Diagnostik bei Entwicklungsstörungen und seltenen ...
Sie wollen auch ein ePaper? Erhöhen Sie die Reichweite Ihrer Titel.
YUMPU macht aus Druck-PDFs automatisch weboptimierte ePaper, die Google liebt.
Fortbildung | „Artikel des Monats“<br />
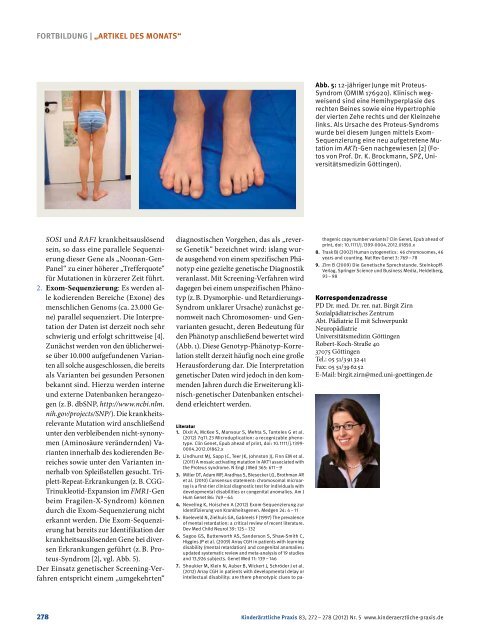
Abb. 5: 12-jähriger Junge mit Proteus-<br />
Syndrom (OMIM 176920). Klinisch wegweisend<br />
sind eine Hemihyperplasie des<br />
rechten Beines sowie eine Hypertrophie<br />
der vierten Zehe rechts <strong>und</strong> der Kleinzehe<br />
links. Als Ursache des Proteus-Syndroms<br />
wurde <strong>bei</strong> diesem Jungen mittels Exom-<br />
Sequenzierung eine neu aufgetretene Mutation<br />
im AKT1-Gen nachgewiesen [2] (Fotos<br />
von Prof. Dr. K. Brockmann, SPZ, Universitätsmedizin<br />
Göttingen).<br />
SOS1 <strong>und</strong> RAF1 krankheitsauslösend<br />
sein, so dass eine parallele Sequenzierung<br />
dieser Gene als „Noonan-Gen-<br />
Panel“ zu einer höherer „Trefferquote“<br />
für Mutationen in kürzerer Zeit führt.<br />
2. Exom-Sequenzierung: Es werden alle<br />
kodierenden Bereiche (Exone) des<br />
menschlichen Genoms (ca. 23.000 Gene)<br />
parallel sequenziert. Die Interpretation<br />
der Daten ist derzeit noch sehr<br />
schwierig <strong>und</strong> erfolgt schrittweise [4].<br />
Zunächst werden von den üblicherweise<br />
über 10.000 aufgef<strong>und</strong>enen Varianten<br />
all solche ausgeschlossen, die bereits<br />
als Varianten <strong>bei</strong> ges<strong>und</strong>en Personen<br />
bekannt sind. Hierzu werden interne<br />
<strong>und</strong> externe Datenbanken herangezogen<br />
(z. B. dbSNP, http://www.ncbi.nlm.<br />
nih.gov/projects/SNP/). Die krankheitsrelevante<br />
Mutation wird anschließend<br />
unter den verbleibenden nicht-synonymen<br />
(Aminosäure verändernden) Varianten<br />
innerhalb des kodierenden Bereiches<br />
sowie unter den Varianten innerhalb<br />
von Spleißstellen gesucht. Triplett-Repeat-Erkrankungen<br />
(z. B. CGG-<br />
Trinukleotid-Expansion im FMR1-Gen<br />
<strong>bei</strong>m Fragilen-X-Syndrom) können<br />
durch die Exom-Sequenzierung nicht<br />
erkannt werden. Die Exom-Sequenzierung<br />
hat bereits zur Identifikation der<br />
krankheitsauslösenden Gene <strong>bei</strong> diversen<br />
Erkrankungen geführt (z. B. Proteus-Syndrom<br />
[2], vgl. Abb. 5).<br />
Der Einsatz genetischer Screening-Verfahren<br />
entspricht einem „umgekehrten“<br />
diagnostischen Vorgehen, das als „reverse<br />
Genetik“ bezeichnet wird: islang wurde<br />
ausgehend von einem spezifischen Phänotyp<br />
eine gezielte genetische <strong>Diagnostik</strong><br />
veranlasst. Mit Screening-Verfahren wird<br />
dagegen <strong>bei</strong> einem unspezifischen Phänotyp<br />
(z. B. Dysmorphie- <strong>und</strong> Retardierungs-<br />
Syndrom unklarer Ursache) zunächst genomweit<br />
nach Chromosomen- <strong>und</strong> Genvarianten<br />
gesucht, deren Bedeutung für<br />
den Phänotyp anschließend bewertet wird<br />
(Abb. 1). Diese Genotyp-Phänotyp-Korrelation<br />
stellt derzeit häufig noch eine große<br />
Herausforderung dar. Die Interpretation<br />
genetischer Daten wird jedoch in den kommenden<br />
Jahren durch die Erweiterung klinisch-genetischer<br />
Datenbanken entscheidend<br />
erleichtert werden.<br />
Literatur<br />
1. Dixit A, McKee S, Mansour S, Mehta S, Tanteles G et al.<br />
(2012) 7q11.23 Microduplication: a recognizable phenotype.<br />
Clin Genet, Epub ahead of print, doi: 10.1111/j.1399-<br />
0004.2012.01862.x<br />
2. Lindhurst MJ, Sapp JC, Teer JK, Johnston JJ, Finn EM et al.<br />
(2011) A mosaic activating mutation in AKT1 associated with<br />
the Proteus syndrome. N Engl J Med 365: 611 – 9<br />
3. Miller DT, Adam MP, Aradhya S, Biesecker LG, Brothman AR<br />
et al. (2010) Consensus statement: chromosomal microarray<br />
is a first-tier clinical diagnostic test for individuals with<br />
developmental disabilities or congenital anomalies. Am J<br />
Hum Genet 86: 749 – 64<br />
4. Neveling K, Hoischen A (2012) Exom-Sequenzierung zur<br />
Identifizierung von Krankheitsgenen. Medgen 24: 4 – 11<br />
5. Roeleveld N, Zielhuis GA, Gabreels F (1997) The prevalence<br />
of mental retardation: a critical review of recent literature.<br />
Dev Med Child Neurol 39: 125 – 132<br />
6. Sagoo GS, Butterworth AS, Sanderson S, Shaw-Smith C,<br />
Higgins JP et al. (2009) Array CGH in patients with learning<br />
disability (mental retardation) and congenital anomalies:<br />
updated systematic review and meta-analysis of 19 studies<br />
and 13,926 subjects. Genet Med 11: 139 – 146<br />
7. Shoukier M, Klein N, Auber B, Wickert J, Schröder J et al.<br />
(2012) Array CGH in patients with developmental delay or<br />
intellectual disability: are there phenotypic clues to pathogenic<br />
copy number variants? Clin Genet, Epub ahead of<br />
print, doi: 10.1111/j.1399-0004.2012.01850.x<br />
8. Trask BJ (2002) Human cytogenetics: 46 chromosomes, 46<br />
years and counting. Nat Rev Genet 3: 769 – 78<br />
9. Zirn B (2009) Die <strong>Genetische</strong> Sprechst<strong>und</strong>e. Steinkopff-<br />
Verlag, Springer Science <strong>und</strong> Business Media, Heidelberg,<br />
93 – 98<br />
Korrespondenzadresse<br />
PD Dr. med. Dr. rer. nat. Birgit Zirn<br />
Sozialpädiatrisches Zentrum<br />
Abt. Pädiatrie II mit Schwerpunkt<br />
Neuropädiatrie<br />
Universitätsmedizin Göttingen<br />
Robert-Koch-Straße 40<br />
37075 Göttingen<br />
Tel.: 05 51/3 91 32 41<br />
Fax: 05 51/39 62 52<br />
E-Mail: birgit.zirn@med.uni-goettingen.de<br />
278<br />
Kinderärztliche Praxis 83, 272 – 278 (2012) Nr. 5 www.kinderaerztliche-praxis.de