Einleitung 1 1. Einleitung 1.1. Interaktionen zwischen ...
Einleitung 1 1. Einleitung 1.1. Interaktionen zwischen ...
Einleitung 1 1. Einleitung 1.1. Interaktionen zwischen ...
Sie wollen auch ein ePaper? Erhöhen Sie die Reichweite Ihrer Titel.
YUMPU macht aus Druck-PDFs automatisch weboptimierte ePaper, die Google liebt.
<strong>Einleitung</strong><br />
Pflanzenzelle vor Effektorinjektion<br />
Effektorprotein<br />
Pflanzenzelle nach Effektorinjektion<br />
Reaktion<br />
A:<br />
B:<br />
C:<br />
D:<br />
E:<br />
V<br />
V<br />
V<br />
R<br />
R<br />
R<br />
R<br />
R<br />
HopAR1<br />
AvrRpt2<br />
AvrRpm1<br />
AvrXa27<br />
PopP2<br />
RPS2<br />
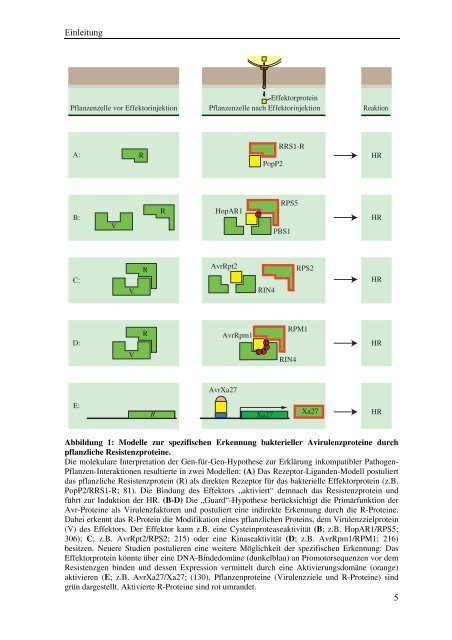
Abbildung 1: Modelle zur spezifischen Erkennung bakterieller Avirulenzproteine durch<br />
pflanzliche Resistenzproteine.<br />
Die molekulare Interpretation der Gen-für-Gen-Hypothese zur Erklärung inkompatibler Pathogen-<br />
Pflanzen-<strong>Interaktionen</strong> resultierte in zwei Modellen: (A) Das Rezeptor-Liganden-Modell postuliert<br />
das pflanzliche Resistenzprotein (R) als direkten Rezeptor für das bakterielle Effektorprotein (z.B.<br />
PopP2/RRS1-R; 81). Die Bindung des Effektors „aktiviert“ demnach das Resistenzprotein und<br />
führt zur Induktion der HR. (B-D) Die „Guard“-Hypothese berücksichtigt die Primärfunktion der<br />
Avr-Proteine als Virulenzfaktoren und postuliert eine indirekte Erkennung durch die R-Proteine.<br />
Dabei erkennt das R-Protein die Modifikation eines pflanzlichen Proteins, dem Virulenzzielprotein<br />
(V) des Effektors. Der Effektor kann z.B. eine Cysteinproteaseaktivität (B; z.B. HopAR1/RPS5;<br />
306); C; z.B. AvrRpt2/RPS2; 215) oder eine Kinaseaktivität (D; z.B. AvrRpm1/RPM1; 216)<br />
besitzen. Neuere Studien postulieren eine weitere Möglichkeit der spezifischen Erkennung: Das<br />
Effektorprotein könnte über eine DNA-Bindedomäne (dunkelblau) an Promotorsequenzen vor dem<br />
Resistenzgen binden und dessen Expression vermittelt durch eine Aktivierungsdomäne (orange)<br />
aktivieren (E; z.B. AvrXa27/Xa27; (130). Pflanzenproteine (Virulenzziele und R-Proteine) sind<br />
grün dargestellt. Aktivierte R-Proteine sind rot umrandet.<br />
5<br />
P<br />
RIN4<br />
P P<br />
P<br />
Xa27<br />
RRS1-R<br />
RPS5<br />
PBS1<br />
RIN4<br />
RPM1<br />
Xa27<br />
HR<br />
HR<br />
HR<br />
HR<br />
HR