Biología de la Conservación de Vella pseudocytisus subespecie ...
Biología de la Conservación de Vella pseudocytisus subespecie ...
Biología de la Conservación de Vella pseudocytisus subespecie ...
Create successful ePaper yourself
Turn your PDF publications into a flip-book with our unique Google optimized e-Paper software.
<strong>Biología</strong> <strong>de</strong> <strong>la</strong> <strong>Conservación</strong> <strong>de</strong> Vel<strong>la</strong> <strong>pseudocytisus</strong> subsp. paui<br />
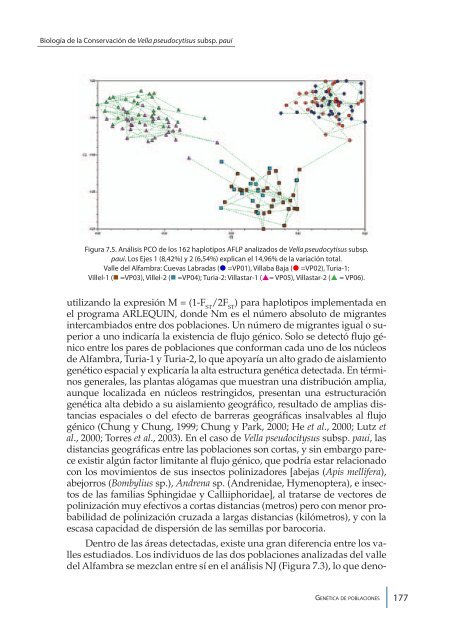
Figura 7.5. Análisis PCO <strong>de</strong> los 162 haplotipos AFLP analizados <strong>de</strong> Vel<strong>la</strong> <strong>pseudocytisus</strong> subsp.<br />
paui. Los Ejes 1 (8,42%) y 2 (6,54%) explican el 14,96% <strong>de</strong> <strong>la</strong> variación total.<br />
Valle <strong>de</strong>l Alfambra: Cuevas Labradas ( =VP01), Vil<strong>la</strong>ba Baja ( =VP02), Turia-1:<br />
Villel-1 (n =VP03), Villel-2 (n =VP04); Turia-2: Vil<strong>la</strong>star-1 (p= VP05), Vil<strong>la</strong>star-2 (p = VP06).<br />
utilizando <strong>la</strong> expresión m = (1-F /2F ) para haplotipos implementada en<br />
st st<br />
el programa arleQuin, don<strong>de</strong> nm es el número absoluto <strong>de</strong> migrantes<br />
intercambiados entre dos pob<strong>la</strong>ciones. un número <strong>de</strong> migrantes igual o superior<br />
a uno indicaría <strong>la</strong> existencia <strong>de</strong> flujo génico. solo se <strong>de</strong>tectó flujo génico<br />
entre los pares <strong>de</strong> pob<strong>la</strong>ciones que conforman cada uno <strong>de</strong> los núcleos<br />
<strong>de</strong> alfambra, turia-1 y turia-2, lo que apoyaría un alto grado <strong>de</strong> ais<strong>la</strong>miento<br />
genético espacial y explicaría <strong>la</strong> alta estructura genética <strong>de</strong>tectada. en términos<br />
generales, <strong>la</strong>s p<strong>la</strong>ntas alógamas que muestran una distribución amplia,<br />
aunque localizada en núcleos restringidos, presentan una estructuración<br />
genética alta <strong>de</strong>bido a su ais<strong>la</strong>miento geográfico, resultado <strong>de</strong> amplias distancias<br />
espaciales o <strong>de</strong>l efecto <strong>de</strong> barreras geográficas insalvables al flujo<br />
génico (Chung y Chung, 1999; Chung y park, 2000; He et al., 2000; lutz et<br />
al., 2000; torres et al., 2003). en el caso <strong>de</strong> Vel<strong>la</strong> pseudocitysus subsp. paui, <strong>la</strong>s<br />
distancias geográficas entre <strong>la</strong>s pob<strong>la</strong>ciones son cortas, y sin embargo parece<br />
existir algún factor limitante al flujo génico, que podría estar re<strong>la</strong>cionado<br />
con los movimientos <strong>de</strong> sus insectos polinizadores [abejas (Apis mellifera),<br />
abejorros (Bombylius sp.), Andrena sp. (andrenidae, Hymenoptera), e insectos<br />
<strong>de</strong> <strong>la</strong>s familias sphingidae y Calliiphoridae], al tratarse <strong>de</strong> vectores <strong>de</strong><br />
polinización muy efectivos a cortas distancias (metros) pero con menor probabilidad<br />
<strong>de</strong> polinización cruzada a <strong>la</strong>rgas distancias (kilómetros), y con <strong>la</strong><br />
escasa capacidad <strong>de</strong> dispersión <strong>de</strong> <strong>la</strong>s semil<strong>la</strong>s por barocoria.<br />
Dentro <strong>de</strong> <strong>la</strong>s áreas <strong>de</strong>tectadas, existe una gran diferencia entre los valles<br />
estudiados. los individuos <strong>de</strong> <strong>la</strong>s dos pob<strong>la</strong>ciones analizadas <strong>de</strong>l valle<br />
<strong>de</strong>l alfambra se mezc<strong>la</strong>n entre sí en el análisis nJ (Figura 7.3), lo que <strong>de</strong>no-<br />
genétIca <strong>de</strong> Pob<strong>la</strong>cIones 177