Genética molecular - IES San Isidro
Genética molecular - IES San Isidro
Genética molecular - IES San Isidro
You also want an ePaper? Increase the reach of your titles
YUMPU automatically turns print PDFs into web optimized ePapers that Google loves.
TEMA 15. GENÉTICA MOLECULAR<br />
1.- El ADN como depositario de la información genética: Experimentos de Griffith (1928)<br />
sobre transformación bacteriana.<br />
En 1928 F. Griffith, trabajó con dos cepas de neumococos productoras de la neumonía humana:<br />
Bacterias no capsuladas formando colonias rugosas R (no virulenta: no mueren los ratones).<br />
Los que presentan un aspecto de superficie áspera (rugosa) R (Rough), eran inofensivos<br />
cuando se inyectaban en ratones. Esto se debe a que la cápsula previene que los<br />
neumococos sean atrapadas por fagocitos y destruidos por células neutrófilas y macrófagos<br />
del cuerpo.<br />
Bacterias capsuladas formando colonias lisas S (virulenta: mueren los ratones). Una<br />
inyección de neumococos del tipo S (Smooth) liso, mata a un ratón en 24 horas. Después<br />
de un cultivo prolongado en un medio artificial, algunas células pierden la capacidad de<br />
formar la cápsula y la superficie de las colonias muestran un aspecto rugoso y áspero R<br />
(Rough).Con la pérdida de esta cápsula, la bacteria también pierde su virulencia.<br />
Griffith observó que si inyectaba a los ratones con neumococos de tipo R (no virulentos) a las 24 horas<br />
seguían vivos mientras que si los inyectaba con neumococos S (virulentos) a las 24 horas los ratones<br />
morían. Entonces decidió calentar los neumococos S (virulentos) para destruirlos y posteriormente<br />
inyectarlos a los ratones, encontrando que los ratones seguían vivos después de 24 horas. Por consiguiente<br />
el calor, destruía el poder infectivo de los neumococos S. Por último, inyectó a los ratones una mezcla de<br />
neumococos R vivos (no virulentos) y de S (virulentos) previamente muertos por calor, encontrando que los<br />
ratones morían a las 24 horas y extrayendo de su sangre neumococos S vivos.<br />
Conclusiones de Griffith: puesto que los neumococos R (no virulentos) nunca mutan a S (virulentos), en el<br />
último experimento se demuestra la existencia de una sustancia presente en los extractos de neumococos S<br />
muertos por calor que es capaz de transformar a los neumocos R vivos en S vivos. Dicha sustancia fue<br />
denominada por Griffith el Principio Transformante y el proceso Transformación bacteriana.<br />
http://www.dnai.org/timeline/index.html En 1920-49 pinchar en Oswald Avery para observar<br />
los experimentos<br />
Candelas Manzano Martín y Mª José Martínez Rodrigo 1
TEMA 15. GENÉTICA MOLECULAR<br />
Algo de las células muertas S había producido un<br />
cambio permanente en las células de R. El proceso fue<br />
llamado Transformación Bacteriana (intercambio<br />
genético producido cuando una bacteria es capaz de<br />
captar fragmentos de ADN de otra bacteria que se<br />
encuentra disperso en el medio en el que vive)<br />
Por el año 1944 Oswald Avery, MacLeod, y McCarty , sus<br />
colegas en el instituto Rockefeller de New York City<br />
repitieron los experimentos de Griffith y demostraron que<br />
el "Principio transformante" que convertía a las bacterias<br />
rugosas en lisas era el ADN, descubrimiento que marcó un<br />
hito importante en la historia de la <strong>Genética</strong>.<br />
2.- Concepto <strong>molecular</strong> de gen. Teorías de "un gen-una cadena polipeptídica" y de "un gen-un<br />
enzima" Beadle y Tatum (1948). A principios de la década de 1940, los estadounidenses, George<br />
W. Beadle y Edward L. Tatum estudiaron las consecuencias de las mutaciones. Comprobaron que<br />
la alteración de un gen suponía una variación fenotípica que consistía en el fallo en el<br />
funcionamiento de un enzima. Propusieron entonces la hipótesis UN GEN = UN ENZIMA. Se<br />
demostró la relación entre la secuencia de nucleótidos y la secuencia de aminoácidos, se aceptó<br />
que el gen podía dividirse en partes más pequeñas. Actualmente, reconocemos que la mínima<br />
unidad de mutación y recombinación es un nucleótido, un par de bases en la doble cadena de<br />
ADN. Por otra parte, muchos enzimas están compuestos por varias cadena polipeptídicas, cada una<br />
determinada por un fragmento de ADN. Además, muchas proteínas no son enzimas y se modificó<br />
la hipótesis para UN CISTRÓN -------- UN POLIPÉPTIDO en la que un cistrón es el equivalente<br />
a un gen.<br />
http://www.dnaftb.org/dnaftb/16/concept/index.html<br />
En 1953, el bioquímico estadounidense James D. Watson y el británico Francis H. C. Crick aunaron sus<br />
conocimientos químicos, utilizaron la información de Rosalind Franklin y Maurice Wilkins obtenida<br />
mediante difracción de rayos X, así como los trabajos de Chargaff sobre composición química del DNA y<br />
elaboraron una hipótesis sobre la estructura del DNA: la DOBLE HÉLICE.<br />
3.- Características de los genes en organismos procariotas y eucariotas.<br />
1. La cantidad de ADN por célula es la misma para cada célula diploide de cada especie, pero las<br />
variaciones entre las diferentes especies es enorme. Los seres humanos tienen 25 veces más pares<br />
de bases que Drosophila, pero no existe una correlación estrecha entre la complejidad del<br />
organismo y la cantidad de ADN. La mayor cantidad de ADN observada hasta el momento con una<br />
cantidad de 40 veces más de ADN que la especie humana es un pez pulmonado.<br />
2. La unidad funcional del ADN es el GEN (cistrón): segmento de ADN que codifica una cadena<br />
polipeptídica o una proteína.<br />
3. El ADN de las células está compuesto por:<br />
Genes Estructurales: Codifican y se transcriben a cadena polipeptídicas, ARN R y ARN T<br />
Secuencias reguladoras: Son señales de inicio o final del gen y codifican proteínas que controlan<br />
la actividad de los genes estructurales.<br />
http://www.wiley.com/legacy/college/boyer/0470003790/animations/animations.htm Síntesis de<br />
proteínas<br />
Candelas Manzano Martín y Mª José Martínez Rodrigo 2
TEMA 15. GENÉTICA MOLECULAR<br />
GENES DE PROCARIOTAS GENES DE EUCARIOTAS<br />
Casi todo el ADN de procariotas es<br />
codificante: todo el ADN se expresa en forma<br />
de proteínas excepto las secuencias<br />
reguladoras<br />
Cada molécula del ADN bacteriano contiene<br />
una sola copia de cada gen<br />
Los genes de procariotas no tienen<br />
INTRONES<br />
Algunos genes contienen información para<br />
proteínas diferentes (Policistrónicos)<br />
En cada célula eucariota parece haber una gran cantidad de ADN cuyas<br />
funciones son desconocidas. Sólo alrededor del 5-10 % consiste en<br />
ADN codificador de proteínas y el resto del genoma consiste en ADN<br />
no codificador, secuencias repetitivas y secuencias reguladoras.<br />
Casi la mitad del ADN consiste en secuencias de nucleótidos que se<br />
repiten centenas y hasta millones de veces.<br />
Las secuencias de genes que codifican proteínas habitualmente no son<br />
continuas sino que están interrumpidas por secuencias no codificadoras<br />
(INTRONES). Las secuencias codificadoras que se expresan se llaman<br />
(EXONES).<br />
No hay genes Policistrónicos.<br />
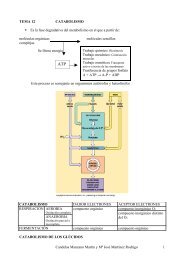
4.-LA TRANSMISIÓN DE LA INFORMACIÓN: EL DOGMA CENTRAL DE LA BIOLOGÍA<br />
5.- REPLICACIÓN DEL ADN:<br />
Finalidad del proceso e importancia biológica. Etapa del ciclo celular donde tiene lugar<br />
La replicación consiste en la síntesis de una copia de una molécula de ADN; es decir, a partir de una<br />
molécula de ADN se obtendrán dos moléculas idénticas. Este proceso está relacionado con la reproducción<br />
y ocurre en la fase S del ciclo celular. De esta forma, después de que ha tenido lugar la división celular,<br />
cada célula hija posee la misma información genética. Se produce en el núcleo en eucariotas y en el<br />
citoplasma en procariotas<br />
Características del mecanismo de replicación. ADN-polimerasa.<br />
Una vez descubierta la estructura del ADN, se plantearon tres hipótesis para tratar de explicar el mecanismo<br />
de la replicación:<br />
• Conservativa. Según esta hipótesis, las dos cadenas de la doble hélice hija se sintetizan de nuevo a<br />
partir del molde de la parental, que permanece.<br />
• Dispersiva. Según esta hipótesis, las dos cadenas tendrían fragmentos de la cadena antigua y<br />
fragmentos recién sintetizados.<br />
• Semiconservativa. La molécula de ADN se separa en sus dos hebras y cada una de ellas sirve de<br />
molde para la síntesis de su complementaria. De esta forma, las dobles hélices resultantes<br />
contienen una hebra antigua o parental y una de nueva síntesis. La hipótesis semiconservativa es el<br />
modelo de replicación confirmado, tanto en eucariontes como en procariontes. Propuesta por<br />
Watson y Crick.<br />
Candelas Manzano Martín y Mª José Martínez Rodrigo 3
TEMA 15. GENÉTICA MOLECULAR<br />
La replicación es:<br />
SEMICONSERVATIVA<br />
BIDIRECCIONAL: comienza por los puntos de origen (O) o locus Ori-C y avanza<br />
bidireccionalmente hacia los puntos de terminación, formando “Burbujas de replicación”<br />
ANTIPARALELA: los nucleótidos complementarios son seleccionados y fijados por la<br />
ADN polimerasa<br />
3’ 5’<br />
5´ 3’<br />
SEMIDISCONTINUA:Una hebra de crecimiento continuo (conductora) y otra de<br />
crcimiento discontinuo (retardada)<br />
Las principales moléculas implicadas en la replicación del ADN en los procariontes son:<br />
• ADN polimerasas. Son los enzimas que se encargan de catalizar la formación de enlaces<br />
fosfodiéster entre dos nucleótidos consecutivos. Los nucleótidos complementarios a los de la<br />
cadena que actúa como molde se añaden solamente por el extremo 3´. Se lee la cadena en<br />
dirección 3´ --- 5´ y se forma la nueva en la dirección 5´---3´<br />
Para llevar a cabo la catálisis necesitan un extremo 3'–OH libre necesario para que la ADN- polimerasa<br />
pueda añadir nucleótidos, por lo que requieren un cebador para iniciar la síntesis. Este cebador es un<br />
fragmento de ARN llamado primer o iniciador.<br />
En E. Coli (PROCARIOTAS) se conocen tres polimerasas:<br />
ADN polimerasa I, que presenta también actividad exonucleasa y se encarga de rellenar<br />
espacios polimerizando ADN<br />
ADN polimerasa II, que interviene en la reparación del ADN<br />
ADN polimerasa III, que sintetiza la mayor parte del ADN durante la replicación.<br />
• Helicasas. Separan las dos hebras de la molécula de ADN mediante la rotura de los puentes de<br />
hidrógeno que las mantienen unidas; de este modo, cada hebra puede actuar de molde para la<br />
síntesis de una nueva cadena.<br />
• Topoisomerasas. (Girasa) Son enzimas encargados de desenrollar la doble hélice de ADN a<br />
medida que se va replicando, para permitir la acción del ADN polimerasa.<br />
• Primasa. Es un ARN polimerasa que sintetiza pequeños fragmentos de ARN llamados cebadores o<br />
"primer".<br />
• Proteínas SSB. Son proteínas estabilizadoras de la cadena sencilla. Una vez que actúa la helicasa<br />
se unen a las cadenas sencillas, estabilizándolas mientras se produce la replicación.<br />
• ADN ligasas. Se encargan de unir fragmentos adyacentes de ADN, que se encuentran<br />
correctamente emparejados con la hebra complementaria.<br />
Candelas Manzano Martín y Mª José Martínez Rodrigo 4
TEMA 15. GENÉTICA MOLECULAR<br />
• ATP y GTP<br />
MECANISMO DE REPLICACIÓN: Inicio de la replicación. Formación de las nuevas<br />
hebras de ADN. Corrección de errores<br />
http://www.wiley.com/legacy/college/boyer/0470003790/animations/animations.htm DNA<br />
replication<br />
1. Inicio: Desenrollamiento y apertura de la doble hélice.<br />
La molécula de ADN se desenrolla por acción de los enzimas Girasas. Las helicasas rompen los puentes<br />
de hidrógeno que mantienen unidas las dos cadenas proporcionando la energía el ATP. Estas se separan y<br />
se forma una horquilla de replicación.<br />
Las hebras se mantienen separadas al unirse las Proteínas SSB que las estabilizan.<br />
2. Síntesis de las nuevas hebras.<br />
Simultáneamente a la separación de las dos hebras, se van sintetizando las nuevas hebras complementarias,<br />
por acción de las ADN polimerasas.<br />
El proceso es el siguiente:<br />
• El ARN polimerasa, denominada primasa, sintetiza una pequeña molécula de ARN que actúa de<br />
cebador, ya que el ADN polimerasa III es capaz de alargar la cadena, pero no de iniciar la síntesis.<br />
El cebador aporta un extremo 3´ OH necesario para que la ADN polimerasa III pueda añadir<br />
nucleótidos.<br />
• El ADN polimerasa III, utilizando como cebador ("primer") el fragmento de ARN, va alargando<br />
la cadena. Este enzima solo es capaz de unir nucleótidos en sentido 5’--- 3'. Como las dos cadenas<br />
que forman el ADN son antiparalelas, las dos hebras se sintetizan de manera diferente:<br />
Hebra continua: (a partir del origen de replicación de la cadena molde 3´---5´) se lee<br />
la cadena en dirección 3´---5´ y la hebra hija que se sintetiza a partir de ella lo hace de<br />
forma continua de 5´--3´.<br />
Hebra retardada: (a partir del origen de replicación de la cadena molde 5´---3´ ) se<br />
replica de forma discontinua y de forma retardada mediante la síntesis de pequeños<br />
fragmentos (1 000 nucleótidos) que crecen en dirección 5'---3', llamados fragmentos de<br />
Okazaki.<br />
• A continuación el ADN polimerasa I elimina los fragmentos de ARN que han actuado de<br />
cebadores ( función exonucleasa) y rellena los huecos entre los fragmentos (función polimerasa)<br />
• Por último, el ADN-ligasa une los extremos de los fragmentos, dando lugar a la molécula completa<br />
utilizando la energía del ATP.<br />
3. Corrección de errores.<br />
En E. coli tanto la ADN polimerasa I como la III tienen capacidad para corregir errores en la incorporación<br />
de nucleótidos con bases incorrectamente apareadas. Esto es posible por la actividad exonucleasa 3´-5´de<br />
estos enzimas, que eliminan los nucleótidos mal colocados. La ADN polimerasa I también tienen actividad<br />
exonucleasa 5´-3´, lo que permite la corrección de otros tipos de errores y la eliminación del ARN cebador.<br />
Candelas Manzano Martín y Mª José Martínez Rodrigo 5
TEMA 15. GENÉTICA MOLECULAR<br />
REPLICACIÓN : PROCESO SEGÚN EL CUAL UNA MOLÉCULA DE ADN DE DOBLE<br />
HÉLICE DA LUGAR A OTRAS DOS MOLÉCULAS DE ADN CON LA MISMA SECUENCIA<br />
DE BASES<br />
Candelas Manzano Martín y Mª José Martínez Rodrigo 6
TEMA 15. GENÉTICA MOLECULAR<br />
SÍNTESIS DE LA HEBRA CONTINUA Y DE LA HEBRA DISCONTINUA<br />
http://web.educastur.princast.es/proyectos/biogeo_ov/2BCH/PDFs/17Replicacion.pdf<br />
Diferencias entre el proceso replicativo en procariotas y en eucariotas.<br />
Las principales diferencias entre la replicación en procariontes y eucariontes se resumen en la siguiente<br />
tabla:<br />
Candelas Manzano Martín y Mª José Martínez Rodrigo 7
TEMA 15. GENÉTICA MOLECULAR<br />
PROCARIONTES EUCARIONTES<br />
Existen tres ADN polimerasas I, II y III.<br />
La replicación la realiza fundasmentalmente la<br />
polimerasa III.<br />
Tienen cuatro ADN polimerasas a, d , b y e, además<br />
de la polimerasa g, que replica el ADN mitocondrial.<br />
La polimerasa a replica la hebra retardada, y la d, la<br />
hebra lider.<br />
Presentan cientos de puntos de origen de la<br />
Presentan un único punto de inicio de la replicación<br />
(oriC).<br />
replicación.<br />
No tienen actividad telomerasa, ya que su ADN es Debido a que las cadenas de ADN son lineales,<br />
circular y no presentan problemas en la terminación presentan actividad telomerasa (enzima del extremo<br />
de la síntesis.<br />
de los cromosomas) para poder completar el extremo<br />
de la hebra una vez quitado el cebador.<br />
Fragmentos de Okazaki mayor (1000 a 2000 Fragmentos Okazaki menores (100 a 200<br />
nucleótidos)<br />
nucleótidos)<br />
Velocidad replicación mayor Velocidad replicación menor (hasta 50 veces)<br />
http://www.uc3m.es/uc3m/serv/GA/SEL/examenes.html preguntas selectividad<br />
Candelas Manzano Martín y Mª José Martínez Rodrigo 8
TEMA 15. GENÉTICA MOLECULAR<br />
http://www.selectividad.tv/ http://www.selectividad.profesores.net/ Selectividad<br />
4) El dogma central de la biología <strong>molecular</strong> se puede representar esquemáticamente de la siguiente manera:<br />
ADN ------- ARN------- proteína.<br />
a) Indique las diferencias que existen entre la composición y estructura del ADN y del ARN (1 punto).<br />
b) Indique el nombre de los procesos ADN ----- ARN y ARN ----- proteína e indique en qué parte de la célula<br />
eucariótica se producen (0,5 puntos).<br />
c) Mencione tres tipos distintos de ARN y cite el proceso que permite el paso de ARN a ADN (0,5 puntos)<br />
En el proceso de duplicación del ADN en bacterias (Escherichia coli):<br />
a) Explique el significado de los siguientes términos: replicación semiconservativa y<br />
replicación bidireccional. (0,5 puntos)<br />
b) Explique brevemente el mecanismo de la síntesis de ADN en la cadena retardada.<br />
(1,5 puntos)<br />
La Replicación del ADN es un proceso importante para las células<br />
¿Cuál es la finalidad de la replicación ¿En qué fase del ciclo celular se produce?¿por qué es tan importante que la<br />
replicación se produzca de forma fiel?<br />
Candelas Manzano Martín y Mª José Martínez Rodrigo 9