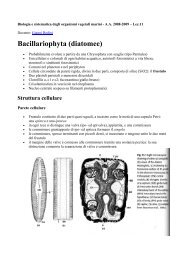
marker genetici per le alghe
marker genetici per le alghe
marker genetici per le alghe
You also want an ePaper? Increase the reach of your titles
YUMPU automatically turns print PDFs into web optimized ePapers that Google loves.
MARCATORI usati <strong>per</strong><br />
la<br />
SISTEMATICA del<strong>le</strong><br />
ALGHE<br />
Bruno Maria, Cimmino Cristina, Ferrario Jasmine<br />
♦ Chlorophyta o <strong>alghe</strong> verdi<br />
(clorofilla a e b)<br />
♦ Chromophyta o <strong>alghe</strong> brune<br />
(clorofilla a e c)<br />
♦ Rhodophyta o <strong>alghe</strong> rosse<br />
(clorofilla a e ficobiline)<br />
taxa<br />
♦ cryptophyta<br />
♦ haptophyta<br />
♦ dinoflagellati<br />
♦ heterokontae<br />
( Abdelghani, 2001)<br />
Classificazione sulla base di:<br />
-pigmenti<br />
- ultrastruttura dei plastidi<br />
- apparato flagellare<br />
- analisi mo<strong>le</strong>colare
70S<br />
80S<br />
30S<br />
50 S<br />
40S<br />
60S<br />
16S RNA<br />
21 proteine<br />
5S RNA<br />
23S RNA<br />
34 proteine<br />
18S RNA<br />
~ 33 proteine<br />
5S RNA<br />
28S RNA<br />
5.8S RNA<br />
~ 49 proteine<br />
RIBOSOMI<br />
(citoplasmatici)<br />
BATTERI<br />
EUCARIOTI<br />
♦ Secondo il modello oggi ritenuto<br />
più plausibi<strong>le</strong>, un eucariote<br />
eterotrofo fagocitò un<br />
cianobatterio senza digerirlo. Le<br />
due cellu<strong>le</strong> si integrarono<br />
gradualmente e il cianobatterio<br />
<strong>per</strong>se molti dei suoi geni e ne<br />
trasferì altri nel nuc<strong>le</strong>o della cellula<br />
ospite (Gray, 1996).<br />
L'eredità include i ribosomi 70S<br />
degli organelli.<br />
♦ Gli RNA del<strong>le</strong> subunità dei<br />
ribosomi del cloroplasto mostrano<br />
maggiori somiglianze con l'RNA<br />
dei cianobatteri rispetto a quello<br />
degli eucarioti.<br />
♦ Nel nuc<strong>le</strong>o i geni <strong>per</strong> gli RNA 18S<br />
e 28S sono separati dal gene <strong>per</strong><br />
l'RNA 5S, ma nei procarioti e nella<br />
maggior parte dei plastidi, i tre geni<br />
si trovano in un unico cluster.
mt DNA<br />
♦ Composizione in GC diversa da quella del DNA nuc<strong>le</strong>are.<br />
♦ Contenuto genico, in termini di numero e funzione, è molto simi<strong>le</strong> nei genomi<br />
mitocondriali di specie anche molto diverse.<br />
♦ La dimensione del genoma mitocondria<strong>le</strong> varia molto da organismo a organismo.<br />
♦ Contiene una grande quantità di DNA che non codifica <strong>per</strong> alcun prodotto genico.<br />
♦ Ha origine materna, ciò fa si che <strong>le</strong> linee di discendenza femmini<strong>le</strong> siano praticamente<br />
uniche. Quindi Consente di studiare <strong>le</strong> relazioni del<strong>le</strong> linee di discendenza femmini<strong>le</strong><br />
tra individui.<br />
♦ Viene usato <strong>per</strong> studi riguardanti la biodiversità, <strong>per</strong> stabilire il grado di variabilità<br />
genetica presente nel<strong>le</strong> popolazioni naturali.<br />
♦ Nel<strong>le</strong> piante si evolve ad un tasso più <strong>le</strong>nto del DNA nuc<strong>le</strong>are.<br />
♦ Viene usato <strong>per</strong> analizzare <strong>le</strong> linee evolutive strettamente imparentate.<br />
(J. Russel,2003)<br />
c DNA<br />
♦ Composizione in GC diversa da quella del DNA nuc<strong>le</strong>are e dell’mt DNA.<br />
♦ Contiene i geni <strong>per</strong> tutti gli rRNA dei cloroplasti (16S; 23S; 4.5S e 5S).<br />
♦ I cloroplasti del<strong>le</strong> piante su<strong>per</strong>iori sono derivati da batteri fotosintetici.<br />
♦ La sintesi proteica nel cloroplasto somiglia al processo di sintesi proteica dei<br />
procarioti (E. coli e cianobatteri).<br />
♦ Molti dei geni del batterio originario sono ora presenti nel genoma nuc<strong>le</strong>are,<br />
dove sono stati trasferiti e conservati stabilmente.<br />
♦ Il genoma dei cloroplasti è stato conservato stabilmente <strong>per</strong> almeno parecchie<br />
centinaia di milioni di anni.<br />
(J. Russel,2003)
Sequenze utilizzate <strong>per</strong> l'analisi filogenetica:<br />
■ 18S rRNA<br />
gene rbcL<br />
SSU rRNA<br />
LSU rRNA<br />
■ SSU rRNA<br />
gene rbcL<br />
(Andersen, 2004)<br />
usati <strong>per</strong> analizzare <strong>le</strong><br />
differenze all'interno del<br />
taxon heterokontae<br />
conclusione = heterokontae gruppo monofi<strong>le</strong>tico<br />
utilizzati <strong>per</strong> analizzare <strong>le</strong><br />
differenze filogenetiche tra i<br />
seguenti gruppi:<br />
- heterokontae<br />
- haptophyta<br />
- alveolate<br />
- cryptophyta<br />
- rodophyta<br />
- 5.8S RNA<br />
- 16S RNA<br />
- 18S RNA<br />
- 23S RNA<br />
- 28S RNA<br />
- gene rbcL<br />
- SSU rRNA<br />
-LSU rRNA<br />
- mt DNA
Da analisi mo<strong>le</strong>colari effettuate su <strong>alghe</strong><br />
eucarotiche e cianobatteri si è giunti alla<br />
conclusione che la sequenza 23S rDNA è un<br />
primer universa<strong>le</strong>. Può essere quindi utilizzato <strong>per</strong><br />
ri<strong>le</strong>vare <strong>le</strong> differenze filogenetiche tra i due gruppi<br />
sopra citati.<br />
( Sherwood, 2007)<br />
Per studi intraspecifici sul<strong>le</strong> <strong>alghe</strong> rosse sono<br />
stati utilizzati marcatori nuc<strong>le</strong>ari e plastidiali del<br />
DNA.<br />
Inoltre l'utilizzo del DNA mitocondria<strong>le</strong> (cox2 e<br />
cox3 codificanti la citocromo ossidasi) è<br />
risultato uti<strong>le</strong> <strong>per</strong> studi filo<strong>genetici</strong> e di<br />
popolazione in quanto ha genotipo aploide e vi è<br />
poca ricombinazione<br />
(Zuccarello, 1999)<br />
□ 18S rRNA ♦ utilizzato <strong>per</strong> analizzare <strong>le</strong> differenze<br />
filogenetiche di varie specie appartenenti al<br />
taxon del<strong>le</strong> rhodophyta.<br />
♦ utilizzato <strong>per</strong> l'identificazione di un nuovo<br />
epifita di rodophyta (disco)<br />
(Huisman, 2003)<br />
□ SSU rRNA<br />
(del nuc<strong>le</strong>o)<br />
(Ragan,1994)<br />
utilizzato <strong>per</strong> analizzare <strong>le</strong> differenze<br />
filogenetice di 39 specie appartenenti a 15<br />
diversi ordini di rodophyta
□ LSU rRNA<br />
SSU rRNA<br />
NM LSU rRNA<br />
utilizzati <strong>per</strong> analizzare <strong>le</strong> relazioni filogenetiche tra:<br />
- chlorarachniophyta<br />
- cryptomonads<br />
- haptophyta<br />
Conclusione = formano un'unica linea indipendente<br />
□ Sulla base di studi su LSU rRNA, <strong>le</strong> <strong>alghe</strong> appartenenti al taxon<br />
heterokontae, risultano formare una linea monofi<strong>le</strong>tica<br />
(Abdelghani,2001)<br />
■ differenze :<br />
Molte profonde divergenze nel regno eucariotico sono state<br />
risolte con la corstruzione fina<strong>le</strong> di alberi filo<strong>genetici</strong> basati<br />
sul<strong>le</strong> sequenze LSU e SSU rRNA<br />
(Abdelghani,2001)<br />
- LSU rRNA forniscono risultati<br />
meno solidi rispetto a SSU rRNA<br />
- LSU rRNA forniscono maggiori<br />
informazioni
CONCLUSIONI<br />
Le varie divergenze filogenetiche possono essere<br />
descritte con l'uso di <strong>marker</strong> mo<strong>le</strong>colari<br />
Mt DNA e NM LSU rRNA sono utili <strong>per</strong> studiare<br />
linee evolutive imparentate tra loro.<br />
Il DNA dei diversi rRNA e' uti<strong>le</strong> <strong>per</strong> studiare linee<br />
evolutive anche molto distanti tra loro.<br />
Lo studio del<strong>le</strong> relazioni filogenetiche e' tutt'ora in<br />
atto<br />
BIBLIOGRAFIA<br />
“Phylogenethic relationships among algae based on comp<strong>le</strong>te large-subunit rRNA<br />
sequences”Abdelghani Ben Ali, Raymond De Baere, Gert Van der Auwera, Ru<strong>per</strong>t De<br />
Wachter and Yves Van de Peer. International Journal of systematic and Evolutionary<br />
Microbiology (2001), 51,737-749<br />
“Biology and systematics of Heterokont and Haptophyte algae” Robert A. Andersen.<br />
American Journal of Botany 91(10):1508-1522. 2004<br />
“Universal primers amplify a 23S rDNA plastid <strong>marker</strong> in eukaryotic algae and<br />
cyanobacteria” Alison R. Sherwood, Gernot G. Presting. Journal of Phycology 43,<br />
605-608 (2007)<br />
“A mitochondrial <strong>marker</strong> for red algae intraspecific relationships” G.C. Zuccarello, G.<br />
Burger, J.A. West and R.J. King. Mo<strong>le</strong>cular Ecology (1999) 8,1443-1447<br />
“Morphology, reproduction and the 18S rRNA gene sequence of Pihiella liagoraciphila<br />
gen.et sp. nov. (rhodophyta), the so-cal<strong>le</strong>d 'monosporangial discs' associated with<br />
members of the liagoraceae(rhodophyta), and proposal of the plhiella<strong>le</strong>s ord. Nov.”<br />
John M. Huisman, Alison R. Sherwood and Isabella A. Abbot. Journal of Phycology<br />
39, 978-987 (2003)<br />
“A mo<strong>le</strong>cular phylogeny of the marine red algae(rhodophyta) based on the nuc<strong>le</strong>ar<br />
small-subunit rRNA gene” Mark A. Ragan, Carolyn J. Bird, El<strong>le</strong>n L. Rice, Robin R.<br />
Gutell, Col<strong>le</strong>en A. Murphy and Rama K. Singh. Plant Biology, Vol. 91, pp. 7276-7280,<br />
July 1994