lezione 7
lezione 7
lezione 7
Create successful ePaper yourself
Turn your PDF publications into a flip-book with our unique Google optimized e-Paper software.
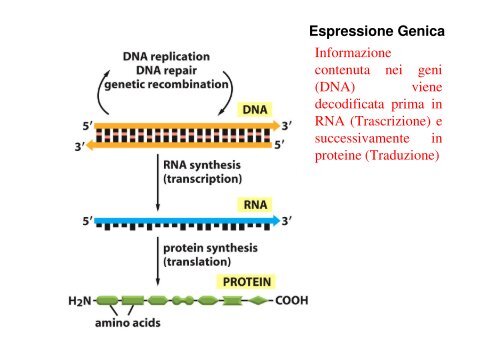
Espressione Genica<br />
Informazione<br />
contenuta nei geni<br />
(DNA) viene<br />
decodificata prima in<br />
RNA (Trascrizione) e<br />
successivamente in<br />
proteine (Traduzione)
Nucleotidi e Ribonucleotidi
L’RNA è costituituito da un singolo filamento<br />
ripiegato in strutture specifiche
Trascrizione….i protagonisti:<br />
•Stampo di DNA<br />
•RNA polimerasi<br />
•Ribonucleotidi trifosfato (ATP, GTP, UTP, CTP)
RNA polimerasi Batterica<br />
RNA polimerasi DNA-dipendente MA NON NECESSITA DI<br />
INNESCO per avviare la trascrizione<br />
nucleoside trifosfato + RNAn ⇄ difosfato + RNAn+1<br />
Procede in direzione 5’-3’
Polimerizzazione dei<br />
ribonucleotidi ad opera<br />
dell’RNA polimerasi<br />
durante la trascrizione<br />
L’RNA polimerasi catalizza<br />
un legame fosfodiestere tra il<br />
gruppo 3’OH libero dell’RNA<br />
nascente ed il fosfato in alfa<br />
del nuovo ribonucleotide
Stadi della Trascrizione<br />
1) Fase di inizio<br />
2) Fase di allungamento<br />
3) Fase di Terminazione
RNA polimerasi batterica<br />
Apoenzima dotato di attività catalitica:<br />
2 subunità α, β e β’<br />
Oloenzima: Apoenzima + subunità σ<br />
La subunità σ “guida”<br />
l’RNA Polimerasi<br />
fino alla sequenza<br />
genica da trascrivere<br />
che si riconosce dal<br />
suo promotore
Il promotore genico…<br />
-è una sequenza di DNA altamente conservata<br />
-si trova a monte del gene da trascrivere<br />
-è riconosciuto dalla RNA polimerasi e dagli altri fattori di<br />
trascrizione<br />
- È coinvolto nella regolazione della trascrizione genica
Sequenze Consenso per i prinicipali Promotori Batterici
Ciclo di trascrizione dell’RNA polimerasi batterica<br />
1) L’oloenzima si lega<br />
al promotore<br />
2) La polimerasi<br />
denatura il DNA al<br />
sito di inizio della<br />
trascrizione.<br />
3) La polimerasi inizia a<br />
trascrivere (Trascrizione<br />
Abortiva)<br />
4-5) Si stacca σ e la<br />
polimerasi diventa più<br />
processiva (Fase di<br />
Allungamento)<br />
6-7) In presenza di segnali<br />
(Terminatori) avviene il<br />
distacco della polimerasi dal<br />
DNA ed il rilascio di RNA
La direzione dell’RNA polimerasi è determinata<br />
dall’orientamento del promotore<br />
L’ RNA polimerasi procede sempre in direzione 5’-3’
RNA polimerasi Eucariotiche<br />
Esistono tre tipi di RNA polimerasi eucariotiche
L’RNA polimerasi II è molto simile all’RNA<br />
polimerasi batterica ma ci sono delle differenze<br />
1) Richede l’intervento dei<br />
fattori trascrizionali basali<br />
2) Tiene conto del<br />
compattamento del DNA<br />
nei nucleosomi<br />
I fattori trascrizionali basali, aiutano l’RNA<br />
I fattori trascrizionali basali, aiutano l’RNA<br />
polimerasi II a posizionarsi correttamente sul<br />
promotore.<br />
Svolgono una funzione analoga alla subunità<br />
σ dell’RNA polimerasi batterica
Fasi di inizio della trascrizione eucariotica<br />
I promotori eucariotici<br />
contengono una sequenza<br />
consensus chiamata TATA<br />
box<br />
La TATA box è<br />
riconosciuta e legata da<br />
TFIID<br />
TFIID è costituita da varie<br />
subunità: TBP e TAFs<br />
TBP è responsabile del<br />
riconoscimento della<br />
TATA box<br />
TFIIB si associa dopo il legame di<br />
TFIID
TFIIF stabilizza<br />
l’interazione dell’RNA<br />
polimerasi II con TFIID e<br />
TFIIB e recluta TFIIE<br />
TFIIE recluta TFIIH
TFIIH è dotata di attività<br />
elicasica e di fosforilazione del<br />
dominio CTD (C-terminal<br />
domain) dell’RNA polimerasi II
Ruoli dei fattori trascrizionali basali
Modifiche post-traduzionali delle code delle proteine<br />
istoniche possono influenzare l’espressione genica<br />
Acetilazione<br />
Istoni H3 ed H4<br />
(Attivazione<br />
genica)<br />
Metilazione Istone<br />
H3 (Inattivazione<br />
genica)<br />
De-acetilazione<br />
Istone H3 ed<br />
H4<br />
(Inattivazione<br />
genica)
Acetilazione, metilazione e fosforilazione degli istoni
Fattori Trascrizionali<br />
Posseggono delle particolari regioni (motivi) che legano il DNA<br />
Stabiliscono un gran numero di contatti con il DNA, coinvolgendo<br />
legami idrogeno, legami ionici ed interazioni idrofobiche<br />
Tali legami determinano un’interazione con il DNA molto forte ed<br />
altamente specifica<br />
I motivi sono generalmente costituiti da alfa eliche e foglietti beta
Motivo ELICA-GIRO-ELICA<br />
L’alfa-elica C-terminale (Rosso) si lega in modo sequenza<br />
specifica al livello del solco maggiore del DNA
Motivo a dita di Zinco
Motivo a cerniera di leucine (“Leucine Zipper”)
Motivo Elica-Ansa-Elica<br />
Tale motivo consiste di una breve alfa-elica connessa da un’ansa ad<br />
una seconda alfa-elica più lunga. La flessibilità dell’ana permette ad<br />
un’elica di ripiegarsi e di compattarsi contro l’altra.<br />
L’elica più lunga di solito è responsabile dell’interazione con il DNA
Dal Gene alla Proteina nei Procarioti ed Eucarioti
Differenze tra i trascritti procariotici e i trascritti eucariotici
Disposizione di Esoni ed Introni in differenti geni
Processo di Splicing
Sequenze consensus che dirigono il taglio e la<br />
poli-Adenilazione del 3’ del trascritto
Passaggi principali nella generazione<br />
dell’estremità 3’ di un mRNA eucariotico<br />
1) Legate al CTD della pol II ci sono<br />
CPSF (Fattore della specificità del taglio e della poli-<br />
Adenilazione) e CsTF (Fattore di stimolazione del taglio)<br />
2) CPSF riconosce il consenus AAUAAA; CstF richiama altri<br />
fattori di taglio che determinano il taglio del trascritto al sito<br />
CA<br />
3) L’enzima PoliA-polimerasi (RNA polimerasi stampoindipendente)<br />
catalizza l’aggiunta di una coda di A (circa 250<br />
nucleotidi) al gruppo 3’OH libero del dinucleotide CA al sito<br />
di taglio<br />
4) La coda di poli-A viene riconosciuta e legata dalle PolyAbinding<br />
protein che stabilizzo il trascritto ed impediscono che<br />
venga degradato da RNasi
I trascritti dopo aver subito le modifiche del:<br />
1) Aggiunta del Cap di 7-metil Guanosina al 5’<br />
2) Splicing<br />
3) Aggiunta di coda di poli-A<br />
Vengono esportati dal nucleo al citoplasma attraverso i pori nucleari
Dal gene alla proteina…..
Traduzione…i protagonisti<br />
•RNA messaggero<br />
•Ribosoma<br />
•tRNA “carichi” di amminoacido<br />
•Amminoacil transferasi<br />
…..sullo sfondo…il codice genetico
Codice genetico<br />
Il codice è letto in gruppi di tre nucleotidi (triplette)<br />
Ciascuna tripletta sull’mRNA è chiamato codone<br />
Il codice è ridondante: codoni diversi possono codificare per un<br />
singolo amminoacido
Per una data sequenza di RNA ci sono tre differenti quadri di lettura<br />
MA solo un quadro di lettura possibili in un mRNA codifica per<br />
una proteina!!!
Struttura del tRNA
Appaiamento codone-anticodone<br />
Fenomeno del vacillamento<br />
La terza base è oscillante
Come si forma amminoacil-tRNA?<br />
Avviene ad opera dell’Amminoacil-tRNA sintetasi mediante un processo a 2 fasi:<br />
1) Attivazione degli amminoacidi<br />
2) Formazione del legame esterico tra il gruppo carbossilico dell’amminoacido<br />
ed il 3’OH libero dell’adenina al livello del CCA all’estremità 3’ del tRNA
Il messaggio contenuto nell’RNA è decodificato dai ribosomi
Ribosoma contiene quattro siti fondamentali: per mRNA, per<br />
amminoacil-tRNA, per peptidil-tRNA e sito di uscita per i tRNA<br />
“scarichi”
1) L’amminoacil tRNA si<br />
lega al sito A del ribosoma<br />
2) Formazione del legame<br />
peptidico catalizzata dalla<br />
peptidil-transferasi
3) La subunità maggiore del<br />
ribosoma trasloca rispetto<br />
alla subunità minore. Ciò<br />
porta il peptidil-tRNA al<br />
livello del sito A e il tRNA<br />
“scarico” al livello del sito E<br />
nella subunità maggiore<br />
(posizione ibrida)
4)La subunità minore si sposta<br />
di una tripletta lungo l’mRNA.<br />
Ora il peptidil-tRNA è passato<br />
completamente nel sito P e il<br />
tRNA “scaricato” nel sito E
5) Un nuovo<br />
amminoaciltRNA<br />
entra nel<br />
sito A mentre il<br />
tRNA scarico<br />
esce dal sito E
Una tipica sequenza<br />
consensus di inizio della<br />
traduzione è ACCAUGG<br />
La subunità minore scorre<br />
lungo l’mRNA alla ricerca<br />
del codone AUG<br />
il tRNA iniziatore che<br />
porta legato una Metionina<br />
si lega al livello del sito P<br />
della subunità minore del<br />
ribosoma
Nei batteri il tRNA<br />
iniziatore porta una<br />
Metionina modificata<br />
(Formil-Metionina)
Fase di terminazione della traduzione<br />
Quando un codone di stop viene “letto dal<br />
ribosoma” si posizionano nel sito A del<br />
ribosoma i Fattori di rilascio della traduzione<br />
(RF)<br />
Codoni di stop:<br />
UAA, UAG, UGA