Spleissen - ZMBH
Spleissen - ZMBH
Spleissen - ZMBH
- Keine Tags gefunden...
Sie wollen auch ein ePaper? Erhöhen Sie die Reichweite Ihrer Titel.
YUMPU macht aus Druck-PDFs automatisch weboptimierte ePaper, die Google liebt.
<strong>Spleissen</strong> Dezember 2009Elmar Schiebel, <strong>ZMBH</strong>
Eines der Experimente von Roberts und Sharp:Analyse von DNA-mRNA Hybride mittels EM.
Spleißen...Die typische prä-mRNA enthält wenige kleine Exons vor einem Hintergrundriesiger intronischer Bereiche. Das korrekte Zusammenfügender Exons in der korrekten Reihenfolge erfordert eine aufwendigeMaschinerie (das „Spleißosom“, über 50 Proteine, 5 RNAs !!!).mittlere Gengrößemittlere Zahl an Exonenpro Genmittleres Exon27 000 Basenpaare9145 Basenpaare1 bis 178Die Spleißreaktion muss auf das Nukleotid genau durchgeführtwerden, da es sonst zu Verschiebungen im Leseraster kommen würde.Bei der Translation würde dann ein Protein mit völlig falscher Aminosäuresequenzentstehen. Daher sind die zu spleißenden Stellen auf derprä-mRNA sehr genau markiert.
Punktmutationen (Austausch eines einzigen Nukleotids gegen einanderes) an Spleißgrenzen sind sehr häufig die Ursache von Erbkrankheiten(15% aller genetisch bedingten Krankheiten).Ein Defekt der Spleißreaktion verursacht die KrankheitBeta-Thallasämiie. Ein normales beta-Globin Gen ist bei der Person Avorhanden. B, C sind zwei mutierte Gene, die Beta-Thallasämiieverursachen. Die Pfeile zeigen die Position der Spleißmutationen an. Diegestrichelten Linien verbinden die Segmente, die durch die Spleißreaktionverbunden werden.
Wozu mag das Spleißen gut sein?1) Evolutionäre Argumentation: Exons kodieren Protein-Domänen,welche im Baukastenprinzip zu neuen Proteinen rekombiniertwerden können.2) Das Codierungspotential des Genoms wird durch alternativesSpleißen stark erhöht -- ein Gen kann verschiedene Genproduktecodieren.
Pyruvate Kinase,ein Protein bestehend ausdrei Domänen.
Alternatives Spleißen...
Gewebeabhängiges alternatives Spleißen des Tropomyosin-Gens
Sequenzmerkmale an Intron-Exon-Grenzen (Säugetierzellen):Drei Stellen müssen für den Spleißvorgang markiert sein:1. 5‘-Spleißstelle2. Verzweigungsstelle3. 3‘-SpleißstelleKnippers, Abb 14.1Nur die Folge GU am Anfang und AG am Ende des Introns sind invariant,alle anderen Elemente des Konsensus können im Einzelfallabweichen. Das macht die genaue Vorhersage der Spleißstellen sehrschwierig (für einen Wissenschaftler! -- die Zelle weis bescheid...).
Basierend auf den Sequenzdaten von genomischer DNA, ist es häufig schwierigein Gen zu identifizieren. Dies ist jedoch äußerst wichtig, bedenkt man, dass in derZwischenzeit sehr viele Genome sequenziert sind.Wissenschaftler haben deshalb Computerprogramme entwickelt, die es erlaubenGenen vorherzusagen. Diese machen Vorhersagen für lange Offene Leserahmen(ORFs), Promotoren, Intron/Exon Spleißstellen, und Polyadenylierungs-Stellen.GENSCANDieses Computerprogramm erlaubt es Intron/Exon Spleißstellen, Promotoren undPolyadenylierungs Sequenzen vorherzusagen. Es verbindet zudem die verschiedenenExons, um ein langes translatiertes Protein zu erzeugen, das wiederum dazubenutzt wird, um eine Protein-Datanbanken zu durchsuchen (interne Kontrolle).http://genes.mit.edu/GENSCAN.html“Simply paste the genomic DNA sequence you wish to analyze into the box labeledDNA sequence and select Run GENSCAN.”
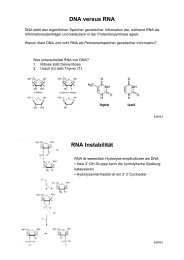
Die Chemie des Spleißens ist einfach:Während des Spleißens laufen zwei Transester-Reaktionen ab.1) Die Phosphodiester-Verknüpfung am 5‘-Ende des Introns wird gelöstund gleichzeitig bildet sich eine ungewöhnliche 2‘ -> 5‘ Phosphodiester-Verknüpfungam Verzweigungspunkt. Das in sich vernetzteZwischenprodukt wird auch als „Lariat*-Zwischenform“ bezeichnet.Das nun freie 3‘-Ende des vor dem Intron liegenden Exons greift diePhosphodiester-Verknüpfung am 3‘-Ende des Introns an.2) Eine normale 5‘ -> 3‘ Phosphodiester-Verknüpfung verbindet nun diebeiden Exons miteinander. Das Intron wird als Lariat freigesetzt.Die Chemie der Spleiß-Reaktion benötigt keine Energie-Zufuhr inForm von ATP- oder GTP-Hydrolyse. Trotzdem läuft das Spleißenunter hohem Energieverbrauch ab, was die ungeheure Präzisionermöglicht. *„Lariat“ ist ein ungewöhnliches Wort für „Lasso“.
Der Zwei-Schritt-Prozess:VerzweigungsstelleExon 15’Exon 2Exon 13’Exon 2Exon 15’Exon 15’IntronExon 23’Knippers, Abb 14.2
IntronDie Lariat-Zwischenform:Ungewöhnlich ist dieVerzweigung eines Polynukleotids,die auf einer Phosphodiester-Bindungan der 3‘- undder 2‘-OH-Gruppe eines Ribose-Bausteins beruht.Exon 2VerzweigungsstelleKnippers, Abb 14.3
Der Zwei-Schritt-Prozess(noch mal!):3’ Spleißstelle5’ Spleißstelle
TGGE: Temperature gradient gel electrophoresiserlaubt den Nachweis von Lariat Strukturen20ºC 60ºC
Die Chemie des Spleißens ist einfach,.... der Gesamtprozess hochkomplex:Das Kunststück besteht darin, die Exon-Intron-Grenzen richtig zuerkennen und die beiden Transester-Reaktionen an der richtigenStelle zu katalysieren. Dieses Kunstück vollbringt eine zelluläreMaschine, die das Spleißosom genannt wird.Die eigentliche Erkennung der relevanten Stellen wird von kleinenRNA-Molekülen durchgeführt. Die heißen snRNAs (small nuclearRNAs). Sie sind mit vielen Proteinen assoziiert; diese RNA-Protein-Komplexe nennt man snRNPs (small ribonucleoprotein, Snurps!).Der wichtige Punkt: Während der Spleißreaktion werden diese kleinenRNAs benutzt, um wiederholt die Sequenz an den bearbeitetenStellen „nachzuprüfen“ (d.h. nur wenn sie sich entsprechend mit derbearbeiteten Stelle paaren können, läuft die Reaktion weiter).Weiterhin bilden sie das katalytische Zentrum des Spleißosoms.
Die an der Spleißreaktion beteiligten snRNAs:Knippers, Tab 14.1
snRNPDie beiden essentiellen Komponenten von snRNPs sind Proteinmoleküle(RNP) und RNA. Der RNA Anteil wird auch als “small nuclear RNA” odersnRNA bezeichnet. snRNAs sind ungefähr 150 Nukleotide lang. snRNAhat eine direkte und strukturelle Rolle.Struktur derU2 snRNP
13 2457U58U26U2Das Spleißen von RNA wird durch eindynamisches Assemble von snRNPskatalysiert, die zusammen mitweiteren Proteinen das Spleißosombilden.Das Spleißosom erkennt dieSpleißsignale auf einem der prämRNAMolekül, führt die beidenEnden des Introns zusammmen undstellt die enzymatische Aktivitätfür die beiden Transester-Reaktionen.1. BBP (branch point binding protein)+ U2AF erkennen den Branch point.2. BBP/U2AF werden durch U2-snRNPersetzt.3. U1-snRNP bindet an 5’ Spleißstelle.4. U4/U6+U5 binden.5. Umlagerung: U4 löst sich. U6 verdängt U1von der 5’ Spleißstelle.6. Transester-Reationen laufen ab.7. Fusion von Exon 1-Exon 28. Freisetzung von Lariot, U2-U5-U6.
Umlagerungen im SpleißosomSchritt 5: U6 bindet an die5’ Spleißstelle und interagiert mit U2.Erste Transester-Reaktion.Schritt 6: Freigesetztes 5‘ Exon 1interagiert mit U5. Umlagerung vonExon 2. Zweite Transester-Reaktion.
Ein Großteil der beim Spleißen verbrauchten Energie (in Form vonATP-Hydrolyse) fließt in die Aktivität von RNA-Helikasen. Diesetrennen die RNA-Doppelstränge, die für die Erkennung der relevantenSequenzen auf dem Substrat und beim Aufbau des katalytischenZentrums gebildet werden.
Wir erinnern uns noch mal, dass das Spleißen schon während derTranskription an der entstehenden prä-mRNA beginnt!Pollard, EarnshawIm Rückblick wird klar, dass alternatives Spleißen auf einer zelltypspezifischenRegulation der Spleiß-Reaktion beruhen muss (die Detailssparen wir uns für später...).
Herstellung von cDNA:Das polyA-Ende von mRNAskann für die selektiveAnreicherung dieser (seltenen)RNA-Spezies genutzt werden.Ein oligo-dT-Primer kann dannbenutzt werden, um die RNAmit der reversen Transkriptasein cDNA umzuschreiben. Deranschliessende RNaseH-Verdauhinterlässt RNA-Fragmente, diewiederum als Primer für dieZweitstrangsynthese dienen undim Verlauf der DNA-Syntheseabgebaut werden. Jetzt liegteine doppelsträngige cDNA vor.
Eine Gen-Bibliothek ist eine (zufällige) Mischung von DNA-Fragmenten, die in einen Vektor kloniert sind. Der Vektorkann ein Plasmid oder ein Bakteriophagen-Derivat (wie z.B.M13) sein, oder Elemente von beiden enthalten ("Phagemid").Eine gute genomische Biobliothek sollte die gesamtegenetische Information eines Organismus umfassen.
Genomische DNA Bibliothek
Zwei verschiedene Typen von Genbibliothek im Vergleich:Genomische DNA BibliothekcDNA Gen-BibliothekWann sinvoll ?Vorteile ?Nachteile ?