Revista de la Sociedad de Endodoncia de Chile Nº 23 Abril 2011
Revista de la Sociedad de Endodoncia de Chile Nº 23 Abril 2011
Revista de la Sociedad de Endodoncia de Chile Nº 23 Abril 2011
Create successful ePaper yourself
Turn your PDF publications into a flip-book with our unique Google optimized e-Paper software.
Análisis estadístico<br />
Los análisis estadísticos <strong>de</strong> RT-PCR (qRT-PCR)<br />
fueron realizados usando el Software Stat view 5.0 (Abacus<br />
Concepts, Berkeley,CA). Comparaciones entre 2 grupos fueron<br />
realizadas con el test Stu<strong>de</strong>nt`s T y los valores p menores a<br />
0.05 fueron consi<strong>de</strong>rados estadísticamente significativos.<br />
RESULTADOS<br />
Establecimiento <strong>de</strong> un Mo<strong>de</strong>lo Experimental Animal <strong>de</strong><br />
Lesiones Periapicales<br />
El protocolo experimental se lo muestra en <strong>la</strong> Fig. 1.<br />
Para confirmar <strong>la</strong> presencia <strong>de</strong> <strong>la</strong> reacción inf<strong>la</strong>matoria<br />
en el área periapical se realizó un examen radiográfico<br />
(Fig. 2) e histológico (Fig. 3). Se observó que existió una<br />
reacción inf<strong>la</strong>matoria dramática, infiltración <strong>de</strong> leucocitos y<br />
engrosamiento <strong>de</strong>l ligamento periodontal 4 semanas <strong>de</strong>spués<br />
<strong>de</strong> <strong>la</strong> exposición pulpar en el área periapical. La reacción<br />
inf<strong>la</strong>matoria disminuyó a <strong>la</strong>s 4 semanas <strong>de</strong>spués <strong>de</strong> <strong>la</strong><br />
obturación radicu<strong>la</strong>r. De estos resultados se categorizaron<br />
los estadios inf<strong>la</strong>matorios alre<strong>de</strong>dor <strong>de</strong> <strong>la</strong> región periapical<br />
en 3 grupos (sano, inf<strong>la</strong>matorio, y <strong>de</strong> reparación). No hubo<br />
diferencias significativas en el peso <strong>de</strong> <strong>la</strong>s ratas entre los<br />
grupos durante el transcurso <strong>de</strong> los experimentos.<br />
Figura 3 (Modificada <strong>de</strong> Arias Martinez Z. et al ; Gene Profiles during Root Canal<br />
Treatment in Experimental Rat Periapical Lesions. JOE, 33 No 8 August 2007)<br />
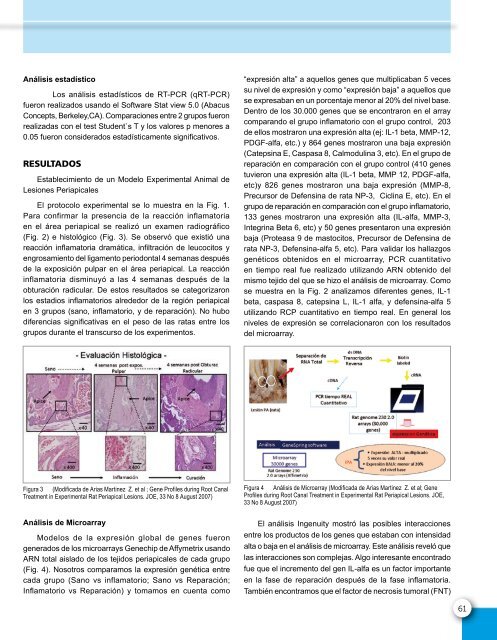
Análisis <strong>de</strong> Microarray<br />
Mo<strong>de</strong>los <strong>de</strong> <strong>la</strong> expresión global <strong>de</strong> genes fueron<br />
generados <strong>de</strong> los microarrays Genechip <strong>de</strong> Affymetrix usando<br />
ARN total ais<strong>la</strong>do <strong>de</strong> los tejidos periapicales <strong>de</strong> cada grupo<br />
(Fig. 4). Nosotros comparamos <strong>la</strong> expresión genética entre<br />
cada grupo (Sano vs inf<strong>la</strong>matorio; Sano vs Reparación;<br />
Inf<strong>la</strong>matorio vs Reparación) y tomamos en cuenta como<br />
“expresión alta” a aquellos genes que multiplicaban 5 veces<br />
su nivel <strong>de</strong> expresión y como “expresión baja” a aquellos que<br />
se expresaban en un porcentaje menor al 20% <strong>de</strong>l nivel base.<br />
Dentro <strong>de</strong> los 30.000 genes que se encontraron en el array<br />
comparando el grupo inf<strong>la</strong>matorio con el grupo control, 203<br />
<strong>de</strong> ellos mostraron una expresión alta (ej: IL-1 beta, MMP-12,<br />
PDGF-alfa, etc.) y 864 genes mostraron una baja expresión<br />
(Catepsina E, Caspasa 8, Calmodulina 3, etc). En el grupo <strong>de</strong><br />
reparación en comparación con el grupo control (410 genes<br />
tuvieron una expresión alta (IL-1 beta, MMP 12, PDGF-alfa,<br />
etc)y 826 genes mostraron una baja expresión (MMP-8,<br />
Precursor <strong>de</strong> Defensina <strong>de</strong> rata NP-3, Ciclina E, etc). En el<br />
grupo <strong>de</strong> reparación en comparación con el grupo inf<strong>la</strong>matorio,<br />
133 genes mostraron una expresión alta (IL-alfa, MMP-3,<br />
Integrina Beta 6, etc) y 50 genes presentaron una expresión<br />
baja (Proteasa 9 <strong>de</strong> mastocitos, Precursor <strong>de</strong> Defensina <strong>de</strong><br />
rata NP-3, Defensina-alfa 5, etc). Para validar los hal<strong>la</strong>zgos<br />
genéticos obtenidos en el microarray, PCR cuantitativo<br />
en tiempo real fue realizado utilizando ARN obtenido <strong>de</strong>l<br />
mismo tejido <strong>de</strong>l que se hizo el análisis <strong>de</strong> microarray. Como<br />
se muestra en <strong>la</strong> Fig. 2 analizamos diferentes genes, IL-1<br />
beta, caspasa 8, catepsina L, IL-1 alfa, y <strong>de</strong>fensina-alfa 5<br />
utilizando RCP cuantitativo en tiempo real. En general los<br />
niveles <strong>de</strong> expresión se corre<strong>la</strong>cionaron con los resultados<br />
<strong>de</strong>l microarray.<br />
Figura 4 Análisis <strong>de</strong> Microarray (Modificada <strong>de</strong> Arias Martinez Z. et al; Gene<br />
Profiles during Root Canal Treatment in Experimental Rat Periapical Lesions. JOE,<br />
33 No 8 August 2007)<br />
El análisis Ingenuity mostró <strong>la</strong>s posibles interacciones<br />
entre los productos <strong>de</strong> los genes que estaban con intensidad<br />
alta o baja en el análisis <strong>de</strong> microarray. Este análisis reveló que<br />
<strong>la</strong>s interacciones son complejas. Algo interesante encontrado<br />
fue que el incremento <strong>de</strong>l gen IL-alfa es un factor importante<br />
en <strong>la</strong> fase <strong>de</strong> reparación <strong>de</strong>spués <strong>de</strong> <strong>la</strong> fase inf<strong>la</strong>matoria.<br />
También encontramos que el factor <strong>de</strong> necrosis tumoral (FNT)<br />
61