TEMA 16. EL ADN Y LA INGENIERÃA GENÃTICA - BiologÃa El Valle
TEMA 16. EL ADN Y LA INGENIERÃA GENÃTICA - BiologÃa El Valle
TEMA 16. EL ADN Y LA INGENIERÃA GENÃTICA - BiologÃa El Valle
- No tags were found...
Create successful ePaper yourself
Turn your PDF publications into a flip-book with our unique Google optimized e-Paper software.
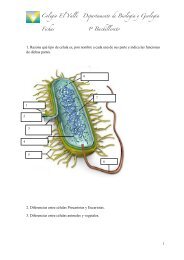
Colegio <strong>El</strong> <strong>Valle</strong> Departamento de Biología<strong>TEMA</strong> <strong>16.</strong> <strong>EL</strong> <strong>ADN</strong> Y <strong>LA</strong> INGENIERÍA GENÉTICA. DE <strong>LA</strong> BIOTECNOLOGÍA A <strong>LA</strong> INGENIERÍA GENÉTICA Ingeniería genética: conjunto de métodos y técnicas que permiten el acceso y la manipulación del <strong>ADN</strong>. Biotecnología: ciencia que utiliza a los organismos vivos o sus componentes en la obtención de productos útiles para las personas. 1. OBTENCIÓN DE FRAGMENTOS DE <strong>ADN</strong>. 1.1. Tecnología del <strong>ADN</strong> recombinante. Técnica que permite cortar la molécula de <strong>ADN</strong> de un organismo en múltiples trozos y aislar alguno de los fragmentos obtenidos para, mediante un vector, introducirlo en otro organismo. Se desarrolló a partir del descubrimiento de las enzimas de restricción o endonucleasas de restricción, que reconocen pequeñas bases secuenciadas, llamadas sitios de restricción, para luego cortar las dos cadenas de nucleótidos dentro de ese sitio, la mayoría de los cuales son simétricos (secuencias palindrómicas). Las enzimas de restricción se dividen dejando extremos lisos o extremos cohesivos o pegajosos, que pueden unirse por puentes de hidrógeno con una secuencia complementaria). Estas uniones se pueden hacer permanentes en presencia de la enzima ligasa, dando lugar al <strong>ADN</strong> recombinante o trasgén. 1.2. Formación de un <strong>ADN</strong> complementario por hibridación. • Se desnaturalizan dos hebras de <strong>ADN</strong> correspondientes a dos organismos distintos (pH > 13; Tª > 100°C). • Se renaturalizan dos de estas hebras manteniendolas a 65ºC. Las hebras pueden tener orígenes distintos, siempre que exista una secuencia complementaria. Cuanto más relacionados estén los <strong>ADN</strong>, ,ás recombinación se producirá. Esta técnica se utiliza para detectar cadenas complementarias, localizar genes relacionados en distintas poblaciones o diagnosticar enfermedades genéticas debidas a la alteración de la secuencia del <strong>ADN</strong>. 1.3. Síntesis de un <strong>ADN</strong>c utilizando como molde el ARNm. Los <strong>ADN</strong> de eucariotas presentan intrones y exones, y el ARN que se transcribe es sometido a un proceso de maduración para eliminar los intrones. Esto no puede ocurrir en procariotas. Por ello, a partir de un molde de ARN maduro se sintetiza un <strong>ADN</strong> de cadena sencilla, llamada <strong>ADN</strong> complementario (<strong>ADN</strong> c). Se consigue gracias a la transcriptasa inversa. Este <strong>ADN</strong> c servirá como molde para la síntesis de otro <strong>ADN</strong> c de doble cadena, sin intrones, y por lo tanto puede ser traducido por bacterias. • Se aisla un fragmento de ARN m maduro y se le añade un corto oligonucleótido de timina que hibrida con las adeninas de la cola poliA, y actúa como cebador de la transcriptasa inversa. <strong>El</strong> ARN m actúa como molde para la síntesis de <strong>ADN</strong> c de cadena sencilla. • Se sintetiza el <strong>ADN</strong> complementario, al que se añade una disolución de NaOH que hidroliza el ARNm. Se forma una horquilla en el <strong>ADN</strong> c, la cual sevirá como cebador para que la <strong>ADN</strong> polimerasa I sintetice la segunda hebra de <strong>ADN</strong>. • Se elimina la horquilla de replicación con la nucleasa S1, que sólo actúa en zonas de cadena sencilla. Finalmente se obtiene un <strong>ADN</strong>c de doble cadena. 1
Colegio <strong>El</strong> <strong>Valle</strong> Departamento de Biología2. Clonación del <strong>ADN</strong>. -‐ Permite la producción de múltiples copias de un gen específico o de un fragmento de <strong>ADN</strong> en el interior de un organismo que hace las veces de hospedador, el cual presenta las siguientes características: • Tener un crecimiento rápido. Se obtienen muchas generaciones en poco tiempo. • No ser patógeno, para que no produzca infecciones. • Favorecer la entrada del trasgén en su interior e incorporar el gen de interés en su genoma. • Ser un organismo ampliamente conocido y fácilmente manipulable, como la bacteria Escherichia coli. -‐ Para incorporar el gen de interés en el hospedador se emplean vectores de clonación, que son moléculas de <strong>ADN</strong> que transportan <strong>ADN</strong> extraño y se replican en el interior del hospedador. Todos ellos deben tener su propio origen de replicación y deben portar unos genes marcadores que sirvan para ser identíficados fácil y rápidamente. Los vectores más utilizados son: • Plásmidos. • Virus bacteriófagos. Infectan bacterias, y transportan genes bacterianos de una célula a otra, de forma que algún gen de la bacteria huésped puede incorporarse al genoma del bacteriófago, y al infectar a otra bacteria puede transferirle los genes de la primera. <strong>El</strong> más empleado es el bacteriófago lambda (λ), que puede llevar mayor cantidad de <strong>ADN</strong> que un plásmido. • Cósmicos. Son vectores híbridos entre el fago λ y un plásmido, de manera que se <strong>ADN</strong> puede replicarse en una célula como un plásmido, o puede empaquetarse como un fago. Tienen un origen de replicación plasmídico, genes marcadores que le dan resistencia a algunos antibióticos y sitios de restricción donde puede insertarse el <strong>ADN</strong> foráneo. La ventaja de los cósmidos frente a los plásmidos es que la porción de <strong>ADN</strong> foráneo que llevan puede ser mucho mayor. Clonación de un gen humano en un plásmido bacteriano. A. Se inserta uno o más genes marcadores en el plásmido que se utiliza como vector. Por ejemplo, el gen amp (que da resistencia a la bacteria al antibiótico ampicilina) y el gen lacZ (codifica la enzima β-‐galactosidasa, que hidroliza lactosa y un compuesto llamado X-‐gal, el cual sintetiza un compuesto de color azul). B. Se aísla el <strong>ADN</strong> humano y el plásmido y se cortan con las mismas enzimas de restricción. <strong>El</strong> <strong>ADN</strong> humano se rompe en muchos fragmentos y el plásmido sólo en el gen lacZ, donde se encuentra el sitio de restricción. C. Se mezclan los fragmentos de <strong>ADN</strong> humano con los plásmidos cortados. Como han sido cortados con la misma enzima de restricción, existen extremos cohesivos que hacen que se unan por complementariedad de bases, dando lugar a los plásmidos recombinantes y muchos otros no recombinantes. D. Se mezclan los plásmidos con bacterias que portan una mutación en el gen lacZ que les impide hidrolizar la lactosa. Habrá bacterias que incorporen plásmidos recombinantes que lleven el gen de interés. Otras incorporan plásmidos sin recombinar. Otras incorporan plásmidos recombinantes con un <strong>ADN</strong> sin interés. E. Se siembran las bacterias sobre una placa Petri con ampicilina y se incuban las bacterias hasta hacerse visibles. Se apreciarán tres tipos de bacterias: • Bacterias sin el plásmido recombinante. Mueren, ya que son sensibles a la ampicilina. • Bacterias transformadas con plásmidos no recombinante. Crecerán, ya que son resistentes a la ampicilina. Además sus colonias son azules debido a que presentan actividad galactosidasa, y transforman la sustancia X-‐Gal en un compuesto de color azul. 2
Colegio <strong>El</strong> <strong>Valle</strong> Departamento de Biología• Bacterias transformadas con plásmidos recombinantes. Crecen, puesto que son resistentes a la ampicilina. Sus colonias son de color blanco, ya que no tienen actividad β-‐galactosidasa, ya que el gen lacZ está desactivado. -Almacenamiento de los genes clonados. Genotecas: lugares donde se almacenan miles de clones que portan el gen de interés, mezclados con otros miles que no lo llevan. 3. Identificación de clones mediante sondas. Para localizar el <strong>ADN</strong> recombinante que lleva el gen de interés en una genoteca se utilizan sondas de <strong>ADN</strong>. Las sondas son pequeños oligonucleótidos de cadena sencilla, formados por unos 20 nucleótidos que se hibridarán con cualquier <strong>ADN</strong> recombinante que tenga una secuencia de bases complementaria. -Selección de clones con una sonda de ácidos nucleicos. 1. Se transfieren las bacterias con los plásmidos recombinantes a una placa Petri para que formen colonias visibles. 2. Se presiona la muestra con un filtro absorbente y las bacterias que forman los distintos clones se transfieren a éste, obteniendo una imagen especular de las colonias, que a continuación serán numeradas. 3. Se trata el filtro de nitrocelulosa con un detergente para romper las membranas celulares y que las células liberen su contenido, las moléculas de <strong>ADN</strong> se desnaturalicen y se unan fuertemente a la nitrocelulosa. 4. <strong>El</strong> filtro se incuba con una sonda de <strong>ADN</strong> preparada específicamente para que se hibride únicamente con los <strong>ADN</strong> recombinantes que hayan incorporado el gen de interés. <strong>El</strong> resto se eliminan mediante lavado. 5. Se coloca el filtro debajo de una película fotográfica que luego se revela. Quedará ennegrecida en los lugares donde se ha producido la hibridación. 6. Se compara la película revelada con la placa original, para así localizar las colonias portadoras del gen de interés. 4. Reacción en cadena de la polimerasa (PCR). Técnica in vitro que permite obtener de forma rápida un elevado número de copias de un fragmento pequeño de <strong>ADN</strong>. Materiales necesarios: • Muestra de <strong>ADN</strong>. • Secuencia de nucleótidos (cebador). • Fuente de calor. • <strong>ADN</strong> polimerasa resistente al calor. • Nucleótidos. <strong>El</strong> proceso se desarrolla por ciclos: 1. Desnaturalización por calor de la doble cadena de <strong>ADN</strong> para separarla en dos hebras. Cada hebra servirá de molde para formar una nueva cadena complementaria. 2. Hibridación. Se enfría la muestra para permitir que los cebadores se unan mediante puentes de hidrógeno a cada uno de los extremos de las dos hebras de <strong>ADN</strong>. 3. Extensión. Gracias a la <strong>ADN</strong> polimerasa se van agregando nucleótidos en el extremo 3´ del cebador y se forman las dos hebras nuevas. - Aplicaciones de la PCR • Fragmentos de <strong>ADN</strong> antiguos. Se ha amplificado el <strong>ADN</strong> de un mamut lanudo congelado que vivió hace 40.000 años, para compararlos con genes semejantes de organismos actuales y hecer estudios evolutivos. Otro ejemplo son las momias de antiguas civilizaciones, para estudiar, por ejemplo, enfermedades genéticas. • <strong>ADN</strong> obtenido en la escena de un crimen. Se recoge en pequeñas cantidades de sangre, tejidos, pelo o semen. Se aplica en medicina forense para identificar al autor de un delito. 3
Colegio <strong>El</strong> <strong>Valle</strong> Departamento de Biología• <strong>ADN</strong> de células embrionarias. Para diagnósticos prenatales de trastornos genéticos. • <strong>ADN</strong> de genes virales. Por ejemplo, el VIH. 5. Secuenciación del <strong>ADN</strong>. Técnica que permite conocer el orden de los nucleótidos que forman parte, no sólo de un gen concreto, sino también de los que forman el genoma completo de un organismo. -Método didesoxi de Sanger. Emplea didesoxirribonucleótidos, que son nucleótidos modificados que han perdido el grupo -‐OH situado en el carbono 3´. Esto implica que la síntesis de <strong>ADN</strong> se detiene en ese nucleótido concreto, ya que la <strong>ADN</strong> polimerasa no puede añadir otro debido a la ausencia del extremo 3´. Materiales necesarios: • Fragmento de <strong>ADN</strong> a secuenciar. • Cebador o primer, formado por pocos nucleótidos complementarios a los situados en el extremo 3´de la cadena de <strong>ADN</strong>. • <strong>ADN</strong>-‐polimerasa. • Los cuatro desoxirribonucleótidos • Los cuatro didesoxirribonucleótidos marcados con una molécula fluorescente, específica para cada uno de ellos. Proceso: 1. Aumentar la cantidad de <strong>ADN</strong> que queremos estudiar. 2. Desnaturalizar el <strong>ADN</strong> para separarla en dos cadenas simples que se incubanen tubos de ensayo con el resto de componentes. 3. Se mezclan todos los componentes de la reacción, formándose nuevas cadenas de <strong>ADN</strong>. La síntesis comienza en el extremo 3´ del cebador. Se obtienen varias cadenas de <strong>ADN</strong> de longitud variada que llevan en su extremo 5´ alguno de los nucleótidos marcados con la molécula fluorescente. 4. Se separan las cadenas marcadas al hacer pasar la muestra por un gel de poliacrilanina, de manera que las más cortas se mueven con mayor rapidez. Gracias a un detector de fluorescencia se registra el color de cada marca fluorescente. <strong>El</strong> color de la marca indica qué nucleótido ocupa el extremo de la cadena de <strong>ADN</strong>. 5. Se obtiene un espectograma con una impresora, que al leerlo desde abajo hacia arriba nos dará las bases complementarias de la cadena molde. Este método de secuenciación es útil para secuenciar fragmentos de <strong>ADN</strong> de hasta 800 pares de bases. Es un método muy rápido que permite secuenciar unas 450 bases por hora. Para secuenciar todo el genoma humano se necesitaron técnologías más rápidas. GENÓMICA Y PROTEÓMICA. • Genómica: conocimiento del genoma completo y las interacciones de sus genes. - Identificación de genes que codifican proteínas. Hay genes que no sé sabe que proteínas codifican, denominados genes dudosos o genes candidaos. Para identificarlos se usan programas de ordenador que rastrean señales de inicio y detención de transcripción y traducción, así como lugares de corte y empalme en el ARN. - Genes que interactúan entre sí. Para evaluar la expresión de un genoma se utilizan los ensayos de micromatrices de <strong>ADN</strong>, chips de <strong>ADN</strong> o microarray. Permite conocer mejor enfermedades y elaborar técnicas de diagnóstico y terapia. Ejemplo: comparar tumores y tejidos no cancerosos. 1. Se aísla el ARNm de una muestra de tejido de un organismo. 2. Se sintetiza <strong>ADN</strong>c por transcripción inversa con nucleótidos marcados con fluorescente. 3. Se mezcla de <strong>ADN</strong>c con fragmentos de <strong>ADN</strong> procedentes del organismo en una micromatriz para que se hibriden por complementariedad de bases. 4. Se elimina el <strong>ADN</strong> por lavado. Cada punto con fluorescencia representa un gen expresado de la muestra de tejido. - Comparación entre genomas de diferentes especies. Permite establecer relaciones evolutivas 4
Colegio <strong>El</strong> <strong>Valle</strong> Departamento de Biología• Proteómica: disciplina que realiza un estudio sistemático del conjunto completo de proteínas codificadas por el genoma de un organismo. INGENIERÍA GENÉTICA Y MEDICINA A. Obtención de productos farmacéuticos. La insulina, el interferón, la hormona del crecimiento o el factor VIII de coagulación son proteínas que se producen en cantidades muy pequñas, y actualmete se pueden fabricar mediante ingeniería genéttica. La insulina está formada por el polipéptido A y el B, y para producirlas hubo que sintetizar, en primer lugar, los genes que expresan estas dos cadenas de polipéptidos A y B. Los genes sintéticos se insertan por separado y junto al gen que codifica la β-galactosidasa, en plásmidos de E.coli, obteniéndose plásmidos recombinantes. Estos plásmidos se insertan en distintas cepas de E.coli, donde se expresan y se forma una proteína de fusión muy estable, llamada β-‐gal-‐insulina. Estas proteínas de fusión se procesan químicamente para separar los polipéptidos A y B, y tras una posterior renaturalización y oxidación de las cisteínas, se unen para obtener insulina activa. B. Medicina forense. Los humanos sólo diferimos en un 0,1%, y estas diferencias no se distribuyen al azar, sino que se localizan en regiones cromosómicas concretas, pudiendo ser usadas como marcadores genéticos. La identificación de estas regiones hace que estas diferencias actúen como huella genética que se utiliza en medicina forense y en pruebas de paternidad. Uno de los marcadores más utilizados son las repeticiones cortas en tandem, que son secuencias cortas de bases nitrogenadas que se repiten consecutivamente. Lo que diferencia a las personas es el número de repeticiones que hay en cada uno. Para elaborar una huella genética se emplea un método denominado Southern blot, en el que se comparan fragmentos de restricción obtenidos a partir de muestras distintas de <strong>ADN</strong>. 1. Si la muestra de <strong>ADN</strong> es pequeña, se amplifica con una PCR. Se corta con enzimas de restricción y se somete a una electroforesis en gelnpara separar los fragmentos resultantes de la restricción. Se va a obtener un patrón de bandas característico que aún no es visible. 2. Se aplica una solución alcalina para desnaturalizar el <strong>ADN</strong> y transferir los fragmentos de restricción a una lámina de papel de nitrocelulosa llamada blot. 3. <strong>El</strong> blot se introduce en una bolsa de plástico sellada y se expone a sondas radiactivas de <strong>ADN</strong> específicas para determinados marcadores genéticos. La sonda se hibrida con los fragmentos de restricción que lleven los marcadores, y posteriormente se aplica sobre el blot una película fotográfica en la que la radiactividad de las sondas de <strong>ADN</strong> dejará una imagen que refleja las bandas formadas por el <strong>ADN</strong> apareado con la sonda. C. Diagnóstico de enfermedades. Se pueden detectar los genes responsables de enfermedades genéticas como la anemia falciforme, la hemofilia, la fibrosis quística o la enfermedad de Huntington. Para ello, se clona el gen mediante PCR, se secuencia el producto amplificado y se determina la mutación causante de la enfermedad. Las personas afectadas se pueden identificar incluso antes de nacer, e incluso se pueden detectar los individuos portadores que no desarrollan la enfermedad. D. Terapia génica. 5
Colegio <strong>El</strong> <strong>Valle</strong> Departamento de BiologíaEs una técnica terapéuticamediante la cual se inserta un gen funcional en las células de un paciente humano para corregir un defecto genético o para dotar a las células de una nueva función. Se emplea para curar enfermedades hereditarias y adquiridas, y en ambos casos deben ser provocadas por un gen recesivo, descartando las que están determinadas por muchos genes o las producidas por alteraciones cromosómicas. La terapia génica se puede aplicar a células germinales (se transmiten a la descendencia) o a células somáticas (no se transmiten a la descendencia). En el caso de la línea germinal, está prohibida en la mayoría de países, ya que se plantean problemas éticos relacionados con la modificación del acervo génico de la especie humana y con la eugenesia, al seleccionar artificialmente genes que confieran características especiales a sus portadores. La terapia génica en células somáticaspuede realizarse de varias maneras: • Ex vivo. Se extraen células del paciente, se corrige el defecto genético en el laboratorio y se reintegran al organismo. • In vivo. Los genes terapéuticos se transfieren directamente al paciente mediante un vector, por ejemplo, mediante una inyección intravenosa. • In situ. Se realiza directamente en las células del paciente, introduciendo los genes funcionales edn el órgano afectado. Se usa en casos donde las células son difíciles de extraer y de implantar posteriormente. La terapia génica se empleó por primera vez con los niños burbuja, que están afectados por el síndrome de inmunodeficiencia combinada severa. Se debe a la deficiencia de una enzima, la adenosín desaminasa, implicada en el metabolismo de laspurinas en células madre de médula ósea. INGENIERÍA GENÉTICA Y AGRICULTURA Desde siempre, el hombre ha intentado mejorar el rendimiento de los cultivos de distintas maneras, como or ejemplo, seleccionando para la reproducción, aquelloas plantas más útiles, o aquellas que se adaptan mejor a determinadas condiciones. • Resistencia a herbicidas. <strong>El</strong> glifosato es un herbicida no selectivo que mata las plantas debido a que inactiva una enzima implicada en el metabolismo de los aminoácidos. <strong>El</strong> gen de la resistencia al glifosato se obtuvo de cepas de E.coli resistentes, se clonó y se introdujo en plantas, las cuales sólo sufrirían daños con concentraciones muy grandes de herbicida. • Mejora del producto. Se puede mejorar el valor nutritivo de algunas plantas empleadas en la alimentación humana. Un ejemplo es el arroz transgénico, que produce granos amarillos debido a que lleva beta-‐caroteno, que nuestro organismo utiliza para sintetizar vitamina A. Este arroz podría ayudar a paliar el défict de vitamina A de muchas poblaciones que se alimentan básicamente de arroz. • Plantas farmacéuticas. Se usan para producir proteínas humanas para uso médico, proteínas virales como vacunas o anticuerpos (planticuerpos). Resultan una ventaja frente al cultivo de microorganismos, ya que es más económico y además las plantas tienen los mecanismos adecuados para la fase postraduccional. Obtención de plantas transgénicas <strong>El</strong> vector de clonación más utilizado es el plásmido Ti, procedente de una bacteria del suelo, Agrobacterium tumefaciens. Este plásmido posee un segmento llamado T-‐<strong>ADN</strong> que se integra en el genoma del hospedador. Ambos son cortado por la misma enzima de restricción, formándose posteriormente plásmidos recombinantes, algunos de ellos con el gen de interés, que serán introducidos en céluas vegetales, originando plantas transgénicas. 6
Colegio <strong>El</strong> <strong>Valle</strong> Departamento de BiologíaINGENIERÍA GENÉTICA Y AMBIENTE • Biorremediación. Algunas bacterias degradan la materia orgánica. Mediante ingeniería genética pueden ser modificadas para mejorar su rendimiento. Se utilizan en la limpieza de vertidos de hidrocarburos. • Bioadsorción. Bacterias genéticamente modificadas pueden adsorber (fijar en la superficie celular) ciertos metales pesados que contaminan el suelo. INGENIERÍA GENÉTICA Y GANADERÍA Pretende crear ovejas que den más lana o mejor calidad, animales que crezcan rápidamente, que den carne menos grasa, más leche, etc. También se obtienen sustancias de interés médico, como medicamentos y vacunas. Se ha conseguido expresar el gen de interés junto a otro que promueve la síntesis de una proteína de la leche. <strong>El</strong> producto únicamente se forma en las glándulas mamarias y se secreta a través de la leche, de donde se extrae y purifica. Ejemplo: Activador tisular del plasminógeno, el cual disuelve los trombos sanguíneos producidos en los infartos de miocardio. Ejemplo: Factor VIII de coagulación, que se obtiene de células ováricas del hamster chino y se emplea para combatir la hemofilia. Obtención de un animal transgénico portador de un gen de interés. • Se extraen óvulos de una hembra y se fertilizan in vitro. • Se clona el gen de interés foráneo y posteriormente se inserta el <strong>ADN</strong> clonado directamente en el núcleo de los óvulos fertilizados. Algunos óvulos insertan el transgen en su genoma y pueden expresarlo. • Se implantan los embriones manipulados genéticamente en el útero de una madre sustituta que parirá animales transgénicos que llevan en su dotación genética un gen foráneo, incluso de una especie distinta. • Se obtiene leche de estos descendientes que contiene la proteína de interés. • Se fraccionan y purifican las proteínas de la leche, obteniéndose entre ellas la proteína de interés. CLONACIÓN TERAPÉUTICA Y REPRODUCTIVA EN ANIMALES La clonación terapéutica consiste en la creacción de embriones, por clonación, para utilizarlos como materia prima en distintas terapias. La clonación reproductiva consigue nimales genéticamente idénticos a partir de una célula adulta. <strong>El</strong> método más utilizado es la transferencia nuclear. Clonación d animales por transferencia nuclear • Se obtiene una célula somática del individuo que se quiere clonar. • Se extrae un óvulo de una hembra donante y se elimina el núcleo. • Se reemplaza este núcleo por el de la célula somática. • Se desarrolla la célula creada hasta formar un embrión. • Se implanta en el útero de una hembra de la misma especie, donde culmina su desarrollo. 7
Colegio <strong>El</strong> <strong>Valle</strong> Departamento de BiologíaPROYECTO GENOMA HUMANO. 1. Situar genes y marcadores moleculares en mapas genéticos de alta resolución de cada cromosoma. Este tipo de mapas permite el ordenamiento relativo de genes u otro tipo de secuencia identificable en el <strong>ADN</strong>. 2. Caracterizar y localizar físicamente fragmentos de <strong>ADN</strong> clonados para crear así mapas físicos de cada cromosoma, que se elaboran cortando el <strong>ADN</strong> en fragmentos de restricción para luego determinar su orden en el <strong>ADN</strong> cromosómico. 3. Secuenciar los pequeños fragmentos en los que se ha cortado el <strong>ADN</strong> para luego ensamblarlos y obtener la secuencia genómica completa, generando un mapa completo de secuencias de cada cromosoma. Las características principales del proyecto son: • <strong>El</strong> ser humano posee de 20.000 a 25.000 genescodificadores de proteínas. Más del 40% de los genes no tiene función conocida. • Los seres humanos son idénticos en un 99,9% de los genes. • La mayor parte de estas diferencias se localizan en los polimorfismos de un único nucleótido, que son variaciones que sólo afectan a un par de basaes nitrogenadas. • <strong>El</strong> <strong>ADN</strong> no codificante, que es la mayor parte del genoma, se llama <strong>ADN</strong> basura, porque se creyó que no tenía ninguna función. Actualmente se cree que regula la expresión diferencial de los genes, como la capacidad de agarrar y manipular objetos. 8