iTRAQ - Inserm
iTRAQ - Inserm
iTRAQ - Inserm
Create successful ePaper yourself
Turn your PDF publications into a flip-book with our unique Google optimized e-Paper software.
Fractionnement, LC-Maldi, iTraq<br />
Amélioration de la Couverture Protéomique des<br />
Milieux très Complexes<br />
Catherine Guette<br />
Centre Régional de Lutte Contre<br />
le Cancer Paul Papin<br />
Centre INSERM Régional<br />
de Recherche sur le Cancer U892
2D-DIGE (électrophorèse 2D différentielle)<br />
<strong>iTRAQ</strong><br />
Deux techniques de<br />
protéomique quantitative<br />
100<br />
50<br />
0<br />
111.2 114.4 117.6 120.8<br />
Mass (m/z)
Oxaliplatine : un sel de platine de<br />
NH 3 +<br />
NH 3 +<br />
Pt<br />
Cl<br />
Cl<br />
3 ème génération<br />
NH 3 +<br />
NH 3 +<br />
Cisplatine Carboplatine<br />
Pt<br />
C<br />
C<br />
CO<br />
CO<br />
NH 2<br />
NH 2<br />
Pt<br />
O<br />
O<br />
Oxaliplatine<br />
• Seul sel de platine actif dans les cancers colorectaux métastasés<br />
• Synergie avec le 5-FU, utilisation en première ligne thérapeutique<br />
• Activité dans les cancers du sein et des ovaires<br />
CO<br />
CO
Toxicité de l’oxaliplatine<br />
• Absence de toxicité rénale<br />
cardiaque<br />
auditive<br />
• Faibles toxicités hématologique<br />
digestive<br />
• Toxicité limitante neurologique<br />
De type neuropathies périphériques :<br />
- Neurotoxicité aiguë<br />
- Neurotoxicité chronique<br />
AIGUE : transitoire et dose-dépendante (85 à 95% des<br />
patients). Troubles sensitifs: Crampes, fourmillement<br />
parfois laryngospasmes<br />
CHRONIQUE : évolution progressive<br />
Difficulté à marcher, écrire, se boutonner<br />
Dans 20% des cas pas de régression après 8 mois d’arrêt
Protéomique quantitative<br />
Modèle cellulaire :<br />
neuroblastome SH-SY5Y<br />
CTRL<br />
Ox 5 µM<br />
Ox 10 µM<br />
4 réplicats biologiques<br />
indépendants<br />
Ox 50 µM, 24 h<br />
DIGE-2D <strong>iTRAQ</strong><br />
Cy2, Cy3, Cy5<br />
12,5%, pH 3-10, 18 cm<br />
MALDI-TOF/TOF<br />
4-plex et 8-plex<br />
OFFGEL 24 fractions, nano-LC-C 18<br />
MALDI-TOF/TOF
Cy2<br />
Cy3<br />
Cy5<br />
std interne<br />
éch. 1<br />
éch. 2<br />
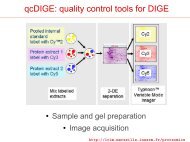
DIGE-2D : principe<br />
+<br />
Mélange des extraits marqués Séparation Scan Analyse
2D-DIGE : Résultats<br />
–pI
1592 protéines<br />
82 protéines<br />
∆ +/- 30 %<br />
Analyse par DIGE-2D
Les approches
<strong>iTRAQ</strong> : Principe chimique<br />
Ross, P. L. (2004) Mol. Cell. Proteomics 3: 1154-1169
Premiers essais avec le 8-plex<br />
8-plex <strong>iTRAQ</strong>
Le réactif <strong>iTRAQ</strong><br />
114 31 Peptide A échantillon 1<br />
115 30 Peptide A échantillon 2<br />
116 29 Peptide A échantillon 3<br />
117 28 Peptide A échantillon 4<br />
Réactif spécifique de la fonction amine des extrémités<br />
N-terminales des peptides et des chaînes latérales des<br />
lysines
Marquage<br />
Quantification<br />
117<br />
116<br />
115<br />
114<br />
31<br />
30<br />
29<br />
28<br />
<strong>iTRAQ</strong><br />
114 31 Peptide A échantillon 1<br />
115 30 Peptide A échantillon 2<br />
115 30 Peptide A échantillon 2<br />
116 29 Peptide A échantillon 3<br />
116 29 Peptide A échantillon 3<br />
116 29 Peptide A échantillon 3<br />
117 28 Peptide A échantillon 4<br />
117 28 Peptide A échantillon 4<br />
117 28 Peptide A échantillon 4<br />
117 28 Peptide A échantillon 4<br />
Fragmentation par collision<br />
Identification
Marquage <strong>iTRAQ</strong><br />
Échantillon 1 Échantillon 2 Échantillon 3 Échantillon 4<br />
Digestat 1 Digestat 2<br />
Réduction, alkylation, digestion<br />
iTraq 114 iTraq 115 iTraq 116 iTraq 117<br />
Digestat 114 Digestat 115 Digestat 116<br />
Digestat 117<br />
Mélange des peptides<br />
Séparation 2D:<br />
1ère dimension: SCX ou OFFGEL<br />
2ème dimension : nanoLC sur C 18<br />
Digestat 2 Digestat 2<br />
Analyse MS/MS sur MALDI TOF/TOF
Couplage LC-MALDI MS/MS<br />
SCX<br />
1614 MS<br />
8585 MS/MS<br />
42 heures
100<br />
90<br />
80<br />
70<br />
60<br />
50<br />
40<br />
30<br />
20<br />
10<br />
Les spectres MS et MS/MS<br />
1176.5342(R12665,S1920)<br />
Final - Shots 2000 - 2LC5; Label 1077<br />
1459.5674(R14251,S833)<br />
1532.6335(R14770,S434)<br />
1767.6576(R15965,S175)<br />
0<br />
699.0 1361.8 2024.6 2687.4 3350.2 4013.0<br />
Mass (m/z)<br />
MS/MS<br />
100<br />
90<br />
80<br />
70<br />
60<br />
50<br />
40<br />
30<br />
20<br />
10<br />
1410.4<br />
114.1301<br />
70.0783<br />
112.1062<br />
175.1482<br />
MS<br />
Final - Shots 2000 - 2LC5; Label 1077 Prec = 1176.5000<br />
636.4142<br />
0<br />
9 256 503 750 997 1244<br />
Mass (m/z)<br />
713.4863<br />
1020.7413<br />
1022.6471<br />
1002.6669<br />
8.0E+4
Zoom sur la zone des étiquettes iTraq<br />
100 114.1146 116.1154<br />
90<br />
80<br />
70<br />
60<br />
50<br />
40<br />
30<br />
20<br />
10<br />
0<br />
110.0883<br />
112.0933<br />
108.0 111.2 114.4 117.6 120.8 124.0<br />
Mass (m/z)<br />
120.0905<br />
4177.5
100<br />
90<br />
80<br />
70<br />
60<br />
50<br />
40<br />
30<br />
20<br />
10<br />
Final - Shots 2000 - 2LC5; Run #4; Label 1076 Prec = 1533.0422<br />
114.1038<br />
116.1008<br />
698.3973<br />
272.1489( 948.4424<br />
472.2503(<br />
0<br />
9.0 331.4 653.8 976.2 1298.6 1621.0<br />
Mass (m/z)<br />
QUANTIFICATION<br />
Résultats<br />
974.4841<br />
100<br />
90<br />
80<br />
70<br />
60<br />
50<br />
40<br />
30<br />
20<br />
10<br />
1102.9<br />
Final - Shots 2000 - 2LC5; Run #4; Label 1076 Prec = 1533.0422<br />
112.0804(<br />
114.1038<br />
115.1052<br />
IDENTIFICATION<br />
117.1096<br />
120.0793 129.1108(<br />
0<br />
110 115 120 125 130 135<br />
Mass (m/z)<br />
1102.9
100<br />
90<br />
80<br />
70<br />
60<br />
50<br />
40<br />
30<br />
20<br />
10<br />
Final - Shots 2000 - 2LC5; Run #4; Label 1348 Prec = 1418.9219<br />
117.1147<br />
115.1078<br />
145.1094<br />
258.2011(<br />
371.2890<br />
569.4044<br />
687.3405<br />
0<br />
9.0 307.2 605.4 903.6 1201.8 1500.0<br />
Mass (m/z)<br />
QUANTIFICATION<br />
Résultats<br />
833.3831<br />
949.4618<br />
100<br />
90<br />
80<br />
70<br />
60<br />
50<br />
40<br />
30<br />
20<br />
8766.7<br />
Final - Shots 2000 - 2LC5; Run #4; Label 1348 Prec = 1418.9219<br />
112.0830<br />
114.1084<br />
115.1078<br />
IDENTIFICATION<br />
116.1122<br />
117.1147<br />
10<br />
110.0714 119.0998<br />
0<br />
110.0 113.2 116.4 119.6 122.8 126.0<br />
Mass (m/z)<br />
8766.7
Poids<br />
(µg)<br />
Identification des protéines :<br />
<strong>iTRAQ</strong> vs non-marqué<br />
Marquage<br />
<strong>iTRAQ</strong><br />
Fractionnement<br />
(1ère dimension)<br />
Nombre de<br />
protéines<br />
200 non<br />
SCX 116 (159)<br />
200<br />
oui<br />
900<br />
800<br />
700<br />
600<br />
500<br />
400<br />
300<br />
200<br />
100<br />
0<br />
SCX 310 (472)<br />
K R<br />
non-labeled<br />
<strong>iTRAQ</strong>
Affinité protonique et <strong>iTRAQ</strong><br />
L’affinité protonique des amines<br />
tertiaires est supérieure à celle des<br />
amines primaires (50 kJ/mole)<br />
Peptides nonmarqués<br />
1273<br />
Peptides<br />
<strong>iTRAQ</strong><br />
7438<br />
L’<strong>iTRAQ</strong> augmente l’affinité<br />
protonique des peptides
Evaluation de la concentration des<br />
protéines par calcul du PAI<br />
PAI= nombre de peptides identifiés/nombre de peptides théoriques<br />
Number of identified proteins<br />
60<br />
50<br />
40<br />
30<br />
20<br />
10<br />
0<br />
200 microg, µg, SCX, SCX, non-marqués non-labeled vs <strong>iTRAQ</strong><br />
PAI<br />
1<br />
PAI<br />
label-free<br />
<strong>iTRAQ</strong>
Nombre de protéines identifiées<br />
Poids<br />
(µg)<br />
200<br />
400<br />
en fonction de la quantité<br />
80<br />
70<br />
60<br />
50<br />
40<br />
30<br />
20<br />
10<br />
0<br />
Marquage<br />
<strong>iTRAQ</strong><br />
oui<br />
Fractionnement<br />
(1ère dimension)<br />
oui SCX<br />
Nombre de<br />
protéines<br />
SCX 310<br />
<strong>iTRAQ</strong>, SCX, 200microg vs 400 microg<br />
429<br />
1<br />
PAI<br />
(472)<br />
(492)<br />
200 µg<br />
400 µg
Fractionnement en fonction du<br />
pI des peptides
Poids<br />
(µg)<br />
400<br />
Identification des protéines :<br />
Marquage<br />
<strong>iTRAQ</strong><br />
oui<br />
400 oui<br />
180<br />
160<br />
140<br />
120<br />
100<br />
80<br />
60<br />
40<br />
20<br />
0<br />
SCX vs OFFGEL<br />
Fractionnement<br />
(1ère dimension)<br />
Nombre de<br />
protéines<br />
SCX 429 (492)<br />
OFFGEL 739 (879)<br />
1<br />
PAI<br />
SCX<br />
OFFGEL
Efficacité de la séparation OFFGEL<br />
Total identified peptides (%)<br />
90%<br />
80%<br />
70%<br />
60%<br />
50%<br />
40%<br />
30%<br />
20%<br />
10%<br />
0%<br />
non-marqués<br />
<strong>iTRAQ</strong><br />
1 2 3 4<br />
Nombre de fractions contenant un peptide unique<br />
Introduction d’un paramètre<br />
supplémentaire, le pI, pour<br />
valider l’identification des<br />
protéines
Classification des protéines en<br />
The human mammary epithelial cell proteome.<br />
Liu et al Proteomics. 2005 (5):1263-73.<br />
35<br />
30<br />
25<br />
20<br />
15<br />
fonction du pI<br />
Protéome humain<br />
(NCBI human protein database)<br />
10<br />
Classification of total identified proteins (A, C) and the human proteome (B, D) based on physiochemical<br />
5<br />
properties. pI distributions were plotted for the total identifications (A) and all human proteins (B) using<br />
0<br />
predicted pI values extracted from 3,7<br />
NCBI human protein database, over increments of 0.1 pH units. MW<br />
distributions were plotted for the total Isoelectric identifications point (0.2 (C) unit andincrements) all human proteins (D) using 20 kDa<br />
increments.
Classification des protéines en<br />
fonction de la masse<br />
The human mammary epithelial cell proteome.<br />
Liu et al Proteomics. 2005 (5):1263-73.<br />
Number of proteins<br />
250<br />
200<br />
150<br />
100<br />
50<br />
0<br />
300
Conclusion<br />
L’<strong>iTRAQ</strong> favorise l’ionisation des peptides en MALDI<br />
Identification de 947 protéines (1111) à partir de 400 µg<br />
Le fractionnement OFFGEL permet d’introduction d’un filtre<br />
supplémentaire , le pI, pour la validation de l’identification des<br />
protéines<br />
E. Ernoult, E. Gamelin, C. Guette, Proteome Science, sous presse.
Nombre<br />
d’expériences<br />
Améliorations technologiques<br />
et injections multiples<br />
Quantité Marquage Séparation<br />
1ère Dimension<br />
103<br />
104<br />
152<br />
245<br />
1406<br />
Séparation<br />
2ème Dimension<br />
3 400 µg <strong>iTRAQ</strong> OFFGEL nanoLC-C18<br />
N° Nombre de<br />
protéines<br />
1 1449<br />
2 1608<br />
3 1637<br />
Total 2791<br />
244<br />
89
<strong>iTRAQ</strong><br />
∆ +/- 30%<br />
141<br />
Comparaison<br />
2D-DIGE / marquage <strong>iTRAQ</strong><br />
186 protéines<br />
(6,6%)<br />
45<br />
37<br />
82 protéines<br />
(5,1%)<br />
2D-DIGE<br />
∆ +/- 30%
Le plasma sanguin humain<br />
22 protéines ~ 99% poids total<br />
Dynamique de concentration ~10 11
Traitement du plasma<br />
Conservation des échantillons<br />
Déplétion<br />
Digestion<br />
Marquage <strong>iTRAQ</strong> (4 ou 8 plasmas simultanément, soit 400 µg)<br />
Fractionnement OFFGEL 24 fractions<br />
Analyse LC-MS/MS<br />
Multiple Affinity Removal Spin<br />
Cartridge – Human 14<br />
Proteominer<br />
Résultats : identification des protéines et quantification
Quantity<br />
(µg)<br />
400<br />
400<br />
400<br />
400<br />
400<br />
Score :<br />
Résultats<br />
Déplétion <strong>iTRAQ</strong> Fractionation Protein<br />
number<br />
no<br />
no<br />
7 prots<br />
7 prots<br />
14 prots<br />
95%<br />
no<br />
no<br />
no<br />
yes<br />
yes<br />
2DLC<br />
(SCX/C18)<br />
OFF-GEL<br />
(12 fractions)<br />
OFF-GEL<br />
(24 fractions)<br />
OFF-GEL<br />
(24 fractions)<br />
OFF-GEL<br />
(24 fractions)<br />
148<br />
127<br />
Peptide<br />
Number<br />
2787<br />
2852<br />
140 7630<br />
184 11 651<br />
216 16 156
Quantité<br />
(µg)<br />
Comparaison<br />
Immunoaffinité / banque de peptides<br />
Déplétion <strong>iTRAQ</strong> Séparation Nombre<br />
Protéines<br />
400 MARS-14 oui OFFGEL<br />
(24 fractions)<br />
3 expériences<br />
400 ProtéoM oui OFFGEL<br />
(24 fractions)<br />
3 expériences<br />
Nombre<br />
Peptides<br />
326 26120<br />
374<br />
275 16987<br />
367
…et la gamme de concentration
Equipe “Protéomique’’<br />
Catherine Guette<br />
Emilie Ernoult<br />
Anthony Bourreau<br />
Catherine Hameline<br />
Muriel Bahut