Einleitung 1. Einleitung 1.1. Bakterielle Polysaccharide Die meisten ...
Einleitung 1. Einleitung 1.1. Bakterielle Polysaccharide Die meisten ...
Einleitung 1. Einleitung 1.1. Bakterielle Polysaccharide Die meisten ...
Erfolgreiche ePaper selbst erstellen
Machen Sie aus Ihren PDF Publikationen ein blätterbares Flipbook mit unserer einzigartigen Google optimierten e-Paper Software.
10 20 30 40 50<br />
. . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . |<br />
RcsA_EC MSTIIMDLCSYTRLGLTGYLLSRGVKKREINDIETVDDLAIACDSQRPSV 50<br />
RcsA_ST MSTIIMDLCSYTRLGLSGYLVSRGVKKREINDIETVDELAIACGAHQPSV 50<br />
RcsA_KP MSTMIMDLCSYTRLGLTGYLTSRGIKKQEIVEVNSAADLQKHCTSCCPAV 50<br />
RcsA_KA MSTMIMDLCSYTRLGLTGYLTSRGIKKQEIVEVNSAADLQKHCTSCCPAV 50<br />
RcsA_EA MPTIIMDSCNYTRLGLTEYMTVKGVKKKNISLINDIAQLQNKCQQLKPGV 50<br />
RcsA_PS MPTIIMDSCNYTRLGLSDYMSSKGVKKKNITSVSDIEQLQQRCEQLKPGV 50<br />
60 70 80 90 100<br />
. . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . |<br />
RcsA_EC VFINEDCFIHDASNSQRIKLIINQHPNTLFIVFMAIANVHFDEYLLVRKN 100<br />
RcsA_ST VFINEDCFIHTPSDSQQIKQIINQHPDTLFIVFMAIANVHFDEYLLVRKN 100<br />
RcsA_KP VFLNEDCFVHDDESNGIIRQIITQNPATLFVIFMSLANIHFDRYLRVRKN 100<br />
RcsA_KA VFLNEDCFVHDDESNGIIRQIITQNPATLFVIFMSLANIHFDRYLRVRKN 100<br />
RcsA_EA VLINEDCFIHESDASERIRKIILQHPDTLFFIFMAISNIHFEEYLYVRNN 100<br />
RcsA_PS VFINEDCFIHETDSSERIRSIINQHPETLFFIFMAISNIHFEEYLYVRKN 100<br />
110 120 130 140 150<br />
. . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . |<br />
RcsA_EC LLISSKSIKPESLDDILGDILKKETTITS----FLNMPTLSLSRTESSML 146<br />
RcsA_ST LLISSKSIKPDSLDTLLGDILKKESGISG----TINLPTLSLSRTESSML 146<br />
RcsA_KP LLISSKSITPKDLDVILVNYLKYKNTSVG----QLTLPTLSLSKTESNML 146<br />
RcsA_KA LLISSKSITPKDLDVILVNYLKYKNTSVG----QLTLPTLSLSKTESNML 146<br />
RcsA_EA LIITSKAIKISTLDSLLNGYFQKKLNLSVRHGTHSEVHPLTLSQTESNML 150<br />
RcsA_PS LIITSKAIKPSTLDSLLSTYLQKKLNMSPRISSGLDVHPLTLSQTESNML 150<br />
160 170 180 190 200<br />
. . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . |<br />
RcsA_EC RMWMAGQGTIQISDQMNIKAKTVSSHKGNIKRKIKTHNKQVIYHVVRLTD 196<br />
RcsA_ST RMWMEGQGTIQISDRMNIKAKTVSSHKGNIKRKIKTHNKQVIYHVVRLTD 196<br />
RcsA_KP QMWMAGHGTSQISTQMNIKAKTVSSHKGNIKKKIQTHNKQVIYHIVRLTE 196<br />
RcsA_KA QMWMAGHGTSQISTQMNIKAKTVSSHKGNIKKKIQTHNKQVIYHIVRLTE 196<br />
RcsA_EA KMWMSGHDTIQISDKMQIKAKTVSSHKGNIKRKIKTHNKQVIYHVVRLTD 200<br />
RcsA_PS KMWMSGHDTIQISDKMQIKAKTVSSHKGNIKRKIKTHNKQVIYHVVRLTD 200<br />
210<br />
. . . . | . . . . | .<br />
RcsA_EC NVTNGIFVNMR 207<br />
RcsA_ST NVTNGIFANMR 207<br />
RcsA_KP NITSGIQVNMR 207<br />
RcsA_KA NITSGIQVNMR 207<br />
RcsA_EA NVTSGIYVNER 211<br />
RcsA_PS NVTSGIYVNVR 211<br />
17<br />
10 20 30 40 50<br />
. . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . |<br />
RcsB_EC MNNMNVIIADDHPIVLFGIRKSLEQIEWVNVVGEFEDSTALINNLPKLDA 50<br />
RcsB_ST MNNMNIIIADDHPIVLFGIRKSLEQIEWVNVVGEFEDSTALINNLPKLDA 50<br />
RcsB_EA MNNLNVIIADDHPIVLFGIRKSLEQIEWVNVVGEFEDSTALINSLSKLDA 50<br />
60 70 80 90 100<br />
. . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . |<br />
RcsB_EC HVLITDLSMPGDKYGDGITLIKYIKRHFPSLSIIVLTMNNNPAILSAVLD 100<br />
RcsB_ST HVLITDLSMPGDKYGDGITLIKYIKRHFPSLSIIVLTMNNNPAILSAVLD 100<br />
RcsB_EA NVLVTDLSMPGEKYGDGITLIKYIKRHYPDLSIIVLTMNNNPAILSAVLD 100<br />
110 120 130 140 150<br />
. . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . |<br />
RcsB_EC LDIEGIVLKQGAPTDLPKALAALQKGKKFTPESVSRLLEKISAGGYGDKR 150<br />
RcsB_ST LDIEGIVLKQGAPTDLPKALAALQKGKKFTPESVSRLLEKISAGGYGDKR 150<br />
RcsB_EA LDIEGIVLKQGAPTDLPKALAALQKGKKYTPESVAKLLEKISAGGYGDKR 150<br />
160 170 180 190 200<br />
. . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . |<br />
RcsB_EC LSPKESEVLRLFAEGFLVTEIAKKLNRSIKTISSQKKSAMMKLGVENDIA 200<br />
RcsB_ST LSPKESEVLRLFAEGFLVTEIAKKLNRSIKTISSQKKSAMMKLGVENDIA 200<br />
RcsB_EA LSPKESEVLRLFAEGFLVTEIAKKLNRSIKTISSQKKSAMMKLGVDNDIA 200<br />
210<br />
. . . . | . . . . | . . . . | .<br />
RcsB_EC LLNYLSSVTLSPADKD 216<br />
RcsB_ST LLNYLSSVTLSPTDKE 216<br />
RcsB_EA LLNYLSSVSMTPVDK- 215<br />
<strong>Einleitung</strong><br />
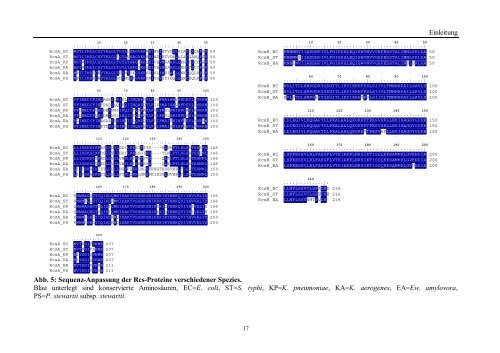
Abb. 5: Sequenz-Anpassung der Rcs-Proteine verschiedener Spezies.<br />
Blau unterlegt sind konservierte Aminosäuren, EC=E. coli, ST=S. typhi, KP=K. pneumoniae, KA=K. aerogenes, EA=Ew. amylovora,<br />
PS=P. stewartii subsp. stewartii.