Manual rápido de uso de Tools for Population Genetics (TFPGA)
Manual rápido de uso de Tools for Population Genetics (TFPGA)
Manual rápido de uso de Tools for Population Genetics (TFPGA)
Create successful ePaper yourself
Turn your PDF publications into a flip-book with our unique Google optimized e-Paper software.
4.- Genetic distance<br />
Calcula la distancia genética entre las poblaciones y/o subpoblaciones. Este análisis nos va a mostrar una tabla<br />
<strong>de</strong> distancias genéticas entre todos los pares <strong>de</strong> poblaciones y/o subpoblaciones <strong>de</strong> nuestra muestra. Una distancia <strong>de</strong> 0<br />
indica que las poblaciones comparten el 100% <strong>de</strong> sus alelos, es <strong>de</strong>cir que son idénticas y una distancia gran<strong>de</strong> (generalmente<br />
<strong>de</strong> 1) indica que no comparten ninguno <strong>de</strong> sus alelos, es <strong>de</strong>cir, que son completamente diferentes. Nota: este parámetro se<br />
entien<strong>de</strong> e interpret mejor luego <strong>de</strong> hacer el UPGMA (árbol <strong>de</strong> distancias o representación gráfica entre las distancias)<br />
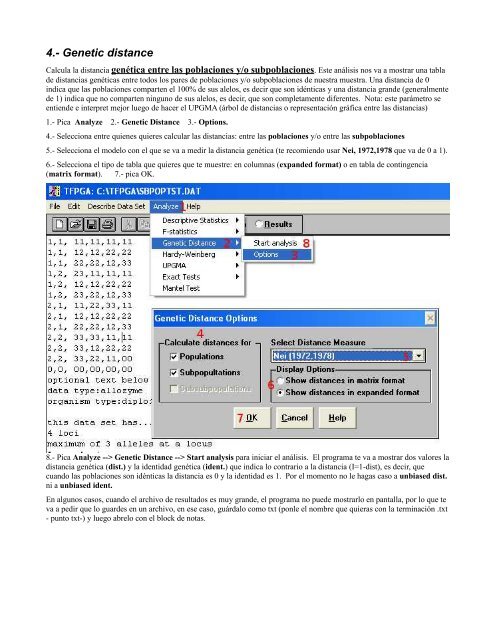
1.- Pica Analyze 2.- Genetic Distance 3.- Options.<br />
4.- Selecciona entre quienes quieres calcular las distancias: entre las poblaciones y/o entre las subpoblaciones<br />
5.- Selecciona el mo<strong>de</strong>lo con el que se va a medir la distancia genética (te recomiendo usar Nei, 1972,1978 que va <strong>de</strong> 0 a 1).<br />
6.- Selecciona el tipo <strong>de</strong> tabla que quieres que te muestre: en columnas (expan<strong>de</strong>d <strong>for</strong>mat) o en tabla <strong>de</strong> contingencia<br />
(matrix <strong>for</strong>mat). 7.- pica OK.<br />
8.- Pica Analyze --> Genetic Distance --> Start analysis para iniciar el análisis. El programa te va a mostrar dos valores la<br />
distancia genética (dist.) y la i<strong>de</strong>ntidad genética (i<strong>de</strong>nt.) que indica lo contrario a la distancia (I=1-dist), es <strong>de</strong>cir, que<br />
cuando las poblaciones son idénticas la distancia es 0 y la i<strong>de</strong>ntidad es 1. Por el momento no le hagas caso a unbiased dist.<br />
ni a unbiased i<strong>de</strong>nt.<br />
En algunos casos, cuando el archivo <strong>de</strong> resultados es muy gran<strong>de</strong>, el programa no pue<strong>de</strong> mostrarlo en pantalla, por lo que te<br />
va a pedir que lo guar<strong>de</strong>s en un archivo, en ese caso, guárdalo como txt (ponle el nombre que quieras con la terminación .txt<br />
- punto txt-) y luego abrelo con el block <strong>de</strong> notas.