Manual rápido de uso de Tools for Population Genetics (TFPGA)
Manual rápido de uso de Tools for Population Genetics (TFPGA)
Manual rápido de uso de Tools for Population Genetics (TFPGA)
You also want an ePaper? Increase the reach of your titles
YUMPU automatically turns print PDFs into web optimized ePapers that Google loves.
6.- UPGMA<br />
Este análisis calcula las distancias genéticas <strong>de</strong> igual <strong>for</strong>ma que el apartado 4 (Genetic distance), pero a<strong>de</strong>más las<br />
representa mediante un <strong>de</strong>ndograma <strong>de</strong> distancias mediante el método UPGMA, es <strong>de</strong>cir, que te va a dar un árbol <strong>de</strong><br />
distancias entre las poblaciones, subpoblaciones o sub-subpoblaciones.<br />
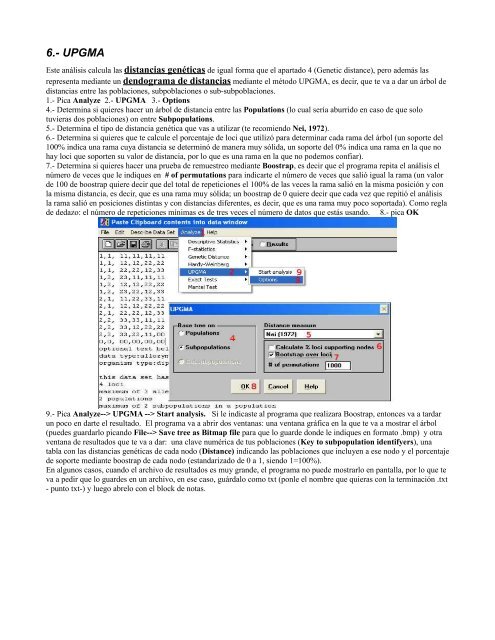
1.- Pica Analyze 2.- UPGMA 3.- Options<br />
4.- Determina si quieres hacer un árbol <strong>de</strong> distancia entre las <strong>Population</strong>s (lo cual sería aburrido en caso <strong>de</strong> que solo<br />
tuvieras dos poblaciones) on entre Subpopulations.<br />
5.- Determina el tipo <strong>de</strong> distancia genética que vas a utilizar (te recomiendo Nei, 1972).<br />
6.- Determina si quieres que te calcule el porcentaje <strong>de</strong> loci que utilizó para <strong>de</strong>terminar cada rama <strong>de</strong>l árbol (un soporte <strong>de</strong>l<br />
100% indica una rama cuya distancia se <strong>de</strong>terminó <strong>de</strong> manera muy sólida, un soporte <strong>de</strong>l 0% indica una rama en la que no<br />
hay loci que soporten su valor <strong>de</strong> distancia, por lo que es una rama en la que no po<strong>de</strong>mos confiar).<br />
7.- Determina si quieres hacer una prueba <strong>de</strong> remuestreo mediante Boostrap, es <strong>de</strong>cir que el programa repita el análisis el<br />
número <strong>de</strong> veces que le indiques en # of permutations para indicarte el número <strong>de</strong> veces que salió igual la rama (un valor<br />
<strong>de</strong> 100 <strong>de</strong> boostrap quiere <strong>de</strong>cir que <strong>de</strong>l total <strong>de</strong> repeticiones el 100% <strong>de</strong> las veces la rama salió en la misma posición y con<br />
la misma distancia, es <strong>de</strong>cir, que es una rama muy sólida; un boostrap <strong>de</strong> 0 quiere <strong>de</strong>cir que cada vez que repitió el análisis<br />
la rama salió en posiciones distintas y con distancias diferentes, es <strong>de</strong>cir, que es una rama muy poco soportada). Como regla<br />
<strong>de</strong> <strong>de</strong>dazo: el número <strong>de</strong> repeticiones mínimas es <strong>de</strong> tres veces el número <strong>de</strong> datos que estás usando. 8.- pica OK<br />
9.- Pica Analyze--> UPGMA --> Start analysis. Si le indicaste al programa que realizara Boostrap, entonces va a tardar<br />
un poco en darte el resultado. El programa va a abrir dos ventanas: una ventana gráfica en la que te va a mostrar el árbol<br />
(pue<strong>de</strong>s guardarlo picando File--> Save tree as Bitmap file para que lo guar<strong>de</strong> don<strong>de</strong> le indiques en <strong>for</strong>mato .bmp) y otra<br />
ventana <strong>de</strong> resultados que te va a dar: una clave numérica <strong>de</strong> tus poblaciones (Key to subpopulation i<strong>de</strong>ntifyers), una<br />
tabla con las distancias genéticas <strong>de</strong> cada nodo (Distance) indicando las poblaciones que incluyen a ese nodo y el porcentaje<br />
<strong>de</strong> soporte mediante boostrap <strong>de</strong> cada nodo (estandarizado <strong>de</strong> 0 a 1, siendo 1=100%).<br />
En algunos casos, cuando el archivo <strong>de</strong> resultados es muy gran<strong>de</strong>, el programa no pue<strong>de</strong> mostrarlo en pantalla, por lo que te<br />
va a pedir que lo guar<strong>de</strong>s en un archivo, en ese caso, guárdalo como txt (ponle el nombre que quieras con la terminación .txt<br />
- punto txt-) y luego abrelo con el block <strong>de</strong> notas.