Phosphorylierungen bei Signalproteinen erkennen ... - BIOspektrum
Phosphorylierungen bei Signalproteinen erkennen ... - BIOspektrum
Phosphorylierungen bei Signalproteinen erkennen ... - BIOspektrum
Sie wollen auch ein ePaper? Erhöhen Sie die Reichweite Ihrer Titel.
YUMPU macht aus Druck-PDFs automatisch weboptimierte ePaper, die Google liebt.
270 WISSENSCHAFT · SPECIAL: PROTEOMICS<br />
Posttranslationale Modifikationen<br />
<strong>Phosphorylierungen</strong> <strong>bei</strong><br />
<strong>Signalproteinen</strong> <strong>erkennen</strong> und deuten<br />
GERALD RADZIWILL, ADRIAN FISCHER, BETTINA WARSCHEID<br />
INSTITUT FÜR BIOLOGIE II, BIOCHEMIE – FUNKTIONELLE PROTEOMIK UND BIOSS<br />
CENTRE FOR BIOLOGICAL SIGNALLING STUDIES, UNIVERSITÄT FREIBURG<br />
Detailed knowledge of phosphorylation events in proteins is an important<br />
key for a better understanding of biological signalling processes in health<br />
and disease. The combined use of traditional biochemical methods and<br />
modern mass spectrometry employing innovative fragmentation technologies<br />
opens up new avenues for detailed functional studies of signalling<br />
proteins.<br />
DOI: 10.1007/s12268-013-0307-z<br />
© Springer-Verlag 2013<br />
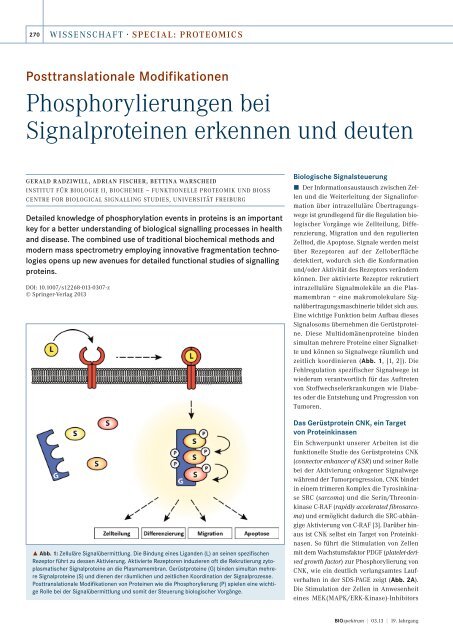
˚ Abb. 1: Zelluläre Signalübermittlung. Die Bindung eines Liganden (L) an seinen spezifischen<br />
Rezeptor führt zu dessen Aktivierung. Aktivierte Rezeptoren induzieren oft die Rekrutierung zytoplasmatischer<br />
Signalproteine an die Plasmamembran. Gerüstproteine (G) binden simultan mehrere<br />
Signalproteine (S) und dienen der räumlichen und zeitlichen Koordination der Signalprozesse.<br />
Posttranslationale Modifikationen von Proteinen wie die Phosphorylierung (P) spielen eine wichtige<br />
Rolle <strong>bei</strong> der Signalübermittlung und somit der Steuerung biologischer Vorgänge.<br />
Biologische Signalsteuerung<br />
ó Der Informationsaustausch zwischen Zellen<br />
und die Weiterleitung der Signalinformation<br />
über intrazelluläre Übertragungswege<br />
ist grundlegend für die Regulation biologischer<br />
Vorgänge wie Zellteilung, Differenzierung,<br />
Migration und den regulierten<br />
Zelltod, die Apoptose. Signale werden meist<br />
über Rezeptoren auf der Zelloberfläche<br />
detektiert, wodurch sich die Konformation<br />
und/oder Aktivität des Rezeptors verändern<br />
können. Der aktivierte Rezeptor rekrutiert<br />
intrazelluläre Signalmoleküle an die Plasmamembran<br />
– eine makromolekulare Signalübertragungsmaschinerie<br />
bildet sich aus.<br />
Eine wichtige Funktion <strong>bei</strong>m Aufbau dieses<br />
Signalosoms übernehmen die Gerüstproteine.<br />
Diese Multidomänenproteine binden<br />
simultan mehrere Proteine einer Signalkette<br />
und können so Signalwege räumlich und<br />
zeitlich koordinieren (Abb. 1, [1, 2]). Die<br />
Fehlregulation spezifischer Signalwege ist<br />
wiederum verantwortlich für das Auftreten<br />
von Stoffwechselerkrankungen wie Diabetes<br />
oder die Entstehung und Progression von<br />
Tumoren.<br />
Das Gerüst protein CNK, ein Target<br />
von Protein kinasen<br />
Ein Schwerpunkt unserer Ar<strong>bei</strong>ten ist die<br />
funktionelle Studie des Ge rüstproteins CNK<br />
(connector enhancer of KSR) und seiner Rolle<br />
<strong>bei</strong> der Aktivierung onkogener Signalwege<br />
während der Tumorprogression. CNK bindet<br />
in einem trimeren Komplex die Tyrosinkinase<br />
SRC (sarcoma) und die Serin/Threonin -<br />
kinase C-RAF (rapidly accelerated fibrosarcoma)<br />
und ermöglicht dadurch die SRC-abhängige<br />
Aktivierung von C-RAF [3]. Darüber hinaus<br />
ist CNK selbst ein Target von Proteinkinasen.<br />
So führt die Stimulation von Zellen<br />
mit dem Wachstumsfaktor PDGF (platelet-derived<br />
growth factor) zur Phosphorylierung von<br />
CNK, wie ein deutlich verlangsamtes Laufverhalten<br />
in der SDS-PAGE zeigt (Abb. 2A).<br />
Die Stimulation der Zellen in Anwesenheit<br />
eines MEK(MAPK/ERK-Kinase)-Inhibitors<br />
<strong>BIOspektrum</strong> | 03.13 | 19. Jahrgang
A<br />
B<br />
˚ Abb. 2: Analyse von Phosphorylierungsereignissen.<br />
A, FLAG-CNK-überexprimierende<br />
HEK293-Zellen wurden mit PDGF<br />
(platelet-derived growth factor) in Anwesenheit<br />
(+) oder Abwesenheit (–) eines<br />
MEK(MAPK/ ERK-Kinase)-Inhibitors stimuliert.<br />
Zelllysate wurden mittels SDS-PAGE<br />
aufgetrennt und CNK in einem Immunoblot<br />
mit Anti-FLAG-Antikörpern detektiert. B,<br />
Affinitätsaufge reinigte CNK-Fragmente aus<br />
Lysaten von CNK-überexprimierenden Zellen<br />
wurden als Substrate für ERK in einer<br />
in vitro-Kinase-Reaktion in Anwesenheit<br />
von [γ- 32 P]ATP eingesetzt. Die Reaktionsprodukte<br />
wurden über SDS-PAGE und<br />
Autoradiografie analysiert. pCNK, phosphorylierte<br />
CNK-Isoform.<br />
bestätigt, dass CNK durch MEK oder ERK<br />
(extracellular signal-regulated kinase) phosphoryliert<br />
wird.<br />
Mithilfe einer in vitro-Kinase-Reaktion in<br />
Anwesenheit von ERK und radioaktiv markiertem<br />
[γ- 32 P]ATP können wir zeigen, dass<br />
CNK ein spezifisches Substrat von ERK ist<br />
(Abb. 2B). Anhand des Autoradiogramms<br />
des SDS-Gels ist zu sehen, dass ERK CNK im<br />
Bereich der Aminosäuren 280 bis 720 phosphoryliert.<br />
Somit ist CNK einerseits ein positiver<br />
Regulator der durch verschiedene<br />
Wachstumsfaktoren stimulierten, zentralen<br />
RAF-MEK-ERK-Signalkette. Andererseits ist<br />
CNK selbst ein Substrat dieser Signalkette,<br />
<strong>BIOspektrum</strong> | 03.13 | 19. Jahrgang<br />
eine Beobachtung, die auf einen möglichen<br />
Rückkopplungsmechanismus hinweist. Ziel<br />
unserer aktuellen Studien ist es, Kinase-vermittelte<br />
Phosphorylierungsereignisse in<br />
CNK umfassend zu entschlüsseln. Moderne<br />
Massenspektrometrie(MS)-basierte<br />
Methoden stellen einen sehr eleganten<br />
Ansatz zur genauen Bestimmung von<br />
in vivo-Phosphorylierungsstellen in <strong>Signalproteinen</strong><br />
sowie zur detaillierten Untersuchung<br />
von Kinase-Substrat-Beziehungen<br />
dar.<br />
Analyse von<br />
Phosphorylierungsmustern<br />
Für die MS-basierte Kartierung von in vivo-<br />
Phosphorylierungsstellen empfiehlt es sich,<br />
das endogen exprimierte Protein in Anwesenheit<br />
von Phosphatase-Inhibitoren affinitätsbasiert<br />
aufzureinigen. Nachfolgend können<br />
eventuell störende Komponenten mittels<br />
einer SDS-PAGE abgetrennt werden. Einen<br />
interessanten Ansatz bietet auch die PhostagTM-SDS-PAGE<br />
[4], mit der phosphorylierte<br />
und nicht phosphorylierte Proteinisoformen<br />
vor der MS-Analyse aufgetrennt werden<br />
können (Abb. 3A). Die Verwendung<br />
eines zweiwertigen Metallionenkomplexes<br />
(Mn2+ -Phos-tag) führt zur spezifischen Bindung<br />
von Phosphomonoester-Dianionen wie<br />
Phosphoserin, Phosphothreonin oder Phosphotyrosin<br />
in Proteinen und damit zur<br />
Retardierung der phosphorylierten Proteinisoformen<br />
während der Gelelektrophorese.<br />
Alternativ bietet sich ein gelfreier Ansatz<br />
an. Das Protein wird hierzu z. B. mit Aceton<br />
präzipitiert und in einem geeigneten<br />
Puffersystem aufgenommen. Nach proteolytischem<br />
Verdau der Proteine in der Gelmatrix<br />
oder in Lösung erfolgt die Analyse<br />
mittels Flüssigkeitschromatografie in direkter<br />
Kopplung mit der Massenspektrometrie<br />
(LC/MS) (Abb. 3A). Die Auswertung der<br />
Daten erfolgt über spezielle Suchalgorithmen.<br />
Fragmentierung zur Lokalisierung<br />
der Phosphatgruppe<br />
Eine umfassende sowie zuverlässige Bestimmung<br />
von Phosphorylierungsstellen beruht<br />
zum einen auf einer möglichst vollständigen<br />
und zum Teil redundanten Sequenzierung<br />
des Proteins durch den Einsatz verschiedener<br />
Proteasen (z. B. Chymotrypsin,<br />
AspN) neben der Standardprotease Trypsin.<br />
Zum anderen ist es erforderlich, positionsspezifische<br />
Fragmentionen in MS/MS-Spektren<br />
zu detektieren, anhand derer Phos-
272 WISSENSCHAFT · SPECIAL: PROTEOMICS<br />
A<br />
B<br />
˚ Abb. 3: Analyse von in vivo-Phosphorylierungsstellen. A, Das affinitätsaufgereinigte Protein<br />
einschließlich seiner phosphorylierten Isoformen wird entweder über SDS-PAGE bzw. Phos-tag-<br />
SDS-PAGE oder direkt aus der Lösung analysiert. Im Anschluss erfolgt ein proteolytischer Verdau<br />
im Gel oder in Lösung. Die generierten Peptide werden mittels LC/MS analysiert. B, Analyse eines<br />
phosphorylierten Peptids mit unterschiedlichen MS/MS-Methoden. Fragmentierung mittels CID<br />
(collision-induced dissociation) und MSA (multistage activation) führt jeweils zum Verlust der<br />
Phosphatgruppe unter Abspaltung von H 3 PO 4 (98 u) als Neutralteilchen. Die Generierung<br />
sequenzspezifischer Fragmentionen ist generell effizienter mit MSA im Vergleich zu CID. ETD<br />
(electron transfer dissociation) erlaubt die Fragmentierung phosphorylierter Peptide ohne Abspaltung<br />
der Phosphatgruppe. Es werden sowohl sequenz- als auch positionsspezifische Fragmentionen<br />
im Spektrum beobachtet.<br />
phatgruppen in der Aminosäuresequenz lokalisiert<br />
werden können. Bei der Fragmentierung<br />
von Peptiden mit phosphorylierten<br />
Serin- oder Threoninresten mittels CID (collision-induced<br />
dissociation) ist jedoch die Spaltung<br />
der Phosphoesterbindung bevorzugt.<br />
Dies führt zum Verlust des Phosphatrestes<br />
plus Wasser (H 3 PO 4 , 98 u) durch β-Elimination<br />
– ein Phänomen, das die Lokalisierung<br />
der Phosphatgruppe in Peptiden mit mehreren<br />
potenziellen Phosphorylierungsstellen oft<br />
erschwert und auf einfach, aber insbesonde-<br />
re mehrfach phosphorylierte Peptide zutrifft<br />
(Abb. 3B).<br />
Der Einsatz neuer MS/MS-Techniken wie<br />
MSA (multistage activation) [5] und HCD<br />
(higher energy collisional dissociation) [6] kann<br />
zu einer verbesserten Fragmentierung und<br />
somit Identifizierung von Phosphopeptiden<br />
führen, jedoch wird der Verlust der Phosphatgruppe<br />
nicht verhindert. Eine vielversprechende<br />
Alternative bietet ETD (electron<br />
transfer dissociation) [7]. Bei dieser Methode<br />
wird die Fragmentierung des Peptids durch<br />
die Übertragung eines Elektrons und Bildung<br />
eines Radikalkations initiiert. Es kommt letztlich<br />
zur Spaltung der N-Cα-Bindung im<br />
Peptid, ohne Verlust der Phosphatgruppe<br />
(Abb. 3B). ETD stellt daher eine leistungsstarke<br />
Methode zur genauen Lokalisierung<br />
von Phosphorylierungsstellen in Proteinen<br />
mit komplexen Modifikationsmustern dar.<br />
Bedeutung für das Verständnis von<br />
Erkrankungen<br />
Die Analyse von Kinase-Substrat-Beziehungen<br />
zeigte, dass lineare Signalketten zu einem<br />
komplexen zellulären Signalnetzwerk verknüpft<br />
sind. Die Funktionstüchtigkeit dieses<br />
Signalnetzwerks muss gewährleitet sein; Störungen<br />
sind die Ursache vieler Erkrankungen.<br />
Mithilfe moderner MS-basierter Ansätze<br />
lassen sich Kinase-vermittelte Phosphorylierungsmuster<br />
genau <strong>erkennen</strong>, deren Deutung<br />
nicht nur die Grundlage für das Verständnis<br />
biologischer Signalprozesse bildet, sondern<br />
auch zum Verständnis der Entstehung von<br />
Erkrankungen <strong>bei</strong>trägt und die Entwicklung<br />
neuer therapeutischer Ansätze ermöglicht.<br />
Danksagung<br />
Die Ar<strong>bei</strong>ten sind von der Deutschen Forschungsgemeinschaft<br />
und der Exzellenzinitiative<br />
des Bundes und der Länder (EXC 294<br />
BIOSS) gefördert. ó<br />
Literatur<br />
[1] Good MC, Zalatan JG, Lim WA (2011) Scaffold proteins:<br />
hubs for controlling the flow of cellular information.<br />
Science 332:680–686<br />
[2] Fritz RD, Radziwill G (2011) CNK1 and other scaffolds for<br />
Akt/FoxO signaling. Biochim Biophys Acta 1813:1971–1977<br />
[3] Ziogas A, Moelling K, Radziwill G (2005) CNK1 is a scaffold<br />
protein that regulates Src-mediated Raf-1 activation.<br />
J Biol Chem 280:24205–24211<br />
[4] Kinoshita E, Kinoshita-Kikuta E, Takiyama K et al. (2006)<br />
Phosphate-binding tag, a new tool to visualize phosphorylated<br />
proteins. Mol Cell Proteomics 5:749–757<br />
[5] Schroeder MJ, Shabanowitz J, Schwartz JC et al. (2004) A<br />
neutral loss activation method for improved phosphopeptide<br />
sequence analysis by quadrupole ion trap mass spectrometry.<br />
Anal Chem 76:3590–3598<br />
[6] Olsen JV, Macek B, Lange O et al. (2007) Higher-energy<br />
C-trap dissociation for peptide modification analysis.<br />
Nat Methods 4:709–712<br />
[7] Syka JE, Coon JJ, Schroeder MJ et al. (2004) Peptide and<br />
protein sequence analysis by electron transfer dissociation<br />
mass spectrometry. Proc Natl Acad Sci USA 101:9528–9533<br />
Korrespondenzadresse:<br />
Prof. Dr. Bettina Warscheid<br />
Prof. Dr. Gerald Radziwill<br />
Institut für Biologie II, Biochemie – Funktionelle<br />
Proteomik<br />
Albert-Ludwigs-Universität Freiburg<br />
Schänzlestraße 1<br />
D-79104 Freiburg<br />
Tel.: 0761-203-2689<br />
Fax: 0761-203-2601<br />
bettina.warscheid@biologie.uni-freiburg.de<br />
gerald.radziwill@bioss.uni-freiburg.de<br />
<strong>BIOspektrum</strong> | 03.13 | 19. Jahrgang
AUTOREN<br />
<strong>BIOspektrum</strong> | 03.13 | 19. Jahrgang<br />
Gerald Radziwill<br />
1980–1985 Biologiestudium,<br />
Universität Heidelberg. 1989<br />
Promotion, Zentrum für<br />
Molekulare Biologie, Heidelberg.<br />
1989–1993 wissenschaftlicher<br />
Mitar<strong>bei</strong>ter, Max-<br />
Planck-Institut für Molekulare<br />
Genetik, Berlin. 1994–2008<br />
Assistent/Oberassistent,<br />
Institut für Virologie, Universität<br />
Zürich. 2008 Habilitation<br />
für das Fach Molekulare<br />
Medizin, Universität Zürich.<br />
Seit 2009 Professurvertretung<br />
für Biochemie, Fakultät für<br />
Biologie, Universität Freiburg.<br />
2012 Ernennung zum außerplanmäßigen<br />
Professor, Universität<br />
Freiburg.<br />
Adrian Fischer<br />
2006–2012 Biologiestudium<br />
an der Universität Freiburg.<br />
Seit 2012 Doktorand in der<br />
Abteilung Biochemie – Funktionelle<br />
Proteomik, Institut für<br />
Biologie II, Universität Freiburg.<br />
Bettina Warscheid<br />
1991–1997 Chemiestudium,<br />
TU Dortmund. 2002 Promotion,<br />
Leibniz-Institut für<br />
Analytische Wissenschaften,<br />
Dortmund. 2002–2003 DFG-<br />
Forschungsstipendium,<br />
University of Maryland, College<br />
Park, MD, USA. 2003–<br />
2010 Gruppenleiterin, Medizinisches<br />
Proteom-Center, Ruhr-<br />
Universität Bochum. 2004–<br />
2009 W1-Professur, Ruhr-Universität<br />
Bochum. 2009–2010<br />
W2-Professur, Universität<br />
Duisburg-Essen. Seit 2010<br />
W3-Professur für Funktionelle<br />
Proteomik, Fakultät für Biologie<br />
und BIOSS Centre for Biological<br />
Signalling Studies, Universität<br />
Freiburg.<br />
273