Phosphorylierungen bei Signalproteinen erkennen ... - BIOspektrum
Phosphorylierungen bei Signalproteinen erkennen ... - BIOspektrum
Phosphorylierungen bei Signalproteinen erkennen ... - BIOspektrum
Sie wollen auch ein ePaper? Erhöhen Sie die Reichweite Ihrer Titel.
YUMPU macht aus Druck-PDFs automatisch weboptimierte ePaper, die Google liebt.
A<br />
B<br />
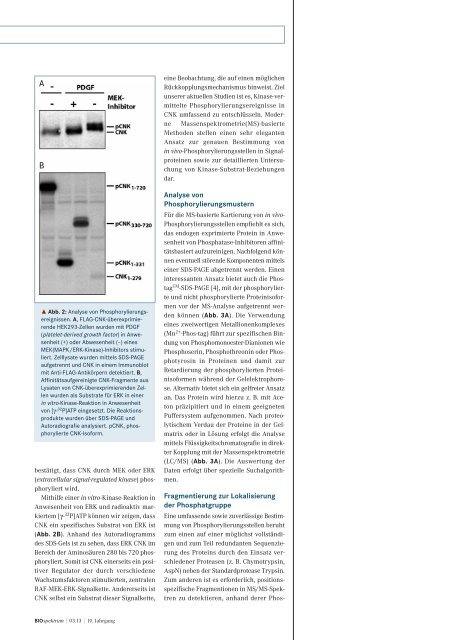
˚ Abb. 2: Analyse von Phosphorylierungsereignissen.<br />
A, FLAG-CNK-überexprimierende<br />
HEK293-Zellen wurden mit PDGF<br />
(platelet-derived growth factor) in Anwesenheit<br />
(+) oder Abwesenheit (–) eines<br />
MEK(MAPK/ ERK-Kinase)-Inhibitors stimuliert.<br />
Zelllysate wurden mittels SDS-PAGE<br />
aufgetrennt und CNK in einem Immunoblot<br />
mit Anti-FLAG-Antikörpern detektiert. B,<br />
Affinitätsaufge reinigte CNK-Fragmente aus<br />
Lysaten von CNK-überexprimierenden Zellen<br />
wurden als Substrate für ERK in einer<br />
in vitro-Kinase-Reaktion in Anwesenheit<br />
von [γ- 32 P]ATP eingesetzt. Die Reaktionsprodukte<br />
wurden über SDS-PAGE und<br />
Autoradiografie analysiert. pCNK, phosphorylierte<br />
CNK-Isoform.<br />
bestätigt, dass CNK durch MEK oder ERK<br />
(extracellular signal-regulated kinase) phosphoryliert<br />
wird.<br />
Mithilfe einer in vitro-Kinase-Reaktion in<br />
Anwesenheit von ERK und radioaktiv markiertem<br />
[γ- 32 P]ATP können wir zeigen, dass<br />
CNK ein spezifisches Substrat von ERK ist<br />
(Abb. 2B). Anhand des Autoradiogramms<br />
des SDS-Gels ist zu sehen, dass ERK CNK im<br />
Bereich der Aminosäuren 280 bis 720 phosphoryliert.<br />
Somit ist CNK einerseits ein positiver<br />
Regulator der durch verschiedene<br />
Wachstumsfaktoren stimulierten, zentralen<br />
RAF-MEK-ERK-Signalkette. Andererseits ist<br />
CNK selbst ein Substrat dieser Signalkette,<br />
<strong>BIOspektrum</strong> | 03.13 | 19. Jahrgang<br />
eine Beobachtung, die auf einen möglichen<br />
Rückkopplungsmechanismus hinweist. Ziel<br />
unserer aktuellen Studien ist es, Kinase-vermittelte<br />
Phosphorylierungsereignisse in<br />
CNK umfassend zu entschlüsseln. Moderne<br />
Massenspektrometrie(MS)-basierte<br />
Methoden stellen einen sehr eleganten<br />
Ansatz zur genauen Bestimmung von<br />
in vivo-Phosphorylierungsstellen in <strong>Signalproteinen</strong><br />
sowie zur detaillierten Untersuchung<br />
von Kinase-Substrat-Beziehungen<br />
dar.<br />
Analyse von<br />
Phosphorylierungsmustern<br />
Für die MS-basierte Kartierung von in vivo-<br />
Phosphorylierungsstellen empfiehlt es sich,<br />
das endogen exprimierte Protein in Anwesenheit<br />
von Phosphatase-Inhibitoren affinitätsbasiert<br />
aufzureinigen. Nachfolgend können<br />
eventuell störende Komponenten mittels<br />
einer SDS-PAGE abgetrennt werden. Einen<br />
interessanten Ansatz bietet auch die PhostagTM-SDS-PAGE<br />
[4], mit der phosphorylierte<br />
und nicht phosphorylierte Proteinisoformen<br />
vor der MS-Analyse aufgetrennt werden<br />
können (Abb. 3A). Die Verwendung<br />
eines zweiwertigen Metallionenkomplexes<br />
(Mn2+ -Phos-tag) führt zur spezifischen Bindung<br />
von Phosphomonoester-Dianionen wie<br />
Phosphoserin, Phosphothreonin oder Phosphotyrosin<br />
in Proteinen und damit zur<br />
Retardierung der phosphorylierten Proteinisoformen<br />
während der Gelelektrophorese.<br />
Alternativ bietet sich ein gelfreier Ansatz<br />
an. Das Protein wird hierzu z. B. mit Aceton<br />
präzipitiert und in einem geeigneten<br />
Puffersystem aufgenommen. Nach proteolytischem<br />
Verdau der Proteine in der Gelmatrix<br />
oder in Lösung erfolgt die Analyse<br />
mittels Flüssigkeitschromatografie in direkter<br />
Kopplung mit der Massenspektrometrie<br />
(LC/MS) (Abb. 3A). Die Auswertung der<br />
Daten erfolgt über spezielle Suchalgorithmen.<br />
Fragmentierung zur Lokalisierung<br />
der Phosphatgruppe<br />
Eine umfassende sowie zuverlässige Bestimmung<br />
von Phosphorylierungsstellen beruht<br />
zum einen auf einer möglichst vollständigen<br />
und zum Teil redundanten Sequenzierung<br />
des Proteins durch den Einsatz verschiedener<br />
Proteasen (z. B. Chymotrypsin,<br />
AspN) neben der Standardprotease Trypsin.<br />
Zum anderen ist es erforderlich, positionsspezifische<br />
Fragmentionen in MS/MS-Spektren<br />
zu detektieren, anhand derer Phos-