MALDI-TOF Massenspektrometrie zur Identifzierung klinisch ...
MALDI-TOF Massenspektrometrie zur Identifzierung klinisch ...
MALDI-TOF Massenspektrometrie zur Identifzierung klinisch ...
Sie wollen auch ein ePaper? Erhöhen Sie die Reichweite Ihrer Titel.
YUMPU macht aus Druck-PDFs automatisch weboptimierte ePaper, die Google liebt.
mikrobiologie<br />
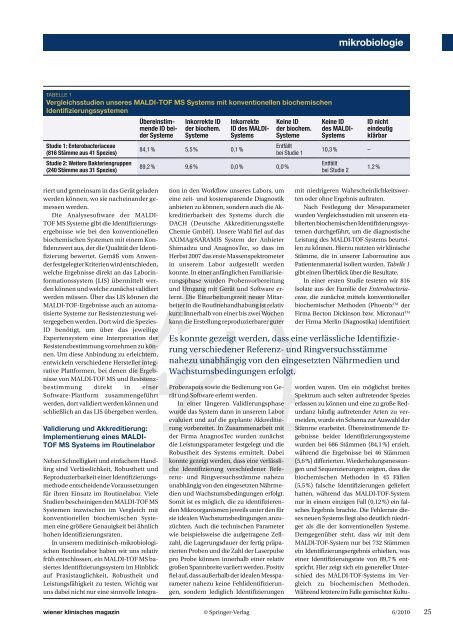
Tabelle 1<br />
Vergleichsstudien unseres <strong>MALDI</strong>-<strong>TOF</strong> MS Systems mit konventionellen biochemischen<br />
Identifizierungssystemen<br />
Studie 1: Enterobacteriaceae<br />
(816 Stämme aus 41 Spezies)<br />
Studie 2: Weitere Bakteriengruppen<br />
(240 Stämme aus 31 Spezies)<br />
worden waren. Um ein möglichst breites<br />
Spektrum auch selten auftretender Spezies<br />
erfassen zu können und eine zu große Redundanz<br />
häufig auftretender Arten zu vermeiden,<br />
wurde ein Schema <strong>zur</strong> Auswahl der<br />
Stämme erarbeitet. Übereinstimmende Ergebnisse<br />
beider Identifizierungssysteme<br />
wurden bei 686 Stämmen (84,1 %) erzielt,<br />
während die Ergebnisse bei 46 Stämmen<br />
(5,6 %) differierten. Wiederholungsmessungen<br />
und Sequenzierungen zeigten, dass die<br />
biochemischen Methoden in 45 Fällen<br />
(5,5 %) falsche Identifizierungen geliefert<br />
hatten, während das <strong>MALDI</strong>-<strong>TOF</strong>-System<br />
nur in einem einzigen Fall (0,12 %) ein falsches<br />
Ergebnis brachte. Die Fehlerrate dieses<br />
neuen Systems liegt also deutlich niedriger<br />
als die der konventionellen Systeme.<br />
Demgegenüber steht, dass wir mit dem<br />
<strong>MALDI</strong>-<strong>TOF</strong>-System nur bei 732 Stämmen<br />
ein Identifi zierungsergebnis erhielten, was<br />
einer Identifizierungsrate von 89,7 % entspricht.<br />
Hier zeigt sich ein genereller Unterschied<br />
des <strong>MALDI</strong>-<strong>TOF</strong>-Systems im Vergleich<br />
zu biochemischen Methoden.<br />
Während letztere im Falle gemischter Kultu<br />
Übereinstimmende<br />
ID beider<br />
Systeme<br />
Inkorrekte ID<br />
der biochem.<br />
Systeme<br />
84,1 % 5,5 % 0,1 %<br />
Inkorrekte<br />
ID des <strong>MALDI</strong>-<br />
Systems<br />
89,2 % 9,6 % 0,0 % 0,0 %<br />
Keine ID<br />
der biochem.<br />
Systeme<br />
Entfällt<br />
bei Studie 1<br />
Keine ID<br />
des <strong>MALDI</strong>-<br />
Systems<br />
10,3 % –<br />
Entfällt<br />
bei Studie 2<br />
ID nicht<br />
eindeutig<br />
klärbar<br />
1,2 %<br />
riert und gemeinsam in das Gerät geladen<br />
werden können, wo sie nacheinander gemessen<br />
werden.<br />
Die Analysesoftware der <strong>MALDI</strong>-<br />
<strong>TOF</strong> MS Systeme gibt die Identifizierungsergebnisse<br />
wie bei den konventionellen<br />
biochemischen Systemen mit einem Konfidenzwert<br />
aus, der die Qualität der Identifizierung<br />
bewertet. Gemäß vom Anwender<br />
festgelegter Kriterien wird entschieden,<br />
welche Ergebnisse direkt an das Laborinformationssystem<br />
(LIS) übermittelt werden<br />
können und welche zunächst validiert<br />
werden müssen. Über das LIS können die<br />
<strong>MALDI</strong>-<strong>TOF</strong>-Ergebnisse auch an automatisierte<br />
Systeme <strong>zur</strong> Resistenztestung weitergegeben<br />
werden. Dort wird die Spezies-<br />
ID benötigt, um über das jeweilige<br />
Expertensystem eine Interpretation der<br />
Resistenzbestimmung vornehmen zu können.<br />
Um diese Anbindung zu erleichtern,<br />
entwickeln verschiedene Hersteller integrative<br />
Plattformen, bei denen die Ergebnisse<br />
von <strong>MALDI</strong>-<strong>TOF</strong> MS und Resistenzbestimmung<br />
direkt in einer<br />
Software-Plattform zusammengeführt<br />
werden, dort validiert werden können und<br />
schließlich an das LIS übergeben werden.<br />
Validierung und Akkreditierung:<br />
Implementierung eines <strong>MALDI</strong>-<br />
<strong>TOF</strong> MS Systems im Routinelabor<br />
Neben Schnelligkeit und einfachem Handling<br />
sind Verlässlichkeit, Robustheit und<br />
Reproduzierbarkeit einer Identifizierungsmethode<br />
entscheidende Voraussetzungen<br />
für ihren Einsatz im Routinelabor. Viele<br />
Studien bescheinigen den <strong>MALDI</strong>-<strong>TOF</strong> MS<br />
Systemen inzwischen im Vergleich mit<br />
konventionellen biochemischen Systemen<br />
eine größere Genauigkeit bei ähnlich<br />
hohen Identifizierungsraten.<br />
In unserem medizinisch-mikrobiologischen<br />
Routinelabor haben wir uns relativ<br />
früh entschlossen, ein <strong>MALDI</strong>-<strong>TOF</strong> MS basiertes<br />
Identifizierungssystem im Hinblick<br />
auf Praxistauglichkeit, Robustheit und<br />
Leistungsfähigkeit zu testen. Wichtig war<br />
uns dabei nicht nur eine sinnvolle Integration<br />
in den Workflow unseres Labors, um<br />
eine zeit- und kostensparende Diagnostik<br />
anbieten zu können, sondern auch die Akkreditierbarkeit<br />
des Systems durch die<br />
1DACH (Deutsche Akkreditierungsstelle<br />
Chemie GmbH). Unsere Wahl fiel auf das<br />
AXIMA@SARAMIS System der Anbieter<br />
Shimadzu und AnagnosTec, so dass im<br />
Herbst 2007 das erste Massenspektrometer<br />
in unserem Labor aufgestellt werden<br />
konnte. In einer anfänglichen Familiarisierungsphase<br />
wurden Probenvorbereitung<br />
und Umgang mit Gerät und Software erlernt.<br />
Die Einarbeitungszeit neuer Mitarbeiter<br />
in die Routinehandhabung ist relativ<br />
kurz: Innerhalb von einer bis zwei Wochen<br />
kann die Erstellung reproduzierbarer guter<br />
Probenspots sowie die Bedienung von Gerät<br />
und Software erlernt werden.<br />
In einer längeren Validierungsphase<br />
wurde das System dann in unserem Labor<br />
evaluiert und auf die geplante Akkreditierung<br />
vorbereitet. In Zusammenarbeit mit<br />
der Firma AnagnosTec wurden zunächst<br />
die Leistungsparameter festgelegt und die<br />
Robustheit des Systems ermittelt. Dabei<br />
konnte gezeigt werden, dass eine verlässliche<br />
Identifizierung verschiedener Referenz-<br />
und Ringversuchsstämme nahezu<br />
unabhängig von den eingesetzten Nährmedien<br />
und Wachstumsbedingungen erfolgt.<br />
Somit ist es möglich, die zu identifizierenden<br />
Mikroorganismen jeweils unter den für<br />
sie idealen Wachstumsbedingungen anzuzüchten.<br />
Auch die technischen Parameter<br />
wie beispielsweise die aufgetragene Zellzahl,<br />
die Lagerungsdauer der fertig präparierten<br />
Proben und die Zahl der Laserpulse<br />
pro Probe können innerhalb einer relativ<br />
großen Spannbreite variiert werden. Positiv<br />
fiel auf, dass außerhalb der idealen Messparameter<br />
nahezu keine Fehlidentifizierungen,<br />
sondern lediglich Identifizierungen<br />
mit niedrigeren Wahrscheinlichkeitswerten<br />
oder ohne Ergebnis auftraten.<br />
Nach Festlegung der Messparameter<br />
wurden Vergleichsstudien mit unseren etablierten<br />
biochemischen Identifizierungssystemen<br />
durchgeführt, um die diagnostische<br />
Leistung des <strong>MALDI</strong>-<strong>TOF</strong>-Systems beurteilen<br />
zu können. Hierzu nutzten wir <strong>klinisch</strong>e<br />
Stämme, die in unserer Laborroutine aus<br />
Patientenmaterial isoliert wurden. Tabelle 1<br />
gibt einen Überblick über die Resultate.<br />
In einer ersten Studie testeten wir 816<br />
Isolate aus der Familie der Enterobacteriaceae,<br />
die zunächst mittels konventioneller<br />
biochemischer Methoden (Phoenix TM der<br />
Firma Becton Dickinson bzw. Micronaut TM<br />
der Firma Merlin Dia gnostika) identifiziert<br />
Es konnte gezeigt werden, dass eine verlässliche Identifizierung<br />
verschiedener Referenz- und Ringversuchsstämme<br />
nahezu unabhängig von den eingesetzten Nährmedien und<br />
Wachstumsbedingungen erfolgt.<br />
wiener <strong>klinisch</strong>es magazin © Springer-Verlag<br />
6/2010 25