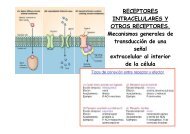
Historia y desarrollo del concepto de cuasiespecies ... - Severo Ochoa
Historia y desarrollo del concepto de cuasiespecies ... - Severo Ochoa
Historia y desarrollo del concepto de cuasiespecies ... - Severo Ochoa
You also want an ePaper? Increase the reach of your titles
YUMPU automatically turns print PDFs into web optimized ePapers that Google loves.
J. Bergadá y J. Jornet), actualmente Pfizer Olot, que suministraron<br />
muestras <strong><strong>de</strong>l</strong> virus. Para los experimentos<br />
con el virus <strong>de</strong> la gripe, colaboramos y obtuvimos muestras<br />
<strong><strong>de</strong>l</strong> entonces Centro Nacional <strong>de</strong> Microbiología,<br />
Virología e Inmunología Sanitarias <strong>de</strong> Majadahonda,<br />
Madrid, actualmente Instituto <strong>de</strong> Salud Carlos III (Rafael<br />
Nájera y Cecilio López-Galín<strong>de</strong>z). Los resultados<br />
con estos dos virus animales confirmaron la dinámica<br />
<strong>de</strong> las <strong>cuasiespecies</strong>, tal como se había observado con el<br />
bacteriófago Qβ y fueron publicados al inicio <strong>de</strong> la década<br />
<strong>de</strong> 1980 [4,22,23,26] .<br />
Cabe <strong>de</strong>stacar que estos primeros estudios generalizaron<br />
la aplicabilidad <strong><strong>de</strong>l</strong> <strong>concepto</strong> <strong>de</strong> <strong>cuasiespecies</strong> en<br />
Virología antes <strong>de</strong> que existieran las herramientas <strong>de</strong><br />
clonaje molecular y <strong>de</strong> secuenciación <strong>de</strong> las que disponemos<br />
ahora. A<strong>de</strong>más, en el caso <strong><strong>de</strong>l</strong> FMDV, se <strong>de</strong>scribió<br />
la coexistencia <strong>de</strong> dos virus (que presentaban un<br />
solo cambio en el patrón <strong>de</strong> huellas dactilares T1) en<br />
el mismo animal enfermo, lo que constituyó la primera<br />
evi<strong>de</strong>ncia <strong>de</strong> <strong>cuasiespecies</strong> in vivo [4] [Figura 5]. (Como<br />
relato histórico <strong>de</strong> estos resultados y sus implicaciones<br />
<strong>de</strong>s<strong>de</strong> el punto <strong>de</strong> vista actual, véanse las referencias<br />
[3,10] ). Merece la pena <strong>de</strong>stacar también unas frases<br />
<strong>de</strong> la discusión <strong><strong>de</strong>l</strong> trabajo <strong>de</strong> Nieves Villanueva (entonces<br />
posdoctoral en el Centro <strong>de</strong> Biología Molecular,<br />
y previamente a su incorporación a Abelló, S. A. y<br />
luego al Centro Nacional <strong>de</strong> Microbiología, Virología<br />
e Inmunología Sanitarias <strong>de</strong> Majadahonda) en el que<br />
se <strong>de</strong>scribió el clonaje molecular y secuenciación <strong><strong>de</strong>l</strong><br />
gen que codifica la proteína VP1, proteína antigénica<br />
principal <strong>de</strong> la cápsida <strong><strong>de</strong>l</strong> virus [28] Figura 5. La primera<br />
evi<strong>de</strong>ncia <strong>de</strong> la<br />
presencia <strong>de</strong> dos<br />
mutantes distintos en<br />
un animal infectado<br />
por el virus <strong>de</strong> la<br />
fiebre aftosa. A y B<br />
son las huellas<br />
dactilares <strong><strong>de</strong>l</strong> ARN <strong>de</strong><br />
dos clones biológicos<br />
aislados <strong>de</strong> un mismo<br />
animal. Los<br />
oligonucleótidos 35 y<br />
35’ difieren en su<br />
posición <strong>de</strong>bido a una<br />
mutación puntual. En<br />
C se representa la<br />
huella <strong>de</strong> una mezcla<br />
<strong>de</strong> los dos ARNs<br />
(Basado en<br />
:<br />
[4] y presentada<br />
en la memoria<br />
<strong>de</strong> toma <strong>de</strong> posesión<br />
<strong>de</strong> Esteban Domingo<br />
como Miembro<br />
Numerario <strong>de</strong> la Real<br />
Aca<strong>de</strong>mia <strong>de</strong><br />
Ciencias Exactas,<br />
Físicas y Naturales [3] ).<br />
Virología | Volumen 16 - Número 1/2013<br />
Artículo original: <strong>concepto</strong> <strong>de</strong> <strong>cuasiespecies</strong> en Virología<br />
7 <br />
Las secuencias <strong>de</strong> nucleótido <strong><strong>de</strong>l</strong> ADN<br />
complementario clonado <strong>de</strong> FMDV representan<br />
una o unas pocas moléculas <strong>de</strong> ARN<br />
<strong>de</strong> entre las presentes en los preparados <strong>de</strong><br />
virus (...) De las estimaciones <strong>de</strong> las frecuencias<br />
<strong>de</strong> mutación para virus ARN, parece<br />
muy improbable que puedan prepararse<br />
microgramos <strong>de</strong> ARN vírico sin una proporción<br />
significativa <strong>de</strong> moléculas variantes.<br />
En el presente estudio, el virus ha sido pasado<br />
en cultivos celulares tan poco como ha<br />
sido posible porque, cuando se realizan 30<br />
pases <strong><strong>de</strong>l</strong> FMDV en cultivos celulares a baja<br />
multiplicidad <strong>de</strong> infección, se incorporan al<br />
azar entre 14 y 57 mutaciones por genoma;<br />
y la población resultante está formada por<br />
reservas <strong>de</strong> mutantes infecciosos (…) A<br />
pesar <strong>de</strong> la heterogeneidad genética, cada<br />
secuencia clonada estará estrechamente relacionada<br />
con los genomas que fluctúan en<br />
la población natural original. En el caso <strong>de</strong><br />
la VP1, la <strong>de</strong>terminación <strong>de</strong> su secuencia<br />
ayudará a establecer correlaciones entre la<br />
estructura <strong>de</strong> la proteína y su comportamiento<br />
antigénico.<br />
Previamente a la <strong>de</strong>mostración <strong>de</strong> la existencia <strong>de</strong> <strong>cuasiespecies</strong><br />
in vivo, la variabilidad genética <strong>de</strong> los virus ya<br />
estaba bien establecida, en particular con los estudios con<br />
el virus <strong>de</strong> la gripe para el que ya se habían <strong>de</strong>finido los<br />
<strong>concepto</strong>s <strong>de</strong> «<strong>de</strong>riva antigénica» y <strong>de</strong> «salto antigénico».<br />
A este respecto, es bien conocida la frase <strong>de</strong> Edwin<br />
D. Kilbourne [20] :<br />
La gripe, ya <strong>de</strong>s<strong>de</strong> 1918 ha sido una enfermedad<br />
notablemente constante –una enfermedad<br />
invariable, tres días <strong>de</strong> fiebre– causada<br />
por un virus variable.<br />
Sin embargo, aunque se sabía que la variación afectaba a<br />
la comparación <strong>de</strong> aislados distintos, no se contemplaba<br />
la heterogeneidad «<strong>de</strong>ntro» <strong>de</strong> un mismo aislado, sugerida<br />
por la tasa <strong>de</strong> error y frecuencias <strong>de</strong> mutación <strong>de</strong>terminadas<br />
para el bacteriófago Qβ, el FMDV y el virus <strong>de</strong><br />
la gripe. De hecho, tanto en monografías <strong>de</strong> la época<br />
como en los planes <strong>de</strong> trabajo <strong>de</strong> las compañías farmacéuticas<br />
en los inicios <strong>de</strong> los años 80, se hacían planes<br />
para clonar cada una <strong>de</strong> las hemaglutininas existentes <strong><strong>de</strong>l</strong><br />
virus <strong>de</strong> la gripe o cada una <strong>de</strong> las VP1 <strong><strong>de</strong>l</strong> FMDV. El <strong>concepto</strong><br />
prevalente en la época era que, una vez diagnosticado<br />
el virus circulante en las primeras fases <strong>de</strong> un brote<br />
<strong>de</strong> enfermedad, se acudiría al <strong>de</strong>pósito <strong>de</strong> genes <strong>de</strong> hemaglutinina<br />
o <strong>de</strong> VP1 para expresar el que correspondía y<br />
obtener, <strong>de</strong> este modo, la vacuna a<strong>de</strong>cuada. Todo esto<br />
suena hoy tremendamente ingenuo, pero lo viví directa-