Les parasites du genre Perkinsus infectant les mollusques : focus ...
Les parasites du genre Perkinsus infectant les mollusques : focus ...
Les parasites du genre Perkinsus infectant les mollusques : focus ...
Create successful ePaper yourself
Turn your PDF publications into a flip-book with our unique Google optimized e-Paper software.
<strong>Les</strong> <strong>parasites</strong> <strong>du</strong> <strong>genre</strong> <strong>Perkinsus</strong> <strong>infectant</strong> <strong>les</strong><br />
<strong>mollusques</strong> :<br />
<strong>focus</strong> sur <strong>les</strong> bénitiers de Polynésie française<br />
Plan de la présentation<br />
1-Contexte<br />
2-Objectifs de l’étude<br />
3-Résultats obtenus<br />
4-Conclusions /Perspectives<br />
D. Saulnier et al., Séminaire « Bénitier »<br />
UPF, 1 er mars 2013<br />
1
1-Contexte<br />
1-Infection à Bonamia ostreae :<br />
2-Infection à Bonamia exitiosa :<br />
3-Infection à Marteilia refringens :<br />
4-Infection à <strong>Perkinsus</strong> marinus :<br />
5-Infection à <strong>Perkinsus</strong> olseni :<br />
6-Infection à Candidatus<br />
Xenohaliotis californiensis :<br />
7-Infection à un pseudo-Herpès :<br />
Santé des Mollusques<br />
Infections à déclaration obligatoire<br />
OIE Code aquatique 2011<br />
et espèces sensib<strong>les</strong><br />
Ostrea e<strong>du</strong>lis, O. angasi, O. puelchana, O. chilensis, O.<br />
denselammellosa, Crassostrea ariakensis (6 espèces, 2 <strong>genre</strong>s)<br />
Ostrea chilensis, O. angasi, O. e<strong>du</strong>lis (3 espèces, 1 <strong>genre</strong>)<br />
Ostrea e<strong>du</strong>lis, O. angasi, O.puelchana, O. chilensis, Mytilus e<strong>du</strong>lis,<br />
M.galloprovincialis (6 espèces, 2 <strong>genre</strong>s)<br />
Crassostrea gigas, C. avirginica, C. ariakensis, C. rhizophorae, C.<br />
corteziensis, Mya arenaria, Macoma balthica (7 espèces, 3 <strong>genre</strong>s)<br />
Austrovenus stutchburyi, Tridacna maxima, T. crocea, Pitar rostrata,<br />
Ruditapes philippinarum, R. decussatus, Haliotis rubra, H. laevigata,<br />
H. cyclobates, H. scalaris, Anadara trapezia, Crassostrea gigas, C.<br />
ariakensis, C. sikamea, Pinctada maxima, P. fucata, P. sugillata, P.<br />
martensii (18 espèces, 10 <strong>genre</strong>s)<br />
Haliotis cracherodii, H. sorenseni, H. rufescens, H. tuberculata, H.<br />
corrugata, H. fulgens, H. wallalensis, H. discus-hannai, H. diversicolor<br />
supertexta (9 espèces, 1 <strong>genre</strong>)<br />
Haliotis diversicolor, H. laevegata, H. rubra, hybrides H. laevegata x H.<br />
rubra (3 espèces, 1 <strong>genre</strong>s)
Impact de maladies infectieuses chez <strong>les</strong> huîtres cultivées de<br />
France métropolitaine
Cycle parasitaire de <strong>Perkinsus</strong> olseni chez la palourde R. philippinarum<br />
D’après Choi et al. 2005<br />
50 µm<br />
Hypnospores de P. olseni dans <strong>les</strong><br />
branchies de l’huître perlière Pinctada<br />
fucata, d’après Sanil et al. 2009<br />
Trophozoites<br />
dans le tissus<br />
conjonctif <strong>du</strong><br />
manteau<br />
(histologie)
Impact de <strong>Perkinsus</strong> olseni sur la santé des espèces sensib<strong>les</strong><br />
1-Croissance des <strong>mollusques</strong> ralentie<br />
2-Effort de repro<strong>du</strong>ction ré<strong>du</strong>it<br />
3-Apparition de nécroses tissulaires /<br />
réactions inflammatoires / no<strong>du</strong><strong>les</strong><br />
4-Episodes de mortalité<br />
Selon Sanil et al, 2009, P. olseni serait responsable <strong>du</strong> déclin de P. fucata dans le<br />
golfe de Mannar en Inde (côte sud-est).<br />
Plus <strong>les</strong> niveaux d’infestation parasitaire sont importants (charge<br />
parasitaire) et plus l’impact de P. olseni sur la santé des espèces<br />
sensib<strong>les</strong> est grand.<br />
Evolution de l’index de développement<br />
gonadique (GSI) chez la palourde R.<br />
philippinarum (Corée) selon la saison et la<br />
charge parasitaire, Park et al. 2006
Carte de répartition des différentes espèces de <strong>Perkinsus</strong><br />
<strong>Les</strong> risques de dissémination <strong>du</strong> parasite<br />
(d’après Petty et al, 2010)<br />
-Transferts d’animaux de zones impactées vers des zones indemnes <strong>du</strong> fait<br />
principalement des activités aquaco<strong>les</strong><br />
-Intervention de vecteurs de transmission : eaux de ballast des navires<br />
commerciaux par exemple, prédateurs, importation de <strong>mollusques</strong> pour la<br />
consommation humaine, pratiques liées à l’aquariophilie (Sheppard et al ,2008)<br />
“Importation policies regarding importation of T. crocea into the USA,<br />
6<br />
… need to be reexamined ».
2-Objectifs de l’étude<br />
Le projet IBENI intègre un volet zoosanitaire qui consiste à évaluer plusieurs<br />
méthodes de diagnostic moléculaire permettant la détection de <strong>Perkinsus</strong> olseni,<br />
un agent infectieux d’importance zoo sanitaire reconnue.<br />
Ce parasite est même à déclaration obligatoire auprès de l’OIE. Il est de plus<br />
réputé capable d’infester des <strong>mollusques</strong> <strong>du</strong> <strong>genre</strong> Tridacna et Pinctada présents<br />
en Polynésie française.<br />
Il était important de savoir si cet agent infectieux était présent ou non en<br />
Polynésie française afin de l’intégrer dans la gestion des transferts de <strong>mollusques</strong><br />
entre zones géographiques indemnes ou pas.<br />
<strong>Les</strong> outils diagnostics utilisés lors de cette étude ont été validés à partir de<br />
prélèvements tissulaires de bénitiers provenant de différentes î<strong>les</strong> de Polynésie<br />
française.<br />
7
Origine des bénitiers et nature des prélèvements tissulaires<br />
D'après Choi et al, 2010 il existe une forte corrélation entre <strong>les</strong> charges parasitaires dans un<br />
organe particulier et la charge parasitaire dans l'animal entier<br />
Branchies, masse viscérale ou manteau (palourde)<br />
8
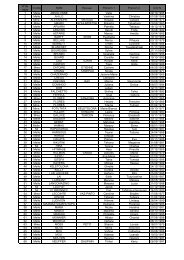
Origine Tahiti Tatakoto Gambier Tubuai Hao Mopelia Manihi Takaroa Mataiva Aratika<br />
Traitement indivi<strong>du</strong>el pool pool pool pool pool pool pool pool pool<br />
Nombre<br />
d’échant./<br />
pool<br />
Traitement des échantillons de tissus pour l’extraction d’ADN<br />
/ 12 12 10 11 10 10 10 10 10 10 10 10 10 10 10 10 10 10 10 10 10<br />
228 bénitiers, 24 extraits ADNs
Evaluation / développement de méthodes diagnostiques,<br />
par biologie moléculaire<br />
Méthodes de référence décrites dans le Manuel de l’OIE se déroulant en deux étapes,<br />
utilisant la technique d’amplification en chaîne par la polymérase(PCR).<br />
1 ère PCR ciblant la région ITS de l’ARN ribosomal de toutes <strong>les</strong> espèces de <strong>Perkinsus</strong>,<br />
excepté P. qugwadi,<br />
2 ème PCR ciblant la région ITS de l’ARN ribosomal de <strong>Perkinsus</strong> olseni uniquement,<br />
la sensibilité de cette méthode est grande puisque 1 cellule parasitaire dans 30mg<br />
de tissus total peut être détectée.<br />
Une deuxième méthode diagnostique, par PCR en temps réel (qPCR), non décrite dans la<br />
littérature scientifique, a dans un deuxième temps été développée au LEP afin de pouvoir<br />
détecter et quantifier <strong>les</strong> niveaux d’infestation <strong>du</strong> bénitier par P. olseni.
3-Résultats obtenus<br />
De l’ADN de <strong>Perkinsus</strong> sp. et de P. olseni est détecté en PCR classique (+)<br />
Origine Identification des PCR de détection d’ADN PCR de détection d’ADN de<br />
échantillons <strong>du</strong> <strong>genre</strong> <strong>Perkinsus</strong> <strong>Perkinsus</strong> olseni<br />
B1 + +<br />
TAHITI<br />
B2 + -<br />
B3 + +<br />
TATAKOTO<br />
Pool 1<br />
Pool 2<br />
-<br />
-<br />
-<br />
+<br />
GAMBIER<br />
Pool 3<br />
Pool 4<br />
+<br />
-<br />
+<br />
-<br />
Pool 5 + -<br />
TUBUAI<br />
Pool 6 + -<br />
Pool 7 + +<br />
HAO<br />
Pool 8<br />
Pool 9<br />
+<br />
+<br />
+<br />
+<br />
MOPELIA<br />
Pool 10<br />
Pool 11<br />
-<br />
-<br />
+<br />
+<br />
Pool 12 - -<br />
MANIHI<br />
Pool 13 - +<br />
Pool 14 - -<br />
Pool 15 - -<br />
TAKAROA<br />
Pool 16 - +<br />
Pool 17 + +<br />
MATAIVA<br />
Pool 18<br />
Pool 19<br />
-<br />
-<br />
-<br />
+<br />
ARATIKA<br />
Pool 20<br />
Pool 21<br />
-<br />
-<br />
-<br />
-<br />
Bilan 24 échantillons 10+/24 = 42% 13+/24 = 54%<br />
11
De l’ADN de P. olseni est détecté par PCR en temps réel et la cible peut être quantifiée<br />
- Entre 20 et 20 millions de copies <strong>du</strong> gène ITS de P. olseni présents dans un<br />
échantillon tissulaire à analyser sont quantifiab<strong>les</strong>, <strong>du</strong> fait de l’excellente efficacité<br />
de la réaction PCRq, proche de 100%.<br />
- Seu<strong>les</strong> 20 copies <strong>du</strong> gène ITS de <strong>Perkinsus</strong> olseni sont détectab<strong>les</strong> par PCRq.<br />
Efficacité de 90.5 %<br />
12
Résultats de la PCRq<br />
Echantillon Origine<br />
Quantification (nb de copies/mg de<br />
tissu analysé)<br />
B1<br />
32<br />
B2<br />
Tahiti<br />
B3 9<br />
Pool 1<br />
Pool 2<br />
Tatakoto<br />
Pool 3<br />
Pool 4<br />
Gambier<br />
Pool 5<br />
63<br />
Pool 6<br />
Pool 7<br />
Tubuai<br />
Pool 8<br />
Pool 9<br />
Hao<br />
1,8E+05<br />
1,0E+04<br />
Pool 10<br />
Pool 11<br />
Pool 12<br />
Mopelia<br />
Pool 13<br />
Pool 14<br />
Pool 15<br />
Manihi<br />
5<br />
Pool 16<br />
Pool 17<br />
Takaroa<br />
Pool 18<br />
Pool 19<br />
Mataiva<br />
341<br />
Pool 20<br />
Pool 21<br />
Aratika
Diagnostic de confirmation de la présence de <strong>Perkinsus</strong> en Polynésie<br />
française<br />
clonage des pro<strong>du</strong>its PCR obtenues avec <strong>les</strong> amorces spécifiques <strong>du</strong> <strong>genre</strong> <strong>Perkinsus</strong> ,<br />
à partir des échantillons de bénitier B1, B2 et B3<br />
Séquençage de 1 à 2 clones recombinants /origine de bénitier = 4 séquences,<br />
Alignement des séquences ITS,<br />
Analyse phylogénétique par l’algorithme NJM, 615 sites nucléotidiques analysées.<br />
0.005<br />
92<br />
97<br />
100<br />
P4 bénitier B2<br />
P1 Bénitier B1<br />
<strong>Perkinsus</strong> mediterraneus isolate11<br />
P6 bénitier B3<br />
<strong>Perkinsus</strong> honshuensis isolate Mie3<br />
P2 Bénitier B1<br />
<strong>Perkinsus</strong> olseni isolate Mie13v<br />
<strong>Perkinsus</strong> marinus isolate Kino0806
4-Conclusions / Perspectives<br />
Démonstration de la présence de <strong>Perkinsus</strong> olseni et <strong>Perkinsus</strong> mediterraneus infestant<br />
le bénitier en Polynésie française.<br />
Démonstration de la capacité d’un laboratoire (LEP) présent en Polynésie française à<br />
pouvoir diagnostiquer P. olseni, 1 des 7 agents responsable d’une maladie chez <strong>les</strong><br />
Mollusques à déclaration obligatoire auprès de l’OIE, par des outils moléculaires.<br />
Développement d’un test de diagnostic quantitatif (PCRq) des infestations à P. olseni<br />
très sensible : 20 copies/mg de tissus de bénitier, et rapide à mettre en œuvre : moins<br />
de 3 heures à partir d’extraits ADNs.<br />
PCRq : outil diagnostic d’intérêt pour <strong>les</strong> futures enquêtes d’intérêt épidémiologique et<br />
l’évaluation de l’impact de la Perkinsiose à P. olseni sur <strong>les</strong> <strong>mollusques</strong> de Polynésie<br />
française. En effet, d’après la littérature, plus <strong>les</strong> niveaux d’infestation parasitaire sont<br />
importants (charge parasitaire) et plus l’impact de P. olseni sur la santé des espèces<br />
sensib<strong>les</strong> est grand.
Existe-t-il d’autres <strong>mollusques</strong> que le bénitier, présents en Polynésie française, pouvant être<br />
infestés par P. olseni : Pinctada margaritifera en particulier?<br />
Quelle est la répartition géographique de P. olseni à l’échèle des nombreuses î<strong>les</strong> de Polynésie<br />
française ?<br />
Quelle est la relation entre la prévalence à P. olseni / niveaux d’infestation parasitaires et la<br />
survenue d’épisodes de mortalité pouvant ou ayant pu survenir dans <strong>les</strong> cheptels de bénitier<br />
présents en Polynésie française ?<br />
Quel est l’impact des co-infections <strong>Perkinsus</strong> olseni / P. mediterraneus sur la santé des<br />
cheptels de bénitier ?<br />
Y a-t-il d’autres espèces de <strong>Perkinsus</strong> également présentes en Polynésie française :<br />
P. marinus notamment ?<br />
Peut-on visualiser en histologie <strong>les</strong> <strong>parasites</strong> <strong>du</strong> <strong>genre</strong> <strong>Perkinsus</strong> ? sachant que la totalité des<br />
échantillons analysés par biologie moléculaire lors de cette étude ont également été traités, et<br />
à un niveau indivi<strong>du</strong>el, afin de permettre des analyses ultérieures en histologie.<br />
Possibilité de traiter en indivi<strong>du</strong>el certains lots de bénitiers trouvés fortement infectés par P.<br />
olseni lors de cette étude et analysés en pool, par ex ceux dont l’origine est Hao.<br />
Valorisation des travaux et analyses déjà réalisés sous forme d’une publication dans une revue<br />
à comité de lecture.
Remerciements<br />
L’essentiel <strong>du</strong> travail de laboratoire a été effectué par Hélène Boulet en CDD à mi temps<br />
à l’U. de Polynésie française (10 sept - 10 déc. 2012) dans le cadre <strong>du</strong> projet IBENI et<br />
accueilli au sein <strong>du</strong> Laboratoire Ecosystème Perlicole (LEP) de l’Ifremer Tahiti.<br />
Cette étude a bénéficié de l’aide d’agents de la DRM et de l’IRSN : campagnes<br />
d’échantillonnage en ce qui concerne certains des lots analysées ou/et traitement des<br />
échantillons permettant de différer et grouper <strong>les</strong> analyses (extraction ADN et<br />
histologie).<br />
Maururu roa