Abschlußbericht zum Projekt „Molekularbiologische ... - UOK
Abschlußbericht zum Projekt „Molekularbiologische ... - UOK
Abschlußbericht zum Projekt „Molekularbiologische ... - UOK
Erfolgreiche ePaper selbst erstellen
Machen Sie aus Ihren PDF Publikationen ein blätterbares Flipbook mit unserer einzigartigen Google optimierten e-Paper Software.
Abschlussbericht FKZ UGV04070803084<br />
Durchführung<br />
Zur Testung des Systems wurden 5 bekannte Proben (Aspergillus fumigatus,<br />
Alternaria alternata, Penicillium commune, Cladosporium sphaerospermum und<br />
Aspergillus flavus) mit dem Kit analysiert. Dazu wurden 1,5 ng/µl DNA auf die 36<br />
verschiedenen, für jeden Pilz spezifischen Detektionsansätze des ERMI sm Kit<br />
gegeben und im LightCycler ® 480 System analysiert. Als Positivkontrolle wird ein<br />
Plasmid mit DNA des Organismus Geotrichum candidum mit dem entsprechenden<br />
Detektionsansatz eingesetzt.<br />
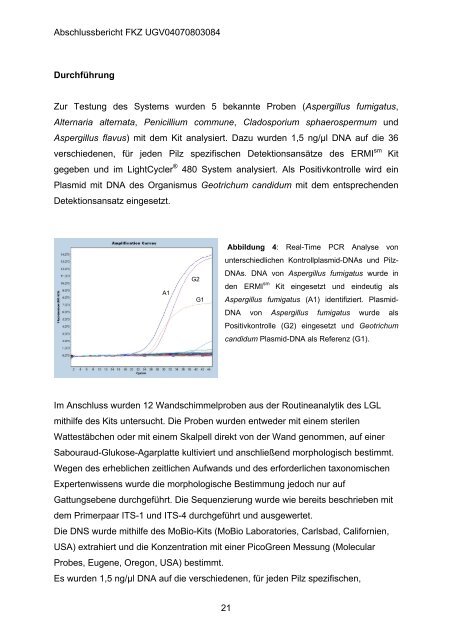
A1<br />
G2<br />
G1<br />
Abbildung 4: Real-Time PCR Analyse von<br />
unterschiedlichen Kontrollplasmid-DNAs und Pilz-<br />
DNAs. DNA von Aspergillus fumigatus wurde in<br />
den ERMI sm Kit eingesetzt und eindeutig als<br />
Aspergillus fumigatus (A1) identifiziert. Plasmid-<br />
DNA von Aspergillus fumigatus wurde als<br />
Positivkontrolle (G2) eingesetzt und Geotrichum<br />
candidum Plasmid-DNA als Referenz (G1).<br />
Im Anschluss wurden 12 Wandschimmelproben aus der Routineanalytik des LGL<br />
mithilfe des Kits untersucht. Die Proben wurden entweder mit einem sterilen<br />
Wattestäbchen oder mit einem Skalpell direkt von der Wand genommen, auf einer<br />
Sabouraud-Glukose-Agarplatte kultiviert und anschließend morphologisch bestimmt.<br />
Wegen des erheblichen zeitlichen Aufwands und des erforderlichen taxonomischen<br />
Expertenwissens wurde die morphologische Bestimmung jedoch nur auf<br />
Gattungsebene durchgeführt. Die Sequenzierung wurde wie bereits beschrieben mit<br />
dem Primerpaar ITS-1 und ITS-4 durchgeführt und ausgewertet.<br />
Die DNS wurde mithilfe des MoBio-Kits (MoBio Laboratories, Carlsbad, Californien,<br />
USA) extrahiert und die Konzentration mit einer PicoGreen Messung (Molecular<br />
Probes, Eugene, Oregon, USA) bestimmt.<br />
Es wurden 1,5 ng/µl DNA auf die verschiedenen, für jeden Pilz spezifischen,<br />
21