Agrégation de peptides amyloïdes par des simulations numériques
Agrégation de peptides amyloïdes par des simulations numériques
Agrégation de peptides amyloïdes par des simulations numériques
You also want an ePaper? Increase the reach of your titles
YUMPU automatically turns print PDFs into web optimized ePapers that Google loves.
38 3 Les petits pepti<strong>de</strong>s amyloï<strong>de</strong>s<br />
faiblement déterminé <strong>par</strong> l’énergie totale du système qui décroît seulement <strong>de</strong> 2 kcal/mol<br />
entre les événements 3000 et 4388. La figure 3.7 (d) souligne que les chaînes explorent es-<br />
sentiellement <strong>de</strong>s arrangements anti<strong>par</strong>allèles durant cette trajectoire. Pour conclure, les<br />
<strong>simulations</strong> qui convergent vers un état tétramérique en feuillet β révèlent néanmoins <strong>de</strong>s<br />
différences importantes d’une trajectoire à l’autre (en <strong>par</strong>tant <strong>de</strong> la même conformation<br />
initiale). Par exemple dans les trajectoires R12 et R25, la région proche <strong>de</strong> -40 kcal/mol<br />
est explorée après plus <strong>de</strong> 5000 événements, certaines chaînes alternent entre une orienta-<br />
tion <strong>par</strong>allèle ou anti<strong>par</strong>allèle et l’intermédiaire oligomérique qui précè<strong>de</strong> la formation du<br />
tétramère natif n’est pas un double dimère mais une conformation trimère-monomère.<br />
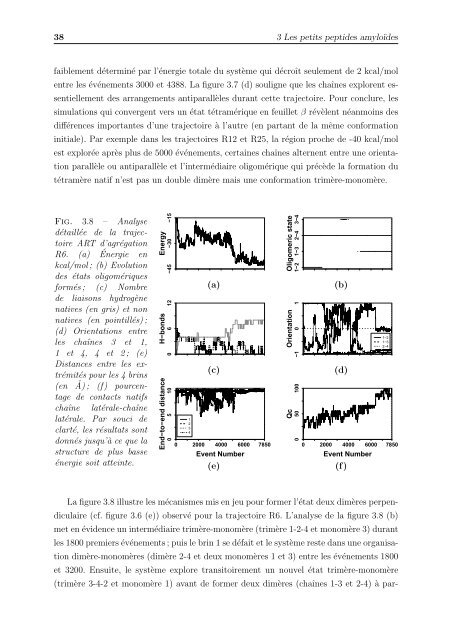
Fig. 3.8 – Analyse<br />
détaillée <strong>de</strong> la trajectoire<br />
ART d’agrégation<br />
R6. (a) Énergie en<br />
kcal/mol ; (b) Evolution<br />
<strong>de</strong>s états oligomériques<br />
formés ; (c) Nombre<br />
<strong>de</strong> liaisons hydrogène<br />
natives (en gris) et non<br />
natives (en pointillés) ;<br />
(d) Orientations entre<br />
les chaînes 3 et 1,<br />
1 et 4, 4 et 2 ; (e)<br />
Distances entre les extrémités<br />
pour les 4 brins<br />
(en ˚ A) ; (f) pourcentage<br />
<strong>de</strong> contacts natifs<br />
chaîne latérale-chaîne<br />
latérale. Par souci <strong>de</strong><br />
clarté, les résultats sont<br />
donnés jusqu’à ce que la<br />
structure <strong>de</strong> plus basse<br />
énergie soit atteinte.<br />
Energy<br />
−45 −30 −15<br />
H−bonds<br />
0 6 12<br />
End−to−end distance<br />
0 5 10<br />
1<br />
2<br />
3<br />
4<br />
Oligomeric state<br />
1−2 1−3 2−4 3−4<br />
(a) (b)<br />
Orientation<br />
−1 0 1<br />
(c) (d)<br />
0 2000 4000 6000 7850<br />
Event Number<br />
Qc<br />
0 50 100<br />
(e) (f)<br />
Event Number<br />
1−2<br />
1−3<br />
2−4<br />
0 2000 4000 6000 7850<br />
La figure 3.8 illustre les mécanismes mis en jeu pour former l’état <strong>de</strong>ux dimères perpen-<br />
diculaire (cf. figure 3.6 (e)) observé pour la trajectoire R6. L’analyse <strong>de</strong> la figure 3.8 (b)<br />
met en évi<strong>de</strong>nce un intermédiaire trimère-monomère (trimère 1-2-4 et monomère 3) durant<br />
les 1800 premiers événements ; puis le brin 1 se défait et le système reste dans une organisa-<br />
tion dimère-monomères (dimère 2-4 et <strong>de</strong>ux monomères 1 et 3) entre les événements 1800<br />
et 3200. Ensuite, le système explore transitoirement un nouvel état trimère-monomère<br />
(trimère 3-4-2 et monomère 1) avant <strong>de</strong> former <strong>de</strong>ux dimères (chaînes 1-3 et 2-4) à <strong>par</strong>-