Biochemie Praktikum I
Biochemie Praktikum I
Biochemie Praktikum I
Erfolgreiche ePaper selbst erstellen
Machen Sie aus Ihren PDF Publikationen ein blätterbares Flipbook mit unserer einzigartigen Google optimierten e-Paper Software.
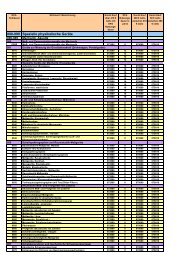
<strong>Praktikum</strong>sskript <strong>Biochemie</strong> für Biologen-Bachelor SS2012<br />
SDS-Elektrophorese<br />
SDS-Elektrophorese<br />
Neben der Gelfiltration wird die Polyacrylamid-Gel-Electrophorese (PAGE) in SDS<br />
(Natriumdodecylsulfat) zur Molekulargewichtsbestimmung von Proteinen benutzt. Zur Trennung<br />
von Proteinen im Polyacrylamidgel wird einerseits der Siebeffekt des Gels, zum anderen die<br />
Ladung der Proteine ausgenutzt. Für die Molekulargewichtsbestimmung in der PAGE muss die<br />
unterschiedliche individuelle Ladung (stammt von den geladenen Aminosäureseitenketten des<br />
jeweiligen Proteins) der Proteine ausgeschaltet werden, damit die elektrophoretische Trennung<br />
allein von der Größe des Moleküls abhängt. Dies wird durch Verwendung des Detergens SDS<br />
erreicht. Durch Bindung von SDS werden Proteine:<br />
1. denaturiert, d.h. von der nativen Struktur zu einer frei beweglichen Kette<br />
entfaltet und<br />
2. durch Anlagerung des hydrophoben Teils des SDS an hydrophobe Bereich der Proteine<br />
wird dieses mit negativen Ladungen beladen, d.h. zu Polyanionen.<br />
+SDS<br />
natives Protein<br />
SDS-beladenes entfaltetes Protein<br />
SDS (Sodiumdodecylsulfate): Na + SO 4 - -CH 2 -(CH 2 ) 10 -CH 3<br />
Da Proteine mit Disulfidbrücken in der SDS-PAGE häufig ein anormales Verhalten zeigen,<br />
werden die Disulfidbrücken vor der Elektrophorese mit β-Mercaptoethanol reduziert. Die<br />
Vorinkubation mit SDS und Mercaptoethanol schafft die Vorrausetzung für die Molekulargewichtbestimmung<br />
im SDS-Gel, da jetzt die Beweglichkeit der Proteine nur eine Funktion der<br />
Größe des Moleküls darstellt.<br />
Für die Molekulargewichtsbestimmung wird die relative Beweglichkeit von Proteinen mit<br />
bekanntem Molekulargewicht in Bezug auf einen internen Standard (meist Bromphenolblau)<br />
verglichen und eine Eichkurve aufgestellt, indem der Logarithmus des Molekulargewichts gegen<br />
den Rf-Wert (relative Beweglichkeit) aufgetragen wird. Aus dem Rf Wert des unbekannten<br />
Proteins kann somit dessen Molekulargewicht ermittelt werden.<br />
24