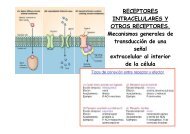
Alineamiento de secuencias biológicas (pdf).
Alineamiento de secuencias biológicas (pdf).
Alineamiento de secuencias biológicas (pdf).
You also want an ePaper? Increase the reach of your titles
YUMPU automatically turns print PDFs into web optimized ePapers that Google loves.
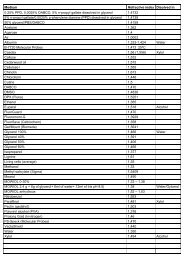
Reducción <strong>de</strong> la Complejidad AlgorítmicaOptimización para reducir tiempos <strong>de</strong> cálculoTabla <strong>de</strong> Dispersión (Hash)Lista con las posiciones <strong>de</strong> lossímbolos <strong>de</strong> la secuenciapos : 1234 5678 901seqX : TCAG ACGA TTG n=11Tabla Hash (seqX)A 3, 5, 8C 2, 6G 4, 7, 11T 1, 9, 10Secuencia a compararpos : 1234567890seqY: ATCGGAGCTG m=10Acumular i<strong>de</strong>ntida<strong>de</strong>s en cada diagonal(d= h - v, si x halínea con y v)y 1(A) en d 2(3-1), d 4(5-1) y d 7(8-1)y 2(T) en d -1(1-2), d 7(9-2) y d 8(10-2)Complejidad : Cada elemento <strong>de</strong> sY vs el número <strong>de</strong> elementos hash para ese símboloO(N*media) media=M / LongTabla (N, M long <strong>de</strong> SeqX y SeqY)k-tuplas : [+] entradas (L=lAlf k ) -> [-] elementos por entradaProteinas : lAlf=20, Si k=2 => L= 20 2 = 400 entradas.Long. Proteina N=400 -> 1 valor por entrada => complejidad O(N+M)Paulino Gomez-PuertasBioinformática.