Estudio retrospectivo de 1.193 componentes monoclonales ...
Estudio retrospectivo de 1.193 componentes monoclonales ...
Estudio retrospectivo de 1.193 componentes monoclonales ...
Create successful ePaper yourself
Turn your PDF publications into a flip-book with our unique Google optimized e-Paper software.
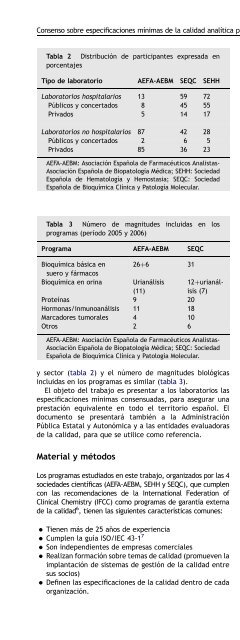
ARTICLE IN PRESSConsenso sobre especificaciones mínimas <strong>de</strong> la calidad analítica para magnitu<strong>de</strong>s hematológicas y <strong>de</strong> bioquímica especial 89Tabla 2 Distribución <strong>de</strong> participantes expresada enporcentajesTipo <strong>de</strong> laboratorio AEFA-AEBM SEQC SEHHLaboratorios hospitalarios 13 59 72Públicos y concertados 8 45 55Privados 5 14 17Laboratorios no hospitalarios 87 42 28Públicos y concertados 2 6 5Privados 85 36 23AEFA-AEBM: Asociación Española <strong>de</strong> Farmacéuticos Analistas-Asociación Española <strong>de</strong> Biopatología Médica; SEHH: SociedadEspañola <strong>de</strong> Hematología y Hemostasia; SEQC: SociedadEspañola <strong>de</strong> Bioquímica Clínica y Patología Molecular.Tabla 3 Número <strong>de</strong> magnitu<strong>de</strong>s incluidas en losprogramas (período 2005 y 2006)Programa AEFA-AEBM SEQCBioquímica básica ensuero y fármacosBioquímica en orinay sector (tabla 2) y el número <strong>de</strong> magnitu<strong>de</strong>s biológicasincluidas en los programas es similar (tabla 3).El objeto <strong>de</strong>l trabajo es presentar a los laboratorios lasespecificaciones mínimas consensuadas, para asegurar unaprestación equivalente en todo el territorio español. Eldocumento se presentará también a la AdministraciónPública Estatal y Autonómica y a las entida<strong>de</strong>s evaluadoras<strong>de</strong> la calidad, para que se utilice como referencia.Material y métodos26þ6 31Urianálisis(11)Proteínas 9 20Hormonas/inmunoanálisis 11 18Marcadores tumorales 4 10Otros 2 612þurianálisis(7)AEFA-AEBM: Asociación Española <strong>de</strong> Farmacéuticos Analistas-Asociación Española <strong>de</strong> Biopatología Médica; SEQC: SociedadEspañola <strong>de</strong> Bioquímica Clínica y Patología Molecular.Se tomaron los datos <strong>de</strong> 2 años (2005 y 2006), en los quese obtuvieron 1.262.623 resultados que correspon<strong>de</strong>n a3.688 laboratorios y 24 períodos.Los resultados que obtuvieron todos los laboratorios participantesen los 3 programas durante los años 2005 y 2006 sealmacenaron en una base <strong>de</strong> datos <strong>de</strong>nominada datum 8 .El trabajo se realizó en varias etapas: en la primera (año2007) se incluyeron 24 magnitu<strong>de</strong>s bioquímicas y losresultados se publicaron previamente 2 . En la segunda (año2008) se añadieron 37 magnitu<strong>de</strong>s biológicas (3 <strong>de</strong> inmunología,5 <strong>de</strong> fármacos, 15 <strong>de</strong> hormonas y marcadorestumorales y 14 <strong>de</strong> hematimetría y coagulación); ésta es laetapa que se <strong>de</strong>scribe en el presente documento. En latercera etapa se estudiarán las magnitu<strong>de</strong>s no comunes a losdiversos programas <strong>de</strong> las 4 socieda<strong>de</strong>s.Las variables <strong>de</strong>l datum fueron i<strong>de</strong>ntificación numérica<strong>de</strong>l laboratorio, ciclo (año), período, nombre <strong>de</strong> la magnitud,resultado <strong>de</strong>l laboratorio y valor <strong>de</strong> comparación. Éstese <strong>de</strong>finió mediante la media global <strong>de</strong> los resultados <strong>de</strong> loslaboratorios, al analizar una misma muestra, en lasmagnitu<strong>de</strong>s con resultados similares entre los diversosmétodos analíticos. En las magnitu<strong>de</strong>s con resultados muydispares entre distintos métodos o instrumentos analíticos,el valor <strong>de</strong> comparación fue la media <strong>de</strong>l grupo específico.En la primera etapa se utilizó un mo<strong>de</strong>lo <strong>de</strong> cálculosencillo (<strong>de</strong>nominado en este artículo como procedimientoA), que se aplicó a cada magnitud y cada resultado conformeal siguiente esquema: Cálculo <strong>de</strong>l error (diferencia absoluta entre el resultado yel valor <strong>de</strong> comparación, expresada en porcentaje) Confección <strong>de</strong> la distribución <strong>de</strong> errores Segregación <strong>de</strong> la distribución <strong>de</strong>l 5% <strong>de</strong> los errores conmayor valor Elección <strong>de</strong>l percentil 95 <strong>de</strong> la distribución <strong>de</strong> erroresrestante, como valor <strong>de</strong> la especificación Redon<strong>de</strong>o <strong>de</strong> la especificación a valores enterosEn la segunda etapa se empleó un mo<strong>de</strong>lo máscomplejo consistente en comparar, para cada magnitud, laDATUMProcedimiento AEspecificacióncandidataLos programas estudiados en este trabajo, organizados por las 4socieda<strong>de</strong>s científicas (AEFA-AEBM, SEHH y SEQC), que cumplencon las recomendaciones <strong>de</strong> la International Fe<strong>de</strong>ration ofClinical Chemistry (IFCC) como programas <strong>de</strong> garantía externa<strong>de</strong> la calidad 6 , tienen las siguientes características comunes:CRITERIOSLa especificación¿Es plausible, parael fin esperado ?SíSe aceptala especificacióncandidata Tienen más <strong>de</strong> 25 años <strong>de</strong> experiencia Cumplen la guía ISO/IEC 43-1 7 Son in<strong>de</strong>pendientes <strong>de</strong> empresas comerciales Realizan formación sobre temas <strong>de</strong> calidad (promueven laimplantación <strong>de</strong> sistemas <strong>de</strong> gestión <strong>de</strong> la calidad entresus socios) Definen las especificaciones <strong>de</strong> la calidad <strong>de</strong>ntro <strong>de</strong> cadaorganización.NoSe <strong>de</strong>scartala especificacióncandidataFigura 1FinEsquema <strong>de</strong> trabajo <strong>de</strong> la primera etapa.