1 Menneskets genom
1 Menneskets genom
1 Menneskets genom
Create successful ePaper yourself
Turn your PDF publications into a flip-book with our unique Google optimized e-Paper software.
18209 01.fm7 Page 32 Friday, March 3, 2006 12:37 PM<br />
1 <strong>Menneskets</strong> <strong>genom</strong><br />
flere typer domæner. Eksempler på det første er<br />
zinkfinger-motiverne (C2H2- og GATA-typerne).<br />
Det er domæner der gør proteinet i stand<br />
til at binde til DNA. Som det fremgår af tabellen<br />
har mennesket mere end 500 gener, hvor<br />
disse domæner forekommer. I bananfluen og<br />
gær forekommer disse domæner i færre antal<br />
gener hhv. 239 og 43.<br />
Disse forskelle er sandsynligvis resultatet af en<br />
evolutionær proces hvor exons fra forskellige<br />
gener er blevet kopieret og splejset sammen, såkaldt<br />
exon shuffling (Figur 1.22). Man har eksperimentelt<br />
påvist at visse af de komplekse proteiner<br />
hos mennesket har domæner som meget<br />
ligner bakteriers simple proteiner således at proteindomæner<br />
med meget grundlæggende biokemiske<br />
funktioner, har en høj grad af sekvenslighed<br />
selv mellem meget simple og meget<br />
komplekse organismer.<br />
Det funktionelle antal gener i et <strong>genom</strong> kan<br />
øges på andre måder, hvoraf man nu kender to<br />
typer usædvanlig gen-organisation: 1) overlappende<br />
gener, og 2) gener-i-gener.<br />
32<br />
NH 2 COOH EGF<br />
NH 2 COOH Chymotrypsin<br />
NH 2 COOH Urokinase<br />
NH 2 COOH Faktor IX<br />
NH 2 COOH Plasminogen<br />
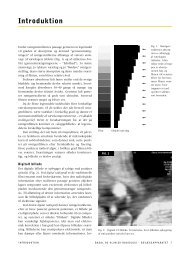
Figur 1.22 Nogle resultater af exon shuffling. Hvert<br />
symbol repræsenterer en familie af proteindomæner,<br />
som hver består af 30-50 aminosyrer. Domænerne repræsenterer<br />
exons som på et tidspunkt i evolutionsforløbet<br />
er forenet for at danne nye, større og mere<br />
komplekse proteiner. Ud for hvert protein er angivet<br />
dets navn.<br />
EGF = epidermal growth factor.<br />
DNA-sekvens<br />
(L-strengen)<br />
Start<br />
Met Leu…<br />
5'–CCAATGCTAA–3'<br />
…Gln<br />
Cys Stop<br />
Gen<br />
ND4<br />
ND4L<br />
Figur 1.23 Overlappende gener i mitokondrie-DNA,<br />
jf. Figur 1.27. Begyndelsen af genet ND4 overlapper<br />
afslutningen af genet ND4L. Aminosyresekvenserne i<br />
hhv. begyndelsen og afslutningen af de to polypeptider<br />
er anført i trebogstavkode. Det ses at læserammerne<br />
for de to gener er forskudt i forhold til hinanden.<br />
Gener der overlapper hinanden har enten<br />
hver sin template-streng eller deres mRNA’er<br />
translateres i overlapsområdet i hver sin læseramme,<br />
dvs aflæsningen af mRNA-sekvenserne<br />
sker med forskellige og faseforskudte startpunkter.<br />
Overlappende gener findes ofte i små kompakte<br />
<strong>genom</strong>er som fx virus<strong>genom</strong>er. De er<br />
sjældne i nukleære <strong>genom</strong>er fra højerestående<br />
eukaryoter. Der er et enkelt eksempel på et beskedent<br />
overlap i det kompakte mitokondrie<strong>genom</strong><br />
(Figur 1.23).<br />
Den anden type, gener-i-gener, er derimod<br />
relativt hyppigt forekommende i nukleære <strong>genom</strong>er.<br />
Et eksempel herpå i det humane <strong>genom</strong><br />
ses i neurofibromatose type 1-genet (NF1) som<br />
i intron 35 indeholder tre små gener, OMG,<br />
EVI2A og EVI2B (Figur 1.24). Hvert af disse<br />
»interne« gener er igen opdelt i egne exons og<br />
introns. Sådanne gener transkriberes ofte omvendt<br />
i forhold til værtsgenets transkriptionsretning<br />
– eller, sagt med andre ord, disse »interne«<br />
gener har værtsgenets ikke-template-streng som<br />
deres egen template-streng. Et andet eksempel er<br />
snoRNA-gener (small nucleolar RNA, som er<br />
ikke-kodende RNA der kemisk modificerer<br />
andre RNA’er) som ligeledes er beliggende i<br />
andre geners intron-sekvenser.