Zellbiologie & Imaging - Laborwelt
Zellbiologie & Imaging - Laborwelt
Zellbiologie & Imaging - Laborwelt
Erfolgreiche ePaper selbst erstellen
Machen Sie aus Ihren PDF Publikationen ein blätterbares Flipbook mit unserer einzigartigen Google optimierten e-Paper Software.
laborwelt<br />
Nr. 3 / 2008 – 9. Jahrgang<br />
<strong>Zellbiologie</strong> & <strong>Imaging</strong><br />
Hochdurchsatz-Two Hybrid-<br />
Screening von Protein-Protein<br />
Interaktionen mit Zellarrays<br />
Marktübersicht:<br />
Cell-based Assays<br />
iTRAQ-Analyse der Caspasevermittelten<br />
Proteinsekretion<br />
in Säugerzellen<br />
Ein neues System für das<br />
automatisierte Screening<br />
von Ionenkanälen
Normalized Normalized Cell Index Cell Index<br />
Cells in Real-Time!<br />
5<br />
Cells in Real-Time!<br />
Control<br />
4<br />
12.5 �M Etoposide<br />
5<br />
3<br />
Control 25 �M Etoposide<br />
4<br />
Treatment<br />
12.5 �M Etoposide<br />
2<br />
3<br />
1<br />
Seed<br />
Cells<br />
Treatment<br />
25 �M Etoposide<br />
50 �M Etoposide<br />
2<br />
Seed<br />
0<br />
Cells<br />
1 0 20 40 60<br />
100 �M Etoposide<br />
50 �M Etoposide 80<br />
0<br />
Time (hours)<br />
100 �M Etoposide<br />
0 20 40<br />
Time (hours)<br />
60 80<br />
xCELLigence Real-Time<br />
Cell xCELLigence Analyzer Real-Time System<br />
Cell Analyzer System<br />
RTCA SP InstrumentNEW<br />
RTCA SP InstrumentNEW<br />
Greater Insight, True Understanding<br />
Greater Insight, True Understanding<br />
Figure 1: Real-time monitoring of cytotoxicity<br />
through DNA damage. Etoposide is a DNA damaging<br />
agent Figure which 1: Real-time induces apoptosis monitoring in high of cytotoxicity concentrations,<br />
while through at lower DNA concentrations damage. Etoposide it leads is a to DNA S-Phase damaging and/or<br />
G2 agent arrest. which induces apoptosis in high concentrations,<br />
while at lower concentrations it leads to S-Phase and/or<br />
G2 arrest.<br />
XCELLIGENCE is a trademark of Roche.<br />
© 2008 Roche Diagnostics GmbH. All rights reserved.<br />
XCELLIGENCE is a trademark of Roche.<br />
© 2008 Roche Diagnostics GmbH. All rights reserved.<br />
www.roche-applied-science.com<br />
www.roche-applied-science.com<br />
Experience the power of dynamic, real-time, label-free cellular<br />
analysis with the xCELLigence System from Roche Applied Science.<br />
Experience the power of dynamic, real-time, label-free cellular<br />
Acquire data that end-point analysis could never realize, throughout<br />
analysis with the xCELLigence System from Roche Applied Science.<br />
your entire experiment. Work label-free to ensure physiologically<br />
Acquire data that end-point analysis could never realize, throughout<br />
relevant data, and choose from flexible throughput options to meet<br />
your entire experiment. Work label-free to ensure physiologically<br />
your needs: 24, 96, or 576 (6 x 96) wells simultaneously.<br />
relevant data, and choose from flexible throughput options to meet<br />
your needs: 24, 96, or 576 (6 x 96) wells simultaneously.<br />
� Capture Real-Time data throughout the entire experiment<br />
� Obtain physiologically relevant data without labels and reporters<br />
� Capture Real-Time data throughout the entire experiment<br />
� Analyze cells across a broad dynamic range<br />
� Obtain physiologically relevant data without labels and reporters<br />
� Perform a wide variety of applications<br />
� Analyze cells across a broad dynamic range<br />
(e.g. proliferation and cytotoxicity, Figure 1)<br />
� Perform a wide variety of applications<br />
(e.g. proliferation and cytotoxicity, Figure 1)<br />
For more information, visit www.xcelligence.roche.com or<br />
contact your local Roche representative today!<br />
For more information, visit www.xcelligence.roche.com or<br />
contact your local Roche representative today!<br />
Roche Diagnostics GmbH<br />
Roche Applied Science<br />
68298 Roche Mannheim, Diagnostics Germany GmbH<br />
Roche Applied Science<br />
68298 Mannheim, Germany
Immer näher an<br />
„in vivo“…<br />
Einen ungeahnten Aufschwung erleben derzeit Ganzzelluntersuchungen,<br />
sogenannte cell-based Assays. Sage und schreibe 51 (!) Anbieter aus<br />
aller Welt hatten, kurz nachdem wir auf die aktuelle Marktübersicht<br />
„cell-based Assays“ hingewiesen hatten, Kontakt zur Redaktion gesucht.<br />
Das Thema trifft derzeit offenbar den Nerv. Denn komplementär zum<br />
klassischen biochemischen Endpunktassay versprechen die zellbasierten<br />
Tests mehr „in vivo“ – quasi einen Realitätstest der in vitro-Daten in<br />
einem lebendigen biologischen Modellsystem.<br />
Einem Realitätstest möchte sich gerne auch die Redaktion von<br />
LABORWELT unterziehen, um die Zeitschrift möglichst nah an den<br />
Bedürfnissen von Ihnen, lieber Leser, zu orientieren: durch eine Leserumfrage.<br />
Für Ihre Anregungen gibt es natürlich auch eine kleine<br />
Belohnung. Gewinnen Sie einen iPOD Nano. Näheres erfahren Sie auf<br />
dem Fragebogen, der dieser Ausgabe von LABORWELT beiliegt. Sollte<br />
Ihnen dieser abhanden gekommen sein, mailen Sie uns einfach unter<br />
laborwelt@biocom.de – wir senden Ihnen die sechs Fragen dann rasch<br />
zu. Wir freuen uns, LABORWELT noch näher an „in vivo“-Bedingungen<br />
– nämlich Ihren Bedürfnissen – zu orientieren.<br />
„In vivo“ ist auch das Thema dieser Ausgabe. Angesichts des Auftauchens<br />
einer Nachfolgefirma der gescheiterten Würzburger TeGenero<br />
AG wirft der „Ex-Würzburger“ Prof. Dr. Holger Reichhardt einen Blick<br />
auf die Forschungsfortschritte seit dem gescheiterten Erstversuch am<br />
Menschen mit TGN1412, dem CD28-Superagonisten von TeGenero. Unternehmensschwerpunkt<br />
des Nachfolgers TheraMAB werden übrigens<br />
molekulardiagnostische Dienstleistungen sein. Die in ein universitäres<br />
Setting eingebettete Forschung an TGN1412 ist nur ein – wenn auch<br />
hochinteressanter – Nebenkriegsschauplatz. Daneben gibt es in dieser<br />
Themenausgabe Aktuelles aus Asssayentwicklung, Ionenkanalscreening,<br />
der klinischen Krebs-, Haut- und Atheroskleroseforschung etc.,<br />
etc. – Viel Lesevergnügen!<br />
Thomas Gabrielczyk<br />
In diesem Heft<br />
Marktübersicht: Cell-based Assays<br />
Sie messen die Wirkung von si- und miRNAs, die Induktion<br />
der Apoptose, zytotoxische Wirkungen und viele andere<br />
biologische Phänomene an lebenden Zellen. Darin sehen viele<br />
einen Vorteil der zellbasierten Assays gegenüber biochemischen<br />
Endpunktbestimmungen. Der Markt für immer neue Testsysteme<br />
und die benötigte Hardware boomt, denn die Nutzer kommen<br />
sowohl aus der Academia als auch aus Firmen (ab Seite 43).<br />
Editorial | Inhalt<br />
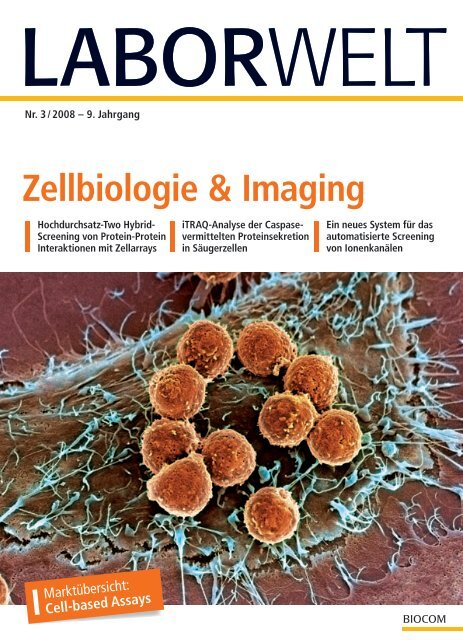
Titel: <strong>Zellbiologie</strong> & <strong>Imaging</strong><br />
Rasterelektronenmikroskopische Aufnahme gegen<br />
Krebsantigene aktivierter T-Lymphocyten (orange) bei<br />
der Attacke einer Krebszelle.<br />
Foto: © Steve Gschmeissner - Science Photo Library<br />
LABORWElT 9. Jahrgang | Nr. 3/2008 | 3<br />
Inhalt<br />
Leserforum Statement<br />
4 Wechselwirkung: Politik und Stammzellforschung<br />
Prof. Dr. Anthony Ho, Medizinische Klinik V, Universität Heidelberg<br />
Wissenschaft Translationale Medizin<br />
6 MIF: ein neuer Protagonist in der Atherosklerose<br />
Prof. Dr. Christian Weber et al., Klinikum der RWTH Aachen<br />
Blitzlicht In-vivo-<strong>Imaging</strong><br />
10 Echtzeit-Tumoranalyse mit rot fluoreszierenden Proteinen<br />
Prof. Dr. Christian Petzelt, MARINPHARM GmbH, Luckenwalde<br />
Blitzlicht Zellkultur<br />
12 Serumfreie Langzeitkultur humaner Hepatozyten<br />
Dr. Dieter Runge et al., PRIMACYT Cell Culture Technology GmbH, Schwerin<br />
Report Tiermodelle<br />
14 Mausmodelle: Therapie-Entwicklung für Epidermolysis bullosa<br />
Dr. Anja Fritsch. Prof Dr. Leena Bruckner-Tuderman et al.,<br />
Universitäts-Hautklinik Freiburg<br />
Report Immunbiologie<br />
18 Tregs, Autoimmunität und CD28-superagonistische Antikörper<br />
Prof. Dr. Holger M. Reichardt et al., Universitätsmedizin Göttingen<br />
Blitzlicht Zell-basierte Assays<br />
24 Gelbasierte Angiogenese-Assays<br />
Dr. Ulf Rädler et al., ibidi GmbH, Martinsried<br />
Blitzlicht Hochdurchsatzanalyse<br />
27 Zellarrays zur Hochdurchsatzanalyse von Proteininteraktionen<br />
Dr. Andrea Fiebitz et al., MPI für molekulare Genetik, Berlin<br />
Blitzlicht Proteomics/<strong>Zellbiologie</strong><br />
34 iTRAQ: Identifikation unkonventionell sekretierter Proteine<br />
Dr. Martin Keller; Dr. Hans-Dietmar Beer, Institut für <strong>Zellbiologie</strong>, ETH Zürich<br />
Blitzlicht Krebs<br />
37 Verstärkung der Chemosensitivität von Tumorzellen<br />
Prof. Dr. Rudolf Fahrig, RESprotect GmbH, Dresden<br />
Blitzlicht Screening<br />
39 Ionenkanalscreening auf den Kopf gestellt<br />
Dr. Dirck Lassen et al., Flyion GmbH, Tübingen<br />
Service Marktübersicht<br />
43 Cell-based Assays<br />
59 Verbände<br />
60 Stellenmarkt<br />
66 Produktwelt<br />
69 Termine<br />
70 Ausblick/Impressum
Statement<br />
Wechselwirkung: Politik<br />
und Stammzellforschung<br />
Prof. Dr. Anthony Ho, Ärztlicher Direktor der Medizinischen Klinik V, Universität Heidelberg<br />
Mit Erleichterung nehmen viele deutsche<br />
Wissenschaftler das Abstimmungsergebnis im<br />
Bundestag vom 11.4.08 auf. 346 Abgeordnete des<br />
Bundestages stimmten für und 228 Abgeordnete<br />
gegen eine Verschiebung des Stichtages für den<br />
Import humaner embryonaler Stammzellen aus<br />
dem Ausland auf den 1. Mai 2007. Denjenigen<br />
Abgeordneten, die zugestimmt haben, gebührt<br />
unser Dank. Die Gegner der embryonalen<br />
Stammzellforschung verdienen allerdings genauso<br />
viel Respekt und Dank dafür, dass sie uns alle<br />
zum Nachdenken angeregt haben, insbesondere<br />
was die Wahrung des Lebensschutzes und die<br />
Menschenwürde anbelangt.<br />
Das Stammzellgesetz vom Juni 2002 hat<br />
deutschen Wissenschaftlern ermöglicht, humane<br />
embryonale Stammzellen für hochrangige<br />
Forschungsarbeiten aus dem Ausland zu importieren,<br />
sofern die Zelllinien vor dem 1. Januar<br />
2002 etabliert worden waren. Seitdem wurde<br />
allerdings die Technik der Stammzellgewinnung<br />
so erheblich verbessert, dass die vor dem Stichtag<br />
etablierten Zelllinien für die Spitzenforschung<br />
inzwischen nicht mehr ausreichen.<br />
Polarisierte Debatte<br />
„Für die Forschung an embryonalen Stammzellen<br />
muss ungeborenes, menschliches Leben<br />
getötet werden!“, behaupteten beharrlich die<br />
Gegner der Erforschung humaner embryonaler<br />
Stammzellen. Dabei haben sie vergessen, dass<br />
beim Prozess der künstlichen Befruchtung<br />
mehrere Eizellen gleichzeitig befruchtet<br />
werden müssen, da die Erfolgschance bei der<br />
Befruchtung und Implantation nur einer gesunden<br />
Eizelle ein gesundes Kind austragen zu<br />
können, etwa 25 bis 30% beträgt. Im Ausland,<br />
aber auch in der Bundesrepublik sind dadurch<br />
eine große Zahl überzähliger Embryonen<br />
entstanden – in den USA zum Beispiel gibt es<br />
mehr als 400.000 tiefgefrorene Embryonen<br />
–, die nach einer gewissen Zeit „entsorgt“<br />
werden. Fast alle gebräuchlichen humanen<br />
embryonalen Stammzelllinien stammen aus<br />
eben solchen überzähligen Embryonen, die bei<br />
der künstlichen Befruchtung erzeugt wurden<br />
und die – statt sie zu „entsorgen“ – von den<br />
Eltern für die Forschung gespendet wurden,<br />
nachdem die künstliche Befruchtung erfolgreich<br />
abgeschlossen war.<br />
Das Ergebnis der Abstimmung im Bundestag<br />
Mitte April hat eine weitreichende Signalwirkung<br />
für die deutsche Wissenschaft: Es zeigt<br />
dass Spitzenforschung in Deutschland wieder<br />
möglich ist, dass Lebensforschung an Stellenwert<br />
gewonnen hat, dass die Wissenschaft das<br />
Vertrauen der Politik und der Gesellschaft wieder<br />
genießt und dass die Wissenschaftler dazu<br />
fähig sind, gute Forschung so verständlich in<br />
die Öffentlichkeit zu transportieren, dass am<br />
Ende Vernunft und Sachlichkeit walten.<br />
Verantwortungsbewusste Forschung<br />
Es herrscht auch Einigkeit darüber – sowohl<br />
bei den Befürwortern als auch den Gegnern<br />
der embryonalen Stammzellforschung –, dass<br />
Embry onen nur für das menschliche Leben<br />
erzeugt werden sollen und dass menschliches<br />
Leben nicht instrumentalisiert werden darf.<br />
Viele – sowohl Befürworter als auch Gegner –<br />
haben in dem Diskursprozess einen noch nie<br />
dagewesenen, tiefgreifenden Dialog geführt<br />
und ein gewisses Verständnis für die jeweils<br />
andere Position gewonnen. Auch das Ausland<br />
verfolgte mit enormer Spannung, ob man uns<br />
als „Land der Ideen und Innovationen“ ernst<br />
nehmen kann und will. Andere Länder wie Großbritannien,<br />
Frankreich, Schweden, Dänemark,<br />
Finnland, die Niederlande, Belgien, Spanien und<br />
Tschechien lassen die embryonale Stammzellforschung<br />
in großem Umfang zu und fassen<br />
deren Grenzen viel weiter als Deutschland. In<br />
diesen europäischen Staaten, die ebenfalls in<br />
der christlichen Tradition verwurzelt sind, ist<br />
eine sorgfältige ethische Wertung der Stammzellforschung<br />
erfolgt, von der sich Deutschland<br />
bisher abgekoppelt hat. Die Abstimmung vom 11.<br />
April hat verhindert, unser Land wissenschaftlich<br />
eine Insel in Europa werden zu lassen.<br />
Übertreibungen meiden<br />
Jedoch sollen wir uns davor hüten, verfrühte<br />
Hoffnungen auf Therapien mit embryonalen<br />
Stammzellen zu wecken – wie bereits vor einigen<br />
Jahren geschehen. Aktuelle Ergebnisse<br />
amerikanischer und japanischer Forschungsgruppen<br />
an tierischen und humanen embryonalen<br />
Stammzellen haben die vier genetischen<br />
Faktoren identifiziert, die für die „Verjüngung“<br />
einer Körperzelle verantwortlich sind. Dieser<br />
Erfolg war nach Aussage der Forscher nur auf<br />
Grundlage der Parallelforschung an humanen<br />
embryonalen und adulten Stammzellen<br />
möglich. Durch Einschleusen dieser vier Gene<br />
in Fibroblasten (Reprogrammierung) wurden<br />
Zellen gewonnen, die teils Eigenschaften<br />
Vita...<br />
Prof. Dr. med. Anthony D. Ho ist seit 1998<br />
Ordinarius und Ärztlicher Direktor der<br />
Medizinischen Klinik V (Schwerpunkte: Hämatologie,<br />
Onkologie und Rheumatologie)<br />
der Universität Heidelberg. Mit Blutstammzellforschung<br />
befasst er sich seit 1990 und<br />
war als Abteilungsleiter an der University<br />
of Ottawa, Canada, und an der University<br />
of California, San Diego, USA, tätig. Im Juli<br />
2002 wurde er als Mitglied der Zentralen<br />
Ethikkommission für Stammzellforschung<br />
des Robert-Koch-Instituts in Berlin bestellt.<br />
Seine spezielle Expertise besteht in der<br />
Blutstammzelltransplantation und der<br />
Therapie bei Leukämien und Lymphomen.<br />
Die Mechanismen der Steuerung von<br />
Stammzelleigenschaften, Selbsterneuerung<br />
und Differenzierung sowie die Wechselwirkung<br />
zwischen Stammzellen und<br />
ihren Nischen stellen die Schwerpunkte<br />
seiner Laborforschung dar.<br />
embryonaler Stammzellen haben (induzierte<br />
pluripotente Stammzellen).Ob und wann diese<br />
reprogrammierten Zellen in der Medizin eingesetzt<br />
werden können, ist noch nicht abzusehen.<br />
Derzeit werden die erforderlichen Reprogrammierungsfaktoren<br />
über Viren eingeschleust, die<br />
genetische Störungen in den Zellen hervorrufen<br />
können. Zudem sind unter den verwendeten<br />
Faktoren auch Onkogene, die tumorauslösend<br />
wirken können.<br />
Gerade um diese Schlüsselprobleme zu überwinden,<br />
ist die Forschung an humanen embryonalen<br />
Stammzellen essentiell. Insbesondere<br />
müssen die molekularen Steuerungsmechanismen<br />
embryonaler Stammzellen verstanden und<br />
auf adulte Zellen übertragen werden. Insofern<br />
stellt die Forschung an embryonalen Stammzellen<br />
eine sehr wichtige Grundlage für die Weiterentwicklung<br />
der adulten Stammzellforschung<br />
dar. Auch bei adulten Stammzellen hat es 20<br />
Jahre gedauert, bis erste Forschungsergebnisse<br />
zu klinischen Erfolgen geführt haben. Das<br />
deutsche Stammzellgesetz wurde im Juli 2002<br />
verabschiedet. Nach nur fünf Jahren ist eine<br />
klinische Umsetzung von Forschungsergebnissen<br />
nicht denkbar. Es muss noch sehr viel mehr<br />
Grundlagenforschung betrieben werden, bevor<br />
eine Umsetzung der Erkenntnisse in Therapien<br />
für die Klinik münden.<br />
4 | 9. Jahrgang | Nr. 3/2008 LABORWElT
Presenting!<br />
Prestige Antibodies <br />
Antibodies that work, right out of the box!<br />
The most highly characterized antibodies in the industry –<br />
1,800 NEW Prestige Antibodies , Powered by Atlas Antibodies.<br />
■ Designed & validated by the Human Proteome Resource (HPR)<br />
■ Standardized in universal protocols<br />
■ Over 500 immunohistochemical images for every antibody<br />
■ Publicly available data on the web<br />
Look even closer. Go to sigma.com/prestige for more information.<br />
Our Innovation, Your Research — Shaping the Future of Life Science<br />
Prestige Antibodies is a trademark of Sigma-Aldrich Biotechnology LP and Sigma-Aldrich Co.<br />
sigma-aldrich.com<br />
Anti-BID Cat No. HPA000722<br />
on A-431 cells shown in green,<br />
nucleus in blue, microtubules in<br />
red and endoplasmic reticulum<br />
(ER) in yellow.<br />
Just one of the over 500 IF,<br />
IHC and WB images available
Wissenschaft Translationale Medizin<br />
MIF – neuer Protagonist<br />
in der Atherosklerose<br />
Dipl.-Biol. Regina Krohn, Prof. Dr. rer. nat. Jürgen Bernhagen, Prof. Dr. med. Christian Weber,<br />
Institut für Molekulare Herz-Kreislaufforschung und Institut für Biochemie,<br />
Universitätsklinikum der RWTH Aachen<br />
Der Makrophagen-Migrations-Inhibitions-Faktor MIF spielt eine wichtige Rolle bei Entzündungserkrankungen<br />
wie der Atherogenese. Kürzlich wurden neben dem bekannten MIF-Rezeptor CD74 die<br />
beiden CXC-Chemokin-Rezeptoren CXCR2 und CXCR4 als funktionelle Rezeptoren für MIF identifiziert.<br />
Die MIF-vermittelte Adhäsion und Chemotaxis von Monozyten und T-Zellen erfolgt spezifisch<br />
durch CXCR2, CXCR4 und CD74. Dabei induzierte MIF eine schnelle Integrin aktivierung und Kalzium-<br />
Mobilisierung. Kompetitionsstudien mit bekannten CXCR2/CXCR4-Liganden bestätigten die Interaktion<br />
von MIF mit diesen Rezeptoren. Darüber hinaus wurden anhand von Co-Immunpräzipitationen<br />
und mittels konfokaler Mikroskopie die direkte Bindung von MIF an CXCR2 und das Vorhandensein<br />
eines CXCR2/CD74-Rezeptorkomplexes nachgewiesen. In Atherosklerose-anfälligen Mäusen verhinderte<br />
MIF-Defizienz den Monozytenarrest an der Aorta-/Arterienwand und CXCR2-Defizienz<br />
die MIF-induzierte Leukozytenadhäsion. MIF-Blockade in Mäusen mit fortgeschrittener Atherosklerose<br />
führte zu einer Regression der Plaquefläche und zu einem stabileren Plaquephänotypen.<br />
Die neu gewonnenen Erkenntnisse über MIF als Regulator der inflammatorischen Zellrekrutierung<br />
und Atherogenese machen das Protein zu einem vielversprechenden therapeutischen Ziel bei der<br />
Behandlung von Patienten mit manifester Atherosklerose.<br />
Die Systemkrankheit Atherosklerose und<br />
ihre klinischen Folgen – wie etwa die Koronare<br />
Herzkrankheit, Periphere Arterielle<br />
Verschlusskrankheit und Schlaganfall – ist<br />
derzeit die häufigste Erkrankung mit Todesfolge<br />
in den entwickelten Ländern 1 . Die<br />
Atherosklerose ist eine chronische Entzündungskrankheit<br />
der Arterienwand, in deren<br />
Verlauf sich immunkompetente Blutzellen<br />
unterhalb des Endothels der Arterienwand<br />
anlagern. Diese sezernieren Zytokine und<br />
spezifische Chemokine, welche wiederum<br />
Abb. 2: MIF-Signaltransduktion über einen funktionellen Rezeptorkomplex. Extrazelluläres<br />
MIF bindet an das Oberflächenprotein CD74 ohne intrazelluläre Signaldomäne,<br />
kann so über das Proteoglykan CD44 Rezeptorkinasen (RTK) der Src-Familie, MAPK/<br />
ERK-Kinasen und den PI3K/Akt-Signalweg aktivieren oder p53-abhängig die Apoptose<br />
inhibieren. MIF bindet und aktiviert die G-Protein-gekoppelten Chemokinrezeptoren<br />
(GPCR) CXCR2 und CXCR4 und ihre Signalwege. Komplexbildung von<br />
CXCR2 mit CD74 kann die GPCR-Aktivierung und Bildung eines GPCR-RTK-artigen<br />
Signalkomplexes erleichtern, welcher einen Calcium-Influx und eine schnelle Integrinaktivierung<br />
vermittelt. Nach Endozytose kann MIF mit JAB-1 interagieren und so<br />
MAPK-Signale hemmen und die AP-1-Aktivität modulieren.<br />
Abb. 1: Dreidimensionale Darstellung des<br />
MIF-Trimers<br />
atherogene Leukozyten zum Entzündungsherd<br />
rekrutieren und durch Aktivierung von<br />
Integrinen deren Adhäsion an die Arterienwand<br />
bewirken. So entsteht nach und nach<br />
ein sogenannter Plaque, der hauptsächlich<br />
aus fettbeladenen Makrophagen, aus Leukozyten,<br />
aber auch glatten Muskelzellen<br />
besteht. Bei fortschreitender Erkrankung<br />
entsteht ein komplexes, fibröses Atherom,<br />
welches instabil werden und – durch noch<br />
nicht aufgeklärte Mechanismen – aufbrechen<br />
kann. Die nachfolgende Thrombusbildung<br />
resultiert in einem Arterienverschluss, der<br />
akute klinische Effekte, wie Herzinfarkt oder<br />
Schlaganfall, zur Folge hat 2 .<br />
MIF ist als Entzündungsmediator<br />
an der Atherogenese beteiligt<br />
Vor etwa 40 Jahren wurde das Zytokin MIF im<br />
Zusammenhang mit der Spättyp-Hypersensitivität<br />
als ein von aktivierten T-Zellen produzierter<br />
Inhibitor der ungerichteten Migration<br />
von Makrophagen entdeckt 3,4 . Später ergab<br />
eine genauere Untersuchung, dass vor allem<br />
Monozyten/Makrophagen MIF produzieren 5 .<br />
In den darauffolgenden Jahren belegten<br />
immer mehr Studien die proinflammatorischen<br />
Eigenschaften von MIF, zum Beispiel<br />
als Gegenspieler von Glucocorticoiden, und<br />
dessen Beteiligung an verschiedenen Autoimmunerkrankungen,<br />
wie etwa der Rheumatoiden<br />
Arthritis 6,7 . Zudem wurde anhand<br />
eines Ratten-Modells der immun induzierten<br />
Glomerulonephritis gezeigt, dass MIF, wie<br />
ein Chemokin, die Leukozytenrekrutierung<br />
stimuliert 8 .<br />
Neuere Forschungsergebnisse deuten<br />
auf eine wichtige Rolle von MIF in der<br />
Atherosklerose hin. Im hyperlipidemischen<br />
Mausmodell führte die Behandlung mit<br />
einem MIF-blockierenden Antikörper nach<br />
drahtvermittelter Denudation der Arteria<br />
carotis zu einer signifikanten Abnahme des<br />
Makrophagen- und Schaumzellengehaltes<br />
in den neointimalen Läsionen. Gleichzeitig<br />
6 | 9. Jahrgang | Nr. 3/2008 LABORWElT
Wissenschaft Translationale Medizin<br />
Abb. 3: Konfokale Mikroskopie der Co-Lokalisation von CXCR2 und CD74 (Overlay: gelborange)<br />
in RAW264.7-CXCR2-Transfektanten. © Bernhagen et al. Nat. Med. 13, 587-596<br />
(2007)<br />
war der Gehalt an glatten Muskelzellen und<br />
fibrillärem Kollagen erhöht, was einem stabileren<br />
Plaque-Phänotypen entsprach. Als<br />
zugrundeliegenden Mechanismus entdeckten<br />
die Autoren die MIF-vermittelte Adhäsion von<br />
Monozyten an Endothelzellen 9 . Danach ist<br />
nach MIF-Blockade eine Reduktion der Makrophagenanzahl<br />
in der Aortawand von Apoe –/– -<br />
Mäusen zu beobachteten. Hinzu kommt eine<br />
Inhibition mehrerer Entzündungsmediatoren,<br />
darunter Matrixmetalloproteinase (MMP)-2<br />
und Tumornekrosefaktor (TNF) 10 .<br />
MIF-Effekte werden über einen<br />
Rezeptor-Kinase-Komplex ausgelöst<br />
Humanes MIF hat ein Molekulargewicht von<br />
12,34 kDa und weist eine 90%ige Homologie<br />
zu murinem MIF auf. Die Röntgenstrukturanalyse<br />
von MIF ergab ein Trimer. Die drei<br />
identischen Untereinheiten formen ein ringförmiges<br />
Protein mit einem hydrophoben<br />
Kanal, dessen Funktion allerdings noch nicht<br />
bekannt ist (Abb. 1). Einer Studie zufolge, in der<br />
Gelfiltration und ‚Cross-linking’-Experimente<br />
durchgeführt wurden, tritt MIF in physiologischer<br />
Lösung als Monomer und als Dimer<br />
auf, die trimere Form kommt dagegen eher<br />
seltener vor 11 . Für MIF wurden sowohl eine<br />
Tautomerase- als auch eine Thiolprotein-<br />
Oxidoreduktaseaktivität beschrieben. Allerdings<br />
konnte diesen enzymatischen Aktiviäten<br />
bisher keine physiologische Bedeutung<br />
zugeordnet werden. Abgesehen von seiner<br />
besonderen Struktur weist das MIF-Monomer<br />
eine auffällige Ähnlichkeit mit dem Dimer des<br />
CXC-Chemokins Interleukin 8 (IL-8) auf, einem<br />
Chemokin, das maßgeblich an der Progression<br />
der Atherosklerose beteiligt ist.<br />
Als erster cytoplasmatischer Interaktionspartner<br />
von MIF wurde das Jab1 (C-Jun<br />
activation domain binding protein-1) mittels<br />
Yeast-Two-Hybrid-Screening entdeckt 12 , eine<br />
Untereinheit des COP-9-Signalosoms. Durch<br />
die Bindung von MIF an Jab1 wird die Aktivierung<br />
der AP-1-vermittelten Genexpression<br />
und die Degradation von p27kip1 verhindert<br />
und so die Zellwachstumsinduktion blockiert<br />
(Abb. 2). Andererseits inhibiert MIF die p53vermittelte<br />
Apoptose. Diese Inhibition ist<br />
Cyclooxygenase (Cox)-2-abhängig 13 , deren<br />
Expression wiederum von AP-1 induziert wird.<br />
MIF könnte hier also auch die AP-1-vermittelte<br />
Expression von Cox-2 fördern, indem es an<br />
Jab1 bindet.<br />
CD74, die MHC-Klasse-II-assoziierte invariante<br />
Kette, wurde als erster Zelloberflächen-<br />
Rezeptor für MIF identifiziert. Es wurde<br />
gezeigt, dass die MIF/CD74-Interaktion eine<br />
Aktivierung der ERK1/2/MAPK-Signaltransduktionskette<br />
zur Folge hat (Abb. 2). Die Studien<br />
der letzten zwei Jahre trugen wesentlich zur<br />
Aufklärung der MIF/CD74-Signalwege bei. Die<br />
Autoren schlugen einen MIF/CD74-Signalkomplex<br />
vor, der über die Kopplung an das Proteoglykan<br />
CD44 und eine Tyrosin-Kinase der Src-<br />
Familie die Induktion der Apoptose blockieren<br />
Abb. 4: Blockade von MIF führte zur Regression und Stabilisierung von fortgeschrittenen<br />
atherosklerotischen Plaques. Nach 12 Wochen fettreicher Diät wurden Apoe –|– -Mäuse<br />
zusätzliche vier Wochen mit blockierenden Antikörpern oder Puffer (Kontrolle) behandelt.<br />
A. Oil-Red-O-Färbung der Aortenwurzeln; B. Datenpunkte repräsentieren die<br />
Plaquefläche nach 12 bzw. 16 Wochen; © Bernhagen et al. Nat. Med. 13, 587-596 (2007)<br />
kann, indem der Phospho-Inositid-3-Kinase<br />
(PI3K)/Akt-Weg aktiviert wird oder auch p53<br />
inhibiert wird 14,15 . Es bedarf noch der Klärung,<br />
ob CD74 die MIF-Internalisierung und/oder<br />
die Interaktion mit Jab1 ermöglicht.<br />
Der CXC-Rezeptorligand MIF vermittelt<br />
inflammatorische Zellrekrutierung<br />
Da nicht alle Zellen, auf die MIF wirkt (z.B<br />
Neutrophile und Fibroblasten), CD74 auf ihrer<br />
Oberfläche exprimieren, lag die Annahme<br />
nahe, dass mindestens ein zusätzlicher Rezeptor<br />
am MIF-Wirkmechanismus beteiligt ist.<br />
Unter Berücksichtigung der chemokinartigen<br />
Wirkweise von MIF und seiner strukturellen<br />
Ähnlichkeit mit dem IL-8-Dimer wurde vermutet,<br />
dass es sich um einen Chemokin-Rezeptor<br />
handeln müsste.<br />
Tatsächlich konnten kürzlich die Rezeptoren<br />
für die CXC-Chemokine IL-8 und SDF-1a,<br />
CXCR2 und CXCR4 als funktionelle Rezeptoren<br />
für MIF identifiziert werden 16 .<br />
In vitro-Adhäsionsversuche in einer laminaren<br />
Flusskammer zeigten, dass MIF einen<br />
Integrin-abhängigen Leukozytenarrest auf<br />
Endothelzellen direkt durch CXCR2 und – zu einem<br />
geringeren Ausmaß – durch CXCR4 sowie<br />
durch den zuvor beschriebenen MIF-Rezeptor<br />
CD74 bewirkte. Durch Expressionsanalysen<br />
und die Anwendung blockierender Antikörper<br />
konnte eine Beteiligung der CXC-Chemokine<br />
IL-8 und SDF-1a ausgeschlossen werden. Auch<br />
die MIF-induzierte Leukozyten-Chemotaxis<br />
wurde durch CXCR2 und CXCR4 vermittelt,<br />
und Kompetitionsstudien mit radioaktiv<br />
markiertem IL-8 und SDF-1a zeigten die Interaktion<br />
von MIF mit diesen Rezeptoren. Letztendlich<br />
belegten Co-Immunpräzipitations-<br />
Experimente und konfokale Mikroskopie die<br />
Existenz eines CD74/CXCR2-Rezeptorkomplexes<br />
(Abb. 3) sowie die direkte Interaktion<br />
von MIF mit CXCR2. Erstaunlicherweise<br />
bewirkte MIF eine rapide CXCR2-abhängige<br />
Calcium-Mobilisierung in Neutrophilen.<br />
Dies bedeutet, dass die Ca 2+ -Mobilisierung<br />
nicht die Kooperation von CD74 erfordert,<br />
da Neutrophile dieses Zelloberflächenprotein<br />
nicht exprimieren. Des Weiteren konnte die<br />
direkte Aktivierung des Integrins aLb2 durch<br />
MIF auf der Monozytenzelllinie MonoMac6<br />
und auf primären Monozyten anhand eines<br />
Antikörpers gegen die aktive Konformation<br />
von aLb2 nachgewiesen werden.<br />
Anhand von Adhäsionsversuchen in explantierten<br />
Mauskarotiden wurden ex vivo<br />
Beweise für die Beteiligung von CXCR2 an<br />
der MIF-induzierten Zellrekrutierung erbracht.<br />
Die Applikation von blockierenden<br />
Antikörpern gegen CD74, CXCR2 oder MIF<br />
führte gleichermaßen zu einer Reduktion<br />
der adhärenten Monozyten. Mittels Intravitalmikroskopie<br />
wurde auch in vivo die Rolle<br />
von CXCR2 dokumentiert. Im Modell der induzierten<br />
Peritonitis wurden MIF-Wildtyp<br />
8 | 9. Jahrgang | Nr. 3/2008 LABORWElT
und MIF-Knock-out-Mäuse mit CXCR2-Knockout-Knochenmark<br />
rekonstituiert. Dies führte<br />
bei den Wildtyp-Mäusen zu einer starken<br />
Abnahme der inflammatorischen Neutrophileninfiltration,<br />
während Transplantation<br />
von Cxcr2 –/– -Knochenmark in MIF-defizienten<br />
Mäusen zu keiner weiteren Abnahme der<br />
Zellrekrutierung führte.<br />
MIF-Blockade führt zu Regression und<br />
stabilisiert fortgeschrittene Plaques<br />
Die Rolle von CXCR2 bei der Atherogenese<br />
wurde bereits beschrieben 17 . Die Autoren beobachteten<br />
bei CXCR2-Deletion eine stärkere<br />
Abnahme der Atherosklerose in LDL-Rezeptor-<br />
Knock-out-Mäusen als bei der Deletion des<br />
Liganden KC/Gro-a. Es musste also noch ein<br />
anderer Faktor, wenigstens teilweise für die<br />
CXCR2-vermittelte Plaquebildung verantwortlich<br />
sein. Versuche zur Atherosklerose-<br />
Regression belegten, dass MIF der gesuchte<br />
CXCR2-Ligand ist und einen erheblichen<br />
Einfluss auf die Plaquegröße und Komposition<br />
in der fortgeschrittenen Atherosklerose hat.<br />
Hierzu wurden Apoe –/– -Mäuse 12 Wochen lang<br />
auf eine fettreiche Diät gesetzt und anschließend<br />
weitere vier Wochen mit Antikörpern<br />
gegen die bekannten murinen CXCR2- und<br />
CXCR4-Liganden, KC- und SDF-1a sowie gegen<br />
den neuentdeckten Liganden MIF behandelt.<br />
Die MIF-Antikörper-Behandlung führte zu<br />
einer signifikanten Reduktion der Läsionen im<br />
Vergleich zu unbehandelten Mäusen. Darüber<br />
hinaus war die Läsionenfläche im Vergleich<br />
mit den Kontrollmäusen 12 Wochen nach der<br />
Antikörperbehandlung signifikant reduziert<br />
(Abb. 4). Zudem waren der Makrophagen- und<br />
T-Zell-Gehalt erniedrigt, was für eine Stabilisierung<br />
des Atheroms spricht.<br />
Fazit<br />
MIF ist ein funktioneller Ligand von CXC-<br />
Chemokin-Rezeptoren und bindet direkt an<br />
den IL-8-Rezeptor CXCR2, der einen Rezeptor-<br />
Komplex mit CD74 bildet. Sobald MIF mit<br />
CXCR2 und/oder CXCR4 interagiert, wirkt es<br />
in einer chemokinartigen Weise und repräsentiert<br />
so eine neue Gruppe von Chemokinen,<br />
die CLF (Chemokine-like-funcion)-Chemokine.<br />
MIF fördert die inflammatorische Leukozytenrekrutierung<br />
durch CXCR2 und von T-Zellen<br />
durch CXCR4. Darüber hinaus konnte für den<br />
Krankheitsverlauf der Atherosklerose eine<br />
enge Kooperation von MIF und CXCR2 nachgewiesen<br />
werden. Die neuesten Erkenntnisse<br />
über MIF machen dieses Protein zu einem<br />
wichtigen neuen therapeutischen Ziel bei<br />
der Behandlung von Atherosklerose. Die Blockade<br />
des MIF-Effektes, zum Beispiel durch<br />
Peptid-Antagonisten, könnte bei Patienten<br />
Wissenschaft Translationale Medizin<br />
mit manifester Atherosklerose zu einem<br />
Rückgang der Krankheit und zur Stabilisierung<br />
von Plaques führen.<br />
Literatur<br />
[1] Libby, P., Nature 420, 868-874 (2002).<br />
[2] Hansson, G.K. New Engl J Med 352, 1685-1695 (2005).<br />
[3] Bloom, B.R. & Bennett, B., Science 153, 80-82 (1966).<br />
[4] David, J.R., Proc Natl Acad Sci U S A 56, 72-77 (1966).<br />
[5] Bernhagen, J., et al., J Exp Med 183, 277-282 (1996).<br />
[6] Calandra, T. & Bucala, R., J Inflamm 47, 39-51 (1995).<br />
[7] Leech, M., et al. , Arthritis Rheum 42, 1601-1608 (1999).<br />
[8] Lan, H.Y., et al. J Exp Med 185, 1455-1465 (1997).<br />
[9] Schober, A., et al., Circulation 109, 380-385 (2004).<br />
[10] Burger-Kentischer, A., et al., Atherosclerosis 184, 28-38 (2006).<br />
[11] Mischke, R., et al., FEBS Lett 427, 85-90 (1998).<br />
[12] Kleemann, R., et al., Nature 408, 211-216 (2000).<br />
[13] Mitchell, R.A., et al., Proc Natl Acad Sci USA 99, 345-350<br />
(2002).<br />
[14] Shi, X., et al., Immunity 25, 595-606 (2006).<br />
[15] Lue, H., et al., Oncogene 26, 5046-5059 (2007).<br />
[16] Bernhagen, J., et al., Nat Med 13, 587-596 (2007).<br />
[17] Boisvert, W.A., et al., Am J Pathol 168, 1385-1395 (2006).<br />
Korrespondenzadresse<br />
Univ.-Prof. Dr. med. Christian Weber<br />
Inst. f. Molekulare Herz-Kreislaufforschung<br />
Universitätsklinikum Aachen<br />
Pauwelsstr. 30, 52074 Aachen<br />
Tel.: +49-(0)241-80-88692<br />
Fax: +49-(0)241-80-82716<br />
cweber@ukaachen.de<br />
����������������������<br />
����������������������<br />
����������������������<br />
����������������������<br />
�����������������������������������������<br />
�������������������������������������<br />
��������������������������������������<br />
�������������������������������������������<br />
������������������������������� �� �<br />
���������� ����������<br />
����������������������<br />
��������������������������������������<br />
��������������������������������������������<br />
�����������������������������������������������<br />
������������������������<br />
������������������������������������<br />
������������������������������������<br />
��������������������������������������<br />
LABORWElT 9. Jahrgang | Nr. 3/2008 | 9<br />
�������������� ����������������������������������������<br />
���������������������������<br />
��������������������
Blitzlicht In vivo-<strong>Imaging</strong><br />
Rot fluoreszierende Zellen<br />
zur Echtzeit-Tumoranalyse<br />
in lebenden Tieren<br />
Prof. Dr. Christian Petzelt; MARINPHARM GmbH, Luckenwalde<br />
Um tieferliegende Tiergewebe visualisieren zu können, ist ein optisches Fenster im nahen Infrarot<br />
vorteilhaft. Ein neues, pH-stabiles, hell leuchtendes und langzeitstabiles, monomeres<br />
rot fluoreszierendes Protein, das mit einer Wellenlänge von 635 nm emittiert, wurde jetzt<br />
stabil in 20 Krebszelllinien transfiziert und eignet sich hervorragend für die Proteinlokalisierung<br />
im lebenden Tier sowie für Fluoreszenz-Resonanz-Energie-Transfer(FRET)-Experimente.<br />
Die lange Fluoreszenz-Lebenszeit des TagRFP, das durch gezielte Mutagenese der für die<br />
Dimerisierung von hellleuchtenden Protein-Sequenzen erhalten wurde, prädestiniert es<br />
für einen Einsatz in der präklinischen Medikamentenentwicklung.<br />
A B<br />
Rot fluoreszierende Melanom-Zellen (A) und rot fluoreszierende Mikrotubuli in Osteosarkom-Zellen<br />
(B)<br />
Lebende Zellen in einem Versuchstier zu<br />
erkennen und zu beobachten, war schon<br />
immer ein Traum und gleichzeitig eine Herausforderung<br />
für die Wissenschaft. Um zum<br />
Beispiel in präklinischen Versuchen neuartige<br />
Substanzen für die Tumortherapie zu testen,<br />
werden geeignete Tumore in den Versuchstieren<br />
erzeugt, indem Krebszellen in das Tier<br />
injiziert werden, die dann sich zu Tumoren<br />
entwickeln, welche zahlreiche Eigenschaften<br />
echter Tumore zeigen. In Mäusen mit einem<br />
supprimierten Immunsystem können sogar<br />
durch Injektion menschlicher Tumorzellen<br />
menschliche Tumore erzeugt werden. Aber wie<br />
werden mögliche therapeutische Effekte einer<br />
neuen Substanz analysiert? Heute werden<br />
meist noch die gleichen analytischen Methoden<br />
wie vor 50 Jahren eingesetzt: Entweder das<br />
Tumorwachstum wird visuell – der Tumor wird<br />
gemessen – oder mit histologischen Methoden<br />
verfolgt – wobei das Tier für die Analyse getötet<br />
werden muss.<br />
Die Entdeckung fluoreszierender Proteine und<br />
die Möglichkeit, ihre Gene in lebende Zellen einzuschleusen,<br />
haben unsere Analysemethoden<br />
komplett verändert. Das Grün Fluoreszierende<br />
Protein (GFP) wird jetzt vielfach eingesetzt,<br />
um lebende Zellen zu markieren. Wenn solche<br />
grün fluoreszierenden Zellen in ein Tier injiziert<br />
werden, sind sie direkt in Echtzeit zu „sehen“,<br />
vorausgesetzt die Zellen bleiben an der Oberfläche<br />
des Tieres. Diese extrem einengende<br />
Beschränkung beruht darauf, dass die Reichweite<br />
des emittierten Fluoreszenzsignals direkt<br />
proportional zu seiner Emissions-Wellenlänge<br />
ist – oder in anderen Worten: je länger die Wellenlänge<br />
des emittierten Lichtes, desto tiefer in<br />
das Tier lässt sich „blicken“.<br />
Vor wenigen Monaten gelang Wissenschaftlern<br />
der russischen Firma Evrogen JSC (Moskau)<br />
ein entscheidender Durchbruch. Sie entdeckten<br />
ein rot fluoreszierendes Protein mit einer Emission<br />
bei 635 nm, also beinahe im infraroten Bereich,<br />
das sich ideal zur Zellmarkierung eignet 1 .<br />
Marinpharm als der Lizenzpartner von Evrogen<br />
JSC (Moskau) ist seit einigen Jahren weltweit<br />
die einzige Firma, die die DNA solcher Proteine<br />
verwendet, um stabil transfizierte Zell-Linien zu<br />
generieren, die diese Fluoreszenzeigenschaften<br />
permanent aufweisen.<br />
Leuchtende Krebszelllinien<br />
Tatsächlich gelang es nur wenige Monate nach<br />
der Publikation 1 des neuen rot fluoreszierenden<br />
Proteins mehr als 20 menschliche Tumor-Zell-<br />
Linien herzustellen, die das Protein exprimieren.<br />
Damit sind ideale Werkzeuge für das in-vivo-<br />
<strong>Imaging</strong> geschaffen. Unter ihnen sind mehrere<br />
metastasierende und nicht-metastasierende<br />
Melanom-Zell-Linien besonders attraktiv für<br />
präklinische Tests (Abb. 1 A). Diese ermöglichen<br />
es erstmals, im lebenden Tier nicht nur die<br />
fluoreszierenden Zellen an der Oberfläche zu<br />
verfolgen, sondern sogar in Organen kleine<br />
Gruppen von Tumorzellen zu lokalisieren, die<br />
deren Invasion erfolgreich durchgeführt haben.<br />
Niemals zuvor war dies mit einer so hohen Auflösung<br />
und Genauigkeit möglich. Dadurch wird<br />
jetzt sowohl die Invasion eines Tumors als auch<br />
die Bildung von Metastasen in Echtzeit im Tier<br />
verfolgbar. Zudem können mögliche Therapieeffekte<br />
verfolgt werden. Eine weitere attraktive<br />
Anwendung: Die DNA-Information für das<br />
neue rot fluoreszierende Protein lässt sich an<br />
die DNA von intrazellulären Proteinen koppeln,<br />
die unentbehrlich für die Zellen sind, um ihren<br />
Zellzyklus zu durchlaufen, zum Beispiel Tubulin<br />
oder Actin. Solche Konstrukte wurden von<br />
Marinpharm erfolgreich benutzt, um humane<br />
Tumor-Zell-Linien zu generieren, die die Tubulin-<br />
und Aktin-Strukturen in brillant-tiefroter<br />
Fluoreszenz zeigen (Abb. 1 B), Zellen, die man<br />
wegen der emittierten langen Wellenlänge als<br />
solche wieder im Tier „sehen“ kann.<br />
Es kann als sicher gelten, dass diese neuen<br />
Werzeuge die präklinische Testung in vielerlei<br />
Hinsicht beschleunigen und verbessern können<br />
und damit dazu beitragen, Zeit und Kosten für<br />
die Entwicklung neuer therapeutischer Substanzen<br />
zu sparen.<br />
Literatur<br />
[1] Merzlyak EM, Goedhart J, Shcherbo D, Bulina ME, Scheglov<br />
AS, Fradkov AF, Gaintzeva A, Lukyanov KA, Lukyanov S,<br />
Gadella TW, Chudakov DM.; Bright monomeric red fluorescent<br />
protein with an extended fluorescence lifetime. Nat<br />
Methods. 2007 Jul;4(7):555-7.<br />
Korrespondenzadresse<br />
Prof. Dr. Christian Petzelt<br />
MARINPHARM GmbH<br />
Im Biotechnologiezentrum TGZ2<br />
14943 Luckenwalde<br />
cpetzelt@marinpharm.com<br />
www.marinpharm.com<br />
10 | 9. Jahrgang | Nr. 3/2008 LABORWElT
A NEW DIMENSION IN SCREENING:<br />
SCREENING STATION FOR LIFE SCIENCE<br />
The Olympus Screening Station for Life Science is<br />
a highly advanced instrument that perfectly combines the<br />
modularity and fl exibility of a microscope-based set-up<br />
with the automation, throughput, reliability and reproducibility<br />
demands of screening applications. Therefore, the<br />
system is equally well suited to handle standard assays as<br />
well as assay development.<br />
By combining world-leading microscope components with<br />
precision automation apparatus, can handle many<br />
different sample formats including multi-well plates, slides<br />
and even custom-built arrays. Furthermore, the unique MT20<br />
illumination system provides high speed and fully controllable<br />
sample illumination. As a result you can capture every detail,<br />
so that even the smallest change can be fully documented.<br />
With an amazing amount of detail extracted from each assay,<br />
brings together superb data handling capabilities with<br />
high-powered, cytometry oriented analysis tools. These allow<br />
for full image processing, object and sub-object detection,<br />
parameter calculation as well as sophisticated gating and<br />
analysis algorithms.<br />
For more information, contact:<br />
Phone: +49 40 23773-4612<br />
E-mail: mikroskopie@olympus.de<br />
www.olympus.de<br />
O L Y M P U S M I C R O S C O P Y
Blitzlicht Zellkultur<br />
Serumfreie Langzeitkultur<br />
humaner Hepatozyten in<br />
lumox TM -Slide-Flasks<br />
Anett Ullrich, Dr. Dieter Runge, PRIMACYT Cell Culture Technology GmbH, Schwerin<br />
Mit HEPAC 2 hat die Primacyt GmbH ein serumfreies Langzeitzellkultursystem mit humanen<br />
Hepatozyten entwickelt, das auch für pharmakologisch-toxikologische Studien im Rahmen<br />
der Arzneimittelentwicklung eingesetzt wird. Dieses System wurde auf ein neues Kultivierungssystem,<br />
der Objektträgerflasche mit dem gasdurchlässigen Folienboden übertragen,<br />
der lumox TM slide flask. Die Harnstofffreisetzung als Parameter für die hepatozelluläre Funktionalität<br />
der Zellen zeigte sich hierbei gegenüber der Kultivierung in einer konventionellen<br />
6-well-Platte deutlich erhöht. Die sehr guten optischen Eigenschaften (geringe Autofluoreszenz,<br />
hohe Transparenz) des lumox TM -Folienbodens stellten zudem eine hervorragende Basis<br />
für zellspezifische Färbungen dar.<br />
Abb. 1: Vergleich der Funktion und Vitalität humaner Hepatozyten in lumox TM slide flask-<br />
(rote Kurve) und 6-well-Kulturplatten (blaue Kurve) eines exemplarischen Experiments:<br />
Freisetzen von Harnstoff in das Zellkulturmedium jeweils innerhalb von 24 Stunden bei<br />
einer Kulturdauer von 26 Tagen.<br />
Die Primacyt Cell Culture Technology GmbH<br />
hat in den vergangenen Jahren ein humanes<br />
Leberzellkultursystem entwickelt. Mit HEPAC 2<br />
werden primäre humane Hepatozyten in einem<br />
chemisch definierten Medium serumfrei über<br />
mehrere Wochen kultiviert, wobei die Zellen<br />
ihre Funktionalität über diesen Zeitraum beibehalten.<br />
Die tägliche Qualitätskontrolle der<br />
Kulturen erfolgt lichtmikroskopisch und über<br />
die Analyse der Vitalität mittels Messung der<br />
Freisetzung des cytoplasmatischen Enzyms<br />
Lactatdehydrogenase sowie der Funktionsparameter<br />
Albumin und Harnstoff 1 . Das System<br />
wird in Wirkstoffprüfungen eingesetzt, die<br />
Übertragbarkeit der gewonnenen Daten auf<br />
die in-vivo-Situation konnte bereits durch eine<br />
klinische Phase II-Studie validiert werden 2 .<br />
In unserem Labor wurde das in einer 6-well-<br />
Kulturplatte entwickelte HEPAC 2 -Langzeitkultursystem<br />
für menschliche Leberzellen auf<br />
die von der In Vitro Systems & Services GmbH<br />
entwickelte lumox TM slide flask übertragen. Die<br />
Hepatozyten wurden in MEM alpha-Medium<br />
ausplattiert. Nach Anheftung wurden die Zellen<br />
in Human Hepatocyte Maintenance-Medium<br />
(HHMM) mit den Wachstumsfaktoren HGF<br />
(Hepatocyte Growth Factor) und EGF (Epidermal<br />
Growth Factor) serumfrei kultiviert. Parallel<br />
dazu wurden die Experimente auf handelsüblichen<br />
6-well-Zellkulturplatten durchgeführt.<br />
Der Zellkulturüberstand wurde täglich komplett<br />
geerntet und zur Analyse der Funktion und<br />
Vitalität der Leberzellen eingesetzt. Zwei Experimente<br />
bestätigten, dass die Abgabe von Harnstoff<br />
durch die Hepatozyten bei der Kultivierung<br />
in lumox TM slide flasks gegenüber der üblichen<br />
6-well-Platte deutlich erhöht ist (Abb. 1). Daher<br />
ist anzunehmen, dass die Zellen in der lumox TM<br />
slide flask einen höheren Differenzierungsgrad<br />
beibehalten können. Die Freisetzung der Lac-<br />
Abb. 2: H+E-Färbung an Tag 26 der Kultur<br />
in lumox TM slide flask, 40-fache Vergrößerung.<br />
tatdehydrogenase zeigte keine signifikanten<br />
Unterschiede zwischen beiden Kulturmethoden<br />
und war über den Kultivierungszeitraum von<br />
fast vier Wochen durchweg niedrig.<br />
Neben der täglichen lichtmikroskopischen<br />
Kontrolle wurde am Ende der Kultivierung eine<br />
Hämatoxylin/Eosin-Färbung durchgeführt.<br />
Hierbei brachte der lumox TM -Folienobjektträger<br />
deutliche Vorteile in der Handhabung<br />
gegenüber den bisher für die Färbung von<br />
Monolayerkulturen verwendeten dünnen<br />
Deckgläschen. Im Anschluss an die Kultur lässt<br />
sich der lumox TM -Objektträger abziehen und<br />
die spezifischen Färbungen direkt auf dem<br />
Objektträger durchführen. Die Morphologie<br />
der Zellen unterschied sich in beiden Kulturmethoden<br />
nicht, soweit dies lichtmikroskopisch<br />
beurteilt werden konnte (Abb. 2).<br />
In weiteren Experimenten ist nun zu untersuchen,<br />
ob die Kultivierung der Leberzellen in<br />
der gasdurchlässigen Objektträgerflasche auch<br />
zur wiederholten Testung von körperfremden<br />
Stoffen eingesetzt werden kann, wie dies mit<br />
HEPAC 2 der Fall ist. Dieses Langzeitzellkultursystem<br />
korreliert mit Ergebnissen aus Phase<br />
II-Studien und stellt ein wichtiges Werkzeug<br />
für die Untersuchung zur Wirkung von Xenobiotika<br />
auf den Menschen dar.<br />
Literatur<br />
[1] Ullrich, A. et al.: Use of a standardized and validated longterm<br />
human hepatocyte culture system for repetitive analyses<br />
of drugs: Repeated administrations of acetaminophen reduces<br />
albumin and urea secretion. ALTEX 2007; 24 (1): 35-40.<br />
[2] Dickens, H. et al.: Anticancer drug cis-4-hydroxy-L-proline: correlation<br />
of preclinical toxicology with clinical parameters of liver<br />
function. Molecular Medicine Reports, accepted for publication<br />
Korrespondenzadresse<br />
Dr. Dieter Runge<br />
PRIMACYT GmbH<br />
Hagenower Str. 73, 19061 Schwerin<br />
Tel.: +49-(0)385-3993-600<br />
Fax: +49-(0)385-3993-602<br />
www.primacyt.de<br />
dieter.runge@primacyt.de<br />
12 | 9. Jahrgang | Nr. 3/2008 LABORWElT
“ N e ve r thought that<br />
real-time PCR could<br />
be so simple!”<br />
Remarkably simple system. Simply remarkable results.<br />
Take your first step into real-time PCR with the afford-<br />
able 48-well StepOne System, and you’ll be surprised<br />
at how easy high performance real-time PCR can be!<br />
Take the second step and upgrade to the 96-well<br />
StepOnePlus System, whenever you are ready.<br />
StepOne System<br />
•<br />
•<br />
•<br />
•<br />
Surprisingly affordable<br />
48 wells for lower throughput demands<br />
3 colours for basic research applications<br />
Easily upgradeable to the StepOnePlus System<br />
It’s remarkable how simple high performance real-time<br />
PCR can be view our free software demonstration at:<br />
info.appliedbiosystems.com/steponeplus<br />
StepOne … a step above the rest!<br />
• High performance real-time PCR<br />
• Remarkably simple to use<br />
• Fast PCR (
Report Tiermodelle<br />
Mausmodelle: Pathogenese<br />
und Therapie-Entwicklung<br />
für Epidermolysis bullosa<br />
Dr. Anja Fritsch, Dr. Johannes Kern, Dr. Stefan Löckermann, Prof. Dr. Leena Bruckner-Tuderman,<br />
Universitäts-Hautklinik Freiburg<br />
Epidermolysis bullosa (EB) ist eine Gruppe seltener, genetisch bedingter Hauterkrankungen,<br />
die durch Blasenbildung und Erosionen an Haut und Schleimhäuten gekennzeichnet sind. Die<br />
zugrundeliegenden Mutationen betreffen Gene, deren Genprodukte wesentlich an der Verankerung<br />
der Epidermis an der darunterliegenden Dermis beteiligt sind. Für viele dieser Gene<br />
existieren bereits Mausmodelle, in denen die Genexpression vollständig inaktiviert wurde.<br />
Diese sind allerdings überwiegend früh nach Geburt letal, was in der humanen Situation nur<br />
bei wenigen EB-Formen der Fall ist. Daher sind sie für eine Analyse der Pathogenese sowie<br />
vor allem für eine Entwicklung kausaler Therapien nur eingeschränkt geeignet. Aus diesem<br />
Grund werden durch konditionelle Inaktivierung der Zielgene durch die Cre-loxP-Technik oder<br />
die Generierung von hypomorphen Allelen neue Mausmodelle etabliert, bei denen die frühe<br />
Letalität des vollständigen Genverlustes vermieden werden kann. Diese stellen gute Modelle der<br />
humanen Erkrankung dar und können zur Analyse der Symptomentwicklung und zur Testung<br />
neuartiger Therapien herangezogen werden.<br />
Abb. 1: Darstellung des Aufbaus des Verankerungskomplexes und der in den verschiedenen<br />
EB-Subtypen auftretenden Mutationen der Keratin-Filamente, Plectin, BP230, Integrina<br />
6 b 4 , Collagen XVII, Laminin-332 und des Kollagen VII als Hauptbestandteil der Verankerungsfibrillen.<br />
Modifiziert nach [1]<br />
Epidermolysis bullosa (EB) bezeichnet eine<br />
Gruppe erblicher Hauterkrankungen, die<br />
durch Fragilität von Haut und Schleimhäuten<br />
und daraus resultierenden Blasen und<br />
Erosionen gekennzeichnet ist. Das Spektrum<br />
der im Allgemeinen schon kurz nach Geburt<br />
auftretenden Symptome reicht von einer<br />
erhöhten Tendenz zu Blasenbildung bei geringer<br />
mechanischer Beanspruchung bis hin<br />
zu schwersten Erkrankungen, die bereits im<br />
Kindesalter tödlich verlaufen. Der EB liegen<br />
Mutationen zugrunde, die die Funktionen<br />
des epidermalen Adhäsionskomplexes – ei-<br />
nes Protein-Netzwerkes an der Basalmembranzone<br />
– beeinträchtigen 1 .<br />
Die Integrität und Funktionalität der Haut<br />
als äußerster Barriere des Körpers ist von Zell-<br />
Zell- und Zell-Matrix-Interaktionen abhängig,<br />
die die sich ständig erneuernde Epidermis stabilisieren<br />
und fest an der darunterliegenden<br />
Dermis verankern, die dem Gewebe mechanische<br />
Stabilität verleiht. Intermediärfilamente<br />
im Zytoskelett der Keratinozyten bilden über<br />
Desmosomen feste Kontakte innerhalb der<br />
Epidermis und werden über hemidesmosomale<br />
Proteine mit der extrazellulären Matrix<br />
verbunden. Wie in Abbildung 1 dargestellt,<br />
fixiert in den Hemidesmosomen ein Proteinkomplex<br />
aus Plektin, Kollagen XVII, BP230 und<br />
Integrin-a 6 b 4 die Intermediärfilamente an der<br />
Plasmamembran der Keratinozyten. Proteine<br />
der extrazellulären Matrix wie Laminin 332<br />
binden an Integrin-a 6 b 4 und werden ihrerseits<br />
durch Verankerungsfibrillen gebunden, die<br />
den Komplex fest an der Dermis verankern 2 .<br />
Die EB wird anhand der Blasenbildungsebene<br />
in drei Gruppen klassifiziert 3 (Abb. 1):<br />
EB simplex (EBS) ist charakterisiert durch<br />
Blasenbildung innerhalb der basalen Keratinozyten<br />
und wird hauptsächlich verursacht<br />
durch Mutationen in den Genen, die für die<br />
Zytoskelett-Proteine Keratin 5 und Keratin 14<br />
kodieren (KRT5, KRT14). Bei der EB junctionalis<br />
(EBJ) bilden sich Blasen unterhalb der basalen<br />
Keratinozyten, entlang der Basalmembran.<br />
Diese Form kann durch eine Vielzahl unterschiedlicher<br />
Mutationen hervorgerufen werden,<br />
zum Beispiel in den Genen für Integrina<br />
6 b 4 (ITGA6, ITGB4), Collagen XVII (COL17A1),<br />
Plektin (PLEC1) sowie den Untereinheiten<br />
von Laminin 332 (LAMA3, LAMB3, LAMC2).<br />
Ursächlich für die EB dystrophica (EBD) sind<br />
Mutationen im Gen für Kollagen VII (COL7A1).<br />
Die Spaltbildung erfolgt unterhalb der Basalmembran,<br />
und die entstehenden Blasen führen<br />
zu Narbenbildung. Das klinische Spektrum<br />
der EBD kann außerdem Pseudosyndaktylie<br />
und die Bildung von Plattenepithelkarzinomen<br />
umfassen 1,4,5 .<br />
EB-Mausmodelle mit konventioneller<br />
Gen-Inaktivierung<br />
Die essentielle Bedeutung vieler Komponenten<br />
des dermal-epidermalen Verankerungskomplexes<br />
wurde in der Vergangenheit durch<br />
Inaktivierung mehrerer beteiligter Gene im<br />
Mausmodell gezeigt. Eine Auswahl der bisher<br />
beschriebenen transgenen Mauslinien<br />
ist in Tabelle 1 zusammengefasst. Auffällig<br />
hierbei ist, dass die murinen Phänotypen<br />
häufig viel stärker ausgeprägt sind und<br />
meist durch Blasenbildung und Erosionen<br />
am ganzen Körper der Tiere gekennzeichnet<br />
sind. Auch die Inaktivierung von Genen, die<br />
beim Patienten keine oder nur eine geringe<br />
Beeinträchtigung der Lebenserwartung mit<br />
sich bringen, ist im Mausmodell in verhältnismäßig<br />
kurzer Zeit nach Geburt letal. Dieser<br />
Unterschied ist höchstwahrscheinlich unter<br />
anderem auch auf die gute medizinische<br />
Versorgung der Neugeborenen im Gegensatz<br />
zu den Versuchstieren zurückzuführen, bedeutet<br />
allerdings, dass die transgenen Tiere<br />
mit konventioneller Gen-Inaktivierung nur<br />
eingeschränkt zur Analyse der Pathogenese<br />
und Symptomentwicklung und zur Therapie-<br />
Testung herangezogen werden können.<br />
Aufgrund dieser Tatsache wurden für einige<br />
EB-Subtypen konditionale Knock-out-Linien<br />
oder Linien mit stark verringerter Genexpres-<br />
14 | 9. Jahrgang | Nr. 3/2008 LABORWElT
Abb. 2: In vivo-Modell für dominante EBS unter Nutzung der inhibitorischen<br />
Effekte der PGK-Neo-Kassette und der Cre-loxP-<br />
Technik (loxP-Sequenzen: rote Dreiecke). Die Verwendung<br />
Hormonrezeptor-gekoppelter Cre-Rekombinasen ermöglicht die<br />
Umgehung des letalen Effektes der Krt14-Inaktivierung (Abb.<br />
modifiziert nach 19).<br />
sion – sogenannte hypomorphe Linien – entwickelt, die eine bessere<br />
Abbildung der humanen Erkrankung erlauben.<br />
In vivo-Modell der dominanten EBS<br />
EBS wird durch Mutationen in den Genen für Keratin 5 und Keratin 14<br />
verursacht. Keratine bilden über ihre zentrale Rod-Domäne Heterodimere,<br />
die dann zu Keratin-Filamenten assembliert werden. Mutationen<br />
innerhalb und insbesondere an den Enden der Rod-Domäne<br />
können über eine Störung der Keratin-Assemblierung dominant<br />
negative Effekte auf die Filament-Stabilität ausüben. Dieser Effekt<br />
wurde in einem eleganten in vivo-Modell nachgestellt 19 , welches sowohl<br />
die Technik der induzierbaren Rekombinase-vermittelten Gen-<br />
Inaktivierung (Cre-loxP-System) als auch die negativen Effekte einer<br />
Selektionskassette auf die Expression des Zielgens nutzt (Abb. 2).<br />
Zunächst wurde die transgene Linie mtK14Neo generiert, bei<br />
der in einem Allel des Keratin 14-Gens eine Punktmutation eingeführt<br />
wurde. Darüberhinaus wurde in Intron 1 eine Kassette zur<br />
Expression der Neomycin-Phosphotransferase unter Kontrolle des<br />
Phosphoglyceratkinase-Promotors (PGK-Neo-Kassette) eingeführt,<br />
die zur Selektion der embryonalen Stammzellen (ES-Zellen) nach<br />
erfolgter homologer Rekombination und Einführung des Transgens<br />
dienen sollte und deren Cre-Rekombinase-vermittelte Exzision über<br />
flankierende loxP-Sequenzen ermöglicht wurde (Abb. 2). Diese PGK-<br />
Neo-Kassette interferierte allerdings stark mit der Expression des<br />
mutierten Allels von Keratin 14. Wurde diese in den transfizierten ES-<br />
Zellen durch die Aktivität der Cre-Rekombinase entfernt, so zeigten<br />
die resultierenden Mäuse einen dominant vererbten, EBS-ähnlichen<br />
Phänotyp mit schwerer Blasenbildung und starben innerhalb der<br />
ersten Lebenswoche. Damit konnte gezeigt werden, dass die Expression<br />
eines mutierten Keratin 14-Allels dieselben Folgen hat wie eine<br />
vollständige Geninaktivierung, allerdings dominant vererbt wird und<br />
damit die humane Situation widerspiegelt.<br />
Die transgene mtK14Neo-Linie bot jedoch auch die Möglichkeit,<br />
die Exzision der PGK-Neo-Kassette nicht in ES-Zellen vorzunehmen,<br />
sondern die Tiere mit einer Mauslinie zu kreuzen, die ein Fusionsprotein<br />
aus Cre-Rekombinase und einem Hormonrezeptor exprimiert. In<br />
diesem Fall wurde eine Linie verwendet, die eine Cre-Rekombinase-<br />
Progesteron-Rezeptor-Fusion trägt, die unter Kontrolle des Keratin<br />
5-Promotors steht und damit in den basalen Keratinozyten produziert<br />
wird. Die topische Applikation des Hormons auf die Pfoten der doppelt<br />
transgenen Tiere führte zur Translokation des Fusionsproteins<br />
Report Tiermodelle<br />
LABORWElT 9. Jahrgang | Nr. 3/2008 | 15
Report Tiermodelle<br />
vom Zytoplasma in den Zellkern, und dort<br />
wurde die PGK-Neo-Kassette entfernt und<br />
die Expression des mutierten Keratin 14<br />
ermöglicht. Dies führte zu Blasenbildung<br />
ausschließlich in den Hormon-behandelten<br />
Arealen, was ein Überleben der Tiere erlaubte.<br />
Folgestudien zeigten unter anderem, dass<br />
die Blasen nach Ende der Hormon-Behandlung<br />
schnell abheilten, was wahrscheinlich<br />
auf eine Einwanderung von epidermalen<br />
Stammzellen in die betroffenen Areale zurückzuführen<br />
ist.<br />
Mit diesem in vivo-Modell der dominanten<br />
EBS steht nun ein überlebensfähiges Tiermodell<br />
der Erkrankung zur Verfügung, welches<br />
die Analyse der Symptompathogenese und<br />
eine Therapieentwicklung ermöglicht.<br />
Der Kollagen VII-Hypomorph als<br />
Modell für EBD<br />
Die Möglichkeit, mit Hilfe einer intronischen<br />
PGK-Neo-Kassette die Expression eines Gens<br />
stark zu verringern, haben wir vor kurzem<br />
auch zur Generierung eines in vivo-Modells<br />
für EBD genutzt 20 . Diese EB-Form kann – abhängig<br />
von der zugrundeliegenden Mutation<br />
im Gen für Kollagen VII (COL7A1) – dominant<br />
oder rezessiv vererbt werden. Kollagen VII als<br />
Hauptbestandteil der Verankerungsfibrillen<br />
ist ein 290 kDa großes Protein mit einer sehr<br />
langen zentralen Tripelhelix, die von zwei<br />
globulären Domänen flankiert wird. Die<br />
schwerwiegendsten klinischen Symptome<br />
werden durch rezessive Mutationen hervorgerufen,<br />
die zu einer starken Reduktion der<br />
Kollagen VII-Synthese bis hin zum vollständigen<br />
Fehlen des Proteins in der Haut führen.<br />
Die betroffenen Patienten leiden nicht nur<br />
unter einer stark erhöhten Tendenz zu Blasenbildung,<br />
sondern entwickeln auch bereits<br />
im Kindesalter starke Narben und schwerwiegende<br />
Mutilationen an den Händen und<br />
Füßen. Außerdem zeigen sie eine erhöhte<br />
Inzidenz von Plattenepithelkarzinomen, die<br />
sich bevorzugt an stark von Blasenbildung<br />
betroffenen Arealen entwickeln und deren<br />
Metastasen die häufigste Todesursache<br />
darstellen 1 .<br />
Zur Generierung eines langlebigen in<br />
vivo-Modells der rezessiven EBD wurde eine<br />
konditionale Inaktivierung des Col7a1-Gens<br />
angestrebt und Exon 2 mit zwei loxP-Sequenzen<br />
flankiert. Diese erlauben die Cre-<br />
Rekombinase-vermittelte Exzision dieses<br />
Exons, was zu einer Leserasterverschiebung<br />
und Generierung von Stop-Codons führt,<br />
die die Proteinsynthese abbrechen. Gleichzeitig<br />
wurde auch eine PGK-Neo-Kassette<br />
Abb. 3: In vivo-Modell der rezessiven EBD. Die Einführung der PGK-Neo-Kassette führt zu stark<br />
verringerter Expression von Kollagen VII (Immunofärbung im Panel „Phänotyp“), was<br />
Blasenbildung verursacht (gekennzeichnet mit Sternchen). Die intradermale Injektion<br />
von Wild-Typ-Fibroblasten kann diesen Phänotyp teilweise revertieren.<br />
zur Selektion der ES-Zellen in Intron 2<br />
eingeführt (Abb. 3). Nach Etablierung der<br />
entsprechenden transgenen Linie konnte<br />
gezeigt werden, dass die PGK-Neo-Kassette<br />
einen starken Einfluß auf die Prozessierung<br />
der Kollagen VII-mRNA hat und die Proteinexpression<br />
auf ca. 10% des Wildtypniveaus<br />
senkt. Die Kollagen VII-hypomorphe Maus<br />
zeigte Blasenbildung der Haut und der<br />
Schleimhäute als klinische Symptome. Die<br />
verbliebenen Kollagen VII-Mengen reichten<br />
aber aus, um die Lebenserwartung der Tiere<br />
gegenüber der Situation bei konventioneller<br />
Gen-Inaktivierung 19 wesentlich zu erhöhen,<br />
auf bis zu sechs Monate. Innerhalb dieses<br />
Zeitraums konnte auch die Entstehung anderer<br />
Symptome der EBD beobachtet und<br />
analysiert werden, vor allem die Bildung von<br />
Mutilationen an den Extremitäten. Es konnte<br />
gezeigt werden, dass die Entwicklung der<br />
Mutilationen auf entzündlichen und darauf<br />
folgenden fibrotischen Prozessen beruht 20 .<br />
Dies ermöglicht es nun, therapeutische Maßnahmen,<br />
wie etwa anti-inflammatorische<br />
oder anti-fibrotische Behandlung der Extremitäten,<br />
in diesem Tiermodell zu testen.<br />
Außerdem eignen sich diese hypomorphen<br />
Mäuse auch für die Testung der Anfälligkeit<br />
für Tumorentstehung, da die fibrotisch veränderten<br />
Pfoten im Zeitverlauf beobachtet<br />
und analysiert werden können.<br />
Aus Sicht der betroffenen Patienten ist die<br />
Testung neuartiger biologisch begründeter<br />
Therapieansätze für EBD am wichtigsten. Die<br />
Effizienz einer Form der Substitutionstherapie<br />
konnte anhand dieses hypomorphen<br />
Mausmodells bereits gezeigt werden. Werden<br />
Fibroblasten aus Tieren mit normaler Kollagen<br />
VII-Synthese in die Rückenhaut der hypomorphen<br />
Tiere injiziert, so wird die Deponierung<br />
von Kollagen VII an der Basalmembran<br />
verstärkt. Außerdem ist bereits sieben Tage<br />
nach der Injektion die Resistenz der Haut<br />
gegenüber mechanischem Stress deutlich<br />
erhöht 20 . Dies zeigt, dass in diesem in vivo-<br />
Modell für EBD eine Analyse der Symptompathogenese<br />
und auch die Entwicklung und<br />
Validierung von Therapieansätzen möglich<br />
ist. Darüber hinaus kann diese transgene Linie<br />
nach Rekombinase-vermittelter Entfernung<br />
der PGK-Neo-Kassette genutzt werden, um<br />
einen konditionalen Knock-out von Kollagen<br />
VII durch Verwendung von Hormonrezeptorgekoppelten<br />
Cre-Rekombinase-Varianten<br />
zu erhalten. Damit wird auch die topische<br />
Gen-Inaktivierung möglich, was es erlaubt,<br />
das Auftreten und die Entwicklung klinischer<br />
Sympotme in einzelnen Geweben zu<br />
beobachten, zum Beispiel der Haut ohne<br />
Beteiligung der Schleimhäute.<br />
Zusammenfassung und Ausblick<br />
Anhand der dargestellten Tiermodelle für<br />
die verschiedenen Formen der Epidermolysis<br />
16 | 9. Jahrgang | Nr. 3/2008 LABORWElT
Tab. 1: Mausmodelle für verschiedene EB-Formen<br />
EB-Subtyp Zielgen Änderung Phänotyp<br />
EBS Krt14 Expression einer verkürzten Keratin-14-Variante Blasenbildung innerhalb der basalen<br />
unter Kontrolle d. endogenen Promoters Keratinozyten, perinatal letal6 Krt14 Ersatz von Krt14 durch eine Vimentin-cDNA Blasenbildung innerhalb der basalen<br />
Keratinozyten, perinatal letal7 Plec Inaktivierung perinatal letal8 bullosa wird deutlich, dass konventionelle<br />
Gen-Inaktivierungen zwar die Bedeutung des<br />
Zielgens für die Funktionen der Haut zeigen<br />
können, darüber hinaus aber als Modelle<br />
für die Erkrankungen nur bedingt geeignet<br />
sind.<br />
Insbesondere die häufig schwerwiegenderen<br />
Phänotypen der transgenen Tiere<br />
im Vergleich zu den klinischen Symptomen<br />
der Patienten machen direkte Vergleiche<br />
und Analysen der Symptompathogenese<br />
schwierig. Die kurze Lebenserwartung der<br />
Knock-out-Tiere macht es außerdem unmöglich,<br />
die Entstehung sekundärer Symptome<br />
einer Erkrankung, wie etwa die Mutilationen<br />
und Tumorbildung bei rezessiver EBD, zu<br />
untersuchen.<br />
Langfristig werden sicherlich auch für weitere<br />
Formen der EB Tiermodelle geschaffen<br />
werden, die eine konditionale Inaktivierung<br />
der Zielgene in einem bestimmten Zelltyp<br />
oder Gewebe areal erlauben. Daneben stellt<br />
auch die Verwendung der PGK-Neo-Kassette<br />
zur Generierung von hypomorphen Tieren<br />
eine elegante Möglichkeit dar, die letalen<br />
Auswirkungen eines vollständigen Fehlens<br />
von Proteinen zu mildern und so überlebensfähige<br />
in vivo-Modelle für humane<br />
Erkrankungen zu erhalten.<br />
Literatur<br />
Plec Inaktivierung via Cre-loxP-system mit Cre-Expression<br />
unter Kontrolle des Keratin 15-Promotors<br />
[1] Has, C., Bruckner-Tuderman, L., J Dermatol Sci 44 (2006),<br />
129-144.<br />
[2] Aumailley, M., Has, C., Tunggal, L., Bruckner-Tuderman, L.,<br />
Expert Rev Mol Med 8 (2006), 1-21.<br />
[3] Fine, J. D., Eady, R. A., Bauer, E. A., Bauer, J. W., Bruckner-<br />
Tuderman, L., Heagerty, A., Hintner, H., Hovnanian, A.,<br />
Jonkman, M. F., Leigh, I., McGrath, J. A., Mellerio, J. E.,<br />
Murrell, D. F., Shimizu, H., Uitto, J., Vahlquist, A., Woodley,<br />
D., Zambruno, G., J Am Acad Dermatol (2008),<br />
in press<br />
[4] Varki, R., Sadowski, S., Pfendner, E., Uitto, J., J Med Genet<br />
43 (2006), 641-652.<br />
Blasenbildung der Haut, perinatal<br />
letal 9<br />
EBJ Itgb4 Inaktivierung Blasenbildung, perinatal letal 10,11<br />
Itgb4 Deletion d. cytoplasmat. Teils d. b4-Integrins Blasenbildung, perinatal letal12 Itga6 Inaktivierung Blasenbildung, perinatal letal13 Col17a1 Ersetzen von Exon 2 durch PGK-Neo-Kassette Blasenbildung, ca. 20% der Tiere<br />
überleben die ersten 8 Wochen14 BP230 Ersetzen der Exons1-3 durch PGK-Neo-Kassette Blasenbildung nach mechanischem<br />
Stress, motorische Defekte15 Lama3 Inaktivierung Blasenbildung, perinatal letal16 Lamc2 Inaktivierung Blasenbildung, perinatal letal17 EBD Col7a1 Inaktivierung Blasenbildung, letal innerhalb der<br />
ersten 14 Tage18 [5] Varki, R., Sadowski, S., Uitto, J., Pfendner, E., J Med Genet 44<br />
(2007), 181-192.<br />
[6] Vassar, R., Coulombe, P. A., Degenstein, L., Albers, K., Fuchs,<br />
E., Cell 64 (1991), 365-380.<br />
[7] Lloyd, C., Yu, Q. C., Cheng, J., Turksen, K., Degenstein, L.,<br />
Hutton, E., Fuchs, E., J Cell Biol 129 (1995), 1329-1344.<br />
[8] Andra, K., Lassmann, H., Bittner, R., Shorny, S., Fassler, R.,<br />
Propst, F., Wiche, G., Genes Dev 11 (1997), 3143-3156.<br />
[9] Ackerl, R., Walko, G., Fuchs, P., Fischer, I., Schmuth, M., Wiche,<br />
G., J Cell Sci 120 (2007), 2435-2443.<br />
[10] Dowling, J., Yu, Q. C., Fuchs, E., J Cell Biol 134 (1996),<br />
559-572.<br />
[11] van der, N. R., Krimpenfort, P., Calafat, J., Niessen, C. M.,<br />
Sonnenberg, A., Nature Genetics 13 (1996), 366-<br />
369.<br />
[12] Murgia, C., Blaikie, P., Kim, N., Dans, M., Petrie, H. T., Giancotti,<br />
F. G., EMBO J 17 (1998), 3940-3951.<br />
[13] Georges-Labouesse, E., Messaddeq, N., Yehia, G., Cadalbert,<br />
L., Dierich, A., Le, M. M., Nat Genet 13 (1996), 370-373.<br />
[14] Nishie, W., Sawamura, D., Goto, M., Ito, K., Shibaki, A., Mc-<br />
Millan, J. R., Sakai, K., Nakamura, H., Olasz, E., Yancey, K. B.,<br />
Akiyama, M., Shimizu, H., Nat Med 13 (2007), 378-383.<br />
[15] Guo, L., Degenstein, L., Dowling, J., Yu, Q. C., Wollmann, R.,<br />
Perman, B., Fuchs, E., Cell 81 (1995), 233-243.<br />
[16] Ryan, M. C., Lee, K., Miyashita, Y., Carter, W. G., J Cell Biol<br />
145 (1999), 1309-1323.<br />
[17] Meng, X., Klement, J. F., Leperi, D. A., Birk, D. E., Sasaki,<br />
T., Timpl, R., Uitto, J., Pulkkinen, L., J Invest Dermatol 121<br />
(2003), 720-731.<br />
[18] Heinonen, S., Mannikko, M., Klement, J. F., Whitaker-<br />
Menezes, D., Murphy, G. F., Uitto, J., J Cell Sci 112 ( Pt 21)<br />
(1999), 3641-3648.<br />
[19] Cao, T., Longley, M. A., Wang, X. J., Roop, D. R., J Cell Biol 152<br />
(2001), 651-656.<br />
[20] Fritsch, A., Loeckermann, S., Kern, J. S., Braun, A., Bosl,<br />
M. R., Bley, T. A., Schumann, H., von, E. D., Paul, D.,<br />
Erlacher, M., Berens von Rautenfeld, D., Hausser, I., Fassler,<br />
R., Bruckner-Tuderman, L., J Clin Invest (2008), in press<br />
Korrespondenzadresse<br />
Prof. Dr. Leena Bruckner-Tuderman<br />
Universitäts-Hautklinik Freiburg<br />
Molekulare Dermatologie<br />
Hauptstr. 7<br />
79104 Freiburg<br />
Tel.: +49-(0)761-270 6716<br />
Fax: +49-(0)761-270 6936<br />
bruckner-tuderman@uniklinik-freiburg.de<br />
www.uniklinik-freiburg.de<br />
Report Tiermodelle<br />
Quantitative<br />
miRNA detection<br />
in less than<br />
one day<br />
miRtect-IT miRNA<br />
Labeling and Detection Kit<br />
Based on splinted-ligation technology<br />
– miRNA is directly labeled by ligation,<br />
then visualized.<br />
• Speed - capture and label miRNA in<br />
just over 2 hours<br />
• Sensitivity - detect miRNA in as little as<br />
50 ng or less of total RNA<br />
• Quantitative Results - accurate miRNA<br />
measurement in 6 hours<br />
Now in Europe<br />
USB Europe GmbH<br />
Hauptstrasse 1<br />
79219 Staufen<br />
Germany<br />
T: +49 (0)76 33-933 40 0<br />
F: +49 (0)76 33-933 40 20<br />
www.usbweb.com<br />
custserv@usbweb.de<br />
LABORWElT 9. Jahrgang | Nr. 3/2008 | 17
��������������������������������������������<br />
����<br />
��������������������<br />
Report Immunbiologie<br />
������<br />
��������<br />
���������������������������������������������������<br />
����������������������<br />
����������������������������������������<br />
����������������������������������<br />
��������������������������������<br />
�������������������������������������<br />
���������������������������������<br />
�����������������������������������<br />
�����������������������������������<br />
������������������������������<br />
���������������������������������<br />
���������������������������������<br />
������������������������������������<br />
�������������������������������������<br />
�����������������������������������<br />
�������<br />
������������������������������������<br />
BIOCOM Verlag GmbH<br />
����������������������<br />
����������������������<br />
�������������<br />
�����������������<br />
������������������������<br />
�����������������������<br />
Treg, Autoimmunität und<br />
CD28-superagonistische<br />
Antikörper<br />
Prof. Dr. Holger M. Reichardt und Dr. Denise Tischner, Universitätsmedizin Göttingen;<br />
Dr. Nora Müller, Universität Würzburg<br />
Autoimmunerkrankungen wie Multiple Sklerose beruhen auf einer Fehlsteuerung des Immunsystems<br />
mit der Folge eines Angriffs auf körpereigene Zellen. Regulatorische T-Zellen<br />
(Treg) zeigen bei der Behandlung solcher Krankheiten neue Perspektiven auf, obgleich ihre<br />
Anwendung beim Menschen mit ernsthaften Schwierigkeiten verbunden ist. Die Infusion<br />
CD28-super agonistischer Antikörper hat sich in verschiedenen Tiermodellen als sehr erfolgreich<br />
erwiesen, ein erster klinischer Test musste aber aufgrund schwerer Nebenwirkungen<br />
abgebrochen werden. In Ratten werden zwei Phasen der T-Zellaktivierung beobachtet. Während<br />
die Lymphozyten kurz nach Infusion des Antikörpers massive Veränderungen in ihrer<br />
Morphologie, Gewebeverteilung und Beweglichkeit erfahren, kommt es nach wenigen Tagen<br />
zur selektiven Vermehrung und Aktivierung der Treg-Zellen. Dagegen tritt beim Menschen<br />
zusätzlich ein massiver Zytokinsturm auf, der eine Anwendung bei Patienten bis auf weiteres<br />
ausschließt. Gleichwohl wird es in Zukunft wichtig sein, das therapeutische Potential von<br />
Treg-Zellen für die Klinik weiter nutzbar zu machen.<br />
Autoimmunkrankheiten haben ihre Basis<br />
in einer Dysregulation des Immunsystems,<br />
wenn Lymphozyten plötzlich damit beginnen,<br />
körpereigene Zellen anzugreifen. Die Therapie<br />
beschränkt sich meist drauf, die autoaggressiven<br />
Zellen in Schach zu halten, wobei dies auf<br />
Kosten teils schwerer Nebenwirkungen und<br />
oft nur mit geringer Effizienz erfolgt. Neue<br />
Behandlungsstrategien zielen daher darauf<br />
ab, einen natürlich vorkommenden Mechanismus<br />
auszunutzen. Bereits vor Jahren wurde<br />
erkannt, dass regulatorische T-Zellen (Treg) sowohl<br />
bei Mäusen als auch beim Menschen dem<br />
Auftreten von Autoimmunreaktionen entgegenwirken.<br />
Aufbauend auf dieser Erkenntnis<br />
wurden seitdem verschiedene Ansätze entwickelt,<br />
um durch Transfer von Treg-Zellen oder<br />
durch deren Vermehrung direkt im Patienten<br />
den schädlichen Wirkungen autoaggressiver<br />
Lymphozyten entgegenzuwirken 1 .<br />
Regulatorische T-Zellen und ihre<br />
therapeutische Anwendung<br />
Treg-Zellen spielen eine wichtige Rolle im Rahmen<br />
pathogener und protektiver Immunreaktionen.<br />
Generell wird zwischen induzierten<br />
und natürlichen Treg-Zellen unterschieden. Zu<br />
ersteren zählen sowohl IL-10-produzierende Tr1<br />
Zellen als auch TGFb-sezernierende TH3-Zellen,<br />
deren Anreicherung nach oraler Verabreichung<br />
von Antigen beobachtet wurde. Im Gegensatz<br />
dazu entwickeln sich natürliche Treg-Zellen<br />
im Thymus und exprimieren den Transkriptionsfaktor<br />
FoxP3. Mutationen im FoxP3-Gen<br />
führen zu einem Funktionsverlust der natür-<br />
lichen Treg-Zellen, was beim Menschen eine<br />
multisymptomatische lymphoproliferative<br />
Erkrankung namens IPEX (Immundysregulation,<br />
Polyendocrinopathy, Enteropathy, Xlinked)<br />
zur Folge hat. Darüber hinaus wurden<br />
auch bei Autoimmunerkrankungen wie der<br />
Multiplen Sklerose (MS) ein Funktionsverlust<br />
oder eine verminderte Anzahl an Treg-Zellen<br />
beobachtet. Die Aktivierung von Treg-Zellen<br />
oder eine Verschiebung ihres Verhältnisses<br />
zu Effektorzellen stellt daher einen potentiell<br />
sehr interessanten Therapieansatz für solche<br />
Erkrankungen dar. Vorteile liegen insbesondere<br />
in der Möglichkeit zur antigenspezifischen<br />
Behandlung ohne gleichzeitige allgemeine<br />
Immunsuppression, der langanhaltenden<br />
physiologischen Regulation in vivo, sowie der<br />
Option zur Entwicklung patientenspezifischer<br />
Therapien. Prinzipiell kann die Aktivierung und<br />
Expansion von Treg-Zellen entweder in vivo im<br />
Patienten oder aber in vitro mit anschließendem<br />
Transfer geschehen.<br />
Treg-Expansion in vitro und in vivo<br />
Bei der Expansion von Treg-Zellen in vitro gibt es<br />
eine Reihe von Problemen, wie deren Isolation<br />
aus humanem peripherem Blut. Zum einen<br />
beträgt der Anteil der Treg-Zellen nur 5-10%<br />
der CD4 + -T-Lymphozyten, zum anderen ist bis<br />
heute kein wirklich spezifischer extrazellulärer<br />
Marker für diese Zellpopulation identifiziert.<br />
FoxP3 kommt als Sortierungskriterium nicht in<br />
Frage, da es sich hier um einen intrazellulären<br />
Transkriptionsfaktor handelt. Zudem wurde<br />
berichtet, dass FoxP3 beim Menschen nach Ak-<br />
18 | 9. Jahrgang | Nr. 3/2008 LABORWElT
tivierung auch in konventionellenCD4 + -T-Zellen<br />
induziert wird und somit dessen Expression<br />
für Treg-Zellen keineswegs spezifisch ist. Die<br />
derzeit effizienteste Aufreinigung wird mittels<br />
Leukapherese und anschließender magnetischer<br />
Aufreinigung der Treg-Zellen aus der Leukozytenfraktion<br />
auf Basis ihrer hohen CD25- und<br />
ihrer geringen CD127-Expression erreicht.<br />
Ein zweites Problem betrifft die Notwendigkeit,<br />
Treg-Zellen in vitro ausreichend stark<br />
zu expandieren. Hierbei besteht die Gefahr,<br />
dass auch kontaminierende CD25 + -Effektor-T-<br />
Zellen proliferieren. Dies würde im Falle eines<br />
Dendritic cell<br />
CD80/CD86<br />
CD80/CD86<br />
Peptide-MHC<br />
class II<br />
inzwischen deutlich reduziert werden. Eine<br />
Phase I-Studie zur Etablierung der Sicherheit von<br />
CD28-superagonistischen Antikörpern musste<br />
dagegen aufgrund massiver Nebenwirkungen<br />
abgebrochen werden, so dass ein weiterer<br />
Einsatz im Menschen bis auf weiteres nicht<br />
abzusehen ist.<br />
Während beim Menschen bislang nur mit<br />
polyklonalen Treg-Zellen gearbeitet wurde, hat<br />
sich in einigen Tiermodellen gezeigt, dass bereits<br />
etablierte Autoimmunerkrankungen nur durch<br />
Transfer antigenspezifischer Treg-Zellen therapiert<br />
werden können. Beim Menschen stellt sich<br />
TCR Signal 1<br />
Signal 2:<br />
Co-inhibition<br />
Naive T H cell<br />
CD28 Signal 2: Co-stimulation<br />
CTLA-4<br />
No response Response:<br />
T-cell activation<br />
Abb. 1: Um T-Zellen zu aktivieren, ist normalerweise eine Co-Stimulation des T-Zellrezeptors<br />
(TCR/CD3) und von CD28 erforderlich. TGN1412 bindet anders an CD28 als das natürliche<br />
Substrat CD80/86 und benötigt daher keine TCR-Bindung zur T-Zell-Aktivierung.<br />
adoptiven Transfers unter Umständen zu einer<br />
Verschlechterung des Krankheitsverlaufes<br />
führen. Um dies zu vermeiden, können Treg-<br />
Zellen in Anwesenheit von Rapamycin kultiviert<br />
werden, was die Proliferation von Effektorzellen<br />
spezifisch inhibiert. Die in vitro-Expansion<br />
der Treg-Zellen wird meist durch klassische<br />
Ko-Stimulation mittels an Beads gekoppelter<br />
monoklonaler Antikörper gegen CD3 und CD28<br />
in Gegenwart hoher Interleukin-2 (IL-2)-Dosen<br />
erreicht. Neben den methodischen Problemen<br />
stellt die Technik der in vitro-Expansion einen<br />
sehr zeit- und kostenaufwendigen Ansatz dar,<br />
insbesondere mit Blick auf die Notwendigkeit<br />
von GMP-Bedingungen. Weiterhin ist bislang<br />
ungeklärt, wie lange Treg-Zellen nach Rücktransfer<br />
im Patienten überleben und inwieweit ihre<br />
Suppressionsfähigkeit erhalten bleibt. Verlässliche<br />
Verfahren zur in vivo-Aktivierung und Expansion<br />
von Treg-Zellen wären daher vorteilhaft.<br />
Im Tiermodell ist es bereits heute möglich, Treg-<br />
Zellen durch gleichzeitige Verabreichung von<br />
Dexamethason und IL-2, durch Infusion niedriger<br />
Dosen eines anti-CD3-Antikörpers sowie mittels<br />
CD28-superagonistischer Antikörper selektiv zu<br />
vermehren. Im Falle von anti-CD3-Antikörpern<br />
wurden positive Effekte auf den Verlauf von<br />
Autoimmunerkrankungen auch in klinischen<br />
Studien nachgewiesen 2 . Die ursprünglich aufgetretenen<br />
schädlichen Begleiterscheinungen<br />
(Cytokine release syndrome) konnten durch<br />
Modifikation der verwendeten Antikörper<br />
���������������������������������������<br />
�������������������������������������������������������������������<br />
�����������������������������������������������������������������������<br />
���������������������������������������������������������������������<br />
���������������������������������������������������������������������<br />
��������� ���������������� ���� ���� ������� ���������������� ����������<br />
������������������������������������������������������������������<br />
��������������������������������������������������������������������<br />
������������ ��������� ���������������� ���� ���������������������������<br />
��������������������������������������������������������������������<br />
������������������������������������������������������������<br />
�������������������<br />
hier das Problem, dass die auslösenden Antigene,<br />
beispielsweise bei MS, häufig unbekannt sind.<br />
Darüber hinaus würde die geringe Anzahl antigenspezifischer<br />
Treg-Zellen im Blut deren Gewinnung<br />
und Expansion in vitro wahrscheinlich<br />
stark erschweren. Bei Krankheitsbildern, denen<br />
hingegen verschiedene Antigene zugrunde liegen,<br />
wie beispielsweise IPEX oder GvHD, ist der<br />
Transfer polyklonaler Treg-Zellen in Bezug auf<br />
die Wirkspezifität sogar vorteilhaft.<br />
CD28-superagonistische Antikörper<br />
Normalerweise benötigen T-Zellen zwei Signale,<br />
um durch Antigene aktiviert zu werden (Abb. 1).<br />
Kommt es zum Kontakt einer Peptid-beladenen<br />
Antigen-präsentierenden Zelle (APC) mit einer<br />
naiven T-Zelle, so wird sowohl der T-Zellrezeptor<br />
(TCR, CD3) als auch das kostimulatorische Molekül<br />
CD28 aktiviert. Nur wenn beide Signale<br />
gleichzeitig vorliegen, wird die T-Zelle zur Teilung<br />
und Differenzierung angeregt. Anders verhält es<br />
sich hingegen bei Stimulation mit CD28-superagonistischen<br />
Antikörpern. Durch die besondere<br />
Art und Weise, wie sie das CD28-Molekül auf<br />
Zellen binden und quervernetzen, reicht dieses<br />
Signal allein bereits aus, um T-Zellen effizient zu<br />
stimulieren 3 . Die Notwendigkeit eines zweiten Signals<br />
wird umgangen, wenngleich die Expression<br />
des T-Zellrezeptors (CD3g) per se erforderlich ist.<br />
Auf diese Weise ist es möglich, T-Zellen sowohl<br />
Report Immunbiologie<br />
������ �����<br />
�������� ��������<br />
�����������������������������������������<br />
��������������������<br />
����������������������������������������<br />
����������������������������������������������������������<br />
�����������������������������������������������������������<br />
��������������������������������<br />
������������������������������������<br />
���������������������������������<br />
��������������������������������<br />
�������������������������������<br />
�����������������������������<br />
�����������������������������<br />
���������������������������������<br />
�������������������������������<br />
�����������������������������������<br />
���������������������������������<br />
���������������������������������<br />
������������������������������������<br />
����������������������������������<br />
��������������������������������<br />
����������������������������������<br />
����������������������������������<br />
�����������������������������������<br />
������������������������������<br />
�������������������������������<br />
����������<br />
���������������������������������<br />
��������������<br />
�������<br />
�����������������������������<br />
����������������������������������<br />
BIOCOM AG Verlag GmbH<br />
����������������������<br />
����������������������<br />
����������������������<br />
����������������������<br />
�������������<br />
�������������<br />
�����������������<br />
�����������������<br />
������������������������<br />
������������������������<br />
�����������������������<br />
�����������������������<br />
LABORWElT 9. Jahrgang | Nr. 3/2008 | 21<br />
����<br />
���������������������������������������<br />
���������������������������������������
Report Immunbiologie<br />
A B C<br />
Abb. 2: Der CD28-superagonistische Antikörper JJ316 bewirkt innerhalb von 12 Stunden nach<br />
Infusion in Ratten eine transiente Veränderung der Morphologie und Größe der<br />
T-Zellen. Darstellung des F-Aktin-Zytoskeletts mittels konfokaler Mikroskopie nach<br />
Färbung mit Phalloidin-Alexa 488. A: T-Zellen vor Antikörperinjektion, B: 12 Stunden<br />
nach Injektion, C: 24 Stunden nach Injektion<br />
in vitro als auch in vivo mit nur einem einzigen<br />
Antikörper zu aktivieren.<br />
Die Fähigkeit T-Zellen direkt zu stimulieren,<br />
ist nicht das einzig Besondere an CD28superagonistischen<br />
Antikörpern. Eine weitere<br />
erstaunliche Eigenschaft ist, dass sie verschiedene<br />
Arten von T-Zellen unterschiedlich gut<br />
zu stimulieren vermögen. Diese Antikörper<br />
sprechen auf längere Sicht Treg-Zellen besser<br />
als konventionelle CD4 + -T-Helferzellen<br />
und andere Lymphozyten an. Folglich haben<br />
CD28-superagonistische Antikörper das Potential,<br />
Treg-Zellen in vivo innerhalb weniger<br />
Tage spezifisch zu expandieren und damit das<br />
Gleichgewicht zugunsten dieser Subpopulation<br />
zu verschieben 4 . Dies wiederum hat sich in<br />
verschiedenen Tiermodellen als therapeutisch<br />
äußerst effizient bei der Behandlung von Autoimmunerkrankungen<br />
erwiesen.<br />
CD28-superagonistischen Antikörpern<br />
wurde eine große Zukunft in der Therapie<br />
zahlreicher Krankheiten vorhergesagt. Präklinische<br />
Studien hatten gezeigt, dass sowohl die<br />
prophylaktische als auch die therapeutische<br />
Behandlung mit JJ316, einem Ratten-spezifischen<br />
CD28-superagonistischen Antikörper,<br />
zu einer signifikanten Reduktion der klinischen<br />
Symptome der Experimentellen Autoimmun-<br />
Enzephalomyelitis (EAE), dem Tiermodell<br />
der MS, führt 5,6 . Ähnliche Ergebnisse wurden<br />
für die Rheumatische Arthritis und den Typ<br />
I-Diabetes Mellitus erhalten. Die positive Wirkung<br />
des Antikörpers wurde vornehmlich den<br />
Treg-Zellen zugeschrieben, da sich der Transfer<br />
solcher Zellen ähnlich auswirkt wie die direkte<br />
Gabe des Antiköpers selbst. Auch wenn dies<br />
Schnelle Effekte CD28-super agonistischer<br />
Antikörper in der Ratte (< 24 Stunden)<br />
Kurzzeitige starke Verminderung der Beweglichkeit<br />
der T-Zellen<br />
möglicherweise nicht der einzige Mechanismus<br />
ist, so bleibt die erstaunliche Effizienz bei der<br />
Behandlung von Autoimmunreaktionen im<br />
Tiermodell unumstritten.<br />
Scheitern im Erstversuch<br />
Im März 2006 erlitten die großen Hoffnungen,<br />
die auf CD28-superagonistischen Antikörpern<br />
ruhten, einen jähen Rückschlag 7 . Sechs Probanden<br />
erhielten im Rahmen einer Phase I-Studie<br />
eine Infusion des humanisierten superagonistischen<br />
CD28-Antikörpers TGN1412. Anders als<br />
bei den zuvor durchgeführten präklinischen<br />
Studien kam es innerhalb kürzester Zeit zum<br />
Auftreten einer fulminanten systemischen<br />
Entzündungsreaktion, gekennzeichnet durch<br />
die Freisetzung großer Mengen an Zytokinen<br />
wie TNFa und IFNg, sowie zum Auftreten einer<br />
massiven Lymphopenie. Die Folgeerscheinungen<br />
des Zytokinsturms normalisierten sich nach<br />
Gabe von Methylprednisolon und monoklonalen<br />
anti-IL-2-Antikörpern sowie intensivmedizinischer<br />
Betreuung im Laufe der folgenden Tage<br />
wieder, so dass die Probanden die Klinik nach<br />
einigen Wochen verlassen konnten.<br />
Biphasische Wirkungen von<br />
CD28-superagonistischen Antikörpern<br />
Weshalb waren in der klinischen Studie von<br />
TGN1412 Wirkungen aufgetreten, die in vergleichbarer<br />
Weise im Tiermodell zuvor nie beobachtet<br />
worden waren? Was das Auslösen eines<br />
Tab. 1: Biphasische Wirkungen CD28-superagonistischer Antikörper in der Ratte<br />
verzögerte Effekte CD28-superagonistischer<br />
Antikörper in der Ratte (>24 Stunden)<br />
Selektive Expansion und Aktivierung der Treg-Zellen<br />
Aktivierung des F-Aktin-Zytoskeletts der Lymphozyten Selektive Veränderung der Morphologie und Größe<br />
der Treg-Zellen<br />
Induktion einer T-Lymphopenie Erhöhte Beweglichkeit der Treg-Zellen<br />
Verstärkte Expression von Aktivierungsmarkern auf<br />
T-Zellen<br />
Erhöhte Adhäsion der T-Zellen<br />
Effiziente Therapie und Prophylaxe von Autoimmunreaktionen<br />
Zytokinsturms durch TGN1412 im Menschen<br />
angeht, so scheint es sich hier tatsächlich um den<br />
wichtigsten Unterschied zwischen Mensch und<br />
Tier zu handeln. Verschiedene Untersuchungen<br />
am Modell der Ratte, derjenigen Spezies welche<br />
eine zentrale Rolle in der präklinischen Forschung<br />
von TGN1412 gespielt hatte, belegen, dass CD28superagonistische<br />
Antikörper im Tiermodell<br />
keine nennenswerte Zytokinsekretion auslösen.<br />
Nach Infusion von JJ316 in Ratten kommt es<br />
zwar zu einer erhöhten mRNA-Expression von<br />
TNFa und IFNg sowie anderen Mediatoren. Die<br />
in das Blut freigesetzten Zytokinmengen sind<br />
hingegen gering 8 . Verschiedene Studien haben<br />
sich mit der Frage nach der Ursache für diesen<br />
Unterschied zwischen Mensch und Tier auseinandergesetzt.<br />
Eine neuere Untersuchung zeigt,<br />
dass bei Anwendung geeigneter experimenteller<br />
Bedingungen zwar humane, nicht jedoch aus<br />
Affen gewonnene T-Zellen durch TGN1412 zur<br />
Freisetzung von Zytokinen angeregt werden 9 .<br />
Eine mögliche Erklärung hierfür liefern jüngste<br />
Erkenntnisse, dass CD28 superagonistische<br />
Antikörper ausschließlich bei humanen T-Zellen<br />
Kalzium-Signale auslösen 10 . Außerdem hat sich<br />
gezeigt, dass T-Zellen beim Menschen im Verlauf<br />
der Evolution inhibitorische Moleküle, so genannte<br />
CD33-verwandte Siglecs, verloren haben 11 . Diese<br />
könnten bei Ratten und Affen (Schimpansen<br />
und Gorillas) eine überschießende Reaktion auf<br />
die Stimulation durch CD28-superagonistische<br />
Antikörper verhindern, während menschlichen<br />
Zellen dieser Schutz fehlt. Auch wenn dieser<br />
Ansatz einiges erklären würde, so fehlt bislang<br />
noch jeglicher Beweis für dessen Gültigkeit.<br />
Es gibt aber auch erstaunliche Parallelen zwischen<br />
den Wirkungen CD28-superagonistischer<br />
Antikörper bei Ratte und Mensch. Insbesondere<br />
gilt dies für die Induktion einer Lymphopenie.<br />
So wurde in der klinischen Studie von TGN1412<br />
beobachtet, dass die Zahl der Lymphozyten<br />
bereits nach acht Stunden im Blut der Probanden<br />
stark vermindert war 6 . Wie sich zeigte, tritt<br />
dieses Phänomen auch nach Infusion von JJ316<br />
bei Ratten auf: nach nur vier Stunden sind kaum<br />
noch T-Zellen im Blut nachzuweisen 8 . Dieser<br />
Zustand ist jedoch vorübergehend, so dass<br />
die Zahl der Lymphozyten im Blut der Ratten<br />
sich innerhalb von zwei Tagen weitgehend<br />
normalisiert. Das Phänomen der Lymphopenie<br />
scheint mehrere Ursachen zu haben. Zum einen<br />
kommt es innerhalb weniger Minuten zu einer<br />
schlagartigen Verminderung der Beweglichkeit<br />
der T-Zellen. Während diese normalerweise in<br />
den lymphatischen Organen umherwandern,<br />
werden sie unmittelbar nach Infusion von JJ316<br />
immobilisiert. Dieser Zustand hält für wenige<br />
Stunden an, bevor die T-Zellen ihre Beweglichkeit<br />
zurückerlangen. Ein weiterer Grund für die<br />
Lymphopenie liegt offensichtlich in der erhöhten<br />
Adhäsivität der T-Zellen nach Behandlung mit<br />
JJ316. Weiterhin verändert sich die Morphologie<br />
der Lymphozyten innerhalb der ersten 24 Stunden<br />
nach Antikörpergabe, begleitet durch eine<br />
kurzzeitige Größenzunahme (Abb. 2). Obgleich die<br />
Beweglichkeit nur übergangsweise verloren geht,<br />
22 | 9. Jahrgang | Nr. 3/2008 LABORWElT
dauert die Lymphopenie deutlich länger an. Dies<br />
liegt vermutlich daran, dass T-Zellen nicht mehr<br />
aus den sekundären lymphatischen Organen<br />
auswandern können. Normalerweise ist hierfür<br />
die Expression des Sphingosin-1-Phosphat-Rezeptors<br />
verantwortlich, der es den Zellen ermöglicht,<br />
auf den im Blut vorkommenden Mediator S1P zu<br />
reagieren und in die Zirkulation zurückzukehren.<br />
Durch die Behandlung mit JJ316 wird die Expression<br />
dieses Rezeptors vermindert, so dass die<br />
Zellen die Lymphknoten nicht mehr verlassen<br />
können. All diese Veränderungen führen letztendlich<br />
dazu, dass es für eine Dauer von 24 bis<br />
48 Stunden zu einer ausgeprägten Lymphopenie<br />
kommt 8 . Anschließend beginnen die T-Zellen zu<br />
proliferieren, wobei insbesondere die Zahl der<br />
Treg-Zellen stark zunimmt und diese ihre Morphologie<br />
in charakteristischer Weise verändern<br />
(Abb. 3). Inwieweit die im Tiermodell auftretenden<br />
Veränderungen alle auch für den Mensch<br />
zutreffen, lässt sich nur spekulieren. Tatsache ist<br />
jedoch, dass die Lymphopenie und die Schnelle<br />
ihres Auftretens Mensch und Ratte gemeinsam<br />
sind. Dies ist jedoch keinesfalls als Nachteil zu<br />
betrachten. Vielmehr muss davon ausgegangen<br />
werden, dass die Induktion einer transienten Lymphopenie<br />
einen positiven Beitrag zur therapeutischen<br />
Effizienz von CD28-superagonistischen<br />
Antikörpern liefern würde. Dies wird durch die<br />
Beobachtung unterstützt, dass JJ316 und FTY720<br />
in Hinblick auf die Lymphopenie in der Ratte einen<br />
vergleichbaren Effekt aufweisen 8 . Bei FTY720<br />
handelt es sich um einen funktionellen Antagonisten<br />
der Sphingosin-1-Phosphat-Rezeptoren,<br />
der sich gegenwärtig in einer fortgeschrittenen<br />
klinischen Studie zur Behandlung von MS befindet.<br />
Es wäre denkbar, dass insbesondere die in<br />
einer frühen Phase eintretende therapeutische<br />
Wirkung von JJ316 bei Ratten der Induktion einer<br />
Lymphopenie zugeschrieben werden kann. Dies<br />
zeigt, dass CD28-superagonistische Antikörper<br />
Abb.3: Innerhalb von drei Tagen nach Infusion<br />
des CD28-superagonistischen<br />
Antikörpers JJ316 kommt es spezifisch<br />
bei Treg-Zellen zu morphologischen<br />
Veränderungen. Einfügung: Treg-Zelle<br />
aus einer unbehandelten Ratte (gleicher<br />
Maßstab). Darstellung der Zellen<br />
mittels Rasterelektronenmikroskopie<br />
möglicherweise über mehrere Mechanismen<br />
einen positiven Einfluss auf den Verlauf von<br />
Autoimmunerkrankungen nehmen und unterstreicht,<br />
ungeachtet der bislang ungelösten<br />
Problematik des Zytokinsturms im Menschen,<br />
deren therapeutisches Potential (Tab. 1).<br />
Fazit<br />
Neue Ansätze zur Behandlung bislang unheilbarer<br />
Autoimmunerkrankungen wie MS oder Rheumatische<br />
Arthritis werden auch in der Zukunft<br />
aufgrund der noch immer sehr beschränkten<br />
Therapiemöglichkeiten von großer Bedeutung<br />
sein. Neben dem Einsatz hochdosierter Glukokortikoide<br />
kommt insbesondere der Entwicklung<br />
neuer monoklonaler Antikörper sowie zellbasier-<br />
ten Ansätzen eine wichtige Rolle zu. Während<br />
verschiedene therapeutische Antikörper bereits<br />
jetzt aus vielen Behandlungsstrategien nicht<br />
mehr wegzudenken sind, wird es noch dauern,<br />
bis Treg-Zellen möglicherweise Einzug in den<br />
klinischen Alltag halten. Auch wenn der Einsatz<br />
CD28-superagonistischer Antikörper beim<br />
Menschen im ersten Anlauf gescheitert ist, so<br />
erscheint das ihnen zugrundeliegende Prinzip<br />
weiterhin vielversprechend.<br />
Literatur<br />
[1] Roncarolo, M.G., Battaglia, M. Nat Rev Immunol 7 (2007),<br />
585-598.<br />
[2] Chatenoud, L., Bluestone, J.A. Nat Rev Immunol 7 (2007),<br />
622-632.<br />
[3] Lühder, F., Huang, Y., Dennehy, K.M., et al. J Exp Med 197<br />
(2003), 955-966.<br />
[4] Hünig, T. Adv Immunol 95 (2007), 111-148.<br />
[5] Beyersdorf, N., Gaupp, S., Balbach, K., et al. J Exp Med 202<br />
(2005), 445-455.<br />
[6] Tischner, D., Weishaupt, A., van den Brandt, J., et al. Brain<br />
129 (2006), 2635-2647.<br />
[7] Suntharalingam, G., Perry, M.R., Ward, S., et al. N Engl J Med<br />
355 (2006), 1018-1028.<br />
[8] Müller, N., van den Brandt, J., Odoardi, F., et al. J Clin Invest<br />
118 (2008), 1405-1416.<br />
[9] Stebbings, R., Findlay, L., Edwards, C., et al. J Immunol 179<br />
(2007), 3325-3331.<br />
[10] Waibler, Z., Sender, L.Y., Merten, C., et al. PLoS ONE 3 (2008), e1708.<br />
[11] Nguyen, D.H., Hurtado-Ziola, N., Gagneux, P., Varki, A. Proc<br />
Natl Acad Sci USA 103 (2006), 7765-7770<br />
Korrespondenz<br />
Report Immunbiologie<br />
Prof. Dr. Holger M. Reichardt<br />
Universitätsmedizin Göttingen<br />
Georg-August-Universität Göttingen<br />
Abt. Zelluläre und Molekulare Immunologie<br />
Humboldtallee 34<br />
37073 Göttingen;<br />
Tel.: +49-(0)551- 393365, Fax: - 395843<br />
hreichardt@med.uni-goettingen.de<br />
LABORWElT 9. Jahrgang | Nr. 3/2008 | 23
Blitzlicht Zellbasierte Assays<br />
Gelbasierte Angiogenese-<br />
Assays – neues Konzept<br />
zur Meniskusproblematik<br />
Dr. Roman Zantl, Dr. Ulf Rädler, Elias Horn, ibidi GmbH, Martinsried<br />
Der Meniskus von Flüssigkeiten, der sich in kleinen Wells an der Wasser-Luftgrenzfläche<br />
bildet, ist bei der Durchführung von gelbasierten Angiogenese-Assays wie zum Beispiel dem<br />
Tube Formation Assay aus drei Gründen störend: Erstens beeinträchtigt er die homogene<br />
Zellverteilung durch die Senke in der Mitte. Zweitens bleibt ein Großteil des teuren Gels<br />
in der Kante zwischen Boden und Seitenwand ungenutzt. Drittens befinden sich die Zellen<br />
auf der gekrümmten Oberfläche teilweise außerhalb des scharf abzubildenden Bereichs. In<br />
dem hier vorgestellten µ-Slide Angiogenesis von ibidi werden diese drei Probleme mit einer<br />
„well-in-a-well“-Struktur gelöst. Die Idee beruht darauf, dass die Oberfläche eines zu 100%<br />
gefüllten Töpfchens exakt eben ist. Im Vergleich zu handelsüblichen 96-well-Platten. erspart<br />
die neue Struktur 90% des einzusetzenden Gels.<br />
Substanzen mit anti-angiogenetischer Wirkung<br />
wird großes Potential bei der unterstützenden<br />
Therapie von Tumorerkrankungen<br />
zugeschrieben. Aufgrund des unkontrollierten<br />
Zellwachstums haben Tumore einen<br />
besonders großen Bedarf an Sauerstoff und<br />
Nährstoffen. Das schnelle Wachstum von Gewebe<br />
ist deshalb auf die Bildung neuer Gefäße<br />
im und um den Tumor herum angewiesen.<br />
Eine medikamentöse Beeinträchtigung der<br />
Angiogenese könnte demnach das Tumorwachstum<br />
verringern und damit die Chancen<br />
A B C<br />
Tube Formation Assay<br />
homogene<br />
Zellaussaat<br />
Gelmatrix<br />
5x<br />
tube<br />
formation<br />
Sprouting Spheroid<br />
Zell Sphäroid<br />
5x<br />
bei der Behandlung mit komplementären<br />
Behandlungsmethoden, wie chirurgischen<br />
Eingriffen, Bestrahlung und Chemotherapie<br />
vergrößern.<br />
Mit Nährstoffen unterversorgtes Gewebe<br />
setzt Signalstoffe frei, die das benachbarte<br />
Gewebe und insbesondere Endothelzellen<br />
dazu bringen, neue Gefäße zu erzeugen.<br />
Dieser Prozess ist meist chemotaktisch getrieben.<br />
Ein vielgenannter Faktor in diesem<br />
Zusammenhang ist das Molekül VEGF (vascular<br />
endothelial growth factor), der Endo-<br />
sprouting<br />
(sprießen)<br />
Stück aus Gefäßwand<br />
12 h 12 h 12 h<br />
Sprouting Aortic Tissue<br />
sprouting<br />
(sprießen)<br />
Abb. 1: Bei gelbasierten Angiogenese-Assays wird die Entwicklung von auf einer Gelmatrix<br />
(gelb) kultivierten Zellen mikroskopisch verfolgt. Beim Tube Formation Assay (A)<br />
bilden vereinzelt ausgesäte Zellen charakteristische, netzartige Strukturen. Im Gegensatz<br />
dazu bilden Endothelzellen aus sphäroidförmigen Zellverbänden oder Gefäßstücken<br />
sprossenartige Strukturen. Dies wird beim Sprouting Spheroid Assay (B) und<br />
beim Sprouting Aortic Tissue Assay (C) genutzt.<br />
24 | 9. Jahrgang | Nr. 3/2008 LABORWElT<br />
5x<br />
thelzellen zur Zellteilung und möglicherweise<br />
zur gerichteten Bildung neuer Gefäße anregt.<br />
Neben einer Behandlung der Tumorzellen ist<br />
deswegen die Unterdrückung der Angiogenese<br />
bei Endothelzellen eine lohnende Strategie<br />
in der Tumorbehandlung.<br />
In vitro-Assays zur Untersuchung<br />
der Angiogenese<br />
Es gibt zahlreiche in vivo- und in vitro-Assays<br />
zur Untersuchung des anti-angiogenetischen<br />
Potentials von Substanzen. Dazu wird die<br />
Wirkung von Stoffen auf die Gefäßneubildung<br />
durch Endothelzellen oder deren<br />
Vorläuferzellen anhand von Modellsystemen<br />
analysiert. Die Möglichkeiten reichen von der<br />
Impfung von Hühnereiern mit Endothelzellen<br />
und verschiedenen Modellsystemen in Tieren<br />
bis zu zellbasierten Untersuchungen. Zu den<br />
prominenten Beispielen der zellbasierten<br />
Assays gehören der „tube formation assay“<br />
und der „sprouting assay“.<br />
Beim tube formation assay werden vereinzelte<br />
Zellen auf eine dünne Gelschicht<br />
aufgetragen und dort für eine bestimmte Zeit<br />
unter Zugabe von aktivierenden Substanzen<br />
kultiviert. Das angiogenetische Potential<br />
wird analysiert, indem die Ausprägung einer<br />
charakteristischen Netzstruktur anhand<br />
verschiedener Parameter (Anzahl der Knotenpunkte,<br />
Anzahl der Verzweigungen pro<br />
Knotenpunkt, Gesamtlänge aller Netzstrukturen,<br />
etc.) mit mikroskopischen Aufnahmen<br />
bestimmt wird. Obwohl die Zellen auf einer<br />
dreidimensionalen Matrix wachsen, sind<br />
die gebildeten Strukturen zweidimensional<br />
und als solche der Mikroskopie gut zugänglich.<br />
Ähnlich ist das Vorgehen beim sprouting<br />
assay, wo ein Sphäroid aus Endothelzellen<br />
oder auch direkt ein Stück des Gefäßes,<br />
also etwa ein ringförmiger Querschnitt des<br />
Gefäßes, auf die Gelmatrix gelegt werden.<br />
Je nach Potential zur Angiogenese sprießen<br />
aus diesen Endothelzellverbänden neue „Gefäße“<br />
stärker oder schwächer aus. Bei beiden<br />
Assays werden keine Gefäße mit einem Lumen<br />
ausgebildet, sondern charakteristische<br />
Strukturen der Gefäßbildung.<br />
Um gute lichtmikroskopische Aufnahmen<br />
zu erhalten, ist es erforderlich, dass sich die<br />
Zellen in einer optischen Ebene befinden –<br />
genauer ausgedrückt: innerhalb des Tiefenschärfebereichs<br />
des verwendeten Objektivs<br />
liegen. Dieser beträgt typischerweise bei<br />
den für die Assays verwendeten 5x- oder 10x-<br />
Objektiven rund 75 µm oder entsprechend<br />
20 µm. Trotzdem liegen die Zellen in den<br />
typischen Bildausschnitten von etwa 4 mm<br />
x 3 mm (5x Objektiv) beziehungsweise 2<br />
mm x 1,5 mm (10x Objektiv) aufgrund der<br />
Meniskusbildung im Allgemeinen nicht im<br />
Tiefenschärfebereich wie in Abbildung 2 C<br />
zu sehen.
�����<br />
���������������������������������<br />
��������������������������������<br />
�������<br />
Detect proteins with the brightest, longest-lasting western blot detection chemistry!<br />
immediate<br />
30 min<br />
60 min<br />
4 hr<br />
24 hr<br />
Western-SuperStar <br />
Detection<br />
marker<br />
0.312 µg<br />
0.625 µg<br />
2.5 µg<br />
5 µg<br />
Competitor’s Kit<br />
marker<br />
0.312 µg<br />
0.625 µg<br />
2.5 µg<br />
5 µg<br />
Go to www.appliedbiosystems.com/westernsuperstar to download the<br />
product bulletin and place your order.<br />
Tropix ® Western-SuperStar Immunodetection System is the most complete<br />
chemiluminescence-based western blotting kit on the market, and offers<br />
five- to ten-fold higher intensity than competitors’ products.<br />
• You choose when you want to expose your blot after adding the substrate<br />
(immediately or up to 4.5 days)<br />
• You choose the membrane (PVDF, nitrocellulose or nylon)<br />
• You choose the imaging method (CCD imager or x-ray film)<br />
• You choose whether you want to strip and reprobe (up to 3 times)<br />
FIND your protein with Western-SuperStar Immunodetection System!<br />
For Research Use Only. Not for use in diagnostic procedures. © 2008 Applied Biosystems. All rights reserved. Applied Biosystems, AB (Design), and Tropix are registered trademarks and Western-SuperStar is a trademark of Applera<br />
Corporation or its subsidiaries in the U.S. and/or certain other countries.
Blitzlicht Zellbasierte Assays<br />
A B<br />
C<br />
5x<br />
Das Problem Meniskusbildung<br />
Sowohl Tube Formation- als auch Sprouting-<br />
Assays werden auf Gelmatrizes durchgeführt,<br />
wobei meist BD Matrigel TM von Becton Dickinson<br />
verwendet wird. Aufgrund der hohen Kosten<br />
für die Gelmatrix und der Parallelisierung<br />
wird angestrebt, den Test in möglichst kleinen<br />
Wells durchzuführen.<br />
Allerdings tritt im Fall kleiner Wells ein<br />
störender Meniskus auf. Dieser führt einerseits<br />
zu einer ungleichmäßigen Zellverteilung<br />
durch Zellkonzentration in der Mitte<br />
des Wells. Andererseits liegen die Zellen<br />
auf einer gekrümmten Oberfläche, was eine<br />
scharfe mikroskopische Abbildung aller Zellen<br />
zugleich unmöglich macht (Abb. 2). Die automatisierte<br />
Bildverarbeitung wird dadurch<br />
drastisch aufwendiger.<br />
Ein möglicher Lösungsansatz ist es, Bilder<br />
in verschiedenen Fokusebenen aufzunehmen,<br />
um danach die jeweils scharfen Bereiche der<br />
Bilder zu einem Bild zusammenzusetzen. Um<br />
das Meniskusproblem selbst zu verringern,<br />
bleibt bisher nur die Verwendung von größeren<br />
Wells, zum Beispiel von 6 Well- oder<br />
35 mm-Petrischalen. In diesem Fall vergrößert<br />
sich allerdings der Matrigeleinsatz pro Well<br />
–in 96-Well-Platten ca. 100 µl – um ein Vielfaches.<br />
Bei der Verwendung von 100 µl pro Well<br />
entstehen bereits Matrigelkosten von rund<br />
300 Euro für eine 96-Well-Platte. Hier gilt es,<br />
zwischen hohen Kosten einerseits und starker<br />
5x<br />
Abb. 2: Bei der Durchführung von Angiogenese-Assays in einfachen Töpfen erzeugt das eingefüllte<br />
Gel einen Meniskus (A), so dass die Zellen in unterschiedlichen Fokusebenen liegen.<br />
Die „well-in-a-well“-Struktur des µ-Slide Angiogenesis (B) verhindert durch vollständiges<br />
Auffüllen des inneren Wells mit Gel die Bildung eines Meniskus. Mikroskopische Bilder, die<br />
in Standard-Wells aufgenommen sind, enthalten ohne Software-seitige Nachbearbeitung<br />
zugleich scharfe und unscharfe Bereiche (C). Wird der Assay in der neuen Struktur durchgeführt,<br />
befinden sich alle Zellen in einer Fokusebene und können dadurch gleichzeitig scharf<br />
abgebildet werden. Bilder m. freundlicher Genehmigung v. Peter Nelson, LMU München<br />
0,8 mm<br />
5x<br />
5x<br />
Meniskusbildung, also schlechter Optik und<br />
inhomogener Zellverteilung andererseits,<br />
einen Kompromiss zu finden.<br />
Zellmedium (50 µl)<br />
Gelmatrix (10 µl)<br />
4 mm<br />
5 mm<br />
26 | 9. Jahrgang | Nr. 3/2008 LABORWElT<br />
1 mm<br />
Abb 3: Das objekträgergroße µ-Slide Angiogenesis<br />
enthält 15 „Well-in-a-Well“-<br />
Strukturen. Wird das kleinere Well<br />
vollständig mit Gel (10 µl) gefüllt,<br />
benötigt das Objektiv einen Arbeitsabstand<br />
von mindestens 1 mm. Sollte der<br />
Meniskus an der Luft-Mediumgrenzfläche<br />
z.B. für Phasenkontrastaufnahmen<br />
stören, kann dieser durch vollständiges<br />
Füllen des großen Töpfchens<br />
(50 µl) vermieden werden. Ohne Gel<br />
können auch hochauflösende Objektive<br />
(z.B. 100x-Ölimmersion) zur Zellbeobachtung<br />
verwendet werden.<br />
Die Lösung: Well-in-a-Well<br />
Mit einem geometrischen Trick lässt sich die<br />
Meniskusbildung des Gels jedoch verhindern<br />
und zugleich 90% des zu verwendenden Gel-<br />
Materials einsparen. Dazu wird ein kleineres<br />
Well mit 4 mm Durchmesser und 0,8 mm<br />
Tiefe – also 10 µl Volumen – in einem größeren<br />
Well mit 5 mm Durchmesser untergebracht.<br />
Wird das kleinere Well komplett mit dem Gel<br />
befüllt, bildet dieses eine flache Oberfläche.<br />
Zellen und sich auf der Geloberfläche bildende<br />
Zellstrukturen liegen damit in derselben<br />
optischen Fokusebene und können einfach<br />
und gleichzeitig scharf abgebildet werden.<br />
Durch Verwendung eines deckglasdicken<br />
Bodens beträgt die Gesamthöhe im Falle des<br />
komplett gefüllten Wells etwa 1 mm und ist<br />
damit mit handelsüblichen 10x-, 20x- und<br />
40x-Objektiven mikroskopierbar.<br />
Für andere Anwendungen können zudem<br />
durch geringere oder größere Füllvolumen<br />
möglicherweise erwünschte Menisken<br />
hergestellt werden, zum Beispiel um wenige<br />
Zellen in der Mitte des kleinen Wells<br />
zu konzentrieren. Dazu würde lediglich<br />
5 µl des Gels eingesetzt werden, um den<br />
Zellen eine biokompatible und schalenförm -<br />
ige Geloberfläche zu präsentieren. Die „Wellin-a-Well“-Idee<br />
könnte auch ihren Platz in<br />
der dreidimensionalen Zellkultur finden,<br />
indem die Zellen nicht auf dem Gel, sondern<br />
in der Gelmatrix ausgesät werden. Dabei ist<br />
die homogene Dicke der Gelmatrix von nur<br />
0,8 mm von Vorteil, weil sie eine gleichmäßige<br />
Versorgung mit Nährstoffen gewähr -<br />
leistet.<br />
Fünfzehn dieser Well-in-a-well-Strukturen<br />
sind im µ-Slide Angiogenesis von ibidi umgesetzt.<br />
Das „well-in-a-well“ Prinzip bietet auch<br />
großes Potential bei der Durchführung von<br />
Screenings im Angiogenesebereich, da sich<br />
gerade hier die Vorteile wie Kosteneinsparung<br />
und vereinfachte Automatisierung der<br />
Bildverarbeitung besonders stark bemerkbar<br />
machen.<br />
Das µ-Slide Angiogenesis wurde in enger<br />
Zusammenarbeit mit Stefan Zahler<br />
vom Center of Drug Research des Instituts<br />
für pharmazeutische Biologie der Ludwig<br />
Maximilians-Universität München entwickelt.<br />
Korrespondenzadresse<br />
Dr. Ulf Rädler<br />
ibidi GmbH<br />
Am Klopferspitz 19<br />
82152 Martinsried<br />
Tel.: +49-(0)89-5204617 31<br />
Fax:+49-(0)89-5204617 59<br />
uraedler@ibidi.de<br />
www.ibidi.de
����������������������������������������������������������������������������������������������������������<br />
����������������������������������������������������������������������������������������������������������<br />
Zellarrays zum<br />
HT-Nachweis von Protein-<br />
Protein-Interaktionen<br />
Dr. Andrea Fiebitz, Prof. Dr. Hans Lehrach, Dr. Michal Janitz, Dr. Dominique Vanhecke,<br />
Max-Planck-Institut für molekulare Genetik, Berlin<br />
Die Erforschung von Protein-Protein-Interaktionen (PPI) ist grundlegend für das Verständnis<br />
von zellulären Funktionen. Mit der Etablierung des „cell array-based protein-protein interaction<br />
assay“ (CAPPIA), eines kostengünstigen und effektiven Hochdurchsatz-Verfahrens<br />
zum Nachweis von Protein-Protein-Interaktionen, steht nun neben dem Hefe- auch ein<br />
hochdurchsatztaugliches Säuger-Zwei-Hybrid-System im Arrayformat zur Verfügung. Es ist<br />
von Vorteil, Interaktionen zwischen Säugerproteinen direkt in Säugerzellen zu untersuchen,<br />
da hier – anders als beispielsweise in Hefezellen – von einer korrekten Abbildung der Proteinexpression<br />
und der post-translationalen Modifikationen ausgegangen werden kann.<br />
Bei dem hier beschriebenen Assay wird eine<br />
große Probenanzahl im Arrayformat auf einem<br />
Objektträger immobilisiert, der dann mit einer<br />
Schicht adhärenter Zellen bedeckt wird. Interagieren<br />
die nach der Transfektion exprimierten<br />
Proteine miteinander, so fluoreszieren die auf<br />
dem Array gewachsenen Zellen infolge eines<br />
durch die Proteininteraktion aktivierten fluoreszenten<br />
Reporters. Die dichte Anordnung<br />
von Proben auf den Zellarrays und der geringe<br />
Blitzlicht Hochdurchsatz-Analyse<br />
Verbrauch von Reagenzien machen CAPPIA<br />
derzeit zu einer der ökonomischsten Hochdurchsatzverfahren<br />
für den Nachweis von<br />
Proteininteraktionen direkt in Säugerzellen.<br />
Vorteile und Funktionsweise<br />
Proteininteraktionen und -komplexe spielen<br />
in biologischen Systemen eine wichtige Rolle.<br />
Eine Standardmethode zum Hochdurchsatz-<br />
Nachweis der Proteininteraktion ist das<br />
Zwei-Hybrid-System 1 , bei dem sogenannte<br />
Bait- und Prey-Expressionsplasmide kotransfiziert<br />
werden. Das Baitprotein ist mit<br />
einer DNA-Bindungsdomäne (DBD) fusioniert,<br />
das Preyprotein mit einer transkriptionalen<br />
Aktivierungsdomäne (AD). Im Falle einer Interaktion<br />
beider Proteine bilden die beiden<br />
Domänen zusammen einen funktionellen<br />
Transkriptionsfaktor, der die Expression eines<br />
Reporterproteins aktiviert. Bisher wurden<br />
Hochdurchsatz analysen mit dem Zwei-Hybrid-<br />
Verfahren fast ausschließlich in Hefe durchgeführt,<br />
auch solche, bei denen Säugerproteine<br />
untersucht werden sollten. Potentielle Interaktionen<br />
müssen aufgrund der hohen Anzahl an<br />
falsch-positiven Signalen anschließend Gen für<br />
Gen im Säugersystem überprüft werden, um<br />
����������� ���������� ������������<br />
����������������������������������������<br />
������������� ����������������������������������������<br />
����������������������������<br />
����������������������������<br />
����������������������������������������������������<br />
����������������������������������������������������<br />
��������������������������������<br />
��������������������������������<br />
�������������������������������<br />
�������������������������������<br />
������������� �������������<br />
�����������������������������������������������������<br />
�����������������������������������������������������<br />
�������������������������������������������������������������<br />
�������������������������������������������������������������<br />
����������������������������������������������<br />
����������������������������������������������<br />
�����������������������������������������������������������������<br />
�����������������������������������������������������������������<br />
����������������������������������������������������������������<br />
����������������������������������������������������������������<br />
���������������������������������������������<br />
���������������������������������������������<br />
�����������������������������������������������������<br />
�����������������������������������������������������<br />
� ���������������������������������������� � ���������������������������������������� �������������� ��������������<br />
� �<br />
� �������������������������������������� � �������������������������������������� ������<br />
� � ����������������������������������<br />
����������������������������������<br />
� ����������������������<br />
� ����������������������<br />
� ���������� � ����������<br />
� ��������������������<br />
� ��������������������<br />
� �����������������������<br />
� �����������������������<br />
� �������������������<br />
� �������������������<br />
� �<br />
��������������<br />
��������������<br />
������������������<br />
������������������<br />
LABORWElT ������������������������<br />
9.<br />
������������������������<br />
Jahrgang | Nr. 3/2008 | 27<br />
����������������������������<br />
����������������������������
Blitzlicht Hochdurchsatz-Analyse<br />
A<br />
B<br />
C<br />
D<br />
Abb. 1: CAPPIA. A. Vorbereiten und Spotten<br />
von Proben mit Bait (X fusioniert an<br />
Aktivierungsdomäne AD) und Prey<br />
(möglicher Interaktionspartner Y<br />
fusioniert mit Bindungsdomäne BD).<br />
B. Adhärente Säugerzellen werden<br />
C. durch die immobilisierter DNA<br />
transfiziert. D. Interagieren Bait und<br />
Prey miteinander, wird der autofluoreszente<br />
Reporter exprimiert (rote<br />
Punkte). EGFP dient der Orientierung<br />
(grüne Punkte).<br />
eventuelle posttranslationale Modifikationen<br />
und beteiligte Co-Faktoren zu berücksichtigen.<br />
Doch obgleich es bereits Zwei-Hybrid-Systeme<br />
für Analysen direkt in Säugerzellen gibt 2,3 , sind<br />
diese bis heute nicht oder nur mit großem<br />
Kosten-, und Geräteaufwand für Hochdurchsatz-Screens<br />
nach neuen Proteininteraktionen<br />
geeignet. Aus diesem Grund wurde ein kostengünstiges<br />
und effizientes System etabliert,<br />
mit dem Säugerproteine direkt in Säugerzellen<br />
untersucht werden können. Hierfür wurde das<br />
Säuger-Zwei-Hybrid-System mit transfizierten<br />
Zell-Microarrays 4 zu einem neuen Verfahren<br />
namens CAPPIA („cell array based proteinprotein-interaction<br />
assay“) kombiniert.<br />
Nachdem die vektorkonstruierten Proben<br />
von Bait-, Prey- und Reporterplasmiden im<br />
Arrayformat auf Objektträgern fixiert worden<br />
sind, werden die so entstandenen Arrays mit<br />
humanen adhärenten Zellen bedeckt. Die Zellen<br />
setzten sich einschichtig auf der gesamten<br />
Oberfläche fest. Aber nur diejenigen, die direkt<br />
auf den aufgetragenen DNA-Spots wachsen,<br />
werden durch die Proben transfiziert und beginnen,<br />
spezifische Bait- und Preyproteine zu<br />
exprimieren. Sobald diese chimären Proteine<br />
miteinander in Wechselwirkung treten, kommt<br />
es zur Expression des Reporterproteins – hier:<br />
eines autofluoreszierenden Proteins –, infolge<br />
der Bildung des funktionellen Transkriptionsfaktors<br />
aus der DNA-Bindedomäne des Baits<br />
und der Aktivierungsdomäne des Preys. Die aus<br />
der Proteininteraktion resultierenden Signale<br />
werden mit Hilfe von Fluoreszenznachweisen<br />
analysiert, ohne dass die Objektträger weiter<br />
bearbeitet werden müssten, wie etwa bei Immunfluoreszenzfärbungen<br />
oder enzymatischen<br />
Nachweisen (Abb. 1)<br />
Für die Herstellung von Microarrays nach dem<br />
CAPPIA-Prinzip werden nur Nanoliter- Volumina<br />
von Lösungen zum Spotten der Objektträger benötigt.<br />
Sie enthalten neben dem Transfektionsreagenz,<br />
die Bait- und Prey-Expressionsplasmide<br />
sowie ein autofluoreszentes Reporterplasmid.<br />
Grundlage für die Konstruktion der Proben sowie<br />
für Positiv- und Negativkontrollen, war das<br />
Mammalian Two-Hybrid Assay-Kit (Stratagene).<br />
Im Vergleich verschiedener Reporter- und Nachweisverfahren<br />
erwies sich der autofluoreszente<br />
GAL4-pZsGreen-Reporter – ein Eigenkonstrukt<br />
aus der GAL4 upstream activating sequence<br />
des pGAL/lacZ (Invitrogen) und dem Plasmid<br />
pZsGreen1-1 (Clontech) – als am besten geeignet.<br />
Von 12 unterschiedlichen, in den Vergleich<br />
einbezogenen Objektträgern erwies sich eine<br />
selbsthergestellte Variante mit einer Kombination<br />
von Poly-L-Lysin und VECTABOND (Vector<br />
Labs) als am geeignetsten. Für das automatische<br />
Auftragen der DNA auf die Objektträger<br />
wurde ein sciFLEXARRAYER S5 (Scienion) genutzt<br />
(Abb. 2). Für eine gute Haftung auf den Objektträgern<br />
und eine spätere effiziente reverse<br />
Transfektion wurden diverse Bedingungen wie<br />
etwa die finale DNA-Konzentration und die<br />
Gelatinekonzentration optimiert. Die Arrays<br />
können nach dem Spotten über mehrere Monate<br />
hinweg stabil gelagert werden und müssen<br />
dann für die reverse Transfektion im Rahmen<br />
eines CAPPIA-Experiments lediglich mit einem<br />
Zell-Monolayer bedeckt und anschließend analysiert<br />
werden. Eine Reihe von Zelllinien wurde<br />
auf ihre Eignung für die reverse Transfektion<br />
und Proteinexpression im Rahmen von CAPPIA-<br />
Experimenten getestet, als Standardzelllinie<br />
für alle Optimierungsschritte und späteren<br />
Experimente diente HEK293T. Die Auswertung<br />
der Fluoreszenzsignale erfolgte mittels eines<br />
BIOccd Image Readers (PE Applied Biosystems).<br />
Für die graphische Darstellung wurden die<br />
Werte gegen die Signale des autofluoreszenten<br />
Proteins EGFP normalisiert. EGFP diente ferner<br />
der einfacheren Orientierung auf den Arrays.<br />
Aus diesem Grund wurde das Plasmid pcDNA4-<br />
EGFP als Umrandung auf jeden Objektträger<br />
gespottet. Eine detaillierte Beschreibung der<br />
grundlegenden Methoden und Materialien<br />
findet sich in [5].<br />
Screening einer Prey-Bibliothek<br />
und quantitative Detektion<br />
Nach Optimierung der revers transfizierten<br />
Zell-Arrays und Anwendung in verschiedenen<br />
Zelllinien wurde die Leistungsfähigkeit von<br />
CAPPIA mittels Verifizierung einer bekannten<br />
hormonabhängigen Interaktion im Rahmen<br />
einer kleinen Bibliothek von potentiell mit<br />
nuklearen Rezeptorfunktionen assoziierten<br />
cDNAs getestet. Zehn Baits und 17 Preys, also<br />
170 verschiedene Prey-Bait-Kombination,<br />
wurden jeweils dreifach gespottet. Abbildung<br />
3 zeigt das Ergebnis der reversen Transfektion<br />
der Ligandenbindedomäne des androgenen<br />
Rezeptors (LBD-AR) als Bait in Kombination mit<br />
den 17 Preys der cDNA-Bibliothek. Die Interaktion<br />
zwischen LBD-AR und der N-terminalen<br />
Domäne des androgenen Rezeptors (NTD-AR)<br />
konnte dabei mit CAPPIA ebenso nachgewiesen<br />
werden wie die Notwendigkeit der Anwesenheit<br />
eines Androgens (hier des synthetischen<br />
Agonisten R1881) für die beobachtete Interaktion<br />
– beides steht in Übereinstimmung mit<br />
früheren Studien 6-8 . Nach dem so erbrachten<br />
Nachweis der Spezifität von CAPPIA wurde die<br />
quantitative Detektion von Proteininteraktio-<br />
Abb. 2: Automatisches Spotten. A. sciFLEX-<br />
ARRAYER S5. B. Mit einer 70 µm-Nozzle<br />
wurden pro Spot 20 Tropfen (ca. 8 nl<br />
Probe) dispensiert. C. Objektträger<br />
nach Transfektion der Zellen, aufgenommen<br />
mit einer BIOccd-Kamera<br />
(PE Applied Biosystems). Untersucht<br />
wurden unterschiedliche Anzahl von<br />
Tropfen pro Spot und verschiedene<br />
Abstände zwischen den Spots.<br />
28 | 9. Jahrgang | Nr. 3/2008 LABORWElT<br />
A<br />
B<br />
C
Realistische Chancen für Wirtschaft und Kapital<br />
Pharma-Mittelstand und Biotech-KMU –<br />
gemeinsam stärker?<br />
Sponsoren:<br />
13. Juni 2008, 9-16 Uhr, Frankfurt/Main, Industriepark Höchst, Peter-Behrens-Bau<br />
Grußwort<br />
Dr. Bernd Wegener, Vorsitzender des BPI, Vorstandsvorsitzender BRAHMS Aktiengesellschaft<br />
Die Märkte verändern sich: Handlungsbedarf!<br />
Märkte im Umbruch – Zwänge und Möglichkeiten: Dr. Gunter Trojandt, Boston Consulting Group, Berlin<br />
Der Pharmamarkt aus Sicht eines mittelständischen Unternehmens: Prof. Dr. G. Strugala, Apogepha Arzneimittel GmbH, Dresden<br />
Entwicklung der Biotech-Branche – Flops, Erfolge, Wege in die Zukunft: Dr. Ulrich Dauer, 4SC AG, Martinsried<br />
Neue Modelle: erste Beispiele für Kooperationen<br />
Riemser Arzneimittel AG und TVM Capital GmbH; Wilex AG und Laboratorios Del Dr. Esteve, S.A., Barcelona;<br />
Revotar Pharmaceuticals AG: Internationale Kooperationen; Fresenius Biotech GmbH und TRION Pharma AG; und weitere<br />
Zwischen Reaktion und Zukunftsgestaltung: die Rolle der Kapitalmärkte<br />
Pharmamittelstand und Biotech – eine übersehene Chance?: Dr. Axel Polack, TVM Capital GmbH, München<br />
Kapitalmarktinstrumente zur Zukunftssicherung: Tilman Wittershagen, Deutsche Bank AG, Frankfurt<br />
und weitere<br />
Podiumsdiskussion: Visionen und/oder realistische Chancen<br />
Pharma-Mittelstand und Biotech-KMU – ein unterschätztes Erfolgsmodell? Moderation: Andreas Mietzsch, BIOCOM AG<br />
Zwischen den Vortragsblöcken besteht ausreichend Gelegenheit zu Diskussion und Networking.<br />
Abschließend optional Besichtigung des Peter-Behrens-Baus und Cocktail-Empfang.<br />
Bitte registrieren Sie sich unter www.biocom.de/events oder senden Sie uns eine eMail an events@biocom.de.<br />
Eine Veranstaltung der BIOCOM AG in Kooperation mit dem Bundesverband der Pharmazeutischen Industrie e.V.<br />
Organisation: BIOCOM Projektmanagement GmbH | Brunnenstraße 128 | 13355 Berlin<br />
www.biocom.de | events@biocom.de | Tel. +49 (0)30 264921-53 | Fax +49 (0)30 264921-66<br />
Foto: InfraServ Höchst
Blitzlicht Hochdurchsatz-Analyse<br />
A<br />
B<br />
Abb. 3: Detektion einer Interaktion. Kotransfektion der Ligandenbindedomäne des Androgenrezeptors<br />
(AR-LBD) als Bait und jeweils einem von 17 Preys. Jede Kombination<br />
(A – Q) wurde dreifach gespottet, ebenso die Kontrollen. In Gegenwart von R1881,<br />
einem synthetischen Androgen-Liganden, interagiert AR-LBD mit der N-terminalen<br />
Domäne des Androgenrezeptors (Prey O). A. BIOccd-Scannerbild, B. Graph<br />
nen anhand der Dosis-Antwort-Abhängigkeit<br />
der hormonregulierten Wechselwirkungen von<br />
LBD-AR und NTD-AR untersucht. Die Analyse der<br />
Regulation mit Hilfe von R1881 als Agonist bzw.<br />
der inhibierende Effekt von Medroxyprogesteronacetat<br />
(MPA) und Hydroxyflutamid (OH-Flu)<br />
als Antagonisten befand sich im Einklang mit<br />
veröffentlichten Daten 6,9 . Zusammengenommen<br />
zeigen diese Experimente deutlich, dass<br />
Proteininteraktionen mit Hilfe von Zellarrays<br />
auch unter verschiedenen physiologischen<br />
Bedingungen zuverlässig und sogar quantitativ<br />
identifiziert werden können.<br />
Um die Flexibilität und damit die Möglichkeiten<br />
der Anwendung von CAPPIA zu erhöhen<br />
wurden zusätzlich zu der hier beschriebenen<br />
Dreifach-Transfektion (Bait + Prey + Reporter)<br />
zwei weitere Transfektionsstrategien mit CAP-<br />
PIA getestet. Hierfür wurden Objektträger ohne<br />
Bait gespottet (sogenannte PR-Objektträger).<br />
Um nach Interaktionspartnern für die gespotteten<br />
Preys zu suchen, wird das zu testende<br />
Bait dann vor der reversen Transfektion in die<br />
Zellen integriert, entweder mittels stabiler<br />
oder transienter Transfektion. Die sogenannten<br />
„PR-stable Bait“- sowie die „PR-trans Bait“-<br />
Zellarrays erbrachten eine vergleichbar spezifische<br />
Trans-Aktivierung der Reporterexpression.<br />
Da PR-Objektträger mit jedem beliebigen<br />
Bait kombiniert werden können, sind sie für<br />
die Suche nach unbekannten Interaktionen<br />
gegenüber Objektträgern mit bereits gespotteten<br />
Baits (PRB-Objektträger) im Vorteil. Große<br />
Prey-Bibliotheken können gespottet und die<br />
entsprechenden Objektträger gelagert oder an<br />
andere Forschungsgruppen verschickt werden,<br />
die nach Interaktionspartnern für ein bestimmtes<br />
als Bait einsetzbares Protein suchen.<br />
Fazit<br />
Im Vergleich zu dem gängigen Zwei-Hybrid-<br />
Assay in Hefe bietet CAPPIA den Vorteil, Interaktionen<br />
zwischen Säugerproteinen im korrekten<br />
zellulären Kontext zu testen, was die Rate an<br />
falsch-positiven Signalen deutlich verringern<br />
sollte, zumal phänotypisch sehr unterschiedliche<br />
Zelltypen verwendet werden können.<br />
Zusammenfassend kann gesagt werden, dass<br />
das große Leistungsvermögen der Zellarrays<br />
zusammen mit der Flexibilität, dem geringen<br />
Verbrauch an Reagenzien und der lange<br />
Lagerfähigkeit der gespotteten Objektträger<br />
CAPPIA derzeit zu einem der ökonomischsten<br />
Hochdurchsatz-Assays für die Detektion von<br />
Protein-Protein-Interaktionen in Säugerzellen<br />
macht. Vor allem der Verbrauch an Transfektionsreagenz<br />
– einer der größten Kostenquellen<br />
bei solchen Assays – ist im Vergleich zu Transfektionen<br />
im Well-Format deutlich reduziert.<br />
Durch die Verwendung eines autofluoreszenten<br />
Reporters, der Immunfluoreszenzfärbung oder<br />
einen enzymbasierten Reporterassay überflüssig<br />
macht, wird die Kosteneffizienz weiter<br />
erhöht. Im Gegensatz zu Mikrowell-basierten<br />
Methoden zum Auffinden von Proteininteraktionen<br />
wie beispielsweise nach Suzuki 3 ist zudem<br />
keine teure Geräteinfrastruktur für den Hochdurchsatzbetrieb<br />
mit einem solchen Format<br />
nötig. Derzeit können bis zu 900 verschiedene<br />
Preys als einzelne Spots auf CAPPIA-Objektträgern<br />
gespottet werden. Das entspricht etwa<br />
neun 96-Mikrowellplatten. Durch die Analyse<br />
mehrerer identischer Objektträger können ohne<br />
großen Mehraufwand replikative Datenpunkte<br />
zusammengelegt analysiert werden. Die lange<br />
Lagerfähigkeit der gespotteten Arrays und die<br />
Möglichkeit, diese an andere Forschungsgruppen<br />
zu verschicken, erhöht die Flexibilität und<br />
Praktikabilität dieses Assays.<br />
Nach erfolgter Optimierung und Verifizierung<br />
der Leistungsfähigkeit von CAPPIA wird<br />
nun die Identifizierung unbekannter PPI angestrebt.<br />
Mit der Schaffung großer Bait-Prey-<br />
Bibliotheken zum Screenen mittels CAPPIA<br />
könnten mehr als 15.000 bereits vorhandene<br />
Open Reading Frame-Klone auf Proteininteraktionen<br />
getestet werden. Ferner kann CAPPIA<br />
durch die gezielte Suche nach Liganden, die<br />
bestimmte Interaktionen modulieren oder.<br />
trennen, für die Identifizierung von Arzeimittelkandidaten<br />
genutzt werden.<br />
Literatur<br />
[1] S. Fields and O. Song, Nature 340 (1989) 245-6.<br />
[2] C.V. Dang, J. Barrett, M. Villa-Garcia, L.M. Resar, G.J. Kato and<br />
E.R. Fearon, Mol Cell Biol 11 (1991) 954-62.<br />
[3] H. Suzuki, Y. Fukunishi, I. Kagawa, R. Saito, H. Oda, T. Endo, S.<br />
Kondo, H. Bono, Y. Okazaki and Y. Hayashizaki, Genome Res 11<br />
(2001) 1758-65.<br />
[4] J. Ziauddin and D.M. Sabatini, Nature 411 (2001) 107-10.<br />
[5] A. Fiebitz, L. Nyarsik, B. Haendler, Y.H. Hu, F. Wagner, S. Thamm,<br />
H. Lehrach, M. Janitz and D. Vanhecke, BMC Genomics 9<br />
(2008) 68.<br />
[6] P. Doesburg, C.W. Kuil, C.A. Berrevoets, K. Steketee, P.W. Faber,<br />
E. Mulder, A.O. Brinkmann and J. Trapman, Biochemistry 36<br />
(1997) 1052-64.<br />
[7] I. Ahrens-Fath, O. Politz, C. Geserick and B. Haendler, Febs J<br />
272 (2005) 74-84.<br />
[8] E. Langley, Z.X. Zhou and E.M. Wilson, J Biol Chem 270 (1995)<br />
29983-90.<br />
[9] J.A. Kemppainen, E. Langley, C.I. Wong, K. Bobseine, W.R. Kelce<br />
and E.M. Wilson, Mol Endocrinol 13 (1999) 440-54.<br />
Korrespondenzadresse<br />
Dr. Andrea Fiebitz<br />
Max-Planck-Institut für molekulare Genetik<br />
Ihnestr. 73, D-14195 Berlin<br />
Tel.: +49-(0)30-8413-1238<br />
Fax: +49-(0)30-8413-1128<br />
fiebitz@molgen.mpg.de<br />
www.molgen.mpg.de<br />
30 | 9. Jahrgang | Nr. 3/2008 LABORWElT
Optimale Überwachung<br />
von Laboranlagen<br />
Will Coulter, Perkin Elmer, One Source Business Leader Europe, Beaconsfield, UK<br />
Pharmazeutische, biotechnische und zahlreiche andere Labore sehen sich stetig wachsenden<br />
Herausforderungen gegenüber, wenn es darum geht, die Produktivität zu erhöhen, das<br />
Compliance-Risiko einzugrenzen und zugleich Kosten zu senken, um damit wettbewerbsfähig<br />
oder für ihre Aktionäre attraktiv zu bleiben. Eine Lösung, auf die zahlreiche Labore<br />
zurückgreifen, um diese Ziele zu erreichen, ist die Vergabe des Wartungsmanagements für<br />
Laborgeräte. Wenn Programme zum Anlagen- und Instandhaltungsmanagement professionell<br />
mit den entsprechenden Ressourcen implementiert werden, können Labore von erheblichen<br />
Kostensenkungen und Leistungssteigerungen profitieren.<br />
Einfach ausgedrückt besitzen oder leasen<br />
Labore Anlagen (Geräte, Instrumente oder<br />
Systeme), die aus finanztechnischen Gründen<br />
und wegen der Einhaltung gesetzlicher<br />
Vorschriften abverfolgt werden müssen.<br />
Wenn Ereignisse, wie etwa eine Installation,<br />
vorbeugende Wartung (PM), Reparatur (CM),<br />
Validierung (IQ/OQ/PQ), Laborumzug etc., im<br />
Zusammenhang mit diesen Anlagen eintreten,<br />
müssen diese Ereignisse und ihre Auswirkungen<br />
ebenfalls dokumentiert werden,<br />
um nachvollziehbar zu sein. Zahlreiche Labore<br />
verwirklichen diese Aufgabe mit minimalem<br />
Aufwand in Form von Anlagenregistern und<br />
Gerätelogbüchern. Diese Daten sind jedoch<br />
nicht zentralisiert oder im gesamten Unternehmen<br />
zugänglich. Zudem sind sie häufig<br />
veraltet, dies gilt insbesondere für Anlagenregister.<br />
Schlimmer noch: Einige Labore<br />
riskieren, Auflagen nicht zu erfüllen, wenn sie<br />
den Termin für vorbeugende Wartungen und<br />
Qualifizierungen (OQ) nicht einhalten.<br />
Zahlreiche Softwareunternehmen bieten<br />
eigenständige Softwarepakete an, um La-<br />
Das BioMedizinZentrumDortmund BMZ bietet als Einrichtung des<br />
TechnologieZentrumDortmund optimale Bedingungen für Start-Ups<br />
und junge Unternehmen im Bereich der Life Sciences.<br />
Für die Bereiche Biomedizin, Bioinformatik und Biomikrostrukturtechnik<br />
bietet das BMZ seinen Nutzern folgende Dienstleistungen an:<br />
• Bereitstellung moderner Büro- und Laborinfrastruktur<br />
• Technisches Facility Management<br />
• Unterstützung bei der Geschäftsentwicklung<br />
• Nationales & internationales Networking<br />
• PR & Marketing-Maßnahmen<br />
Innerhalb des BMZ bietet das Zentrum<br />
für Angewandte Proteomik ZAP<br />
in Zusammenarbeit mit den Universitäten<br />
Bochum und Dortmund<br />
State-of-the-art-Technologien in<br />
folgenden Bereichen:<br />
• Quantitative Proteomik<br />
• 2D-DIGE Technologien<br />
• Glykoanalytik<br />
• Protein Biochips<br />
• Biostatistik & Bioinformatik<br />
info@zap-do.de<br />
info@bmz-do.de<br />
Innerhalb des BMZ bietet das Zentrum<br />
für Angewandte Chemische Genomik ZACG<br />
in Zusammenarbeit mit der Universität<br />
Dortmund und des Max-Planck-Instituts<br />
Dortmund Expertise in folgenden Bereichen:<br />
• Antibiotikaforschung<br />
• Biotechnologische Syntheseoptimierung<br />
• Adressierbare mikro- und nano-Arrays<br />
• Membran-Protein-Wechselwirkungen<br />
• Wirkstoffbibliotheken<br />
Blitzlicht Labor-Wartungsmanagement<br />
bore beim Nachhalten dieser Informationen<br />
zu unterstützen. Diese Software lässt sich<br />
unter Umstände sogar mit dem aktuellen<br />
Warenwirtschaftssystem (ERP) der Labore<br />
verknüpfen. Der Einsatz solcher Pakete setzt<br />
erhebliche Investitionen in Ressourcen und<br />
Zeit voraus, um die Gerätedatenbank zu pflegen<br />
und die Serviceaufzeichnungen auf dem<br />
neuesten Stand zu halten. Für einige Labore<br />
ist dieser Aufwand schlichtweg zu hoch,<br />
mit dem Ergebnis, dass die erfassten Daten<br />
unvollständig und damit unbrauchbar sind.<br />
Selbst wenn die entsprechende Software<br />
richtig eingesetzt wird, fehlen den meisten<br />
Unternehmen die Zielstrebigkeit und das<br />
Fachwissen für eine optimale Nutzung der<br />
erfassten Daten.<br />
Eine Lösung, die einerseits diese Problematik<br />
addressiert und andererseits erhebliche<br />
Verbesserungen in der Effizienz und<br />
Nutzung der Geräte mit sich bringt, ist die<br />
Partnerschaft mit einem Dienstleister, der<br />
Geräte verschiedenster Hersteller sowie die<br />
Dienstleistungen anderer Anbieter betreut.<br />
Verglichen mit dem Abschluss zahlreicher<br />
Verträge bei verschiedenen Anbietern bietet<br />
solch ein konsolidiertes Servicemanagement,<br />
bei dem das Labor alle Leistungen<br />
aus einer Hand erhält, aufgrund des geringeren<br />
Verwaltungsaufwandes unmittelbare<br />
Kosteneinsparungen und Leistungsverbesserungen.<br />
PerkinElmer bietet mit seinen<br />
info@zacg-do.de<br />
LABORWElT 9. Jahrgang | Nr. 3/2008 | 31<br />
Kontakt: BMZ · Otto-Hahn-Straße 15 · 44227 Dortmund · Telefon + 49 231 97 42-130 · info@bmz-do.de · www.bmz-do.de
Blitzlicht Labor-Wartungsmanagement<br />
Abb. 1: Schema des Labormanagement-Angebotes One Source<br />
OneSource®Professional Services diese<br />
Dienstleistungen für Labore an.<br />
Maßgeschneiderte Lösung<br />
OneSource® ist, wie der Name schon sagt,<br />
ein umfassendes Lösungspaket aus einer<br />
Hand für die Geräte-Instandhaltung und Anlagenverwaltung<br />
im Labor. Mit OneSource®<br />
bietet PerkinElmer umfassende Geräteinventarisierungs-<br />
und Prüfungsfunktionen, die<br />
eine exakte Bestandsaufnahme aller Geräte<br />
(und Anlagen) ermöglichen. Die Details zum<br />
Anlagenbestand werden in eine Datenbank<br />
zur Anlagenverwaltung eingegeben, wo sie<br />
die Grundlage für die Datenerfassung und<br />
das Berichtswesen bilden (Abb. 1).<br />
Nach Abschluss der Bestandsaufnahme<br />
erarbeitet PerkinElmer gemeinsam mit den<br />
Anwendern im Labor und den Qualitätssicherungsteams<br />
eine Dokumentation über<br />
Serviceanforderungen, Qualitätsverfahren,<br />
Service Level Agreements (SLAs) und Leistungskennzahlen<br />
(KPIs) für die einzelnen<br />
Instrumente. Qualitätssicherungsverfahren<br />
werden mit den bestehenden Qualitätssystemen<br />
im Labor abgestimmt, um die<br />
Einhaltung einschlägiger Vorschriften sicherzustellen.<br />
Anforderungen zur vorbeugenden<br />
Wartung und Einhaltung von Vorschriften<br />
werden definiert und nachgehalten, um<br />
eine automatische Planung zu ermöglichen<br />
und das Risiko der Nichteinhaltung zu verringern.<br />
Schnelle Problembehebung<br />
Im nächsten Schritt werden Dienstleister mit<br />
der Durchführung der Arbeiten (die Ereignisse)<br />
an den Anlagen betraut. PerkinElmer<br />
bietet direkte technische Unterstützung<br />
für eine Reihe von Geräten verschiedener<br />
Originalgerätehersteller in unterschiedlichen<br />
Technologiebereichen, unter anderem HPLC,<br />
GC, LC/MS, GC/MS und Liquid Handling. Indem<br />
PerkinElmer selbst eine größere Anzahl<br />
an Geräten direkt wartet, in zahlreichen<br />
Fällen durch die Stationierung von Servicetechnikern<br />
vor Ort am Kundenstandort,<br />
erzielt PerkinElmer für den Kunden erhebliche<br />
Verbesserungen in Bezug auf die Reaktionszeit<br />
und die Geräteverfügbarkeit. Während<br />
es normalerweise zwei bis drei Tage dauert,<br />
bis Servicetechniker am Standort des Labors<br />
auftauchen und bis zu vier Tagen benötigen,<br />
um eine Reparatur durchzuführen, reagieren<br />
vor Ort stationierte Teams innerhalb einer<br />
Stunde, und die Reparatur wird in der Regel<br />
innerhalb von vier Stunden durchgeführt,<br />
da vor Ort ein Lagerbestand der wichtigsten<br />
kritischen Ersatzteilkomponenten vorhanden<br />
ist. Vor-Ort-Support aus einer Hand für eine<br />
umfangreiche Palette an Geräten liefert<br />
dem Labor eine höhere Effizienz im Hinblick<br />
auf Betriebszeiten und Verfügbarkeit der<br />
Anlagen.<br />
Optimale Ersatzteillogistik-<br />
und -qualität<br />
Wenn Dienstleister Teile verschiedener<br />
Hersteller verwenden, kann dies zu einem<br />
Problem für das Labor werden. Zum einen<br />
ist die Verfügbarkeit von Originalteilen nicht<br />
sichergestellt, zum anderen werden anstelle<br />
der Originalteile möglicherweise Teile minderer<br />
Qualität verwendet. Zur Lösung dieses<br />
Problems setzt PerkinElmer eigens hierfür<br />
vorhandene Ressourcen ein, um verschiedene<br />
Beschaffungskanäle einzurichten bzw.<br />
zu nutzen. Damit wird sichergestellt, dass<br />
Teile jederzeit verfügbar sind und dass diese<br />
– soweit möglich – von der Quelle bezogen<br />
werden, die auch die Originalgerätehersteller<br />
nutzen. In jedem Fall werden alle Teilestücklisten<br />
im Vorfeld überprüft und mit dem<br />
Kunden abgestimmt, um sicherzustellen, dass<br />
die vom Kunden geforderte Qualität erreicht<br />
oder übertroffen wird, bevor die Teile in die<br />
Ersatzteilversorgung (Abb. 2) aufgenommen<br />
werden.<br />
Zuweilen ergeben sich Fragen hinsichtlich<br />
des Compliance-Risikos, wenn der<br />
Service von einem anderen Anbieter als<br />
dem Originalgerätehersteller erbracht wird.<br />
PerkinElmer hat daher mehrere Millionen<br />
US-Dollar in die Ausstattung verschiedener<br />
Schulungszentren weltweit investiert, in<br />
denen OneSource®-Techniker an Produkten<br />
diverser Hersteller mit demselben Niveau<br />
geschult werden, wie die Techniker, die mit<br />
der Wartung von Perkin Elmer-Produkten<br />
betraut sind. Jeder Techniker muss am Ende<br />
des Kurses eine Abschlussprüfung bestehen,<br />
um ein Ausbildungszertifikat zu erhalten. Kopien<br />
dieser Zertifikate sind am Standort des<br />
Kunden verfügbar, um Qualitätsprüfungen<br />
Abb.2: Beschaffungslogistik innerhalb des One Source-Dienstleistungsangebotes<br />
32 | 9. Jahrgang | Nr. 3/2008 LABORWElT
im Rahmen des gemeinsam vereinbarten Qualitätsmanagementsystems<br />
zu unterstützen.<br />
Stetige Verbesserung der Kundenunterstützung<br />
Durch den jüngsten Erwerb von LabMetrix Technologies, dem<br />
größten unabhängigen Lösungsanbieter für die Qualifizierung metrologischer<br />
Analyseinstrumente verschiedener Hersteller, liefert<br />
PerkinElmer seinen Kunden jetzt eine optimierte Qualifizierungsumgebung,<br />
die herstellerunabhängig für zahlreiche Laborinstrumente<br />
und -technologien erhältlich ist.<br />
Für den Fall, dass PerkinElmer nicht über das Fachwissen verfügt,<br />
um den Service an den Laborgeräten selbst zu erbringen, stehen<br />
dem Kunden bewährte Tools zur Verfügung, um die Erbringung<br />
durch andere, vom Labor benannte Anbieter, zu veranlassen. Der<br />
große Vorteil für die Anwender liegt dabei in der zentralen Koordination.<br />
Wann immer ein Instrument einen Service benötigt, werden<br />
alle Anforderungen – unabhängig vom Gerätehersteller – an das<br />
OneSource® Customer-Care-Center geleitet. Dieses Customer-Care-<br />
Center kann je nach Einsatzaufkommen vor Ort beim Kunden oder<br />
dezentral eingerichtet werden. Der Kundendienstspezialist nimmt<br />
die Kundenanforderung entgegen und steuert anschließend alle<br />
Phasen des Serviceprozesses von der Reparaturbeauftragung bis hin<br />
zur erfolgreichen Behebung des Problems.<br />
Verlässliche Dokumentation<br />
Abschließend werden alle Daten des Serviceeinsatzes in das Anlagenverwaltungssystem<br />
durch das OneSource®-Team eingetragen. Neben<br />
Angaben, wie Servicebeschreibung, verwendete Teile, Arbeitszeit und<br />
entstandene Kosten, werden alle im Rahmen des Einsatzes aufgetretenen<br />
Änderungen in der Anlageninformation (neuer Standort oder<br />
Anwender, Änderung des Kostenträgers oder des Wartungsplans)<br />
erfasst und im Verwaltungssystem für die Anlagenwartung aktualisiert,<br />
um die Datenintegrität zu gewährleisten. Die Daten werden<br />
von erfahrenen Fachleuten unter Verwendung von Lean- und Six<br />
Sigma-Tools geprüft und analysiert, um für den Kunden eine kontinuierliche<br />
Verbesserung in der Diestleistung und Effizienz zu erzielen<br />
sowie eine dauerhafte Kostensenkung zu erreichen. Diese Analysen<br />
und Ergebnisse werden gemeinsam mit den Standardberichten über<br />
Leistungskennzahlen (KPIs) dem Kunden zur Verfügung gestellt, um<br />
weitere Entscheidungsprozesse zu unterstützen und kontinuierlichen<br />
Mehrwert zu schaffen. Auf diese Weise wird sichergestellt, dass der<br />
Kunde und die Labore dauerhaft von den Vorteilen der Outsourcing-<br />
Partnerschaft profitieren.<br />
PerkinElmer hat sich mit seinem OneSource®-Angebot einen<br />
Ruf als zuverlässigen Serviceanbieter erworben und arbeitet mit<br />
zahlreichen führenden Laboren aus dem Pharma-, Biotechnologie<br />
und Umweltprüfungs-Sektor zusammen. Seit der Einführung von<br />
OneSource® in den neunziger Jahren hat PerkinElmer beträchtliche<br />
Investitionen in Personal, Prozesse und Systeme getätigt und bietet<br />
die derzeit auf dem Markt umfassendsten Laborservicelösungen für<br />
Geräte verschiedenster Hersteller an.<br />
Korrespondenzadresse<br />
Blitzlicht Labor-Wartungsmanagement<br />
STOP<br />
SPECULATING.<br />
* START<br />
QUANTIFYING.<br />
Will Coulter<br />
One Source Business Leader<br />
PerkinElmer Life and Analytical Sciences<br />
Chalfont Road<br />
CREATING KNOWLEDGE<br />
Seer Green, Beaconsfield Bucks, HP9 2FX, UK<br />
FOR HEALTH<br />
will.coulter@perkinelmer.com<br />
www.perkinelmer.co.uk<br />
sales@biocrates.com | www.biocrates.com<br />
LABORWElT 9. Jahrgang | Nr. 3/2008 | 33<br />
*<br />
*<br />
*<br />
AbsoluteIDQ Kit<br />
With a sample volume of only 10µL, you can now identifiy<br />
and quantify over 150 metabolites in 4 compound classes<br />
in a matter of minutes, and keep your proprietary data<br />
in-house. Change your views of metabolomics with the<br />
intelligent new AbsoluteIDQ Kit.<br />
*<br />
*
Blitzlicht Proteomics/<strong>Zellbiologie</strong><br />
iTRAQ: Identifizierung<br />
unkonventionell<br />
sekretierter Proteine<br />
Dr. Martin Keller und Dr. Hans-Dietmar Beer, Institut für <strong>Zellbiologie</strong>, ETH Zürich<br />
Die klassische Sekretion von Proteinen aus Säugerzellen beruht auf dem Erkennen aminoterminaler<br />
Signalpeptide und dem Transport dieser Proteine durch das Endoplasmatische Retikulum<br />
(ER) und den Golgi-Apparat. Die proinflammatorischen Zytokine Pro-Interleukin(IL)-1a und -b<br />
und eine Reihe anderer Proteine werden jedoch ohne ein solches Signalpeptid aus der Zelle<br />
geschleust. Dies geschieht durch die nur in Ansätzen verstandene ‚unkonventionelle Sekretion‘.<br />
Die durch Stress in Inflammasomkomplexen aktivierte Protease Caspase-1 ist essentiell für die<br />
Aktivierung von proIL-1b, und Zellen ohne Caspase-1 schleusen kein IL-1b, zugleich aber auch<br />
weniger IL-1a aus. Um die Funktion von Caspase-1 bei der unkonventionellen Sekretion zu untersuchen,<br />
haben wir das Sekretom nach Aktivierung von Caspase-1 mittels iTRAQ-Proteomanalyse<br />
charakterisiert. Dabei zeigte sich, dass Caspase-1 für die Sekretion zahlreicher Proteine ohne<br />
Signalpeptid verantwortlich ist. Deren Eigenschaften sprechen dafür, dass die von Caspase-1<br />
regulierte unkonventionelle Sekretion ein inflammatorischer Weg ist, der die Entzündung des<br />
Gewebes direkt mit dessen Regeneration und dem Überleben der die Entzündung auslösenden<br />
Zellen verbindet 1 .<br />
Die Cysteinprotease Caspase-1 ist essentiell für<br />
die Aktivierung des proinflammatorischen Zytokins<br />
proIL-1b in Makrophagen. Makrophagen<br />
ohne Caspase-1 schleusen deshalb kein IL-1b<br />
aus. Überaschenderweise sekretieren diese<br />
Zellen aber auch weniger IL-1a, obwohl dieses<br />
Zytokin kein Substrat des Enzyms ist und auch<br />
in der Pro-Form an den IL-1-Rezeptor binden<br />
kann 2 . Caspase-1 wird initial als ein inaktives<br />
Vorläuferprotein hergestellt, die Aktivierung<br />
des Prä-Enzyms findet in sogenannten Inflammasomkomplexen<br />
statt 3 (vgl. Hintergrund<br />
1). Keratinozyten, welche die äußerste Haut-<br />
<strong>Laborwelt</strong> Hintergrund 1<br />
schicht (Epidermis) aufbauen, sekretieren nach<br />
UV-Bestrahlung große Mengen an IL-1b, was<br />
Sonnenbrand hervorruft. Erst kürzlich wurde<br />
gezeigt, dass dies ebenfalls von Caspase-1 und<br />
Inflammasomproteinen abhängt 4,5 . In Caspase-<br />
1-Knock-out-Mäusen ist der Sonnenbrand<br />
deutlich schwächer ausgeprägt 4,5 . Interessanterweise<br />
werden nicht nur IL-1a und -b ohne<br />
eine Signalsequenz über den unkonventionellen<br />
Mechanismus aus der Zelle geschleust 6,7 . Auch<br />
Caspase-1 selbst und andere Inflammasomproteine<br />
besitzen keine Signalsequenz, befinden<br />
sich aber nach UV-Bestrahlung trotzdem im<br />
Aktivierung von Inflammasomen führt zur Entzündung<br />
Inflammasome (hier: NALP3-Inflammasom)<br />
sind Proteinkomplexe, die für die Aktivierung<br />
der Protease Caspase-1 benötigt werden 3 .<br />
Caspase-1 kann dann das proinflammatorische<br />
Zytokin proIL-1b aktivieren, dessen Ausschleusung<br />
durch Rekrutierung und Aktivierung<br />
inflammatorischer Zellen zur Entzündung<br />
führt. Inflammasome werden durch bakterielle<br />
Toxine, virale DNA und bakterielle RNA<br />
aktiviert 3 . Zytoplasmatische DNA – und damit<br />
auch jede Transfektion – führt ebenfalls zu<br />
einer Aktivierung der Caspase-1 9 . Dies gilt<br />
auch für Harnsäurekristalle, die für die Gicht<br />
verantwortlich gemacht werden 3 , und Asbest-<br />
oder Silikonpartikel 10 . Auch Keratinozyten exprimieren<br />
Inflammasomkomplexe, die durch<br />
UV-Strahlen aktiviert werden 4,5 .<br />
Abb. 1: Sekretomanalyse von Keratinozyten<br />
nach UVB-Bestrahlung<br />
Überstand der Zellen 4,5 . Diese Ergebnisse lassen<br />
eine Funktion des Inflammasoms und insbesondere<br />
von Caspase-1 bei der unkonventionellen<br />
Sekretion möglich erscheinen.<br />
Die Sekretion von IL-1a und FGF-2<br />
hängt von Caspase-1 ab<br />
Unkonventionell sekretierte Proteine besitzen keine<br />
Signalsequenz und werden nicht glykosyliert.<br />
Da der Mechanismus dieses Sekretionsweges<br />
nicht verstanden ist, kann die unkonventionelle<br />
Sekretion weder verfolgt noch inhibiert werden 6 .<br />
Deshalb ist es nur unter Schwierigkeiten möglich,<br />
die unkonventionelle Sekretion von Proteinen zu<br />
beweisen und diese von der passiven Freisetzung<br />
durch Zelllyse zu unterscheiden, insbesondere,<br />
weil die unkonventionelle Sekretion durch<br />
Stress-verursachende Faktoren induziert wird<br />
und deshalb auch immer zur Zelllyse führt 6,7 .<br />
Eine essentielle Kontrolle von Experimenten<br />
zur unkonventionellen Sekretion stellt deshalb<br />
der Nachweis von cytoplasmatischen Proteinen<br />
im Überstand und in den aktivierten Zellen dar,<br />
da deren Quantifizierung eine Abschätzung der<br />
Zelllyse erlaubt. Lactatdehydrogenase (LDH) und<br />
b-Aktin sind hierfür besonders geeignet 1 . Wenn<br />
die Caspase-1-Aktivität in Keratinozyten inhibiert<br />
oder die Expression der Protease reduziert wird,<br />
dann sekretieren auch diese Zellen nach UV-<br />
Bestrahlung signifikant weniger proIL-1a 1 .<br />
Fibroblast Growth Factor 2 (FGF-2), der eine<br />
wichtige Rolle bei der Reparatur des Gewebes<br />
und bei der Angiogenese spielt, besitzt ebenfalls<br />
keine Signalsequenz und wird unkonventionell<br />
sekretiert 6 . Interessanterweise hängt die<br />
Menge des von UVA-bestrahlten Fibroblasten<br />
sekretierten FGF-2 ebenfalls von der Expression<br />
und der Aktivität von Caspase-1 ab 1 .<br />
Identifizierung der Caspase-1-<br />
abhängigen Sekretion mittels iTRAQ<br />
UV-bestrahlte Keratinozyten bieten sich wegen<br />
ihrer Robustheit zur systematischen Identifizierung<br />
von Proteinen an, die abhängig von der<br />
Caspase-1-Aktivität und -Expression sekretiert<br />
werden. Zusätzlich lässt sich in diesen Zellen die<br />
34 | 9. Jahrgang | Nr. 3/2008 LABORWElT
<strong>Laborwelt</strong> Hintergrund 2<br />
Vergleich von Proteinen in verschiedenen Proben durch iTRAQ<br />
Die kürzlich entwickelte iTRAQ-Methode –<br />
eine Weiterentwicklung der ICAT-Methode –<br />
erlaubt den Vergleich von Proteinen in mehreren<br />
Proben ohne Verwendung von 2-D-Gelen 8 .<br />
iTRAQ beruht auf der unterschiedlichen chemischen<br />
Markierung (Tag) der Aminotermini<br />
von Peptiden, die durch Trypsin-Verdau aus<br />
Proteinen mehrerer Proben hervorgegangen<br />
sind. Die markierten Peptide werden dann<br />
vereint, fraktioniert und mit MALDI-MS/MS<br />
analysiert. Durch Datenbankvergleich werden<br />
die korrespondierenden Proteine identifiziert.<br />
Die Fragmentierung der an die Peptide gebundenen<br />
Tags führt zu einer Generierung von<br />
Reporterionen, die jeweils spezifisch für die<br />
Marker-Tags sind. Die Messung der Intensität<br />
dieser Reporterionen erlaubt eine relative<br />
Quantifizierung der durch Trypsinverdau<br />
erhaltenen Peptide. Damit lässt sich das Vorkommen<br />
von Proteinen in mehreren Proben<br />
relativ zueinander quantifizieren.<br />
Expression oder Aktivität von Caspase-1 mittels<br />
siRNA oder Inhibitoren sehr efffizient und schonend<br />
reduzieren 4 . Gleichwohl muss erwartet<br />
werden, dass sich auch im Überstand dieser<br />
Zellen eine große Menge an intrazellulären Proteinen<br />
findet, die von lysierten Zellen freigesetzt<br />
wurden. Zusätzlich werden natürlich sekretierte<br />
Protein mit Signalsequenz ganz unabhängig<br />
von der Caspase-1-Aktivität ausgeschleust. Also<br />
geht es darum, eine Methode zur Identifizierung<br />
von Proteinen zu verwenden, die trotz eines<br />
starken Auftretens vieler Proteine in gleichen<br />
Mengen (Caspase-1-unabhängige Sekretion und<br />
Freisetzung wegen Zelllyse) in beiden Proben<br />
eine Identifizierung der differentiell sekretierten<br />
Proteine (Caspase-1-abhängige Sekretion)<br />
erlaubt. Diese Voraussetzungen erfüllt das<br />
iTRAQ-Verfahren (isobaric tags for relative and<br />
absolute quantitation) 8 (vgl. Hintergrund 2).<br />
Primäre Keratinozyten wurden entweder<br />
mit Caspase-1-Inhibitoren behandelt, oder die<br />
Expression der Protease wurde mit Hilfe von<br />
siRNAs in einem unabhängigen Experiment<br />
reduziert (siehe Abb. 1). Vier Stunden nach<br />
der UV-Bestrahlung dieser Zellen sowie von<br />
Kontrollzellen wurden die Überstände isoliert,<br />
konzentriert und nach einem Verdau der Proteine<br />
mit Trypsin mit den iTRAQ-Tags markiert.<br />
Nach der Vereinigung beider Proben und einer<br />
Fraktionierung erfolgte die Analyse mittels<br />
MALDI-MS/MS (vgl. Hintergrund 2). Wie erwartet,<br />
ist die Sekretion oder passive Freisetzung<br />
der meisten Proteine nicht abhängig von der<br />
Caspase-1-Aktivität oder -Expression (iTRAQ-<br />
Verhältnis um 1). Aber eine Reihe von Proteinen,<br />
die im Überstand der Kontrollzellen in deutlich<br />
größerer Menge auftraten (iTRAQ-Verhältnis<br />
>1), wurde identifiziert (siehe Tabelle 1, Seite 36).<br />
Die iTRAQ-Ergebnisse wurden für verschiedene<br />
Proteine mittels Western-blot in Keratinozyten<br />
und aktivierten Makrophagen verifiziert 1 .<br />
Schlussfolgerungen: iTRAQ<br />
Obwohl die überwiedende Mehrzahl der<br />
Proteine in gleichen Mengen im Überstand<br />
der Caspase-1-inhibierten Keratinozyten und<br />
Kontrollzellen zu finden war, gelang mit der<br />
iTRAQ-Methode die Identifizierung einer Reihe<br />
von Proteinen, deren Sekretion von der Caspase-1-Expression<br />
und -Aktivität abhängt. Für<br />
alle untersuchten iTRAQ-Kandidaten gelang<br />
eine Verifizierung der Caspase-1-abhängigen<br />
Sekretion sowohl in Keratinozyten als auch in<br />
Makrophagen. Damit hat sich iTRAQ als eine<br />
der Aufgabenstellung gewachsene Methodik<br />
erwiesen, die zudem experimentell leicht und<br />
schnell durchzuführen ist.<br />
Schlussfolgerungen: Caspase-1<br />
und unkonventionelle Sekretion<br />
Von einer Reihe der mit dem iTRAQ-Verfahren<br />
identifizierten Proteine (siehe Tabelle 1) war<br />
eine unkonventionelle Sekretion bereits<br />
bekannt 6,7 , nicht dagegen die Abhängigkeit<br />
ihrer Sekretion von Caspase-1. Zudem wurden<br />
Proteine identifiziert, die trotz des Fehlens<br />
einer Signalsequenz verstärkt von Kontroll-<br />
Keratinozyten ausgeschleust wurden. Einige<br />
dieser Proteine (z.B. Bid) haben keine bekannte<br />
extrazelluläre Funktion, spielen jedoch eine<br />
wichtige intrazelluläre Rolle in der Apoptose. Die<br />
Sekretion dieser Proteine wie auch die Sekretion<br />
LABORWElT 9. Jahrgang | Nr. 2/2008 | 35
Blitzlicht Proteomics/<strong>Zellbiologie</strong><br />
Protein Funktion Subz. Lokalisation SP Sekretion Caspase-1 siRNA YVAD<br />
Death-associated protein 1 (P51397) Induktion TNF-assoziierter Apoptose ZP – unbekannt 2.747<br />
Synaptotagmin-2 (A0FGR8) regul. Freisetzung v. Neurotransmitter TMP – unbekannt 2.460(±0.884)<br />
Testin (Q9UGI8) Tumorsuppressor; induziert Apoptose ZP – unbekannt 2.114 1.211<br />
Galectin-1 (P09382) reguliert Apoptose, Differenzierung u.<br />
Immunantwort<br />
ZP, EZ – unkonventionell 1.930(±0.274) 0.939(±0.136)<br />
Urokinase (Q5SWW8) aktiviert Plasminogen – unbekannt<br />
Bid (P55957) Apoptose ZP, mitoch. Membran – unbekannt 1.889<br />
Annexin A4 (Q6LES2) bindet Phospholipide unbekannt – unbekannt 1.687(±0.270 1.332(±0.250)<br />
IL-1alpha (P01583) proinflammatorisches Zytokin ZP, EZ – unkonventionell 1.666(±0.324)<br />
Destrin (P60981) Aktin-Depolymerisation ZP – unbekannt 1.631(±0.181) 0.867(±0.095)<br />
Maspin (P36952) Protease-Inhibitor; Matrix-Degeneration ZP, EZ – unbekannt 1.527(±0.244)<br />
LDL receptor (P01130) bindet LDL TMP + klassisch 1.472(±0.100) 1.093(±0.123)<br />
Thioredoxin-1 (P10599) Redox-Regulation ZP, EZ – unkonventionell 1.426(±0.073) 0.957(±0.257)<br />
ASC (Q9ULZ3) Komponente des Inflammasoms ZP, EZ – unkonventionell 1.420 0.991(±0.038)<br />
Galectin-3 (P17931) Lektin, welches IgE bindet Nukleus, EZ – unkonventionell 1.327(±0.152) 1.220(±0.095)<br />
IL-1Ra (P18510) inhibiert IL-1R Typ I; IL-1-Antagonist ZP, EZ + klassisch 1.221(±0.096) 1.220(±0.201)<br />
LDH (P00338) Metabolismus; Lyse-Marker ZP – nein 1.096(±0.083) 1.071(±0.204)<br />
Gelsolin Zellmotilität ZP, EZ + klassisch 1.077(±0.135)<br />
Thymosin beta-10 (P63313) zytoskelettale Organisation ZP, EZ - unkonventionell 0.919(±0.150) 1.255(±0.194)<br />
Cystatin-A (P01040) inhibiert Cathepsin ZP, EZ - unkonventionell 0.827(±0.105) 1.251<br />
GAPDH (P04406) Metabolismus ZP - unbekannt 0.464(±0.026) 0.699(±0.050)<br />
EPLIN (Q59FE8) Aktindynamik ZP - unbekannt 1.069(±0.166) 0.742(±0.089)<br />
Tab. 1: Funktion und Eigenschaften ausgewählter mittels iTRAQ (letzte beide Spalten) identifizierter Proteine. Keratinozyten wurden vor der<br />
UV-Bestrahlung mit Caspase-1-Inhibitor (YVAD) oder siRNA gegen Caspase-1 (siRNA) behandelt. Angegeben sind die Mittelwerte und<br />
das 95%ige Vertrauensintervall aller identifizierten und quantifizierten Peptide eines Proteins. Abkürzungen: ZP, Zytoplasma; EZ, extrazellulär;<br />
TMP, Transmembranprotein, SP, Signalpeptid.<br />
von Caspase-1 selbst könnte ein Überleben<br />
der Zellen erleichtern. Die Aktivierung von<br />
Caspase-1 in Inflammasom-Komplexen führt<br />
also nicht nur zur Aktivierung von proinflamma<br />
torischen Zytokinen wie proIL-1b, sondern<br />
auch zur Induktion der unkonventionellen<br />
Abb 2: Modell der Caspase-1-abhängigen Protein-Sekretion.<br />
Sekretion anderer Proteine (siehe Tabelle 1).<br />
Diese Proteine modulieren die Entzündung<br />
(MIF), spielen eine Rolle bei der Gewebereparatur<br />
und Angiogenese (FGF-2), befreien<br />
das Gewebe von reaktiven Sauerstoffspezies<br />
(ROS) (Thioredoxin-1, Peroxiredoxin-1) oder<br />
sind proapoptotisch (Bid) (vgl. Abbildung 2).<br />
Caspase-1 und Inflammasome spielen also eine<br />
deutlich vielschichtigere Rolle bei der Entzündung<br />
als bisher angenommen.<br />
Literatur<br />
[1] Keller, M., Rüegg, A., Werner, S., Beer, H.-D., Cell 132 (2008),<br />
818-831<br />
[2] Kuida, K., Lippke, J.A., Ku, G., Harding, M.W., Livingston, D.J.,<br />
Su, M.S., Flavell, R.A., Science 267 (1995), 2000-3<br />
[3] Ogura, Y., Sutterwala, F.S., Flavell, R.A., Cell 126 (2006),<br />
659-662<br />
[4] Feldmeyer, L., Keller, M., Niklaus, G., Hohl, D., Werner, S., Beer,<br />
H.-D., Curr. Biol. 17 (2007), 1140-1145<br />
[5] Faustin, B., Reed, J.C. Trends Cell Biol. 18 (2008), 4-8<br />
[6] Nickel, W. Eur. J. Biochem. 270 (2003), 2109-2119<br />
[7] Rubartelli, A., Curr. Med. Chem. 4 (2005), 133-40<br />
[8] Ross, P.L., Huang, Y.N., Marchese, J.N., Williamson, B., Parker,<br />
K., et al., Mol. Cell. Proteomics 3 (2004), 1154-1169<br />
[9] Muruve, D.A., Petrilli, V., Zaiss, A.K., White, L.R., Clark, S.A.,<br />
Ross, P.J., Parks, R.J., Tschopp, J., Nature 452 (2008), 103-7<br />
[10] Dostert, C., Petrilli, V., Van Bruggen, R., Steele, C., Mossman,<br />
B.T., Tschopp, J., Science (2008), Apr 10, Epub ahead of print<br />
Korrespondenzadresse<br />
Dr. Hans-Dietmar Beer<br />
ETH Zürich-Institut für <strong>Zellbiologie</strong><br />
Schafmattstr. 18<br />
CH-8093 Zürich<br />
Tel.: +41-(0)44-633-3405<br />
Fax: +41-(0)44-633-1174<br />
dietmar.beer@cell.biol.ethz.ch<br />
www.cell.biol.ethz.ch<br />
36 | 9. Jahrgang | Nr. 3/2008 LABORWElT
Verstärkung der<br />
Chemosensitivität von<br />
Tumorzellen mittels RP101<br />
Prof. Dr. Rudolf Fahrig, RESprotect GmbH, Dresden<br />
Die Chemotherapie ist derzeit die Standardtherapie für Krebserkrankungen. Die für die<br />
Therapie eingesetzten Zytostatika treiben Tumorzellen in den Selbstmord. Leider ist die erfolgreiche<br />
Behandlungsperiode mit den gegenwärtig auf dem Markt befindlichen Zytostatika<br />
meist nicht lang genug, um den Tumor gänzlich zu vernichten. Der Tumor entwickelt nämlich<br />
Abwehrmechanismen gegenüber der Behandlung; er wird resistent. Die von RESprotect<br />
entwickelten Arzneimittel verhindern diese Resistenzbildung von Tumorzellen gegenüber<br />
Chemotherapien und erhöhen die Immunabwehr der Patienten. Im Gegensatz zu den seit<br />
Jahrzehnten bekannten und meist erfolglosen Versuchen, existierende Chemoresistenzen<br />
zu umgehen oder zu mindern, gibt es für diesen technologischen Ansatz weltweit keine<br />
Konkurrenz. Das erste von uns entwickelte Arzneimittel RP101 zeigte in Kombination mit<br />
Zytostatika in zwei kleinen klinischen Studien mit Bauchspeicheldrüsen-Krebspatienten<br />
eine lebensverlängernde Wirkung, welche jene bisheriger Therapien bei weitem übertraf.<br />
Darüber hinaus führte die RP101-Behandlung zu keinen schädlichen Nebenwirkungen, wie<br />
sie bei Behandlung mit Zytostatika immer zu beklagen sind. Da RP101 potentiell mit jedem<br />
Zytostatikum und bei allen Krebsarten wirken sollte, eröffnen sich neue Möglichkeiten für<br />
die Chemotherapie und damit für die Patienten.<br />
Die von RESprotect genutzte Technologie, die<br />
Chemogenomik, besteht in der Verwendung<br />
kleiner Moleküle (small molecules), welche<br />
die Expression bestimmter Gene in einer für<br />
den Patienten günstigen Weise beeinflussen.<br />
Die Konzentration auf bestimmte biologische<br />
Prozesswege führt zu einem besseren<br />
Verständnis der Wirkung eines Arzneimittels.<br />
Auf diese Weise ist die Entdeckung von<br />
Arzneimitteln möglich, deren Wirkung auf<br />
die Ursachen und nicht die Symptome einer<br />
Krankheit gerichtet ist. Die Arzneimittel von<br />
RESprotect werden zusätzlich zur Standard-<br />
Chemotherapie verabreicht. Die Chemotherapie<br />
beruht darauf, dass Tumorzellen in den<br />
Selbstmord (Apoptose) getrieben werden.<br />
Hauptproblem der Chemotherapie ist das<br />
More Modes<br />
Molecular Devices’ multi-detection readers:<br />
SpectraMax M5 e (top) and FlexStation 3<br />
with integrated fluid transfer (bottom).<br />
now part of MDS Analytical Technologies<br />
tel. +44-118-944-8000 | www.moleculardevices.com<br />
Molecular Devices is the #1 supplier of tunable microplate readers. Our two dual-<br />
monochromator multi-detection readers—the SpectraMax ® M5 e and FlexStation ® 3<br />
with integrated compound addition—give you superior results in all modes.<br />
Widest spectrum of absorbance, fluorescence, and luminescence applications in<br />
microplates or cuvettes<br />
FP performance that is unparalleled by any tunable instrument<br />
TRF, HTRF ® , IMAP ® TR-FRET, and other TR-FRET assay capabilities<br />
Integrated plate stacker option for SpectraMax M5e Direct reagent transfer with 8- or 16-channel pipettor option on FlexStation 3 to<br />
increase kinetic assay throughput<br />
Complete SpectraTest hardware and SoftMax ® Pro GxP software validation tools<br />
Whether you’re screening at high throughput or developing assays in cuvettes or 6-<br />
to 384-well microplates, the SpectraMax M5 e and FlexStation 3 can address a wider<br />
array of assays.<br />
Blitzlicht Krebs<br />
Auftreten von Chemoresistenz, welche die<br />
Apoptose verhindert.<br />
Die RESprotect-Technologie, die Chemogenomik,<br />
führt zur Identifizierung von<br />
Zielgenen, die an der Entwicklung der Chemoresistenz<br />
beteiligt sind.<br />
Unser erstes Arzneimittel RP101 unterdrückt<br />
die Überexpression Apoptose-hemmender<br />
Gene, die durch die Behandlung mit<br />
Zytostatika induziert wird.<br />
RP101-Effekt auf Pankreaskrebszellen<br />
In Pankreaskrebszellen (Bauchspeicheldrüsenkrebszellen)<br />
wird das Onkogen, das<br />
heißt krebserzeugende Gen STAT3 häufig<br />
überexprimiert und durch die Zytostatika-<br />
Abb. 1: Wirkung von RP101 auf STAT3-Proteinmenge<br />
(1,2 und 3 stellen die Ergebnisse<br />
von drei unabhängig durchgeführten<br />
Versuchen dar).<br />
behandlung noch weiter hochreguliert. Hierdurch<br />
wird die durch Zytostatika induzierte<br />
Apoptose (Selbstmord der Tumorzellen nach<br />
Schädigung) verhindert und die Krebszellen<br />
zeigen dementsprechend keinerlei Reaktion<br />
auf eine Chemotherapie.<br />
Die RP101-Ko-Behandlung verhindert<br />
jedoch die Hochregulierung von STAT3 und<br />
dessen Zielgen VEGF und macht die Zellen<br />
hierdurch empfindlich gegenüber der Chemotherapie.<br />
Behandlung mit Zytostatika führt in<br />
Krebszellen zur Schädigung der Erbsubstanz<br />
DNA und treibt die Zelle hierdurch zum<br />
Selbstmord. Tumorzellen, insbesondere<br />
Pankreaskrebszellen können dem Selbstmord<br />
(Apoptose) durch Überexpression des<br />
DNA-Reparaturgens APEX/ref-1 entgehen<br />
und hierdurch resistent gegenüber der Chemotherapie<br />
werden. RP101-Ko-Behandlung<br />
verhindert diese Überexpression und erhält<br />
auf diesem Wege die Empfindlichkeit der<br />
Tumorzellen gegenüber der Zytostatikabehandlung<br />
aufrecht.<br />
Spezifisch in Pankreaskarzinomzellen wird<br />
durch RP101-Ko-Behandlung die Hochregulierung<br />
der Uridin-Phosphorylase (UPase)<br />
verhindert.<br />
Die Expression der UPase scheint durch<br />
Onkogene, Tumorsuppressor-Gene und<br />
Zytokine kontrolliert zu werden, und die<br />
Aktivität der UPase ist in Tumoren meist<br />
höher als in gesundem Gewebe. Patienten<br />
mit erhöhtem UPase-Spiegel haben eine<br />
schlechte Prognose.<br />
LABORWElT 9. Jahrgang | Nr. 3/2008 | 37<br />
More Modes ad rev D half-pg A4 122.5x90 mm—LWM.indd 1 5/1/08 4:32:53 PM
Blitzlicht Krebs<br />
Tab. 1: Genregulation in Pankreaskrebszellen als Resultat der RP101-Behandlung –<br />
Quantitative real-time PCR<br />
Genregulation als Resultat<br />
der RP101-Behandlung<br />
uridine phosphorylase, UPase BxPC-3<br />
AsPC-1<br />
natural killer cell transcript 4,<br />
NK 4, IL-32<br />
lymphotoxin-alpha, LTB<br />
tumor necrosis factor C<br />
lymphotoxin beta, LTA, tumor<br />
necrosis factor beta<br />
tumor necrosis factor LIGHT,<br />
TNFSF14<br />
Hum. Pankreas-<br />
Adeno karzinom-<br />
Zellinie<br />
CAPAN-2<br />
AsPC-1<br />
CAPAN-2<br />
AsPC-1<br />
CAPAN-2<br />
AsPC-1<br />
CAPAN-2<br />
AsPC-1<br />
ICAM-1 CAPAN-2<br />
AsPC-1<br />
VEGF, vascular endothelial<br />
growth factor<br />
APEX, ref-1, apurinic<br />
endonuclease<br />
CAPAN-2<br />
AsPC-1<br />
Spezifisch in Pankreaskarzinomzellen werden<br />
durch RP101-Ko-Behandlung die Lymphotoxine<br />
alpha und beta, das „natural<br />
killer cell trancript 4“, der „tumor necrosis<br />
factor LIGHT“ und ICAM-1 hochreguliert.<br />
Die Produkte dieser Gene erhöhen die Anti-<br />
Tumor-Immunität. Die Wirkungen von RP101<br />
in Pankreaskrebszellen auf Genebene finden<br />
sich in Tabelle 1.<br />
In Abbildung 2 ist zu sehen, dass Kobehandlung<br />
mit RP101 die Wirkung von „killer cells“<br />
erhöht, also einen positiven Einfluss auf die<br />
Immunabwehr ausübt. Die gelben Flächen<br />
zeigen die durch Abtötung der Tumorzellen<br />
entstandenen Lücken im Zellrasen.<br />
Wirkung auf Krebspatienten<br />
In zwei voneinander unabhängigen Phase I/<br />
II-Studien mit metastasierten Bauchspeicheldrüsenkrebspatienten<br />
wurde RP101 in Kombination<br />
mit Gemcitabin, der gegenwärtigen<br />
Standardtherapie, sowie mit Gemcitabin<br />
+ Cisplatin eingesetzt. Diese Ergebnisse<br />
wurden kürzlich veröffentlicht 1 . Die RP101-<br />
Ko-Behandlung führte zu einer ungefähren<br />
Verdoppelung der medianen Überlebenszeit.<br />
Darüber hinaus lebten vier- beziehungsweise<br />
fünfmal mehr Patienten mit Fernmetastasen<br />
(Stadium IV) ein Jahr oder länger im Vergleich<br />
zu den Patienten, die mit Chemotherapie<br />
allein behandelt worden waren.<br />
Phase II – Pilotstudie<br />
Dreizehn Patienten mit Metastasen (neun<br />
mit Fernmetastasen in Stadium IV und<br />
vier mit Nahmetastasen in Stadium III der<br />
Krankheit) wurden mit in einer klinischen<br />
Phase II-Studie mit Gemcitabin, Cisplatin<br />
und RP101 behandelt. 77% der Patienten mit<br />
RP101 vs. DMSO<br />
Kontrol. (%)<br />
keine Veränderung<br />
q 64<br />
q 160<br />
q 50<br />
q 180<br />
q 370<br />
q 20<br />
q 70<br />
q 10<br />
q 30<br />
q 32<br />
q 53<br />
Q 37<br />
Q6<br />
Gemcitabine + RP101<br />
vs. GEM allein (%)<br />
Q 66<br />
Q 73<br />
q 125<br />
q 63<br />
q 224<br />
q 760<br />
q 76<br />
q94<br />
q 85<br />
q 200<br />
q 54<br />
q 135<br />
Q 34<br />
Q 27<br />
CAPAN-2 keine Veränderung Q 36<br />
Fernmetastasen lebte länger als ein Jahr nach<br />
Start der Behandlung. Dies bedeutet, dass<br />
von den Patienten in Stadium IV viermal mehr<br />
ein Jahr überlebten als Patienten, die nur mit<br />
Zytostatika behandelt wurden.<br />
Phase II – Dosis-Findungs-Studie<br />
Eine Phase II-Dosis-Findungs-Studie mit<br />
22 metastasierten Patienten wurde in drei<br />
Zentren in Deutschland durchgeführt. Die<br />
Ergebnisse stimmten mit denen der Pilotstudie<br />
überein. Die Ein-Jahres-Überlebensrate<br />
betrug 43%.<br />
Alle diese Patienten waren in Stadium IV,<br />
hatten also Fernmetastasen (Leber, Lunge<br />
etc.). Von diesen Patienten mit der normalerweise<br />
geringsten medianen Überlebenszeit<br />
erlebten fünfmal mehr ein Jahr als Stadium<br />
IV-Patienten der Kontrollgruppe. Die jetzt<br />
A<br />
D<br />
Abb. 2: Wirkung von RP101 auf die zytolytische Aktivität von NK-92-„natural killer<br />
cells“ . (a) CAPAN-2-Zellen+GEM. (b) CAPAN-2-Zellen+NK-92-Zellen. (c) CAPAN-2-<br />
Zellen+GEM+NK-92-Zellen. (d) CAPAN-2-Zellen+GEM+RP101. (e) CAPAN-2-Zellen<br />
+RP101+NK-92-Zellen. (f) CAPAN-2-Zellen +GEM+RP101+NK-92-Zellen.<br />
38 | 9. Jahrgang | Nr. 3/2008 LABORWElT<br />
B<br />
E<br />
begonnene klinische Studie schließt aus<br />
diesem Grund ausschließlich Stadium IV-<br />
Patienten ein.<br />
Es zeigte sich hierbei außerdem die Tendenz,<br />
dass sich die Lebensqualität der behandelten<br />
Patienten durch RP101-Ko-Behandlung<br />
verbesserte. Es konnten keine negativen<br />
Nebenwirkungen festgestellt werden. Eine<br />
Phase II/III Studie begann im Oktober 2007<br />
in den USA und Europa. Etwa 55 Zentren und<br />
153 Patienten mit Fernmetastasen werden<br />
involviert sein.<br />
Tab.2: Überlebenszeit bei Standardtherapie,<br />
in der Dosisfindungs- und Pilotstudie<br />
Therapie 6 Monate 12-Monate<br />
Chemotherapie allein<br />
ohne RP101<br />
45 % 22 %<br />
RP101 + Gemcitabin 71 % 43 %<br />
RP101 + Gemcitabin +<br />
Cisplatin<br />
92 % 77 %<br />
Literatur<br />
[1] Fahrig, R., Heinrich, J.C., Nickel, B., Wilfert, F., Leisser,<br />
C., Krupitza, G., Praha, C., Sonntag, D., Fiedler, B.,<br />
Scherthan, H., and Ernst, H. Cancer Res. 63 (2003) 5745<br />
-5753.<br />
[2] Fahrig, R., Quietzsch, D., Heinrich, J-C., Heinemann, V.,<br />
Boeck, S., Schmid, R.M., Praha, C., Liebert, A., Sonntag,<br />
D., Krupitza, G., and Haenel, M. Anti-Cancer Drugs 17<br />
(2006) 1045-1056.<br />
Korrespondenzadresse<br />
Prof. Dr. Rudolf Fahrig<br />
RESprotect GmbH<br />
Fiedlerstr. 34<br />
01307 Dresden<br />
Tel.: +49-(0)351-450-3201<br />
Fax: +49-(0)351-450-3210<br />
fahrig@resprotect.de<br />
www.resprotect.de<br />
C<br />
F
Ionenkanal-Screening<br />
auf den Kopf gestellt<br />
Martina Knirsch, Albrecht Lepple-Wienhues, Dirck Lassen, flyion GmbH, Tübingen;<br />
Emmanuel Bourinet, IGH CNRS, Institute de Genomique Fonctionelle, Montpellier<br />
Pharmakologische Studien an Ionenkanälen gewinnen in der Arzneimittelforschung zunehmend<br />
an Bedeutung. In den vergangenen Jahren konnten immer häufiger Zusammenhänge zwischen<br />
Ionenkanalproteinen, einer Vielzahl von Erkrankungen und pharmakologischen Nebenwirkungen<br />
aufgeklärt werden. Die größte Herausforderung bei der Entwicklung ionenkanalspezifischer<br />
Arzneimittel besteht in dem für deren funktionelle Untersuchung notwendigen aufwendigen<br />
Messverfahren. Als beste Methode hierfür hat sich die Patch Clamp-Technik weltweit etabliert.<br />
Da die manuelle Handhabung der Patch Clamp-Technik ein hohes Maß an Erfahrung und Fingerspitzengefühl<br />
erfordert und zudem der Messdurchsatz limitiert ist, gab es in den vergangenen<br />
fünf Jahren zahlreiche Ansätze, diese Methode zu automatisieren und zu vereinfachen. Dennoch<br />
erweisen sich viele automatisierte Systeme im Vergleich zum manuellen Patch Clamp hinsichtlich<br />
ihrer Erfolgsraten sowie der Messqualität als unzureichend. Das im Folgenden vorgestellte<br />
vollständig automatisierte Patch Clamp-System basiert auf der „Flip-the-Tip“-Technologie,<br />
einer Invertierung des klassischen Patch Clamp-Verfahrens unter Verwendung der bewährten<br />
Glaspipetten. Diese Methode zeichnet sich durch maximale Erfolgsraten bei hoher Messqualität<br />
unter Verwendung von herkömmlichen physiologischen Lösungen ohne Zusatz von Fluorid<br />
sowie durch extrem schnellen Lösungswechsel (~50 ms) bei kleinsten Applikationsvolumina<br />
(
Blitzlicht Patch Clamp<br />
A<br />
(TRPP, Zystenniere) und Mukolipidose IV<br />
(TRPML) 4 führt.<br />
Automationsansätze<br />
Es gibt heute eine ganze Reihe von Methoden,<br />
um Kandidaten für Ionenkanalwirkstoffe zu<br />
testen. Hierzu zählen elektrophysiologische<br />
Messungen (Patch Clamp), Bindungs-Assays,<br />
radioaktive Flux-Assays, membranpotentialabhängige<br />
Fluoreszenzfarbstoffe und<br />
spannungsabhängiger Fluoreszenzresonanzenergietransfer<br />
(FRET). Jedoch lediglich das<br />
Patch Clamp-Verfahren erlaubt es, winzige<br />
Ionenströme (10 –12 A) durch Kanalproteine<br />
in intakten Zellen mit physiologischer<br />
Membran orientierung unter vollständiger<br />
Spannungskontrolle und exakter Beeinflussung<br />
der chemischen Zusammensetzung<br />
auf beiden Membranseiten in Echtzeit zu<br />
messen.<br />
Bei der traditionellen Patch Clamp-Technik<br />
wird eine Glaspipette mit einer sehr kleinen<br />
Öffnung an einer Zellmembran angesetzt<br />
und ein geringer Unterdruck zum Inneren<br />
der Pipette angelegt. Durch den Sog bil-<br />
Abb. 2: Der vollautomatische Patch Clamp-<br />
Roboter Flyscreen® 8500<br />
B<br />
Abb. 1: A. Konventionelles Patch Clamp: eine einzelne Zelle bildet einen Gigaseal an der Spitze<br />
einer Glasspipette. B: Flip-the-Tip-Patch Clamp: eine Zelle versiegelt die Pipette von<br />
innen und bildet dort einen Gigaseal.<br />
det die Zelle eine stabile Versiegelung mit<br />
der Glasoberfläche der Pipette aus (eine<br />
sogenannte Gigaohm-Versiegelung oder<br />
Gigaseal, da der elektrische Widerstand<br />
ein Gigaohm übersteigt). Der anschließend<br />
applizierte Unterdruck ist notwendig, um<br />
einen Teil der Zellmembran aufzureißen<br />
und so Zugang zum Intrazellularraum zu erhalten<br />
(Abb. 1A). Folglich können über einen<br />
an die Pipette angeschlossenen Verstärker<br />
Spannungspulse an der Zellmembran angelegt<br />
und der aus dem transmembranären<br />
Ionenfluss resultierende elektrische Strom<br />
gemessen werden.<br />
Leider ist diese konventionelle manuelle<br />
Technik sehr langsam und erfordert ein<br />
hohes Maß an Erfahrung und Fingerspitzengefühl.<br />
Daher wurden in den vergangenen<br />
fünf Jahren verschiedene Versuche unternommen,<br />
das Verfahren zu automatisieren<br />
und zu vereinfachen. Dabei haben viele automatisierte<br />
Geräte unerwartete Probleme,<br />
die zu unakzeptabel niedrigen Erfolgsraten<br />
bei den Messungen sowie zu Abweichungen<br />
der pharmakologischen Ergebnisse füh ren.<br />
Die Flip-the-Tip ® -Technologie<br />
Die meisten automatischen Verfahren führen<br />
das Patch Clamp-Experiment in einem<br />
Messgefäß durch, das einem planaren Chip<br />
gleicht, in den ein Loch gebohrt wurde. Aus<br />
diesem Aufbau resultieren Schwierigkeiten,<br />
die teilweise auf das verwendete Chip-Material<br />
zurückzuführen sind, aber auch systembedingt<br />
auf die planare Anordnung.<br />
Um diese Probleme zu vermeiden, haben wir<br />
uns gegen einen planaren Versuchsaufbau<br />
entschieden und statt dessen das klassische<br />
Experiment einfach auf den Kopf gestellt<br />
(Flip-the-Tip) 5 . Dabei wird das Patch Clamp-<br />
Experiment innerhalb einer herkömmlichen<br />
Patch-Pipette durchgeführt, indem eine<br />
Zellsuspension direkt in die Spitze der Pipette<br />
gespült wird. Der hierbei entstehende Seal<br />
im Gigaohmbereich entsteht äußerst schnell,<br />
ist extrem stabil sowie langlebig und wird<br />
weder durch ein Bewegen der Pipette noch<br />
durch das Einspülen von Lösungen beeinträchtigt<br />
(Abb. 1B).<br />
Im vollautomatischen Flyscreen®-Roboter<br />
(Abb. 2) und in der halbautomatischen<br />
PatchBox wurde diese Technologie umgesetzt.<br />
Während im Flyscreen die Ausbildung<br />
des Seals, die Etablierung der Whole Cell-<br />
Konfiguration und die Applikation von Substanzen<br />
auf drei bis sechs parallelen Kanälen<br />
vollautomatisiert durchführbar ist, verfügt<br />
dessen „kleiner Bruder“, die PatchBox, ebenfalls<br />
über eine integrierte Automatisierung<br />
des Seal- und Whole Cell-Vorgangs, erlaubt<br />
jedoch zugleich den Einsatz selbstgezogener<br />
Glasspipetten und erfordert eine manuelle<br />
Handhabung der Lösungen.<br />
An die verwendeten Materialien werden<br />
gerade bei pharmakologischen Anwendungen<br />
hohe Anforderungen gestellt. So zeigen die in<br />
planaren Chip-Systemen eingesetzten Kunststoffmaterialien<br />
insbesondere mit lipophilen<br />
Substanzen häufig unerwünschte Wechselwirkungen.<br />
Aus diesem Grund stehen Zellen, Lösungen<br />
und applizierte Substanzen in den von<br />
Flyion entwickelten Systemen ausschließlich<br />
mit Pipettenoberflächen aus Glas in Kontakt.<br />
Im Gegensatz zu anderen automatisierten<br />
Patch Clamp-Systemen, die den Einsatz von<br />
Fluorid in der Intrazellulärlösung erfordern,<br />
um stabile Gigaseals zu erzielen, werden in<br />
dem hier vorgestellten System ausschließlich<br />
physiologische Lösungen verwendet. Dies<br />
hat den Vorteil, dass damit unerwünschte<br />
Nebeneffekte wie Chelatbildungen des Fluorids<br />
mit Kalzium- und Magenesiumionen<br />
oder im schlimmsten Fall das Präzipitieren als<br />
CaF 2 und MgF 2 ausbleiben. Gravierender noch<br />
Abb. 3: Eine Quarzkapillare mit 170 µm Außendurchmesser<br />
wird in die Patch-<br />
Pipette eingeführt und appliziert<br />
dort die Wirkstofflösungen. Die<br />
einzelnen Videobilder wurden im<br />
Abstand von 100 ms aufgenommen<br />
und illustrieren den laminaren Fluss<br />
beim Lösungswechsel.<br />
sind die nicht kontrollierbaren Auswirkungen<br />
des Fluorids auf von G-Proteinen gesteuerte<br />
Prozesse, Phosphatasen und Kinasen sowie die<br />
irreversible Bindung an zytosolische Proteine 6 ,<br />
was eine Verwendung fluoridhaltiger Lösungen<br />
für die Analyse zahlreicher Ionenkanäle<br />
ausschließt.<br />
40 | 9. Jahrgang | Nr. 3/2008 LABORWElT
Ebenfalls problematisch für pharmakologische Untersuchungen<br />
sind unerwünschte Wechselwirkungen mit einer zu hohen Anzahl<br />
applizierter Zellen, da diese aufgrund ihrer Membranlipide und<br />
Bindungsstellen die Abläufe an der zu untersuchenden Zelle beeinflussen<br />
können. In unseren Patch-Pipetten werden nur etwa 30<br />
bis 40 Zellen in die Spitze abgegeben und der Seal der ersten die<br />
„Seal Area“ erreichenden Zelle durch den anliegenden Unterdruck<br />
zuverlässig innerhalb von Sekunden etabliert. Die zu untersuchende<br />
Substanz wird über eine Kapillare nur 200 µm entfernt direkt auf die<br />
Zelle appliziert, was einen raschen und kompletten Austausch der<br />
Lösungen sicherstellt (Abb. 3).<br />
Dieser in anderen Patch Clamp-Technologien für die Stabilität des<br />
Seals oftmals kritische Prozess hat in den von Flyion entwickelten<br />
ChipTip-Pipetten aufgrund der Strömungseigenschaften innerhalb<br />
der Pipette einen Seal-stabilisierenden Effekt, was sich in den hohen<br />
Erfolgsraten von mehr als 70% widerspiegelt. Ebenso rasch und zu-<br />
Abb. 4: hERG: Dosiswirkungskurven für die Substanzen Propafenon<br />
und E4031<br />
verlässig können applizierte Substanzen wieder ausgewaschen und<br />
Kon trollmessungen durchgeführt werden.<br />
Im Verlauf klassischer manueller Patch Clamp-Ableitungen ist nach<br />
dem Erreichen der Whole Cell-Konfiguration häufig eine Erhöhung<br />
des Serienwiderstandes durch störende Mem branfragmente zu beobachten.<br />
Durch das Invertieren des Systems tritt dieser unerwünschte<br />
Anstieg des Zugriffswiderstandes bei der Flip-the-Tip-Methode<br />
dagegen niemals auf. Der Serienwiderstand beträgt hier weniger<br />
als 5 MΩ und bleibt während der Ableitungen stabil. Ein weiterer<br />
Vorteil dieser inversen Technologie zeigt sich in Perforated Patch-<br />
Experimenten. Der während des Sealvorgangs angelegte Unterdruck<br />
verhindert zunächst einen Kontakt der „Seal Area“ und der Zelle mit<br />
perforierender Intrazellulärlösung. Ist der Seal etabliert, wird der<br />
chemische Zugang zum Zellinneren extrem schnell hergestellt.<br />
Die Untersuchung physiologischer Prozesse erfordert oft temperaturabhängige<br />
Messungen. Diesen Anforderungen wird der Flyscreen-<br />
Roboter durch eine temperaturkontrollierte Innenkammer gerecht.<br />
Es können dabei Temperaturen in einem weiten Bereich innerhalb<br />
von ± 0,2°C stabil kontrolliert werden. Die Einsatzmöglichkeiten<br />
der Flip-the-Tip-Technik werden im Folgenden an drei Beispielen<br />
demonstriert.<br />
hERG-Screening<br />
Der spannungsaktivierte auswärtsgleichrichtende hERG(Human Ether-<br />
A-Go-Go Related Gen)-Kanal spielt beim Aktionspotential des Herzens<br />
für die Repolarisation eine entscheidende Rolle. Eine Blockade dieses<br />
Kanals kann daher zu einer Verlängerung des Aktionspotentials führen,<br />
dem Long QT-Syndrom. Zahlreiche Substanzen und Arzneimittel<br />
mussten in den vergangenen 10 Jahren vom Markt genommen werden,<br />
da sie als unerwünschte Nebenwirkungen die Funktionsweise des<br />
hERG-Kanals beeinflussten.<br />
Der mit dem Flyscreen Roboter-entwickelte Assay erlaubt ein<br />
Substanz-Screening des hERG-Kanals, wofür eine stabil transifizierte,<br />
induzierbare CHO (Chinese Hamster Ovary) TRex-Zellinie zur<br />
GE Healthcare<br />
Blitzlicht Patch Clamp<br />
Drop. Measure. Done.<br />
NanoVue, the easy-to-use<br />
spectrophotometer.<br />
www.gelifesciences.com/tryNanoVue<br />
LABORWElT 9. Jahrgang | Nr. 3/2008 | 41<br />
GE Healthcare Bio-Sciences AB, Björkgatan 30, 751 84 Uppsala, Sweden<br />
© 2008 General Electric Company – All rights reserved.<br />
GE33-07. First published November 2007
Blitzlicht Patch Clamp<br />
Tab. 1: Vergleich der mit dem Flip-the-Tip-<br />
Patch-Clamp-Verfahren bestimmten<br />
IC50-Werte mit den im konventionellen<br />
Patch Clamp ermittelten Werten<br />
Verfügung steht. Aufgrund des schnellen<br />
Lösungswechsels in ChipTips konnten schnell<br />
und langsam wirkende Arzneimittelsubstanzen<br />
unterschieden werden. Abbildung 4<br />
(Seite 41) zeigt Dosis-Wirkungskurven von<br />
hERG unter Einfluss von Propafenon und<br />
E4031. Sämtliche auf diese Weise erhaltene<br />
IC50-Werte zeigen eine klare Übereinstimmung<br />
mit den Daten, die durch manuelle<br />
Patch Clamp-Messungen gewonnen wurden<br />
(Tabelle 1).<br />
T-Typ-Kanäle<br />
Flyscreen ® 8500<br />
APC (nM)<br />
E4031 41 34<br />
terfenadine 184 35<br />
verapamil 548 378<br />
quinidine 889 915<br />
propafenone 1060 2000<br />
Manual Patch<br />
Literature<br />
(nM)<br />
Wird eine Zelle depolarisiert, so aktiviert<br />
dies in neuronalen Zellen Herzmuskelzellen<br />
und in der glatten Muskulatur spannungsgesteuerte<br />
T-Typ-Kalzium-Kanäle, die durch<br />
den Einstrom von Kalziumionen intrazelluläre<br />
Prozesse wie die Kontraktion, Sekretion,<br />
Ausschüttung von Neurotransmittern und<br />
Genexpression steuern. Eine wichtige Rolle<br />
spielen T-Typ-Kanäle bei zellulären Schrittmacherprozessen,<br />
bei der Schmerzwahrnehmung<br />
und Sensitivierung. Bislang ist kein<br />
selektiver T-Typ-Kanal-Inhibitor bekannt.<br />
Eine gezielte Blockierung der T-Typ-Kanäle<br />
wäre im Hinblick auf kardiovaskuläre Erkrankungen,<br />
einige Formen von Epilepsie und in<br />
der Schmerztherapie von großer klinischer<br />
Bedeutung. Dieses Ziel lässt sich jedoch nur<br />
mit einem umfassenden Hochdurchsatz-<br />
Screening dieser Kanäle verfolgen.<br />
Die hier für den T-Typ-Kanal Ca V 3.2<br />
präsentierten Daten wurden an stabil<br />
transfizierten CHO-Zellen mit Hilfe des<br />
Abb. 5: Ca V 2.3: Zeitlicher Verlauf der Strom-<br />
Antwort auf die Einspülung von 20<br />
mM Ca 2+ -Lösung, gefolgt durch anschließende<br />
Auswaschung mit physiologischer<br />
Ca 2+ -Lösung.<br />
Flyscreen-Roboters im Perforated Patch<br />
gewonnen. Die Abbildungen 5 und 6 zeigen<br />
den raschen Effekt einer Applikation hoher<br />
Ca 2+ -Konzentrationen (20 mM) und die zuverlässige<br />
und unmittelbare Auswaschung<br />
durch Einspülen einer Lösung mit physiologischer<br />
Ca 2+ -Konzentration. Die Messungen<br />
zeichnen sich durch ein optimales Signal-<br />
Rausch-Verhältnis aus, was insbesondere<br />
für die Aufzeichnungen dieser kleinen Ca 2+ -<br />
Kanalströme wichtig ist.<br />
Ligandenaktivierte Rezeptoren<br />
Nikotinische Acetylcholinrezeptoren finden<br />
sich im zentralen und peripheren Nervensystem<br />
und werden durch die Bindung<br />
des Neurotransmitters Acetylcholin (Ach)<br />
aktiviert. Die Schließung des jeweiligen<br />
Kanals erfolgt spontan auch in Anwesenheit<br />
von Acetylcholin. Die Aktivierung des<br />
Kanals führt durch den Einstrom von Na +<br />
und Ca 2+ zu einer Depolarisation der Zelle.<br />
Elektrophysiologische Untersuchungen an<br />
ligandenaktivierten Ionenkanälen wie dem<br />
nikotinischen Acetylcholinrezeptor erfordern<br />
eine präzise Applikation des Liganden<br />
sowie eine gute zeitliche Auflösung der<br />
Daten.<br />
Die hier vorgestellten Daten zeigen Messungen<br />
des nAch-Rezeptors in der humanen<br />
Zellinie TE671, die eine vollentwickelte Form<br />
dieses Kanals exprimiert (Abb. 7). Hierfür wurden<br />
Experimente mit ChipTips im Perforated<br />
Patch durchgeführt und dabei 10 µM Ach in<br />
Abständen von 12 s appliziert. Die Abbildungen<br />
zeigen eine gute Auflösung dieser kleinen<br />
Amplituden und die präzise Applikation des<br />
Liganden.<br />
Fazit<br />
Die automatisierte Flip-the-Tip-Methode in<br />
Verbindung mit den speziell entwickelten<br />
ChipTip-Pipetten überwindet die Einschränkungen<br />
planarer Patch Clamp-Systeme. Es<br />
können stabile Gigaseals und Whole-Cell-<br />
Konfigurationen erhalten werden. Unspezifische<br />
Bindung von Substanzen an verwendete<br />
Materialien wird durch den Einsatz von Glas<br />
vermieden. Für eine einzelne Messung sind<br />
weniger als 50 Zellen notwendig, und die Zugabe<br />
von Wirkstofflösungen kann auf Volumina<br />
von wenigen µl beschränkt werden. Die<br />
systembedingten Vorteile des experimentellen<br />
Aufbaus sind extrem schnelle Lösungswechsel<br />
(< 50 ms), die selbst die Analyse<br />
schnell desensitisierender ligandengesteuerter<br />
Kanäle erlauben, und die Möglichkeit,<br />
die Geometrie und Spitzendurchmesser der<br />
Pipetten für spezielle Experimente maßzuschneidern,<br />
um beispielsweise Ionenkanäle<br />
subzellulärer Organellen und Bakterien zu<br />
untersuchen.<br />
Abb. 6: Ca V 2.3: Optimales Signal-Rausch-<br />
Verhältnis<br />
Abb. 7: nAch-Rezeptor : Überlagerung von<br />
vier wiederholten Einspülungen einer<br />
10 µM Acetylcholin-Lösung auf<br />
eine TE671-Zelle, jeweils gefolgt von<br />
anschließender Auswaschung. Der<br />
schwarze Balken repräsentiert das<br />
Zeitfenster der Einspülung.<br />
Literatur<br />
[1] Sanguinetti, M.C.; Jiang, C.; Curran, M.E.; Keating, M.T. 1995,<br />
Cell 81, 299.<br />
[2] Moss, A.J.; Zareba, W.; Kaufman, E.S.; Gartman, E.; Peterson,<br />
D.R.; Benhorin, J.; Towbin, J.A.; Keating, M.T.; Priori, S.G.;<br />
Schwartz, P.J.; Vincent, G.M.; Robinson, J.L.; Andrews, M.L.;<br />
Feng, C.; Hall, W.J.; Medina, A.; Zhang, L.; Wang, Z. 2002,<br />
Circulation, 105, 794.<br />
[3] Cox, J.J.; Reimann, F.; Nicholas, A.K.; Thornton, G.; Roberts, E.;<br />
Springell, K.; Karbani, G.; Jafri, H.; Mannan, J.; Raashid, Y.; Al-<br />
Gazali, L.; Hamamy, H.; Valente, E.M.; Gorman, S.; Williams,<br />
R.; McHale, D.P.; Wood, J.N.; Gribble, F.M.; Woods, C.G. 2006,<br />
Nature, 444, 894.<br />
[4] Nilius, B.; Owsianik, G.; Voets, T.; Peters, J.A. 2007, Physiol.<br />
Rev. 87, 165.<br />
[5] Lepple-Wienhues, A.; Ferlinz, K.; Seeger, A.; Schäfer, A. 2003,<br />
Receptors and Channels, 9, 13<br />
[6] Yatani, A; Brown, A.M. J. Biol. Chem. 1991, 266, 22872.<br />
Korrespondenzadresse<br />
Dr. Dirck Lassen<br />
Flyion GmbH<br />
Waldhäuser Str. 64, 72076 Tübingen<br />
Tel./Fax: +49-(0)7071-6888-30/-68883099<br />
info@flyion.com, www.flyion.com<br />
42 | 9. Jahrgang | Nr. 3/2008 LABORWElT
Marktübersicht Cell-based Assays<br />
Marktübersicht: Zell-basierte Assays<br />
Einen solchen Ansturm auf eine Marktübersicht haben wir seit langem nicht erlebt – die Zell-basierte Untersuchung aller erdenklichen biologischen<br />
Vorgänge ist mit den zahlreichen optisch auslesbaren Assays, den immer besseren Messverfahren und GFP-Reporterproteinen schier<br />
unüberschaubar geworden. Wegen der zahlreichen qualifizierten Rückmeldungen seitens der Anbieter – insgesamt 33 Firmen gaben einen<br />
Einblick in Ihr derzeitiges Angebot – haben wir kurzentschlossen den Umfang dieser LABORWELT-Ausgabe um 16 Seiten erweitert, um die<br />
Information an Sie, liebe Leser, weiterzugeben. Doch selbst das reichte nicht, um alle Informationen unterzubringen. Das Ergebnis sollte aber<br />
eine gute Orientierung bieten, was derzeit am Markt angeboten wird. Auch wenn wir zum Teil etwas straffen und abkürzen mussten, hoffen<br />
wir eine interessante Informationssammlung zusammengestellt zu haben, die Ihnen einen ersten Hinweis auf interessante Produkte und für<br />
Kaufentscheidungen bietet. Bitte nutzen Sie auch die Gelegenheit, an der Optimierung der Marktübersichten mitarbeiten zu können und<br />
nehmen Sie an unserer Leserbefragung teil – der Fragebogen liegt dieser Ausgabe bei.<br />
AMS Biotechnology<br />
(Europe) Ltd.<br />
63B Milton Park<br />
Abingdon, OX14 4RX, UK<br />
William Booth<br />
Tel.: +49-(0)69-779099<br />
Fax: +49-(0)69-13376880<br />
info@amsbio.com<br />
www.amsbio.com<br />
AMS Biotechnology<br />
(Europe) Ltd.<br />
63B Milton Park<br />
Abingdon, OX14 4RX, UK<br />
William Booth<br />
Tel.: +49-(0)69-779099<br />
Fax: +49-(0)69-13376880<br />
info@amsbio.com<br />
www.amsbio.com<br />
Advanced CellSystems GmbH<br />
Mülheimerstr. 26<br />
53840 Troisdorf<br />
Horst Fuchs<br />
Tel.: +49-(0)2241-1651634<br />
info@advancedcellsystems.com<br />
www.advancedcellsystems.com<br />
Firma/Kontakt Advanced CellSystems GmbH<br />
Mülheimerstr. 26<br />
53840 Troisdorf<br />
Horst Fuchs<br />
Tel.: +49-(0)2241-1651634<br />
info@advancedcellsystems.com<br />
www.advancedcellsystems.com<br />
Universal PARP Assay Kit<br />
Produktname AST-2000 3D Vollhautmodell EST-1000 Epidermal Skin Test High Throughput PARP<br />
Apoptosis Assay Kit<br />
Inhibitor Screening using purified PARP<br />
(for R&D, Pharma, Basic Research)<br />
PARP activity before and during<br />
apoptosis (for R&D, Pharma, Basic<br />
Research)<br />
Substanztestung auf Hautkorrosion nach<br />
OECD-Guideline TG 431; Haut irritation;<br />
Genotoxizität (REACH)<br />
Einsatzgebiete Substanztestung auf Sensibilisierung<br />
und Wundheilung<br />
PAR detection via Biotin-NAD (Enzymatic<br />
Activity); 5 hour protocol (preparation<br />
plus assay)<br />
HT PAR ELISA (our most sensitive<br />
ELISA)<br />
EST-1000 ist ein humanes epidermales<br />
in vitro-Hautmodell. Die Testsubstanzen<br />
werden entsprechend der OECD-Guideline<br />
appliziert und ausgewertet.<br />
AST-2000 ist ein humanes Vollhautmodell.<br />
Die Testsubstanzen werden<br />
topikal appliziert und via Vitalitätstest<br />
ausgewertet.<br />
Beschreibung des<br />
Assays<br />
Colorimetric or chemiluminscent Colorimetric or chemiluminscent<br />
Vitalitätstest (MTT), Bestimmung der<br />
IL-1 alpha-Konzentration<br />
Messprinzip/Endpunkt Vitalitätstest (MTT), Bestimmung<br />
der diverser Zytokinkonzentrationen,<br />
Histologie<br />
LABORWElT 9. Jahrgang | Nr. 3/2008 | 43<br />
96-well strip well format suitable for<br />
automation<br />
96 well plate format suitable for<br />
automation<br />
Das Testsystem kann vollständig automatisiert<br />
werden.<br />
Automationsgrad Das Testsystem kann vollständig automatisiert<br />
werden<br />
Sensitivity: 0.01 U PARP/well = 50,000<br />
cells/well<br />
Sensitivity: 0.1 mU PARP or 500<br />
cells/well<br />
Das Testsystem zeichnet sich durch eine<br />
sehr hohe Spezifität und Sensitivität aus.<br />
Das Testsystem zeichnet sich durch<br />
eine sehr hohe Spezifität und Sensitivität<br />
aus.<br />
Dynamischer Bereich/<br />
Sensitivität<br />
Recombinant Human PARP Enzyme<br />
(HSA); 4-amino-1;8-naphthalimide; anti<br />
PAR rabbit polyclonal antibody<br />
Besonderheiten/Extras Auftragstestung in house Auftragstestung in house Recombinant Human PARP<br />
Enzyme (HSA); 4-amino-1;8naphthalimide;<br />
anti PAR rabbit<br />
polyclonal antibody<br />
525 Euro (excluding carriage) 525 Euro (excluding carriage)<br />
Preis Preis auf Anfrage, Staffelung nach Anzahl<br />
der Hautmodelle
Marktübersicht Cell-based Assays<br />
BioCat GmbH<br />
Im Neuenheimer Feld 581, 69120 Heidelberg<br />
Dr. Elke Gamer<br />
Tel.: +49-(0)6221-7141516<br />
Fax: +49-(0)6221-7141529<br />
info@biocat.de, gamer@biocat.de<br />
www.biocat.de<br />
BD Biosciences, Discovery Labware<br />
Tullastr. 8-12<br />
69126 Heidelberg<br />
Dr. Peter Weiser<br />
peter_weiser@europe.bd.com<br />
Axiogenesis AG<br />
Nattermannallee 1<br />
50829 Köln<br />
Dr. Ralf Kettenhofen<br />
Tel. +49-(0)221-998818-0<br />
info@axiogenesis.com<br />
www.axiogenesis.com<br />
Firma/Kontakt Axiogenesis AG<br />
Nattermannallee 1<br />
50829 Köln<br />
Dr. Ralf Kettenhofen<br />
Tel. +49-(0)221-998818-0<br />
info@axiogenesis.com<br />
www.axiogenesis.com<br />
Multiple Substrate Array MSA<br />
Produktname Cor.At ® ready-to-use Cardiomyocytes µEST BD BioCaotTM Tumor Invasion Systems | BD Bio-<br />
CoatTM Angiogenesis Systems<br />
Microarray-basierte Zelladhäsionsanalyse, Vergleich<br />
behandelte-unbehandelte Zellen, Analyse von Zellphänotpyen<br />
(z.B. Morphologie) oder Zelldifferenzierung,<br />
die durch Proteine der extrazellulären Matrix<br />
(ECM) induziert werden, Untersuchung der Spezifität<br />
von Zell-Matrix-Interaktionen<br />
Tumor-Invasion Assays, Screening von anti-metastatischen<br />
Verbindungen | Angiogenese-Assays<br />
(Migration, Invasion, Tube-Formation, Screening v.<br />
anti-angiogenetischen Verbindungen)<br />
in vitro-Test zum Nachweis von Embryotoxizität<br />
und Teratogenität<br />
Einsatzgebiete Arzneimittelentwicklung , Sicherheitspharmakologie,<br />
Elektrophysiologie, Allo- und Xenotransplantation,<br />
RNA-Präparation<br />
Multiple Substrate Array MSA ist ein Glasträger<br />
(slide) mit 10 Substrat-Microarrays (14 verschiedene<br />
ECM-Proteine pro Microarray, je vierfach gespottet)<br />
und 2 Kontroll-Microarrays (nur Poly-L-Lysin). Durch<br />
Aufklipsen des ProPlate Multiarray Slide-Moduls auf<br />
d. Glasträger entsteht pro Microarray eine Inkubationskammer<br />
für d. Zellen. Diese werden in d. Inkubationskammern<br />
ausgesät, inkubiert, nicht-adhärierte<br />
Zellen weggewaschen und die verbleibenden Zellen<br />
analysiert. Die Zellen können entweder lebend oder<br />
nach Fixierung und Anfärben mit Farbstoffen oder<br />
über indirekte Immunmarkierung analysiert werden.<br />
Zellkultur-Inserts (klare oder BD FluoroBlokTM- Membranen) f. Migrations u. Invasions-Studien, z.T.<br />
Beschichtung m. extrazellul. Matrix (BD MatrigelTM :<br />
Invasion; Tube Formation; Fibronectin: Migration)-<br />
Zellkultur-Inserts (klare oder BD FluoroBlokTM- Membranen) f. Migrations- und Invasions-Studien<br />
– automatisiertes <strong>Imaging</strong> (Endothelial Tube<br />
Formation)<br />
Differenzierung von embryonalen Stammzellen<br />
zu Herzgewebe als Marker für normale Embryonalentwicklung<br />
| Quantifizierung der Menge an<br />
Herzzellen über Expression eines Reportergens<br />
unter Kontrolle eines herzspezifischen Promotors<br />
| Vergleich der Differenzierung von behandelten<br />
Zellen zu unbehandelten Kontrollen | Abweichung<br />
der Differenzierung bei den behandelten Zellen ist<br />
der Marker für gestörte Embryonalentwicklung<br />
| Vergleich der spezifischen embryotoxischen<br />
Wirkung mit möglicher zytotoxischer Wirkung der<br />
Substanz, Aussage über Prädiktionsmodell<br />
Cor.At ® Cells: Untersuchungen an Kardiomyocyten,<br />
bei denen Zellen hochrein und in reproduzierbarer<br />
Form benötigt werden. Zellen sind gebrauchsfertig,<br />
nur Auftauen erforderlich. | Cor.At ® Tox: Testkit<br />
zum Nachweis d. Kardiotoxizität (96-Well-Format)<br />
mit allen notwendigen Kontrollen | Cor.At ® Mod:<br />
Modell z. induzierbaren Hypertrophie | Cor.At®<br />
Patch: Gebrauchsfertige Zellen auf Coverslips für<br />
die Elektrophysiologie<br />
Beschreibung des<br />
Assays<br />
Live cell tracking mit Fluoreszenzfarbstoffen;<br />
Kinetik und Endpunkt möglich, je nach verwendetem<br />
Farbstoff (für Kinetik: DiI; Calcein AM nur<br />
Endpunkt)<br />
Quantifizierung der Menge an Herzzellen über<br />
fluorometrischen Nachweis des Reportergenproduktes<br />
Messprinzip/Endpunkt Alle gängigen Messmethoden (wie z. B. Elektrophysiologie,<br />
PatchClamp) und Nachweissysteme<br />
(wie z. B. NRU, MTT, Troponin-ELISA, RT-PCR)<br />
anwendbar.<br />
44 | 9. Jahrgang | Nr. 3/2008 LABORWElT<br />
Analyse per Mikroskop<br />
Mikrotiterplatten-Maßstab<br />
96-well-Platten verfügbar (Tumor Invasion, Endothelial<br />
Cell Migration, Endothelial Cell Tube Formation),<br />
daneben 24-well-Systeme (Endothelial Cell Invasion,<br />
Tumor Invasion, Endothelial Cell Migration).<br />
Das Testsystem kann vollständig automatisiert<br />
werden.<br />
Automationsgrad Zellen für automatisierte Verfahren (z. B. HTS)<br />
bestens geeignet<br />
nur 5.000 bis 20.000 Zellen benötigt<br />
abhängig v. diversen Faktoren: Zelltyp, Fluoreszenzfarbstoff,<br />
screening compounds, Instrument<br />
Das Testsystem zeichnet sich durch eine sehr<br />
hohe Spezifität (87%) und Sensitivität (88%)<br />
aus.<br />
Der Phänotyp der Cor.At ® -Zellen ist sehr gut charakterisiert.<br />
Hohe Aussagekraft und Spezifizät der<br />
Ergebnisse (z. B. im Nachweis von kardiotoxischen<br />
Substanzen)<br />
Dynamischer Bereich/<br />
Sensitivität<br />
| Microarray-Format für die Analyse von Zelladhäsion<br />
| Es wird nur eine geringe Anzahl Zellen benötigt<br />
(5.000 bis 20.000 Zellen pro Microarray) | die Adhäsion<br />
von 10 verschiedenen Zelltypen an jeweils<br />
14 verschiedene ECM-Proteine kann gleichzeitig<br />
analysiert werden<br />
Individual Inserts verfügbar, klare und BD Fluoro-<br />
BlokTM-Membranen, 3.0 µm (Tumor Invasion) und<br />
8.0 µm (Endothelial Cell Migration & Invasion)<br />
Porengröße; HUVEC-Zellen f. Angiogenese-Assays,<br />
Calcein AM- und DiI-Farbstoffe f. Fluoreszenzbasierte<br />
Assays. BD FluoroBlokTM-Membran ermögl.<br />
Ausl. d. Platten o.zusätzl. Waschschritte<br />
µEST eignet sich besonders für den Einsatz<br />
in frühen Phasen der Substanzentwicklung<br />
und zur Priorisierung von Substanzen, da nur<br />
geringe Substanzmengen benötigt werden (5<br />
mg) und der Test schnelle Ergebnisse liefert. Der<br />
Test ist in der Lage, die teratogene Wirkung von<br />
Thalidomid spezifisch nachzuweisen.<br />
Besonderheiten/Extras Von embryonalen Stammzellen abgeleitete Kardiomyocyten<br />
mit hoher physiologischer Relevanz<br />
in Bezug auf adulte humane Kardiomyocyten | Die<br />
Qualität des Produkts wird durch den Hersteller<br />
zertifiziert | Hohe Reinheit der Zellen (99,9 %) |<br />
Keine arbeitsintensive Zellpräparation notwendig |<br />
Zellen transfizierbar<br />
560 Euro für 2 slides mit je 10 Substrat-Microarrays<br />
Preis auf Anfrage, Staffelung nach Anzahl der zu<br />
testenden Substanzen.<br />
Preis Preisliste ist über CellSystems (Tel.<br />
+49-(0)26449512-0) erhältlich.
Biomol GmbH<br />
22769 Hamburg<br />
Waidmannstr. 35<br />
Dr. Edgar Lipsius<br />
Tel.: +49-(0)40-853260-37<br />
www.biomol.de<br />
Firma/Kontakt BioCat GmbH<br />
Im Neuenheimer Feld 581,<br />
69120 Heidelberg<br />
Dr. Elke Gamer<br />
Tel.: +49-(0)6221-7141516<br />
Fax: +49-(0)6221-7141529<br />
info@biocat.de, gamer@biocat.de<br />
www.biocat.de<br />
Produktname CytoSelect 24-well Cell Migration Assay (8 um), Colorimetric Cignal TM Reporter Assay Kits<br />
quantitative Erfassung der Aktivierung oder Hemmung von Signalwegen durch Proteine, niedermolekulare<br />
oder organische Substanzen, siRNA, shRNA, miRNA; im 96-Well-Format<br />
Einsatzgebiete Der CytoSelect 24-well Cell Migration Assay (8 um), Colorimetric wird für die Analyse des Migrationsverhaltens<br />
von Zellen eingesetzt.<br />
über spezifische Transkriptionsfaktoren induzierbare Reporter-Konstrukte werden in gewünschte Zell-<br />
Linien transfiziert | durch Co-Expression oder Stimulierung der Zellen verursachte Effekte werden mittels<br />
des dualen Luciferase-Reporter-Systems gemessen | für 18 Signalwege/Transkriptionsfaktoren<br />
Der Assay enthält Polycarbonatmembraneinsätze (Porengröße 8 µm) in einer 24-well-Platte, die die Vertiefungen<br />
der 24-well-Platte in eine obere und eine untere Kammer teilen und als Barriere zur Separation/<br />
Unterscheidung migratorischer u. nicht-migratorischer Zellen dienen. Migratorische Zellen bilden Fortsätze<br />
in Richtung des chemischen Lockstoffes in d. unteren Kammer und wandern durch d. Poren der Membran<br />
dorthin. Nicht-migratorische Zellen verbleiben auf der Membran i. d. oberen Kammer. Migratorische Zellen<br />
werden angefärbt und quantifiziert.<br />
Beschreibung des<br />
Assays<br />
Messprinzip/Endpunkt Colorimetrische Messung im ELISA-Reader (beim fluorometrischen Format im Fluorometer) Bestimmung der Luciferase-Aktivität im Luminometer (Endpunktmessung)<br />
Automationsgrad Mikrotiterplatten-Maßstab in hohem Maße automatisierbar<br />
Colorimetrischer Assay: 5.000 Zellen (Fluorometrischer Assay: 2.000 Zellen) exzellentes Signal-to-Noise-Verhältnis: bis zu 250:1 | destabilisierte Luciferase und optimierte Promotoren<br />
bewirken maximale Reduktion des Hintergrunds | breiter dynamischer Bereich<br />
Dynamischer Bereich/<br />
Sensitivität<br />
jeder Kit enthält fertig einsetzbare Mischungen aus induzierbaren und konstitutiv exprimierenden Reporter-Plasmiden<br />
sowie Neagtiv-und Positiv-Kontrollen | auch als 10 Pathway Suites für cross-talk-Studien<br />
erhältlich | auch als GFP-Reporter-Assays für Zeitkurven und Einzelzell-Messungen erhältlich (Detektion<br />
per Fluoreszenz-Mikroskop oder Durchflusszytometer)<br />
Besonderheiten/Extras Assay-Dauer < 6h | Quantitativer Assay o. manuelles Auszählen | Verschiedene Formate: für colorimetrische<br />
oder fluorometrische Auswertung | 24-well oder 96-well-Format | als Haptotaxis-Assays beschichtet<br />
mit Collagen, Fibronectin | spezieller Leukocyte Migration-Assay<br />
LABORWElT 9. Jahrgang | Nr. 3/2008 | 45<br />
Preis 325 Euro CignalTM Reporter Assay Kit: 440 Euro<br />
CignalTM Reporter Assay 10-Pathway Suite: 1159 Euro
Marktübersicht Cell-based Assays<br />
CCS Cell Culture Service GmbH<br />
Falkenried 88<br />
20251 Hamburg<br />
Susan Friedemann<br />
Tel.: +49-(0)40-47196575<br />
Friedemann@cellcultureservice.com<br />
CCS Cell Culture Service GmbH<br />
Falkenried 88<br />
20251 Hamburg<br />
Susan Friedemann<br />
Tel.: +49-(0)40-47196575<br />
Friedemann@cellcultureservice.com<br />
Biopharm GmbH<br />
Czernyring 22<br />
69115 Heidelberg<br />
Renate Kron<br />
Tel.: +49-(0)6221-5383-0<br />
services@biopharm.de<br />
www.biopharm.de<br />
Firma/Kontakt Bionas GmbH<br />
Friedrich-Barnewitz-Str. 3<br />
18119 Rostock<br />
Dr.Michael Schulze<br />
Tel.: +49-(0)381-5196-241<br />
Fax: +49-(0)381-5196-246<br />
bionas@bionas.de<br />
www.bionas.de<br />
HEK-AP1-SEAP recombinant Reporter Cell<br />
Line<br />
A549-NFB-SEAP, Recombinant NFB Reporter<br />
Cell Line<br />
Methodenentwicklung und -validierung nach GMP/GLP<br />
zum Nachweis der Wirksamkeit<br />
Produktname Bionas ® 2500 analyzing system Familie,<br />
Bionas ® metabolic chip SC100<br />
GLP konforme und Non-GLP CRO-Services im firmeneigenen<br />
Labor<br />
Screening for AP1-activation<br />
Screening of inflammatory and anti-inflammatory<br />
drugs<br />
Stabilitätsstudien, Freigabeuntersuchung, Qualitätskontrolle<br />
Einsatzgebiete Drug Discovery | Drug Development, Toxikologie,<br />
Krebsforschung/ Therapieoptimierung, Zelltherapien,<br />
Bioproduktion, Zellprodukte, Zellkulturmedien, Nahrungsmittelherstellung,<br />
Umweltmonitoring<br />
Assay for screening of substances for their<br />
activation of AP1 by quantifying SEAP expression<br />
in 96-well formate ready to use<br />
Assay for screening of substances for their<br />
activation of NFB by expression of SEAP<br />
(Secreted Embryonic Alkaline Phosphatase)<br />
reporter gene.<br />
Proliferationsassays, Cytoxitätsassays, Angiogenese-<br />
Assay, Differenzierungsassays u.v.m.<br />
Online-Monitoring lebender Zellen. Das Bionas ® analyzing<br />
system analysiert Effekte d. Zellstoffwechsels<br />
und d. Signaltransduktion auf das Zellkulturmedium.<br />
nicht-invasive, labelfreie, kontinuierliche Beobachtung<br />
d. Zellstoffwechsels in Abhängigkeit v. Testsubstanzen<br />
u. Kulturbedingungen.<br />
Beschreibung des<br />
Assays<br />
measuring of SEAP expression by CSPD<br />
chemo-luminescent substrate<br />
Absorption, Floureszenz, Lumineszenz, Zellzählung measuring of SEAP concentration by chemoluminescent<br />
(CSPD) or fluorescent (MUP)<br />
substrate without the need of lyse the cells.<br />
Messprinzip/Endpunkt Real-time-Zellanalyse metabolisch relevanter Parameter<br />
wie O -Verbrauch, Ansäuerungsrate, Zelladhäsion auf<br />
2<br />
Basis v. Zell-Silikon-Hybriden. Drei Sensortypen pro<br />
Chip. Messung parallel u. kontinuierlich (Stunden -<br />
Tage) u. label-frei. Dadurch keine Beeinflussung d.<br />
Zellphysiologie.<br />
Automationsgrad Vollautomat (6 unabhängige Kanäle) mit langer walkaway-Zeit,<br />
Semi-Automat (2 bzw. 1 Kanal), Sicherheitsfeatures<br />
ermöglichen unbeaufsichtigtes Arbeiten über<br />
Nacht/am Wochenende<br />
46 | 9. Jahrgang | Nr. 3/2008 LABORWElT<br />
Cedex Cell Counter Yes Yes<br />
Sensitivität: 0,5 ng/ml, Signal : Background<br />
=14.000<br />
Hohe Sensitivität über weiten Messbereich Sensitivität: 0,2 ng/ml, Signal : Background<br />
=7500<br />
abhängig v. Aktivität d. untersuchten Zellen. Messprinzip<br />
beinhaltet Relativ-Messung v. Änderungen d.<br />
Zellstoffwechsels nach Wirkstoffzugabe. Startpunkt wird<br />
auf 100% normiert. Beispielhaft können minimal 0,05<br />
pH-Einheiten unterschieden werden. Typ. Standardabweichung<br />
< 15%.<br />
Dynamischer Bereich/<br />
Sensitivität<br />
Flexibilität, Termintreue<br />
Besonderheiten/Extras Patentiertes Fluidiksystem versorgt Zellen/Gewebe<br />
kontinuierlich m frischem Medium dadurch Langzeitmessungen<br />
adhärenter, suspendierter Zellen u. von<br />
Geweben mgl.<br />
18.000 Euro 18.000 Euro<br />
Preis Bionas ® analyzing systeme: 19.900 Euro-92.000 Euro.<br />
GLP- und Non-GLP-Dienstleistungen nach Aufwand und<br />
Fragestellung
Cytocentrics AG<br />
Joachim-Jungius-Str. 9<br />
18059 Rostock<br />
Tel.: +49-(0)381-4059640<br />
info@cytocentrics.com<br />
www.cytocentrics.com<br />
Cytocentrics AG<br />
Joachim-Jungius-Str. 9<br />
18059 Rostock<br />
Tel.: +49-(0)381-4059640<br />
info@cytocentrics.com<br />
www.cytocentrics.com<br />
CellSystems Biotechnologie Vertrieb GmbH<br />
Hummelsbergerstr. 11<br />
53562 St. Katharinen<br />
Dr. Peter Frost<br />
Tel.: +49-(0)2644-9512 -0<br />
info@cellsystems.biz<br />
www.cellsystems.biz<br />
Firma/Kontakt CellSystems Biotechnologie Vertrieb GmbH<br />
Hummelsbergerstr. 11<br />
53562 St. Katharinen<br />
Dr. Peter Frost<br />
Tel.: +49-(0)2644-9512 -0<br />
info@cellsystems.biz<br />
www.cellsystems.biz<br />
Produktname MucilAir AngioKit CytoPAQ hERG-HEK 293 Instant Cells Kit hERG Screening Service (unter GLP und non-<br />
GLP)<br />
Sicherheitspharmakologie, Early Profiling<br />
Manuelle und automatisierte Patch Clamp-Technik,<br />
Sicherheitspharmakologie, hERG-Screening,<br />
fluoreszenzbasierte Assays.<br />
in vitro-Cokultursystem für Substanztestung auf<br />
stimulierende und inhibitorische Effekte im Bereich<br />
Angiogenese<br />
Einsatzgebiete Substanztestung auf toxikologische Effekte im Respirationstrakt;<br />
NanoTox<br />
Die Wirkung von Prüfsubstanzen auf eine<br />
mögliche Inhibierung des hERG (human Ether<br />
a-go-go-Related Gene)-Ionenkanals wird mit<br />
der Patch Clamp-Technik untersucht (manuell<br />
oder automatisiert). Hintergrund: Eine Hemmung<br />
des hERG-Ionenkanals in der Herzzelle durch<br />
Gabe bestimmter Arzneimittel kann zu lebensbedrohenden<br />
Rhythmusstörungen (Torsades de<br />
Pointes) führen. Durch Patch Clamp-Messungen<br />
in der präklinischen Phase der Medikamententestung<br />
werden Hinweise auf derartige Nebenwirkungen<br />
erbracht.<br />
HEK 293-Zellen, die den hERG (human Ether<br />
a-go-go-Related Gene)-Ionenkanal exprimieren,<br />
können 15 min nach Auftauen im Assay<br />
verwendet werden. Zellkulturarbeiten entfallen.<br />
Kit besteht aus qualitätsgesicherten, elektrophysiologisch<br />
validierten hERG- HEK293 Zellen,<br />
intra- und extrazellulärer Lösung und dem Cell<br />
Reservoir.<br />
Cokultursystem aus Endothelzellen und anderen<br />
Primärzellen<br />
Applikation der Testsubstanzen akut und chronisch<br />
(bis zu 12 Monaten)<br />
Beschreibung des<br />
Assays<br />
Durchführung des hERG-Screenings am manuellen<br />
Patch Clamp-Setup und am CytoPatch-<br />
Automaten unter GLP- oder non-GLP-Bedingungen.<br />
Qualitätssicherung jeder Charge durch manuelle<br />
Patch Clamp-Technik. Qualitäts-Zertifikat<br />
standardmäßig mit elektrophysiologischen<br />
Parametern (Sealrate, Stromamplitude, Rundown<br />
and Rate der erfolgreich beendeten Ganzzellableitungen)<br />
Messprinzip/Endpunkt Vitalitätstest (MTT), TEER Quantifizierung der Menge an Gefäßen, Verzweigungspunkten<br />
und Länge der Gefäße via Oberflächenmarker<br />
oder ELISA<br />
Je nach Kundenwunsch wird das Screening am<br />
manuellen Patch Clamp-Setup oder am eigens<br />
von Cytocentrics entwickelten Patch Clamp-<br />
Automaten CytoPatch durchgeführt.<br />
Marktübersicht Cell-based Assays<br />
LABORWElT 9. Jahrgang | Nr. 3/2008 | 47<br />
Zellen können 15 Minuten nach dem Auftauen<br />
für Assays verwendet werden<br />
Das Testsystem kann vollständig automatisiert<br />
werden.<br />
Automationsgrad Das Testsystem kann vollständig automatisiert<br />
werden.<br />
Vitalität der Zellen: >90% | Sealrate: >90% |<br />
Whole Cell Rate: >90% | Stabilität über 25 min:<br />
>60% der Zellen | Stromamplitude: 1000 pA<br />
Das Testsystem zeichnet sich durch eine sehr hohe<br />
Spezifität und Sensitivität aus.<br />
Das Testsystem zeichnet sich durch eine sehr hohe<br />
Spezifität und Sensitivität aus.<br />
Dynamischer Bereich/<br />
Sensitivität<br />
Cytocentrics bietet Assay-Entwicklung und<br />
-Screening mit Kundenzelllinien an<br />
Besonderheiten/Extras Auftragstestung bei Kooperationspartnern möglich Auftragstestung in house möglich Die Zusammensetzung der Kits erfolgt in Absprache<br />
mit dem Kunden und wird an dessen<br />
Bedürfnisse angepasst. Cytocentrics adaptiert<br />
weiterhin Zelllinien von Kunden an das CytoPAQ<br />
Instant Cells System.<br />
Preis auf Anfrage, Staffelung nach Format<br />
(24well/96well), Staffelung nach Anzahl der zu testenden<br />
Substanzen.<br />
Preis Preis auf Anfrage, Staffelung nach Anzahl der Modelle
Marktübersicht Cell-based Assays<br />
Guava Technologies, Inc<br />
Kanalstr. 19/1, 72631 Aichtal<br />
Dr. Michael Liebler<br />
Tel.: +49-(0)7127 953133<br />
Fax: +49-(0)7127 953130<br />
mliebler@guavatechnologies.com<br />
www.guavatechnologies.com<br />
Guava Technologies, Inc<br />
Kanalstr. 19/1, 72631 Aichtal<br />
Dr. Michael Liebler<br />
Tel.: +49-(0)7127 953133<br />
Fax: +49-(0)7127 953130<br />
mliebler@guavatechnologies.com<br />
www.guavatechnologies.com<br />
flyion GmbH<br />
Waldhäuser Str. 64, 72076 Tübingen<br />
Dr. Dirck Lassen<br />
Tel.: +49-(0)7071-6888-30<br />
Fax: +49-(0)7071-6888-399<br />
info@flyion.com, www.flyion.com<br />
Firma/Kontakt Eppendorf Biochip Systems GmbH<br />
Barkhausenweg 1<br />
22339 Hamburg<br />
Dr. Margit Stadler<br />
stadler.m@eppendorf.de<br />
Guava ViaCount Guava Nexin<br />
Produktname TF Chip Stem Cell Kit, TF Chip MAPK Kit | Flyscreen ® 8500 (Patch Clamp-Roboter) | PatchBox<br />
(Patch Clamp-Automat) | Feedback Microforge (vollautomatisches<br />
Mikro-Glasblasegerät )<br />
Apoptose-Messung, in Medienen- und<br />
Zelllinienoptimierung, Target-Validierung,<br />
Wirksamkeitsstudien<br />
Zellzählung und Bestimmung der Viabilität,<br />
in Medienen- und Zelllinienoptimierung,<br />
Target-Validierung, Wirksamkeitsstudien,<br />
allgemeine Zellkultur<br />
Akademische u. pharmazeutische Ionenkanalforschung |<br />
Wirkstofffindungs-Screening | Lead-Optimierung | Sicherheitspharmakologie<br />
| biophysikal. Charakterisierung v.<br />
Ionenkanälen<br />
Einsatzgebiete Nachweis aktivierter Transkriptionsfaktoren in Kernproteinextrakten<br />
aus Zellkulturen<br />
„Mix and Read“, Messung mit Guava-<br />
Durchflusszytometern<br />
„Mix and Read“, volumetrische Messung<br />
mit Guava-Durchflusszytometern<br />
Invertierung d. klassischen Patch Clamp-Experiments (FliptheTip):<br />
Zellen in Suspension werden in mit extrazelluärer<br />
Lösung befüllte Glaspipette gesaugt. Einzelzelle versiegelt<br />
Spitzenöffnung u. bildet elektrisch dichte Verbindung zwischen<br />
Zelle und Glas („Gigaseal“). Zelle wird kontroll. aufgerissen<br />
– dadurch elektr. Zugang zum Zytosol. Verbleibende el. dichte<br />
Membranfläche wird du. Anlegen untersch.Spannungspotentiale<br />
stimuliert. Durch. kontroll. Änderungen d. Membranspannung<br />
werden d. Ionenkanäle in d. Zellmembran aktiviert. Strom<br />
wird über Messverstärker aufgezeichnet.<br />
Doppelstrang-DNA mit Konsensus-Bindungssequenz f. best.<br />
Transkriptionsfaktor wird auf beschichtete Glasoberfläche<br />
gespottet u. mit Kernproteinextrakt inkubiert; d. Nachweis<br />
d. Bindung an Zielsequenz erfolgt via konformationsspezif.<br />
Antikörper. Visualisierung via fluoreszenzmarkierte o. biotinylierte<br />
Sekundärantikörper sowie Anti-Biotin-Nanogold-<br />
Konjugat. Der Goldpartikel katalysiert die Reduktion v.<br />
Silbernitrat zu Silber. Mit TF Chip Kits können bis zu 12 Transkriptionsfaktoren<br />
in einem Assay gemessen werden.<br />
Beschreibung des<br />
Assays<br />
Anfärbung der Zellen mit Annexin V und<br />
7-AAD in einem einzigen Schritt<br />
DNA-Färbung mit Fluoreszenzfarbstoffen<br />
zur Viabilitätsbestimmung und Debris-<br />
Diskriminierung<br />
In intra- o. extrazelluläre Lösung werden Wirkstoffe eingespült<br />
u.ihr Einfluss auf Ionenkanäle bestimmt. Aktivität<br />
wird als Strom/Spannungskurve bzw. Dosis/Wirkungskurve<br />
gemessen. | Messmethoden: Voltage Clamp; Current Clamp<br />
| Voltage clamp-Methoden: „Open Whole Cell“ und „Perforated<br />
Patch“<br />
Messprinzip/Endpunkt Transkriptionsfaktor-Bindungssequenzen als Triplikate<br />
gespottet. Zur Messung unspezif. Bindungen sind mutierte<br />
Sequenzen (keinerlei spezif. Bindung) daneben gespottet.<br />
Weitere Negativ- Kontrollen sind in den Assay integriert.<br />
Quantifizierung d. Silberfärbung mittels def. Konzentrationskurve<br />
mit positiven Detektionskontrollen. Messung d.<br />
fluoreszenzgefärbten Assays via Microarray-Fluoreszenz-<br />
Scanner | Messung d. silbergefärbten Proben via Eppendorf<br />
Silverquant ® -Scanner.<br />
Einzel-Proben und/oder 96-Loch-Platten,<br />
Einbindung in Pipettierstationen verschiedener<br />
Hersteller möglich<br />
Vollautomatisch (Flyscreen) | halbautomatisch (PatchBox) Einzel-Proben und/oder 96-Loch-Platten,<br />
Einbindung in Pipettierstationen verschiedener<br />
Hersteller möglich<br />
48 | 9. Jahrgang | Nr. 3/2008 LABORWElT<br />
Automationsgrad Niedrig; zwei Assays pro Slide, zwei Slides pro Messung im<br />
Silverquant ® -Scanner<br />
Zellkonzentrationen 5x104 bis 2x106 per ml<br />
Zellkonzentrationen 104 bis 2x107 per<br />
ml (und mehr), schon 2.000 Zellen sind<br />
ausreichend<br />
Dynamischer Bereich ≥2 Größenordnungen Durchschnittlicher Seal-Widerstand > 1 GΩ<br />
Durchschnittlicher Whole-Cell-Widerstand > 0,5 GΩ<br />
Pipettenwiderstand 0.8 – 1.3 MΩ<br />
Serienwiderstand < 5 MΩ<br />
Durchschnittliche Whole-Cell-Stabilität ~ 30 min<br />
Erfolgreiche Whole-Cell-Messungen > 70 % (CHO/HEK/LTK)<br />
Dynamischer Bereich/<br />
Sensitivität<br />
Mix and Read ohne Pufferwechsel oder<br />
Waschschritte, besonders zellschonend<br />
| Extrem schneller Lösungsaustausch (~50ms) | Kleinste<br />
Messvolumina (
Invitrogen GmbH<br />
Emmy-Noether-Str. 10<br />
76131 Karslruhe<br />
euroinfo@invitrogen.com<br />
www.Invitrogen.com,<br />
In Vitro Systems & Services GmbH<br />
Rudolf-Wissell-Str. 28<br />
37079 Göttingen<br />
Katrin Esslinger<br />
Tel.: +49-(0)551-50097-329<br />
Fax: +49 (0)551-50097-311<br />
customerservice@ivss.de<br />
www.ivss.de<br />
ibidi GmbH<br />
Am Klopferspitz 19<br />
82152 Martinsried<br />
Dr. Ulf Rädler<br />
Tel.: +49-(0)800-00111128<br />
uraedler@ibidi.de<br />
Firma/Kontakt ibidi GmbH<br />
Am Klopferspitz 19<br />
82152 Martinsried<br />
Dr. Ulf Rädler<br />
Tel.: +49-(0)800-00111128<br />
uraedler@ibidi.de<br />
lumoxTM slide flask SelectScreen Cell-based Kinase/ Nuclear Receptor<br />
Pathway Profiling<br />
Produktname Cell culture Insert Electric Cell-substrate Impedance Sensing<br />
(ECIS)<br />
Zellbasierte Assays, insbesondere Fluoreszenzassays Screening von Substanzen in einer pathwayspezifischen<br />
Zelllinie<br />
Zellwachstum | Zellspreading | Zellmigration<br />
| Zellproliferation und Zytotoxiziät | Funktionelles<br />
Monitoring von Rezeptor-Aktivitäten |<br />
Áutomatisierte Wundheilungsassays<br />
Einsatzgebiete Zellwachstum | Wundheilung | Invasionsassays<br />
Das SelectScreen Cell-based Pathway Profiling<br />
basiert auf Invitrogens Bibliothek von GeneBLAzer ® -<br />
spezif. CellSensor ® -Zelllinien mit GeneBLAzer ® betalactamase<br />
(bla) Reporter-Technologie. Die Reporter-<br />
Assays verwenden ein beta-Laktamase-Gen, kombiniert<br />
mit einem FRET-aktiven Substrat. Bei exprimierter<br />
beta-Laktamase wird d. Substrat umgesetzt – die Zellen<br />
erscheinen blau. Ohne Laktamase: Grünfärbung.<br />
Objektträgerflasche mit gasdurchlässigem Folienboden<br />
mit sehr geringer Autofluoreszenz. Folien-Objektträger<br />
nach der Kultivierung abziehbar und für weitere<br />
Arbeitsschritte einfach zugänglich. Gasdurchlässige<br />
Wachstumsoberfläche in TC-Qualität.<br />
Nicht invasive Echtzeitmessung des zellulären<br />
Zustandes via Impedanz<br />
Nicht invasive Echtzeitmessung der Wundheilung/<br />
Invasion via Mikroskopie | Insert bildet<br />
einen definierten Spalt der Breite 500 µm |<br />
Nach Erreichen der gewünschten Zelldichte<br />
wird der Steg entfernt, und die Echtzeitmessung<br />
beginnt.<br />
Beschreibung des<br />
Assays<br />
Messprinzip/Endpunkt Echtzeitmessung im Mikroskop Echtzeitmessung Zellen werden mit Substrat beladen, das abhängig v.<br />
d. Laktamase-Expression umgesetzt wird. Im Assay<br />
werden Substanzen in einem Panel von den Pathwaysspezifischen<br />
Zelllinien bei d.EC /IC -Bestimmung<br />
50 50<br />
im Aktivierungs- (%) und Inhibierungs- (%) Modus<br />
gescrennt, indem 10 Messpunkte einer Dosis-Responsekurve<br />
bestimmt werden.<br />
Automationsgrad Von Einzelmessungen bis zu 24 well-Platten von 8 well- bis 96 well-Platten in Standard-Plattformen zu automatisieren | Invitrogen<br />
bietet einen Service an. E-Mail an: SelectScreen@<br />
Invitrogen.com<br />
Marktübersicht Cell-based Assays<br />
LABORWElT 9. Jahrgang | Nr. 3/2008 | 49<br />
Flexibler dynamischer Bereich möglich bei hoher Sensitivität<br />
(Fluorezenz-Messung).<br />
Geringe Autofluoreszenz und hohe Transparenz des<br />
lumoxTM-Folienbodens ermöglicht eine verbesserte<br />
Mikroskopierbarkeit und Sensitivität bei fluoreszenzbasierten<br />
Zellassays. Gasdurchlässige Wachstumsfläche für<br />
optimalen O - und CO -Austausch.<br />
2 2<br />
Messungen von Einzellzellen bis zur Konfluenz<br />
des Zellrasens<br />
Messungen von Einzellzellen bis zur Konfluenz<br />
des Zellrasens<br />
Dynamischer Bereich/<br />
Sensitivität<br />
Alle Zelllinien GMP-konform u. auf Mykoplasma<br />
getestet. Response-Elemente werden f. jede Zelllinie<br />
sequenziert. Stimulus/Aktivierung, Small Molecule-<br />
Inhibitoren , RNAi-knockdown, werden – wenn angebracht<br />
– für Assays getestet. Der Assay kann auch im<br />
Flowzytometer durchgeführt werden.<br />
Hervorragende optische Eigenschaften | Hohe Sensitivität<br />
bei Fluoreszenzmessungen | Hintergrundfreie<br />
Bilddarstellung | Homogenes Zellwachstum | Hohe<br />
Oberflächenelastizität<br />
Besonderheiten/Extras Kombinierbar mit Mikroskopie Kombinierbar mit Mikroskopie | Bestimmung<br />
der Membrankapazitäten | Bestimmung der<br />
Barrierefunktion bei konfluenten Zellrasen –<br />
Gut etabliert – mehr als 250 Publikationen<br />
12 Stück 79,- Euro | 96 Stück 600,- Euro<br />
Von ca. 15.000 bis ca. 50.000 Euro (+<br />
MwSt.)<br />
Preis 30 Stück:150 Euro (in Petrischalen ready to<br />
use) | 25 Stück zum Selbsteinsetzen: 80 Euro<br />
(+ MwSt.)
Marktübersicht Cell-based Assays<br />
MDS Analytical Technologies GmbH<br />
Gutenbergstr. 10<br />
85737 Ismaning<br />
Frank.Hafner@moldev.com<br />
MARINPHARM GmbH<br />
Im Biotechnologiepark TGZ 2<br />
14943 Luckenwalde<br />
Prof. Dr. Christian Petzelt<br />
Tel.: +49-(0)3371-681350<br />
Fax: +49-(0)3371-681348<br />
cpetzelt@marinpharm.com<br />
www.marinpharm.com<br />
Lonza Verviers<br />
www.lonza.com<br />
Leon de Bruin<br />
leon.debruin@lonza.com<br />
Firma/Kontakt LI-COR Biosciences GmbH<br />
Siemensstr. 25 A<br />
61352 Bad Homburg<br />
Dr. Sandra Scheuch<br />
Tel.: +49-(0)6172-1717771<br />
Fax: +49-(0)6172-1717799<br />
gmbh@licor.com<br />
www.licor.com<br />
ViaLight ® , ToxiLight ® Fluoreszierende stabil transfizierte Zell-Linien CellKey TM und CellKey TM 384 System<br />
Produktname In-Cell WesternTM Assay | Odyssey Infrared <strong>Imaging</strong><br />
System | Aerius ® Automated Infrared <strong>Imaging</strong> System<br />
funktionaler zellbasierter Assay für endogene<br />
Rezeptoren | Evaluierung von GPCR- und Tyrosin-<br />
Kinase-Rezeptoren und anderen Klassen an<br />
Rezeptoren bzw. Targets | Identifizierung und<br />
Charakterisierung des Signaltransduktionsweges<br />
| Hit-Identifizierung und pharmakologisches<br />
Profiling | Target-Identifizierung und Validierung |<br />
RNAi-Studien zur funktionellen Inhibitierung von<br />
Rezeptoren | zelluläres Primärscreening<br />
Cell culture applications, Drug discovery screening Echtzeitdarstellung von lebenden Zellen<br />
oder Zellorganellen oder Einzelproteinen in<br />
lebenden Zellen, in-vivo-<strong>Imaging</strong> von Tumoren<br />
in Tieren<br />
Einsatzgebiete Direkte Proteindetektion (2 Targets simultan) u. Quantifizierung<br />
in Zellen | Quantitative Analyse zellulärer<br />
Signaltransduktionswege | High Throughput Screening<br />
| Zellbasierte IC -Bestimmung | Inhibitor/-Effektor-<br />
50<br />
Screening<br />
markierungsfreier, biorelevanter zellbasierter<br />
Assay auf 96- und 384-Mikrotiterplatten-Format<br />
Direktes „Sehen“, vom Gewebe im Tier bis<br />
zum einzelnen Protein in der lebenden Zelle<br />
ViaLight ® : quick and accurate analysis of cell proliferation<br />
and cytotoxicity in both adherent and nonadherent<br />
cell types. | ToxiLight ® : measuring the toxicity<br />
of drugs by looking at the release of adenylate<br />
kinase (AK) from damaged eukaryotic cells.<br />
Kultivierung adhärenter Zellen o. Suspensionszellen in<br />
Mikrotiterplatten | Zell-Behandlung (Inhibitor-Inkubation,<br />
Stimulierung, etc.) | Fixierung und Permeabilisierung der<br />
Zellen | Primärantikörper-Inkubation | Inkubation mit 2<br />
Sekundärantikörpern oder einem Sekundärantikörper +<br />
DNA-Färbung | <strong>Imaging</strong> und Quantifizierung<br />
Beschreibung des<br />
Assays<br />
Fluoreszenzmikroskopische Auswertung nicht invasive, Impedanz-basierte, markierungsfreie,<br />
kinetische Messung an lebenden Zellen.<br />
ViaLight ® : bioluminescent detection of ATP, using the<br />
bioluminescent firefly luciferase reaction | ToxiLight ® :<br />
AK actively phosphorylates ADP to form ATP, which is<br />
then measured on a luminometer using the bioluminescent<br />
firefly luciferase reaction<br />
Messprinzip/Endpunkt Zellbasierter immuncytochemischer Assay, Zielprotein<br />
wird in Zellen nachgewiesen, Infrarotfarbstoff-markierter<br />
Sekundärantikörper, direkte Detektion des Fluoreszenzsignals<br />
u. Normalisierung gegen ein zweites<br />
Target ermöglichen präzise quantitative Daten<br />
Online Pipettor zum automatischen Pipettieren<br />
während des Assays. | CellKeyTM ist voll roboterintegrierbar<br />
Automationsgrad Aerius ® Automated Infrared <strong>Imaging</strong> System: Integrierter<br />
Barcode-Scanner. Scannt bis zu 30 Platten pro Lauf<br />
mit optionalem Stacker | Integration in vollautomatische<br />
Arbeitsabläufe möglich – Automatische Analyse<br />
für In-Cell Western- Assays<br />
50 | 9. Jahrgang | Nr. 3/2008 LABORWElT<br />
High-Throughput-Screening je nach Zell-Linie<br />
möglich<br />
Both systems are highly automatable, can be upscaled<br />
to 96 or 384 wells<br />
markierungsfreies Messprinzip ergibt Sensitivität<br />
um einen großen Bereich an endogenen Rezeptoren<br />
messen zu können<br />
breiter Bereich zwischen einem vielzelligen<br />
Tumor im Tier bis hin zu einzelnen Proteinen<br />
in der Zelle, direkt sichtbar<br />
ViaLight ® : Dynamic range of five decades, detects<br />
as few as 10 eukaryotic cells | ToxiLight ® : Dynamic<br />
range of four decades, detects as few as 10 dead<br />
eukaryotic cells per well<br />
Dynamischer Bereich der Odyssey und Aerius ® <strong>Imaging</strong>-Systeme:<br />
5 log-Stufen | hochsensitiv, Nah infrarot-<br />
Technologie | maximale Auflösung: 21 µm<br />
Dynamischer Bereich/<br />
Sensitivität<br />
online Pipettor<br />
Fast, convenient and direct measurements Auf Wunsch Generierung Klienten-spezifischer<br />
Zell-Linien | weltweit konkurrenzlos<br />
Detektion im Nahinfrarot-Bereich (700 nm, 800 nm)<br />
| Min. Autofluoreszenz v. Zellen u. Plastikmaterialien<br />
| Quantifizierung m. automat. Normalisierung auf ein<br />
simultan gemessenes zweites Target | Direkte Detektion<br />
der Proteine im zellulären Kontext (Eliminierung<br />
von Zelllyse-bedingten Artefakten)<br />
Besonderheiten/<br />
Extras<br />
Preis ViaLight ® LT07-221: 500 tests 155 Euro; 1,000<br />
tests 291Euro; 10,000 tests 2501 Euro | ViaLight ®<br />
LT17-221: 500 tests with 5 white TC plates 193 Euro<br />
| ToxiLight ® LT07-217: 500 tests 173 Euro; 1,000<br />
tests 312 Euro | ToxiLight ® LT17-217: 500 tests with<br />
5 white TC plates 207 Euro
Olympus Deutschland GmbH<br />
Mikroskopie + Industrial Insight<br />
Wendenstr.14-18, 20097 Hamburg<br />
Tel.: +49-(0)40-237730<br />
Fax:+49-(0)40-230817<br />
mikroskopie@olympus.de<br />
www.olympus.de<br />
Merck Chemicals Ltd.<br />
Boulevard Industrial Park<br />
Padge Road, Beeston, Nottingham, NG9<br />
2JR, UK<br />
Dr. Martin Finkbeiner<br />
Tel.: +44-(0)115-9430840<br />
Fax: +44-(0)115-115-9430951<br />
techservice@merckbio.eu<br />
www.merckbio.eu<br />
Merck Chemicals Ltd.<br />
Boulevard Industrial Park<br />
Padge Road, Beeston, Nottingham, NG9<br />
2JR, UK<br />
Dr. Martin Finkbeiner<br />
Tel.: +44-(0)115-9430840<br />
Fax: +44-(0)115-115-9430951<br />
techservice@merckbio.eu<br />
www.merckbio.eu<br />
Firma/Kontakt MDS Analytical Technologies GmbH<br />
Gutenbergstr. 10<br />
85737 Ismaning<br />
Frank.Hafner@moldev.com<br />
Screening Station for Life Science scan ®<br />
InnoCyte Laminin-based 96-Well Cell Invasion<br />
Assay<br />
Produktname ImageXpressMICROTM Caspase-3 Detection Kit (FITC-DEVD-FMK) Cat.<br />
No. QIA91<br />
High Content Screening | Zell-basiertes Screening<br />
| Assay-Entwicklung | Grundlagenforschung |<br />
Systembiologie | Pharmazeutische Forschung Drug<br />
Development | Diagnostische Forschung | Routine-<br />
Anwendungen | Toxikologie | Target Identification |<br />
Target Validation<br />
Beurteilung der Invasionsfähigkeit (metastatisches<br />
Potential) von Zellen<br />
Messung von Apoptose in einem frühen Stadium<br />
in lebenden Zellen<br />
Einsatzgebiete zellbasierte Assays mit Fluoreszenzfarbstoffen bis<br />
zum 1.536 Well-Format<br />
Frei adaptierbares u. konfigurierbares Gesamtsystem,<br />
mit d. fast jeder Assay realisiert und Assay-<br />
Entwicklung betrieben werden kann. Hardware<br />
individuell anpassbar. Analyse-Software stellt von<br />
Bildprozessierung über Bildanalyse bis zur Datenanalyse<br />
und Datennavigation eine umfassende/<br />
einfach zu bedienende Gesamtlösung zur Verfügung.<br />
Beispielassays: Zellzyklus, intrazellulärer<br />
Transport, Genexpression, Proteinlokalisation,<br />
RNAi knock-out, Genaktivierung, Membrantransport,<br />
Mitose, Apoptose.<br />
Eine Membran mit 8 µm Porengröße, beschichtet<br />
mit Laminin als effiziente Barriere für nicht-invasive<br />
Zellen, ermöglicht die Identifizierung von Zellen<br />
mit metastatischem Potential und das Screening<br />
anti-metastatischer Wirkstoffe<br />
FITC-DEVD-FMK ist ein zellpermeabler Inhibitor,<br />
der irreversibel an aktivierte Caspase-3 in apoptotischen<br />
Zellen bindet.<br />
High Content Screening, automatisierte wide-field-<br />
Mikroskopie<br />
Beschreibung des<br />
Assays<br />
Vollautomatisierte Weitfeld-Fluoreszenz- und<br />
-Durchlichtmikroskopie, sowohl für End-Point-<br />
Assays mit fixierten Zellen als auch für dynamische<br />
Assays mit lebenden Zellen | Detektor: CCD-<br />
Kamera<br />
Zellen, die durch die Membran gewandert sind,<br />
werden durch Fluoreszenzfärbung (Calcein AM)<br />
quantitativ detektiert (Emission ~520 nm, Anregung<br />
~485 nm)<br />
FITC konjugierter Inhibitor | kann in der Zelle<br />
über Flow Cytometrie (FACS), Fluorometrie oder<br />
Fluoreszenzmikroskopie nachgewiesen werden<br />
(Emission ~535 nm, Anregung ~485 nm)<br />
Messprinzip/Endpunkt Kinetisch oder Endpunkt, Fluoreszenzfarbstoffe,<br />
MetamMorph ® -basierte Software zur Quantifizierung<br />
von Ergebnissen<br />
Automationsgrad Online Pipettor zum automatischen Pipettieren<br />
während des Assays | ImageXpressMICRO TM ist<br />
voll roboterintegrierbar<br />
Marktübersicht Cell-based Assays<br />
LABORWElT 9. Jahrgang | Nr. 3/2008 | 51<br />
Vollautomatisches System, optional mit Laderoboter<br />
und Bar-Code-Reader ausstattbar<br />
Semi-Automatische Detektion in FACS/Fluorometer 96-well high throughput-Format, Detektion im<br />
Fluorometer semi-automatisch<br />
Detektor (CCD) 12 bit. | Intensität der Beleuchtung:<br />
zwischen 1% und 100% regelbar<br />
Es werden nur 40.000 bis 80.000 Zellen pro well<br />
eingesetzt, bereits wenige invasive Zellen sind<br />
fluorometrisch detektierbar<br />
gekühlte CCD-Kamera Quantitative Detektion apoptotischer und nichtapoptotischer<br />
Zellen<br />
Dynamischer Bereich/<br />
Sensitivität<br />
Außerordentliche Flexibilität der Hardware-<br />
Konfiguration | Cytometrisches Interface für die<br />
Datenanalyse | Präzision und Datenqualität<br />
Invasions-Kammer mit Laminin-Membran im<br />
96-well-Format<br />
Methode zur Detektion von aktiver Caspase-3 in<br />
lebenden Zellen<br />
Besonderheiten/Extras Transmitted light, Klimakammer, online Pipettor,<br />
Roboter zur Plattenbeladung<br />
Preis 100 Tests für 546 Euro 96 Tests für 509 Euro
����<br />
�����������������������������������<br />
�<br />
�<br />
��<br />
����<br />
Marktübersicht Cell-based Assays<br />
�������<br />
��������<br />
������������<br />
�����������������<br />
������������������������<br />
BIOCOM<br />
����<br />
������������������<br />
����������������<br />
25.09.2007 12:42:41 Uhr<br />
�������������������������������<br />
��������������������������������<br />
�����������������������������<br />
�������������������������������<br />
����������������������������������<br />
��������������������������������<br />
������������������������������������<br />
��������������������������������<br />
�����������������������������������<br />
��������������������������������<br />
��������������������������������<br />
������������������<br />
�������<br />
����������������������������������<br />
BIOCOM Verlag GmbH<br />
����������������������<br />
����������������������<br />
�������������<br />
�����������������<br />
������������������������<br />
�����������������������<br />
ProBioGen AG<br />
Dr. Gabriele Schneider<br />
Tel.: +49-(0)30-924006-0<br />
info@probiogen.de<br />
www.probiogen.de<br />
Firma/Kontakt PEQLAB Biotechnologie GmbH<br />
Carl-Thiersch-Str. 2B<br />
91052 Erlangen<br />
Dr. Christof Larisch<br />
larisch@peqlab.de<br />
Service für zellbasierte Assays & zelluläre Analytik | Human artificial lymph node (human ALN) • Research,<br />
Entwicklung, Validierung, Prävalidierung, Transfer<br />
Produktname Cellometer Auto T4<br />
Cellometer Auto M10<br />
ProBioGen AG ist eine Contract Manufacturing Organisation (CMO). Wir bieten zellbasierte Assays im<br />
Rahmen der Herstellung von Biopharmazeutika an sowie – über den CMO-Bereich hinaus – zur Charakterisierung<br />
und Optimierung von Wirkstoffen (lead optimisation).<br />
Einsatzgebiete automatischer Zellzähler für Zellen zwischen 5 und 35 µm (Auto T4 | automatischer Zellzähler für Zellen<br />
zwischen 2 und 12 µm (Auto M10 | Trypanblau Lebend-/Totbestimmung (Vitalitätsmessung) | Bestimmung<br />
von Zellgrößen<br />
Mikroskop-basierter, automatischer Zellzähler für unterschiedlichste eukaryotische Zellen Proliferations-/Antiproliferationsassay<br />
Anti-Viraler-Assay (AVA): Charakterisierung von Interferon alpha/beta (Potency Assay)<br />
Reporter-Gen-Assay (IFN alpha/beta) | Antibody dependent Cellular Cytotoxicity’-Assay (ADCC) |<br />
Zellulärer Zytotoxizitäts-Assay (primäre humane NK-Zellen/Zielelle) | Immun-Modulations-Assay (ILA,<br />
primäre humane Leukozyten/Lymphozyten) | MHC-Assay (primäre humane Leukozyten; IFN alpha/beta) |<br />
Humaner artifizieller Lymphknoten (human ALN): Gewebe-/Organoid- Modell zur Immunogenitäts- und<br />
Immunmodulations-Testung (siehe ‚Produktwelt’ in dieser Ausgabe)<br />
52 | 9. Jahrgang | Nr. 3/2008 LABORWElT<br />
Beschreibung des<br />
Assays<br />
Messprinzip/Endpunkt <strong>Imaging</strong>-Cytometrie: Lichtmikroskop mit LED-Lampe, CCD-Kamera mit Festwinkelobjektiv | Bildauflösung<br />
1280 x 960 Pixel | Messung in spezieller Einmal-Zählkammer | Software-basierte Bildanalyse (Zelldatenbank)<br />
Automationsgrad lediglich 20 µl der Zellsuspension in die Einmal-Zählkammer pipettieren, Einmal-Zählkammer in das<br />
Gerät einführen, Messung starten, nach 30 Sekunden werden die Daten erhalten.<br />
Cellometer Auto T4 : Zellzähler für Zellen zwischen 5 und 35 µm | Cellometer Auto M10: Zellzähler für<br />
Zellen zwischen 2 und 12 µm<br />
Dynamischer Bereich/<br />
Sensitivität<br />
Alle R&D-Aktivitäten laufen unter DIN ISO 9001:2000 und DIN EN ISO 13485:2003 Zusätzlich weisen<br />
wir eine GMP-Herstellungserlaubnis auf. | Mit Hilfe des ALN-Modells können immunmodellierbare sowie<br />
immunogene Eigenschaften von Wirkstoffen vorhergesagt werden.<br />
Besonderheiten/Extras Objektive Resultate du. Automation | Ergebnisse in 30 s | Bilder u. Daten werden im PC gespeichert |<br />
keine Verschleißteile, kein Liquid-Handling-System | Minimales Kontaminationsrisiko durch Verwendung<br />
speziell konzipierter Einmal-Zählkammern<br />
Preis
Promega GmbH<br />
Schildkrötstr. 15, 68199 Mannheim<br />
Dr. Steffen Barz<br />
Tel.: +49-(0)621-85010<br />
Firma/Kontakt Promega GmbH<br />
Schildkrötstr. 15, 68199 Mannheim<br />
Dr. Steffen Barz<br />
Tel.: +49-(0)621-85010<br />
Produktname Caspase-Glo ® 3/7 Assay Cytotox-Glo TM Cytotoxicity Assay<br />
Einsatzgebiete Apoptosemessung; Bestimmung von Caspaseaktivität Messung von Zytotoxizität<br />
Lumineszenter Zytotoxizitäts-Assay; hochsensitiv; homogenes Assay-Format; bietet Möglichkeit zum<br />
Multiplexing; lange Signalstabilität<br />
Lumineszenter, hochsensitiver Assay; geeignet zum Multiplexing; homogener Assay; kurze Assayzeiten;<br />
lange Signalstabilität<br />
Beschreibung des<br />
Assays<br />
Messprinzip: Lumineszenter Nachweis einer Protease im Zellkulturüberstand<br />
Endpunkt: Nekrose/Membranschäden<br />
Messprinzip/Endpunkt Assay basiert auf einem modifizierten Luciferin; durch Caspase 3/7 Aktivität wird Luciferin freigesetzt;<br />
Umsetzung des Luciferins setzt Lichtsignal frei, das mit Caspase 3/7 Aktivität korreliert • Endpunkt:<br />
späte Apoptose<br />
Automationsgrad Protokolle zur Automatisierung können unter www.promega.de/automethods/ herunter geladen werden Automatisierbar (durchführbar bis 1-536 Well-Format)<br />
�<br />
Sensitivität: ca. 50 Zellen (abhängig vom Zelltyp) | Dynamischer Bereich: ca. 5 Dekaden Sensitivität: ca. 50 Zellen (abhängig vom Zelltyp) | Dynamischer Bereich: N.A.<br />
����������������������������������������������������������������������<br />
�����������������������������������������������������������������������<br />
�������������������������������������������������������������������������<br />
���������������������������������������������������������������������<br />
����������������������������������������������������������������������<br />
�������������������������������������������������������������������<br />
������������������������������������������������������������������<br />
�������������������������������������������������������������������������<br />
�������������������������������������������������������������������������<br />
�����������������������������������������������������������������������<br />
��������������������������������������������������������������������<br />
������������������������������������������������������������������<br />
����������������������������������������������������������������������������<br />
�������������������������������������������������������������������<br />
������������������������������������������������������������������<br />
���������������������������������������������������������������������<br />
���������������������<br />
�������� ��������� ���� ���� ��� �������������� ������ ������� ��� ����� ���<br />
��������������������������������������������������������������������<br />
���������������������������������������������������������������������<br />
�����������������������������������������������������������������������<br />
��������������������������������������������������������������������<br />
���������������������������������������������������������������������<br />
��������������������������<br />
Dynamischer Bereich/<br />
Sensitivität<br />
Marktübersicht Cell-based Assays<br />
LABORWElT 9. Jahrgang | Nr. 3/2008 | 55<br />
������������������<br />
Besonderheiten/Extras Einfache Anwendung, höchste Sensitivität Zytotoxizitätsmessung über neuen Protease-Biomarker<br />
Preis ab 92 Euro ab 92 Euro<br />
����<br />
������� ���������� ��� ������� ��������<br />
������ ������<br />
�������� ���������<br />
������� ����������<br />
�������<br />
������� ����������<br />
����������<br />
������� ����������<br />
������� ������� ����������<br />
����������<br />
��� ��� ������������� ��������<br />
��� ��� ������������� ��������<br />
��� ��� ������������� ��������<br />
�����������������������������������<br />
����������������������������������<br />
���������������������������������<br />
������������������������������<br />
�������������������������������<br />
��������������������������������<br />
�����������������������������<br />
���������������������������������<br />
�������������������������������<br />
��������������������������������<br />
���������������������������������<br />
����������������������������������<br />
������������������������������������<br />
��������������������������������<br />
��������������������������������<br />
���������������������������������<br />
����������������������������������<br />
��������������������������������<br />
������������������������������<br />
���������������������������������<br />
����������<br />
���������������������<br />
��������������<br />
�������<br />
��� ��� ������������� ������<br />
�����������������������������<br />
���������������������������������<br />
BIOCOM AG Verlag GmbH<br />
����������������������<br />
����������������������<br />
����������������������<br />
����������������������<br />
��� ������� ��������<br />
��� ��� ������������� ��������<br />
�������������<br />
�������������<br />
�����������������<br />
�����������������<br />
������������������������<br />
������������������������<br />
�����������������������<br />
�����������������������<br />
���������������������������������������
Marktübersicht Cell-based Assays<br />
Roche Diagnostics GmbH<br />
Sandhofer Str. 116<br />
68305 Mannheim,<br />
Fachliche Information<br />
Tel.: +49-(0)621-759-8568<br />
mannheim.biocheminfo@roche.com<br />
www.roche-applied-science.com<br />
Roche Diagnostics GmbH<br />
Sandhofer Str. 116<br />
68305 Mannheim,<br />
Fachliche Information<br />
Tel.: +49-(0)621-759-8568<br />
mannheim.biocheminfo@roche.com<br />
www.roche-applied-science.com<br />
PromoCell GmbH<br />
Sickingenstr. 63/65<br />
69126 Heidelberg<br />
Dr. Jürgen Becker<br />
Tel.: +49-(0)6221-64934-0<br />
Fax: +49-(0)6221-64934-40<br />
info@promokine.de<br />
www.promokine.de<br />
Firma/Kontakt PromoCell GmbH<br />
Sickingenstr. 63/65<br />
69126 Heidelberg<br />
Dr. Jürgen Becker<br />
Tel.: +49-(0)6221-64934-0<br />
Fax: +49-(0)6221-64934-40<br />
info@promokine.de<br />
www.promokine.de<br />
Produktname Cell Viability & Cytotoxicity Kits Cell-based Reporter Assays xCELLigence RTCA SP Instrument Cell Proliferation Reagent WST-1<br />
nicht-radioaktive Bestimmung von metabolischer<br />
Aktivität, Zellproliferation, Zellvitalität oder Zytotoxizitäts-Studien<br />
in Zellkulturen im 96 Well-Format<br />
Real Time-Monitoring lebender Zellen. Zeitgleiche<br />
Analyse vieler zellulärer Parameter wie z.B. Zellproliferation,<br />
Adhäsion, Toxizität oder Rezeptorvermittelte<br />
Signaltransduktion über die gesamte<br />
Dauer eines Experiments.<br />
Nachweis und Quantifizierung von Luziferase und<br />
beta-Galaktosidase-Aktivität in eukaryotischen<br />
Zellen<br />
Einsatzgebiete Quantifizierung der Zellviabilität, Zellproliferation<br />
und Zytotoxizität, Zellzahlbestimmung<br />
Mitochondriale Dehydrogenasen vitaler Zellen<br />
spalten Tetrazoliumsalze wie WST-1 zu rötlichen<br />
Formazansalzen. Der Farbumschlag korreliert<br />
direkt mit der Anzahl metabolisch aktiver Zellen in<br />
der Kultur und kann im ELISA Reader vermessen<br />
werden<br />
Zellen werden in E-Plates ® im 96 well-Format<br />
kultiviert. In die wells ist ein mikroelektronischer<br />
Gold-Sensorarray integriert. Durch Anlegen einer<br />
Spannung von 20mV wird die Änderung der Impedanz<br />
gemessen, die ein Maß für Änderungen<br />
zellulärer Parameter wie z.B. Zelloberfläche, Veränderung<br />
der Zellzahl oder Zellmorphologie ist.<br />
Die Aktivität von Luziferase und beta-Galaktosidase,<br />
welche z.B. in mit entsprechenden<br />
Reporterplasmiden transfizierten Zellen exprimiert<br />
werden, wird durch einfache enzymatische Tests<br />
bestimmt und erlaubt eine Quantifizierung der<br />
entsprechenden Enzymexpression und somit auch<br />
des Anteils der mit den jeweiligen Reportergenen<br />
transfizierten Zellen<br />
Nicht-radioaktive Assays, basierend auf der metabolischen<br />
Aktivität zellulärer Enzyme bzw. der<br />
Bildung von ATP in vitalen Zellen (Cell Viability<br />
Assays) oder der Freisetzung von Lactatdehydrogenase<br />
aus toten Zellen (LDH-Zytotoxizitäts-Assay).<br />
Beschreibung des<br />
Assays<br />
Bildung kolorimetrisch messbarer Endprodukte aus<br />
Substraten für die beta-Galaktosidase (beta-Gal<br />
Reporter-Assay) bzw. Messung der freigesetzten<br />
Biolumineszenz (Luziferase-Assay). Nachweis<br />
erfolgt über kolorimetrische oder Biolumineszenz-<br />
Messung.<br />
Messprinzip/Endpunkt Bildung kolorimetrisch und fluorometrisch messbarer<br />
Endprodukte durch aktive Enzyme in vitalen<br />
Zellen bzw. Messung der durch ATP-Synthese<br />
in vitalen Zellen freigesetzten Biolumineszenz<br />
(Luziferase-basierter ATP-Assay). Nachweis erfolgt<br />
über kolorimetrische, fluorometrische oder<br />
Biolumineszenz-Messung.<br />
56 | 9. Jahrgang | Nr. 3/2008 LABORWElT<br />
Kontinuierliche Messung der Impedanz Colorimetrischer ELISA<br />
Vollautomatisierte Messung<br />
Geeignet auch für verschiedene Mikrotiterplatten-<br />
Formate<br />
Automationsgrad Geeignet auch für verschiedene Mikrotiterplatten-<br />
Formate.<br />
Impedance: 20ohm-10kohm Sehr großer linearer Bereich und hohe Sensitivität:<br />
bereits 5 x 102 Zellen/ml sind detektierbar.<br />
25.000 Zellen pro well beta-Galaktosidase-Assay:
Sigma-Aldrich Chemie GmbH<br />
Eschenstr. 5, 82024 Taufkirchen<br />
Dr. Udo Sticher<br />
Tel.: +49-(0)89-6513-1504;<br />
+49-(0)89-6513-0<br />
Fax: +49-(0)89-6513-1169<br />
udo.sticher@sial.com, deorders@sial.com<br />
www.sigmaaldrich.com<br />
Sigma-Aldrich Chemie GmbH<br />
Eschenstr. 5, 82024 Taufkirchen<br />
Dr. Udo Sticher<br />
Tel.: +49-(0)89-6513-1504;<br />
+49-(0)89-6513-0<br />
Fax: +49-(0)89-6513-1169<br />
udo.sticher@sial.com, deorders@sial.com<br />
www.sigmaaldrich.com<br />
S.CO LifeScience GmbH<br />
Boltzmannstr. 11A<br />
85748 Garching (München)<br />
Tel.: +49-(0)89-1214023 40<br />
Fax: +49-(0)89-1214023 44<br />
info@sco-lifescience.com<br />
www.sco-lifescience.com<br />
Firma/Kontakt Roche Diagnostics GmbH<br />
Sandhofer Str. 116<br />
68305 Mannheim,<br />
Fachliche Information<br />
Tel.: +49-(0)621-759-8568<br />
mannheim.biocheminfo@roche.com<br />
www.roche-applied-science.com<br />
CA200-1KT cAMP Enzyme Immunoassay Kit,<br />
Direct<br />
Produktname Cell Death Detection ELISAPLUS S.CORE - Web-based Image Analysis CS0010-1KT beta-Secretase (BACE1) Activity Detection<br />
Kit (Fluorescent)<br />
The EIA Direct cyclic AMP kit is a competitive immunoassay<br />
for the quantitative determination of<br />
cyclic AMP in samples treatedwith 0.1M HCl.<br />
BACE1 Activity Assay Kit is designed for BACE1<br />
inhibitor screening. It provides all the reagents,<br />
including the BACE1 enzyme for use as a positive<br />
control, required for an efficient detection of<br />
BACE1 activity.<br />
Auswertung von biologischen Assays | Zellkultur<br />
| Zellzählung | Tumorforschung | Angiogenese |<br />
Sprouting Assay | Tube Formation Assay | CAM<br />
Assay | estimmung des Vaskularisationsgrades |<br />
Migration Assay | Bestimmung der Transfektionseffizienz<br />
| Colony Forming Assay | Quantitative<br />
Immunhistochemie | Cell Tracking<br />
Einsatzgebiete nach Induktion der Apoptose Detektion fragmentierter<br />
DNA im Zellkulturüberstand, Plasma, Serum,<br />
Zell-Lysaten oder Zellen ex vivo<br />
The kit uses a polyclonal antibody to cAMP to<br />
bind, in a competitive manner, the cAMP in the<br />
sample or an alkaline phosphatase molecule that<br />
has cAMPcovalently attached to it. Samples or<br />
standards, alkaline phosphatase conjugate, and<br />
antibody are simultaneously incubated at room<br />
temperature in a secondary antibody coated multiwell<br />
plate. The excess reagents are then washed<br />
away and substrate is added. After incubation the<br />
enzyme reaction is stopped and the yellow color<br />
generated read on a multiwell plate reader at 405<br />
nm. The intensity of the yellow color is inversely<br />
proportional to the concentration of cAMP in<br />
either the standards or the samples. The measured<br />
optical density is used to calculate the concentration<br />
of cAMP.<br />
The assay is based on a convenient method of<br />
fluorescence resonance energy transfer (FRET)<br />
in which the fluorescence signal enhancement is<br />
observed after the substrate is cleaved by BACE1<br />
Web-basiertes System zur automatischen Bildauswertung<br />
| objektive quantitative Auswertung<br />
komplexer Assays | einfache Bedienung der Applikationen<br />
über ein personifiziertes Internetportal |<br />
Zugriff auf das System von jedem Rechner auf der<br />
Welt, 24 Stunden am Tag<br />
Photometrischer Enzym-Immunoassay zum<br />
qualitativen und quantitativen in vitro-Nachweis<br />
von zytoplasmatischen Histon-assoziierten DNA-<br />
Fragmenten (Mono- und Oligonucleosomen) nach<br />
induziertem Zelltod<br />
Beschreibung des<br />
Assays<br />
Marktübersicht Cell-based Assays<br />
LABORWElT 9. Jahrgang | Nr. 3/2008 | 57<br />
Messprinzip/Endpunkt Colorimetrischer ELISA Software-basiert automatisch Bildauswertung Endpoint Endpoint<br />
Automationsgrad Web-basiert, vollautomatisch No No<br />
Non-Acetylated Version: 0.39 pmol/ml | Acetylated<br />
Version: 0.037pmol/ml<br />
The assay is designed so that the enzyme converts<br />
5–20% of the substrate to a fluorescent product<br />
during 1–2 hours at 37 °C. Sensitivity/Linearity<br />
depends on the fluorometer used to perform the<br />
assay, typical range 30–500 pmol.<br />
äußerst robuste Objekterkennung | Trennung zwischen<br />
Objekten des Interesses und Artefakten<br />
Sehr hohe Sensitivität: bereits 5 x 102 Zellen/ml<br />
können detektiert werden<br />
Dynamischer Bereich/<br />
Sensitivität<br />
The cAMP Direct EIA may be used to assay cAMP<br />
samples that have been treated with hydrochloric<br />
acid to stop endogenous phosphodiesterase activity.<br />
Samples in this matrix can be read directly<br />
without evaporation or further treatment. Samples<br />
with very low levels of cAMP may be acetylated.<br />
Acetylation of the samples increases the sensitivity<br />
of the assay.<br />
BACE1 is a transmembrane protease responsible<br />
for the β site cleavage of the amyloid precursor<br />
protein (APP) to produce amyloid beta peptide (Abeta).<br />
The accumulation of A-beta in the brain is a<br />
primary cause for the progression of Alzheimer‘s.<br />
BACE1 is a target for inhibitor drug discovery.<br />
Free Trial Service | Angebot kostenloser Machbarkeitsstudien<br />
für Individuallösungen | Entwicklung<br />
von maßgeschneiderten individuellen Lösungen<br />
Besonderheiten/Extras One-step ELISA mit hoher Sensitivität, einfache<br />
und schnelle Durchführung (ca. 3h); Positivkontrolle<br />
ist im Kit enthalten; nicht-radioaktiv, kein prelabelling<br />
nötig; auch für High Throughput-Analysen<br />
geeignet<br />
618 Euro/Kit (250 Reaktionen) 510 €/Kit (sufficient for 96 assays)<br />
Preis 360 Euro (96 Tests) zwischen 1 und 5 Euro pro analysiertem Bild, je<br />
nach Komplexität und Anzahl der Bilder
Marktübersicht Cell-based Assays<br />
VWR International GmbH<br />
Hilpertstr. 20a<br />
64295 Darmstadt<br />
Matthias Dornheim<br />
Tel.: +49-(0)6151-39720<br />
Fax: +49-(0)6151-3972450<br />
biotech@de.vwr.com<br />
TTP LabTech Limited<br />
Melbourn Science Park<br />
Melbourn, Herts, SG8 6EE, UK<br />
Thermo Fisher Scientific<br />
Adenauerallee 113<br />
53113 Bonn<br />
Dr. Carolin Kutzki<br />
Tel.: +49-(0)228-9125650<br />
tebu-bio GmbH<br />
Berliner Str. 255<br />
63067 Offenbach<br />
Ingrid Kautner<br />
Tel.: +49-(0)69-801013-0<br />
Fax: +49-(0)69-801013-20<br />
ingrid.kautner@tebu-bio.com<br />
www.tebu-bio.com<br />
Firma/Kontakt tebu-bio GmbH<br />
Berliner Str. 255<br />
63067 Offenbach<br />
Ingrid Kautner<br />
Tel.: +49-(0)69-801013-0<br />
Fax: +49-(0)69-801013-20<br />
ingrid.kautner@tebu-bio.com<br />
www.tebu-bio.com<br />
Produktname PPAR Screening Assay Cell-based ELISA Kits Cellomics HCS Reagent Kits Acumen ® eX3 Origene TissueScan qPCR Array<br />
High content cell-based screening Biomarker-Validierung, SNP-Analyse,<br />
Genexpressionsprofiling<br />
High Content Screening (HCS) and<br />
Analysis (HCA)<br />
Zur Bestimmung des relativen Phosphorylierungsgrades<br />
von Proteinen<br />
Einsatzgebiete Der Assay erlaubt die Identifizierung<br />
von Subtyp-spezifischen Liganden, die<br />
an PPAR-alpha, -beta/delta oder -gamma<br />
binden.<br />
Die cDNA von verschiedensten Krebstypen<br />
und Patienten wurde über RT PCR<br />
gewonnen und auf eine Ready to Use<br />
qPCR-Platte aufgebracht.<br />
Fluorescence-based high content<br />
assays, in-cell Westerns, reporter gene<br />
expression<br />
Validated single- and multiplexed kits<br />
for cell-based screening and analysis<br />
of specific molecular targets and<br />
biological parameters e.g. Cytotoxicity<br />
and Apoptosis, Inflammation and Cell<br />
Stress, Cell Cycle and Proliferation, Cell<br />
Signaling and Transcription Factors or<br />
Morphology<br />
Die zu untersuchenden Zellen werden<br />
in 96-well-Platten ausgesät und nach<br />
der experimentellen Behandlung fixiert.<br />
Der Kit enthält zwei Primärantikörper<br />
von denen einer nur die aktivierte,<br />
phosphorylierte Form des Zielproteins<br />
erkennt, und der andere sowohl das<br />
aktivierte wie auch das nicht modifizierte<br />
Zielprotein. Nach Inkubation mit<br />
HRP-konjugiertem Zweitantikörper und<br />
Zugabe von Substrat erfolgt die Auswertung<br />
im ELISA-Reader.<br />
Assay basiert auf stabilen HeLa-Luciferase-Reporterzelllinien.<br />
Zellen werden<br />
in 96-well-Platten ausgesät und mit<br />
zu testender Substanz für 24 Stunden<br />
inkubiert. Luciferase-Aktivität wird luminometrisch<br />
gemessen, die Ergebnisse<br />
als RLU (relative light units) angegeben.<br />
Als Positivkontrollen dienen Referenzsubstanzen,<br />
die eine max. Luciferaseaktivität<br />
für die jeweilige Zelllinie zeigen,<br />
als NegativkontrolleDMSO.<br />
Beschreibung des<br />
Assays<br />
Messprinzip/Endpunkt Luciferase-Aktivität in RLU. Die RLU<br />
Werte der Referenzliganden werden als<br />
100% Aktivität gesetzt, die RLU-Werte<br />
der Testsubstanzen werden darauf bezogen<br />
angegeben.<br />
58 | 9. Jahrgang | Nr. 3/2008 LABORWElT<br />
Fluorescence detection Fluorescence / live or fixed endpoint qPCR<br />
Kolorimetrische Auswertung im ELISA<br />
Reader<br />
Integration with standard plate stacker<br />
e.g. Thermo RapidStak<br />
Automationsgrad Der Assay ist für HTS geeignet Möglich bis zum HTS Validated on fully automated Thermo<br />
Scientific Array Scan HCS Reader<br />
Keine Herstellerangaben Single-cell detection Multiple laser excitation and high sensitivity<br />
photomultiplier tube detection<br />
Der Assay kann Liganden als „non<br />
agonist“, „total agonist“ und „partial<br />
agonist“ identifizieren.<br />
Dynamischer Bereich/<br />
Sensitivität<br />
Alle Patientenproben sind bestens<br />
dokumentiert (pathologische Befunde,<br />
Berichte über Krankheitsstadien etc.).<br />
Zugang zu allen Ursprungsmaterialen<br />
wie RNA, Protein oder Gewebe.<br />
Choice of laser excitations, plate stacker<br />
for automation, Locator integrated<br />
fluorescence microscope<br />
Einfache Handhabung: keine Extraktionen,<br />
kein Western Blot; Assay ist nicht<br />
radioaktiv; in ca. 3 Stunden durchzuführen<br />
Besonderheiten/Extras Mit den drei zur Verfügung stehenden<br />
Zelllinien können Liganden für PPARalpha,<br />
-beta/delta oder -gamma identifiziert<br />
werden. Die Liganden werden<br />
klassifiziert als „non agonist“, „total<br />
agonist“ oder „partial agonist“.<br />
600 Euro bis 1.500 Euro<br />
£110,000 to £200,000 depending on<br />
specification<br />
416 Euro bis 907 Euro 232 Euros for 96 assays<br />
515 Euros for 5 x 96 assays<br />
Preis Abhängig von der Anzahl der getesteten<br />
Substanzen
Seite bitte abtrennen – per Fax an 030-264921-11<br />
Kontakt zu Verbänden<br />
Die Mitglieder der nachfolgenden Fachgesellschaften erhalten LABORWELT regelmäßig mit freundlicher Empfehlung ihrer Organisationen.<br />
Wer sich darüber hinaus für eine Mitarbeit oder einen Beitritt interessiert, erreicht die Fachgesellschaften unter den folgenden<br />
Kontakt daten:<br />
Ich interessiere mich für<br />
den Beitritt<br />
Unterstützung für Jungwissenschaftler<br />
Interessenvertretung<br />
eine Spende<br />
Fachgruppen im Bereich<br />
Verband (siehe unten, bitte ankreuzen)<br />
DECHEMA<br />
Fachgemeinschaft<br />
Biotechnologie<br />
Theodor-Heuss-Allee 25<br />
60486 Frankfurt am Main<br />
Tel.: +49-(0)-69-7564-0<br />
Fax: +49-(0)-69-7564-169<br />
www.dechema.de<br />
Deutsche Gesellschaft für<br />
Proteomforschung<br />
c/o MPI für Biochemie<br />
Am Klopferspitz 18a<br />
82152 Martinsried<br />
Tel.: +49-(0)-89-1897-9007<br />
Fax: +49-(0)-89-1897-9009<br />
c.kleinhammer@dgpf.org<br />
www.dgpf.org<br />
Österreichische Gesellschaft<br />
für Biotechnologie<br />
c/o Inst. f. Molekulare Biotechnologie,<br />
TU Graz<br />
Petersgasse 14<br />
A-8010 Graz, Austria<br />
Tel.: +43-(0)-316-873-4072<br />
Fax: +43-(0)-316-873-4071<br />
www.oegbt.org<br />
Deutsche Gesellschaft für Hygiene<br />
und Mikrobiologie (DGHM)<br />
c/o Institut für Hygiene und<br />
Mikrobiologie<br />
Josef-Schneider-Str. 2<br />
97080 Würzburg<br />
Tel.: +49-(0)-931-201-46936<br />
Fax: +49-(0)-931-201-46445<br />
www.dghm.org<br />
bts (Biotechnologische Studenteninitiative<br />
e.V.)<br />
c/o BIOCOM<br />
Stralsunder Str. 58-59<br />
13355 Berlin<br />
Tel.: +49-(0)-2649-21-21<br />
Fax: +49-(0)-2649-21-11<br />
www.bts-ev.de<br />
Bitte kontaktieren Sie mich<br />
Name Firma<br />
Tel. Fax<br />
E-Mail<br />
Gesellschaft für Genetik<br />
GESELLSCHAFT FÜR GENETIK<br />
c/o HZM – Deutsches<br />
Forschungszentrum für<br />
Gesundheit/Inst. of Developmental<br />
Genetics<br />
Tel.: +49-(0)-89-3187-2610<br />
Fax: +49-(0)-89-4620<br />
www.gfgenetik.de<br />
Gesellschaft für Signaltransduktion<br />
c/o Prof. Dr. Ralf Hass<br />
Med. Hochschule Hannover<br />
AG Biochemie u. Tumorbiol.<br />
30625 Hannover<br />
Tel.: +49-(0)-511-532-6070<br />
Fax: +49-(0)-511-532-6071<br />
www.sigtrans.de<br />
Gesellschaft für Pharmakologie und<br />
Toxikologie<br />
Geschäftsstelle der DGPT<br />
Achenbachstraße 43<br />
40237 Düsseldorf<br />
Tel.: +49-(0)-211-600-692-77<br />
Fax: +49-(0)-211-600-692-78<br />
mitglieder@dgpt-online.de<br />
www.dgpt-online.de<br />
Nationales Genomforschungsnetz<br />
c/o DKFZ<br />
Im Neuenheimer Feld 580<br />
69120 Heidelberg<br />
Tel.: +49-(0)-6221-424-743<br />
Fax: +49-(0)-6221-423-454<br />
S.Argo@dkfz-heidelberg.de<br />
www.ngfn.de<br />
Deutsche Gesellschaft für<br />
Neurogenetik<br />
Institut für Humangenetik<br />
Calwer Straße 7<br />
72076 Tübingen<br />
Tel.: +49-(0)-7071-2977692<br />
Fax: +49-(0)-7071-295171<br />
peter.bauer@<br />
med.uni-tuebingen.de<br />
www.hih-tuebingen.de/dgng/<br />
Netzwerk Nutrigenomik<br />
RNA-Netzwerk<br />
BIO Deutschland<br />
Service Verbände<br />
Netzwerk Nutrigenomik<br />
Arthur-Scheunert-Allee 114<br />
14558 Nuthetal<br />
Tel.: +49-(0)-33200-88-301<br />
Fax: +49-(0)-33200-88-541<br />
mail@nutrigenomik.de<br />
www.nutrigenomik.de<br />
c/o Prof. Dr. Volker A. Erdmann<br />
Freie Universität Berlin<br />
Thielallee 63, 14195 Berlin<br />
Tel.: +49-(0)-30-8385 6002<br />
Fax: +49-(0)-30-8385 6413<br />
erdmann@chemie.fu-berlin.de<br />
www.rna-network.com<br />
Tegeler Weg 33/<br />
berlinbiotechpark<br />
10589 Berlin<br />
Tel.: +49-(0)-30-264-840-87<br />
Fax: +49-(0)-30-264-840-88<br />
info@biodeutschland.org<br />
www.biodeutschland.org<br />
AG Life Science Research (AG LSR)<br />
c/o VDGH im VCI<br />
Mainzer Landstraße 55<br />
60329 Frankfurt am Main<br />
Tel.: +49-(0)-69-2556-1730<br />
Fax: +49-(0)-69-236650<br />
vdgh@vdgh.de<br />
www.vdgh.de<br />
Sollen die Mitglieder Ihrer<br />
wissenschaftlichen Fachgesellschaft<br />
künftig auch kostenfrei<br />
LABORWELT beziehen? Informationen<br />
erhalten Sie bei:<br />
Andreas Macht<br />
a.macht@biocom.de<br />
LABORWElT 9. Jahrgang | Nr. 3/2008 | 59
Service Stellenmarkt<br />
Akademischer Stellenmarkt<br />
Veröffentlichen Sie Ihre Stellenanzeigen zielgruppengerecht in unserem akademischen Stellenmarkt, der allen nicht-kommerziellen Instituten<br />
für ihre Stellenausschreibungen kostenlos zur Verfügung steht. Bitte senden Sie dazu Ihre Anzeige (1/4 Seite 90 mm breit x 122,5 mm hoch, Logo –<br />
jpg oder tiff, 300 dpi Auflösung) an a.macht@biocom.de. Annahmeschluß für die nächste LABorweLt-Ausgabe 4/08 (erscheinungstermin<br />
18.07.2008) ist der 4. Juli 2008.<br />
1 Doktorarbeit Biologie (BAT IIA/2)<br />
1 Diplom/Masterarbeit Biologie/<br />
Biotechnologie/Biochemie<br />
1 Diplom/Masterarbeit Bioinformatik<br />
Drosophila Group, Department of Cellular Neurology, Hertie-Institute for Clinical<br />
Brain Research,<br />
Center for Neurology, Universitätsklinikum Tübingen<br />
Ziel unserer Arbeitsgruppe, ist es zu einem besseren Verständnis der molekularen<br />
Mechanismen beizutragen, die die Stabilisierung bzw. den Abbau der<br />
Synapsen kontrollieren. Solche Störungen stellen die zelluläre Grundlage von<br />
mentaler Retardierung sowie von Neurodegeneration dar.<br />
Neuromuskuläre Drosophila-Synapsen werden in unserem Labor als Modell<br />
genutzt. Hierbei erlaubt uns ein spezieller Assay, Veränderungen an individuellen<br />
Synapsen über einen Zeitraum von mehreren Tagen in lebenden Tieren<br />
zu verfolgen (Refs. 1+2).<br />
Doktorarbeit sowie Diplomarbeit Biologie:<br />
Deine Aufgabe wäre es, unser Team (1 medizinische + 2 biologische Doktorandinnen<br />
+ mehrere Diplomanden) weiter zu verstärken und mitzuhelfen herauszufinden,<br />
wie genau die identifizierten Gene interagieren um zu „entscheiden“,<br />
welche Synapsen stabilisiert und welche eliminiert werden. Anliegen Deiner<br />
Arbeit ist es, eine „Brücke“ vom mutierten Molekül zum Verhaltensdefekt zu<br />
schlagen.<br />
Methodisch sind Erfahrungen mit Molekularbiologie, Drosophila-Genetik,<br />
Konfokaler Mikroskopie, Elektronenmikroskopie, Bildanalyse/Bioinformatik<br />
und Biochemie von Vorteil, aber keine Voraussetzung für eine erfolgreiche<br />
Bewerbung.<br />
Für eine Promotion solltest Du zu den Top 15% Deines Jahrganges gehören<br />
und idealerweise Erfahrung auf dem Gebiet der Neurobiologie oder Erfahrung<br />
mit Drosophila mitbringen.<br />
Bioinformatik:<br />
Ziel der ausgeschrieben Diplomarbeit ist es, OrthoExpress – ein Programm das<br />
organismenübergreifende Expressionsanalysen aus multiplen Experimenttypen<br />
auf Basis von bestehenden Orthologien ermöglicht – weiterzuentwickeln. Bisher<br />
kann OrthoExpress nur als lokale Applikation genutzt werden. Im Rahmen<br />
einer Publikation soll OrthoExpress nun so modifiziert werden, daß es als<br />
Webapplikation genutzt werden kann. Hierzu sollen u.a. neue Protokolle zum<br />
automatisierten Einlesen und Verwalten von Datensätzen erstellt werden. Es<br />
besteht (falls gewünscht) die Möglichkeit, an der Charakterisierung der erzeugten<br />
Kandidaten mitzuarbeiten.<br />
Du solltest Erfahrung mit DBMS und JAVA mitbringen. Erfahrungen mit HTML<br />
oder XML und Programmiersprachen zur Modellierung von Web-Interfaces sind<br />
von Vorteil, aber nicht unbedingt notwendig.<br />
Bewerbungen und Rückfragen bitte per E-Mail an:<br />
tobias.rasse@medizin.uni-tuebingen.de senden.<br />
(1. Nature Neuroscience (2005) 8: 898-905, (2. Nature Protocols (2007); 2 (12): 3285-3298.<br />
Am Forschungszentrum Borstel<br />
ist ab sofort eine<br />
POST-DOC (Dr. rer. nat) m/w<br />
Stelle im Rahmen des Exzellenzclusters „Inflammation at Interfaces“ neu<br />
zu besetzen.<br />
Als Bestandteil der Zellwand von Gram-negativen und Gram-positiven<br />
Bakterien stellt Peptidoglycan (PG, Murein) eine ideale Zielstruktur für<br />
das Immunsystem dar, um bakterielle Infektionen festzustellen und erste<br />
Abwehrstrategien zu entwickeln. Schwerpunkte des Projektes bilden die<br />
strukturelle und funktionelle Analyse von Nod-Rezeptoren und deren Liganden,<br />
die über eine spezifische Wechselwirkung mit dem programmierten<br />
Zelltod (Apoptose) und dem Angeborenen Immunsystem des Wirtes in<br />
Verbindung stehen.<br />
Das Projekt hat zum Ziel, Struktur und Funktion dieser Bakterienstrukturen<br />
(PAMPs) im Zusammenhang mit den „pathogen related receptors“ (PRR)<br />
auf molekularer Ebene aufzuklären und näher zu charakterisieren. Es ist<br />
integraler Bestandteil der Research Area G [Integrated Research Network<br />
(IRN) „Nod-like receptors“] und erfordert eine enge methodische und inhaltliche<br />
Zusammenarbeit mit anderen Gruppen, sowohl innerhalb dieser<br />
Research Area wie auch dem gesamten Cluster.<br />
Der/die Stelleninhaber/in sollte mit einem breiten Spektrum von biochemisch-anlytischen<br />
Techniken und Methoden (HPLC, MS, NMR), und auch<br />
solchen der angrenzenden Fachgebiete (Molekularbiologie, Biologie,<br />
Biochemie und Immunologie) vertraut sein.<br />
Die Stelle wird zunächst für 3 Jahre ausgeschrieben und kann im Bedarfsfall<br />
für weitere 2 Jahre verlängert werden. Die Bezahlung erfolgt<br />
nach TVÖD 14 (BAT IIa) und hängt von den individuellen Qualifikationen<br />
des Bewerbers/der Bewerberin ab. Das Forschungszentrum Borstel liegt<br />
zwischen Hamburg, Kiel und Lübeck und es besteht die Möglichkeit auf<br />
dem Campus zu wohnen.<br />
Es werden alle im öffentlichen Dienst üblichen Sozialleistungen geboten.<br />
Schwerbehinderte werden bei sonst gleicher Eignung bevorzugt eingestellt.<br />
Bitte schicken Sie Ihre Bewerbung, Lebenslauf und Zeugnisse zusammen<br />
mit allen wichtigen Dokumenten vorzugsweise per E-Mail an<br />
Personalwesen des Forschungszentrums Borstel<br />
Parkallee 2, 23845 Borstel<br />
E-Mail: hpump@fz-borstel.de<br />
60 | 9. Jahrgang | Nr. 3/2008 LABORWElT
Service Stellenmarkt<br />
Als europaweit führendes Forschungszentrum mit der Ausrichtung „Environmental<br />
Health“ (Verknüpfung von Biomedizin und Umweltforschung) analysieren die<br />
Forscherinnen und Forscher des Helmholtz-Zentrums München grundlegende<br />
Prozesse der Krankheitsentstehung, der Schädigung sowie der Abwehr- und<br />
Kompensationsfähigkeit des Organismus. Wir sind eine Forschungseinrichtung<br />
des Bundes und des Freistaats Bayern mit Sitz in Neuherberg, im Norden Münchens, und sind Mitglied der Helmholtz-Gemeinschaft Deutscher Forschungszentren<br />
e.V., der größten öffentlichen Forschungsorganisation Deutschlands. Das Helmholtz-Zentrum München als Träger des Bayerischen Frauenförderpreises sowie des Total<br />
E-Quality-Zertifikates strebt eine Erhöhung des Frauenanteils an und fordert deshalb qualifizierte Interessentinnen auf, sich zu bewerben. Schwerbehinderte werden<br />
bei gleicher Eignung bevorzugt.<br />
Die Abteilung Recht & Technologietransfer sucht für den Bereich Technologietransfer<br />
eine/n<br />
Naturwissenschaftler/in für Erfinder-<br />
beratung und Patente(134/2007)<br />
Ihre Aufgaben<br />
– Erfinderberatung<br />
– Technologie-Scouting<br />
– Management und Betreuung von Patentakten<br />
– Prüfung von wissenschaftlichen Publikationen auf schutzrechtsrelevante<br />
Inhalte<br />
– Durchführung von Recherchen zum Stand der Technik von Patentanmeldungen<br />
– Erstellung und Prüfung von Technologieangeboten für Kooperationsvorhaben<br />
mit der Industrie<br />
Ihre Qualifikation<br />
– Hochschulstudium und Promotion in den Bereichen Chemie, Biochemie,<br />
Physik oder Medizin<br />
– Kenntnisse im Umgang mit Patentschriften<br />
– sehr gutes Englisch<br />
– selbstständige Arbeitsweise<br />
Unser Angebot<br />
– Tätigkeit in einem innovativen, zukunftsorientierten Unternehmen<br />
– umfangreiches Fortbildungsangebot<br />
– zunächst für zwei Jahre befristetes Arbeitsverhältnis und eine Vergütung<br />
nach TVÖD<br />
Wir freuen uns auf Ihre Bewerbung, Martin Reichel<br />
E-Mail: reichel@helmholtz-muenchen.de<br />
Für Rückfragen wenden Sie sich bitte an: Dr. Wolfgang Nagel<br />
Tel.: 089/3187-1210, E-Mail: wolfgang.nagel@helmholtz-muenchen.de<br />
Helmholtz Zentrum München • Deutsches Forschungszentrum für Gesundheit und Umwelt (GmbH) • Institut für Entwicklungsgenetik (IDG)<br />
Postfach 1129, 85758 Neuherberg<br />
An der RWTH Aachen ist im Lehr- und Forschungsgebiet Biomaterialien<br />
zum nächstmöglichen Termin eine<br />
für drei Jahre zu besetzen.<br />
Doktorandenstelle (TV-L13/2)<br />
(Fachrichtung: Biotechnologie/Biologie/Biochemie)<br />
Das Institut für Entwicklungsgenetik (Independent Research Group Neuronal<br />
Circuit Formation, Leitung: Dr. Andrea Huber Brösamle) sucht zum nächstmöglichen<br />
Zeitpunkt eine/n<br />
Naturwissenschaftler/in (93/2008)<br />
Ihre Aufgaben<br />
– Untersuchung der molekularen Mechanismen der Nervenfaserleitung während<br />
der Entwicklung<br />
– Untersuchung der molekularer Prozesse, die die Verdrahtung des Nervensystems<br />
steuern durch Manipulation der Expression von Kandidatenmolekülen<br />
in den Modellsystemen Maus und Hühnchen<br />
Ihre Qualifikation<br />
– Hochschulstudium und Promotion in Biologie oder verwandten Bereichen<br />
– gute Kenntnisse in Entwicklungsneurobiologie<br />
– Erfahrung in anatomischen und molekularbiologischen Methoden<br />
– großes fachliches Interesse<br />
– selbstständige und eigenverantwortliche Arbeitsweise<br />
Unser Angebot<br />
– Tätigkeit in einem innovativen, zukunftsorientierten Unternehmen<br />
– eine anregende Arbeitsumgebung<br />
– umfangreiches Fortbildungsangebot<br />
– zunächst für 2 Jahre befristetes Arbeitsverhältnis und eine Vergütung nach<br />
TVÖD<br />
Wir freuen uns auf Ihre Bewerbung.<br />
Dr. Andrea Huber Brösam<br />
E-Mail: andrea.huber@helmholtz-muenchen.de<br />
Telefon: 089/3187-4117<br />
Das DFG-Projekt „Modifizierte Glykosaminoglykane“ befasst sich mit der kombinierten chemo-enzymatischen Synthese von modifizierten Neo-Glykosaminoglykanen<br />
und beinhaltet die Produktion und Charakterisierung von rekombinanten Enzymen sowie deren Verwendung in der enzymatischen Synthese von Oligosaccharidliganden,<br />
und die biochemische Charakterisierung von biofunktionalisierten Oberflächen. Erfahrungen in der Enzymtechnologie, der Kohlenhydratchemie und der<br />
Biokatalyse wären vorteilhaft.<br />
Voraussetzungen sind ein Diplom/Master in Chemie, Biochemie, Biologie oder Biotechnologie und die Bereitschaft, sich mit Teamfähigkeit und Engagement in neue<br />
Methoden in einem interdisziplinären Arbeitsgebiet einzuarbeiten.<br />
Ihre schriftliche Bewerbung mit Lebenslauf, Zeugnissen und einer Kurzbeschreibung Ihrer bisherigen Forschungstätigkeit senden Sie bitte an:<br />
Univ.-Prof. Dr. Lothar Elling, Lehr- und Forschungsgebiet Biomaterialien, Institut für Biotechnologie und Helmholtz-Institut für Biomedizinische Technik,<br />
Worringer Weg 1, 52074 Aachen, E-Mail: l.elling@biotec.rwth-aachen.de, Tel.: 0241-8028350.<br />
LABORWElT 9. Jahrgang | Nr. 3/2008 | 61
Service Stellenmarkt<br />
Paul-Ehrlich-Institut Paul-Ehrlich-Straße 51 - 59,<br />
63225 Langen, Telefon (06103) 77-11 00<br />
Das Paul-Ehrlich-Institut ist eine wissenschaftliche Einrichtung des Bundes, die<br />
als Bundesoberbehörde für die Zulassung und Chargenprüfung immunbiologischer<br />
Arzneimittel zuständig ist und auf den damit verbundenen Gebieten der<br />
Naturwissenschaften (z.B. Virologie, Bakteriologie, Immunologie, Allergologie,<br />
Medizinische Biotechnologie, Hämatologie) Forschung betreibt.<br />
Im Interesse des Gesundheitsschutzes wurde im Paul-Ehrlich-Institut ein gemäß<br />
DIN EN 17025 und Richtlinie 98/79/EG akkreditiertes Prüflabor für In-vitro-<br />
Diagnostika (IVD) eingerichtet, das mit Benannten Stellen und Herstellern bei der<br />
Prüfung und Bewertung von Diagnostika (HIV-, HTLV-, HBV-, HCV- und Hepatitis<br />
Delta-Testkits sowie Blutgruppenreagenzien) zusammenarbeitet.<br />
Die Aufgaben des Prüflabors bestehen in der praktischen Prüfung der Testkits,<br />
der Beurteilung der Daten und der Kommunikation mit den Auftraggebern.<br />
Im Prüflabor ist zum nächstmöglichen Zeitpunkt die Position einer / eines<br />
Wissenschaftlichen Angestellten<br />
Stellenbewertung: E 13/14 TVöD Bewerbungskennziffer: 21 / 2008<br />
zu besetzen.<br />
Aufgabenprofil:<br />
Die verantwortungsvolle Aufgabe schließt die folgenden Tätigkeiten ein:<br />
– Bewertung von Prüfresultaten<br />
– Bewertung der Leistung von IVDs und Erstellung von Beurteilungsberichten<br />
– Pflege der Standard- und Referenzmaterialien<br />
– Mitarbeit auf dem Gebiet des Qualitätsmanagements<br />
– Fachliche Zusammenarbeit mit anderen Abteilungen des Institutes sowie<br />
externen Behörden und Einrichtungen<br />
– Anleitung der technischen MitarbeiterInnen des Labors<br />
– Mitwirkung bei der Ausbildung von Biologielaboranten und der Fortbildung<br />
ausländischer Behördenmitarbeiter im PEI (Training of Assessors)<br />
Anforderungsprofil:<br />
– Abgeschlossenes naturwissenschaftliches oder (veterinär-)medizinisches<br />
Hochschulstudium<br />
– Diplom/Approbation<br />
– Berufserfahrung in der Virologie, Diagnostik, Hämatologie, Labormedizin<br />
– Fähigkeit zur selbstständigen und eigenverantwortlichen Arbeit<br />
– Teamfähigkeit, Leistungsbereitschaft und Loyalität<br />
– Sehr gute Kenntnisse der englischen Sprache sowie der Standard MS Office<br />
Software<br />
– Bereitschaft zur Einarbeitung in rechtliche Regelwerke<br />
Das Beschäftigungsverhältnis ist zunächst befristet 5 Jahre. Die wöchentliche<br />
Arbeitszeit beträgt 39 Stunden; Teilzeitbeschäftigung ist grundsätzlich<br />
möglich.<br />
Die Eingruppierung erfolgt gemäß den tarifrechtlichen Bestimmungen des<br />
TVöD - Bund.<br />
Auswärtigen Bewerbern ist das Amt bei der Wohnraumbeschaffung behilflich. Trennungsgeld<br />
und Umzugskosten werden nach den gesetzlichen Vorschriften gewährt.<br />
Schwerbehinderte werden bei gleicher Eignung bevorzugt berücksichtigt.<br />
Das Paul-Ehrlich-Institut fördert die Gleichstellung von Frauen und Männern<br />
und ist daher an Bewerbungen von Frauen interessiert.<br />
Bitte richten Sie Ihre vollständigen Bewerbungsunterlagen unter Angabe<br />
der Bewerbungskennziffer bis zum 30. Mai 2008 an das Personalreferat<br />
des Paul-Ehrlich-Instituts.<br />
Postdoc or Ph.D. student position in neurobiology /<br />
protein chemistry of mental diseases<br />
The Institute for Neuropathology, Research Group „Neurodegeneration“ (PD Dr.<br />
C. Korth) invites applications for a postdoc position starting immediately for 2<br />
years, with possibility of extension.<br />
The research focus will be on characterizing proteins and genes associated<br />
with chronic mental diseases like schizophrenia and the affective disorders<br />
with the goal of developing novel ways of diagnosis and therapy. Please read J.<br />
Neuroscience 28: 3839f for a perspective into our research. A strong background<br />
in molecular biology and/or protein chemistry is favorable. The research group<br />
is embedded into several close collaborations with research groups in Europe<br />
and the U.S. on the named topic.<br />
We are looking for a dedicated scholar with excellent skills in protein biochemistry<br />
and molecular biology, fluency in written and spoken English, an open mind<br />
for new approaches and a lot of team spirit.<br />
Further information on the position can be obtained from PD Dr. C. Korth,<br />
0211-8116153, korth@med.uni-duesseldorf.de). Applications please only<br />
electronically to the following address: korth@med.uni-duesseldorf.de.<br />
Applications include CV, publication record, and names and contact information<br />
of three references.<br />
An der Universität Potsdam ist an der Mathematisch-<br />
Naturwissenschaftlichen Fakultät am Institut für Biochemie<br />
und Biologie, Professur für Bioinformatik, in enger<br />
Kooperation mit dem Max-Planck-Institut für Molekulare<br />
Pflanzenphysiologie im Rahmen eines Drittmittelprojekts<br />
möglichst zum 01.07.2008 die Stelle einer/eines<br />
wissenschaftlichen Mitarbeiterin/Mitarbeiters<br />
Entgeltgruppe 13 TV-Länder (Tarifgebiet Ost), Kenn-Nr. 13/2008, mit einer<br />
wöchentlichen Arbeitszeit von 40 Stunden befristet bis zum 30.06.2010 zu<br />
besetzen.<br />
Die Stelle ist in das vom BMBF finanzierte GABI-FUTURE-Verbundvorhaben<br />
“Biomasseproduktion bei Mais: Genomik-basierte und System-orientierte<br />
Pflanzenzüchtung auf Energiemais“ (GABI-ENERGY) eingebettet.<br />
Voraussetzungen:<br />
– abgeschlossenes naturwissenschaftliches Hochschulstudium<br />
– gute Programmierkenntnisse<br />
– Interesse an interdisziplinärer Forschungstätigkeit, insbesondere an Statistik<br />
und biologischen Fragestellungen<br />
– Promotion erwünscht<br />
Aufgaben:<br />
Bioinformatik-Forschungstätigkeit im Bereich der Analyse von komplexen<br />
experimentellen Profildaten über Gene, Metabolite und Proteine, vor allem mit<br />
statistischen Methoden und Verfahren des maschinellen Lernens.<br />
Weitere Auskünfte: Prof. Dr. Joachim Selbig<br />
Tel.: +49 (0)331 5678208, selbig@mpimp-golm.mpg.de<br />
(siehe auch http://www.bio.uni-potsdam.de/ professuren/bioinformatik)<br />
Bewerbungen richten Sie bitte bis an die Universität Potsdam, Dezernat<br />
für Personal- und Rechtsangelegenheiten, Am Neuen Palais 10, 14469<br />
Potsdam.<br />
Die Universität strebt eine Erhöhung des Anteils von Frauen im wissenschaftlichen<br />
Bereich an und fordert deshalb Frauen nachdrücklich zur Bewerbung<br />
auf. Bewerbungen von Schwerbehinderten werden bei gleicher Eignung<br />
bevorzugt.<br />
Für die Rücksendung der Bewerbungsunterlagen bitten wir Sie, einen adressierten<br />
und ausreichend frankierten Briefumschlag beizulegen.<br />
62 | 9. Jahrgang | Nr. 3/2008 LABORWElT
The Dep. Functional Proteomics, Medizinisches Proteom-Center (MPC), Ruhr-<br />
University Bochum advertises the following post:<br />
Full-time Post-Doc position<br />
The MPC is one of the world leading institutes for protein research. The main<br />
scientific focus of the Dep. Functional Proteomics, MPC led by Prof. Dr. Katrin<br />
Marcus is the detailed biochemical protein analysis of biological systems via<br />
state-of-the-art proteomics, molecular biological and cell-based approaches.<br />
Long-term aim is to contribute to the advancement of clinical diagnostics as well<br />
as to the development of therapies concerning major diseases like Alzheimer’s<br />
and Parkinson’s disease (PD).<br />
Responsibilities:<br />
Within an interdisciplinary project in the National Genome Research Network<br />
(NGFNplus) (funding by the Federal Ministry of Education and Research (BMBF)<br />
together with our partners we are interested in the investigation of PD using<br />
modern proteomic methods (HPLC, 2D Fluorescence Difference Gel Electrophoresis<br />
(2D-DIGE), and mass spectrometry) as well as cell-based and molecular<br />
biological approaches. Main focus is the understanding of pathomechanims<br />
underlying PD and the identification of biomarkers.<br />
The candidate will engage a central role in the Brain Proteomics group and<br />
strongly complement the expertise of the team. The position additionally includes<br />
the supervision of students, technical assistants and PhD students.<br />
Requirements:<br />
PhD in Biology, Chemistry, Biochemistry, Pharmacy, or Medicine.<br />
Experience in modern proteome analysis/neuroproteomics and/or Parkinson’s<br />
disease/neurodegeneration is mandatory.<br />
Experience in molecular biology/cell biology is highly desirable.<br />
Personal qualities: Excellent skills in communication and team-oriented work.<br />
An active, professional approach to the work with drive for independent work.<br />
Mobility, flexibility.<br />
Language skills: English business fluent.<br />
The Ruhr-University Bochum aims at increasing the percentage of women<br />
in research positions and strongly encourages women to apply; as an equal<br />
opportunity employer it particularly welcomes applications from individuals<br />
with disabilities.<br />
The position is open from July 1st, 2008 and is initially limited for a period<br />
of 2 years.<br />
Location: Bochum, Germany.<br />
Applicants should send a full CV, a list of publications, names and addresses<br />
of 2 academic referees and a short description of main research interest by<br />
email to<br />
Prof. Dr. Katrin Marcus, Dep. Functional Proteomics<br />
Medizinisches Proteom-Center, Ruhr-University Bochum<br />
Phone.: +49-(0)234-32-28444<br />
Email: katrin.marcus@rub.de, www.funktionelle-proteomik.de<br />
Informal scientific enquiries are always welcome via email or phone.<br />
Service Stellenmarkt<br />
Paul-Ehrlich-Institut Paul-Ehrlich-Straße 51 - 59,<br />
63225 Langen, Telefon (06103) 77-11 00<br />
Das Paul-Ehrlich-Institut ist eine wissenschaftliche Einrichtung des Bundes, die<br />
als Bundesoberbehörde für die Zulassung und Chargenprüfung immunbiologischer<br />
Arzneimittel zuständig ist und auf den damit verbundenen Gebieten der<br />
Naturwissenschaften (z.B. Virologie, Bakteriologie, Immunologie, Allergologie,<br />
Medizinische Biotechnologie, Hämatologie) Forschung betreibt.<br />
Im Fachgebiet “Therapeutische Impfstoffe“ der Abteilung – Immunologie – ist<br />
zum nächstmöglichen Zeitpunkt die Position eines/einer<br />
Technischen Assistenten/<br />
Technischen Assistentin<br />
Stellenbewertung: E 8/ E 9 TVöD<br />
Bewerbungskennziffer: 20/2008<br />
zu besetzen.<br />
Aufgabenprofil:<br />
Es sollen Aufgaben in der Forschung wahrgenommen werden, wo Methoden<br />
der zellulären Immunologie und der Molekularbiologie zur Anwendung kommen.<br />
Forschungsschwerpunkte sind molekulargenetische Untersuchungen transgener<br />
Mauslinien und Analysen zur zellulären Immunität, z. B. gegen Tumore.<br />
Als zusätzliche Aufgaben sind die Herstellung von wässrigen Lösungen,<br />
Zellkulturmedien und hochreines Wasser auf der Basis eines QM-Systems<br />
sowie die Mitwirkung bei der Vorbereitung und Durchführung von Versuchen<br />
zu benennen.<br />
Anforderungsprofil:<br />
– Abgeschlossene Ausbildung als Technische Assistentin/Technischer Assistent<br />
– Erfahrung mit zellbiologischen oder molekularbiologischen Methoden<br />
– Fähigkeit zur Berechnung und Herstellung von Puffern und anderen Rezepturen<br />
– Fähigkeit zum gewissenhaften, selbständigen und verantwortlichen Bearbeiten<br />
der übertragenen Aufgaben<br />
– Teamfähigkeit und Belastbarkeit.<br />
– Kenntnisse der MS Office Software<br />
Das Beschäftigungsverhältnis ist vorerst auf 3 Jahre befristet. Die wöchentliche<br />
Arbeitszeit beträgt 39 Stunden; Teilzeitbeschäftigung ist grundsätzlich<br />
möglich. Die Eingruppierung erfolgt nach den tariflichen Bestimmungen des<br />
TVöD – Bund.<br />
Auswärtigen Bewerbern ist das Amt bei der Wohnraumbeschaffung behilflich.<br />
Trennungsgeld und Umzugskosten werden nach den gesetzlichen Vorschriften<br />
gewährt.<br />
Schwerbehinderte werden bei gleicher Eignung bevorzugt berücksichtigt.<br />
Das Paul-Ehrlich-Institut fördert die Gleichstellung von Frauen und Männern<br />
und ist daher an Bewerbungen von Frauen interessiert.<br />
Fachliche Auskünfte erteilen Ihnen gerne Herr Dr. Thomas Hinz<br />
(06103/77-5001) und Herr Sven Flindt (06103/77-5002).<br />
Bitte richten Sie Ihre vollständigen Bewerbungsunterlagen an das<br />
Personalreferat des Paul-Ehrlich-Instituts.<br />
LABORWElT 9. Jahrgang | Nr. 3/2008 | 63
Service Stellenmarkt<br />
Am Zentrum für Innere<br />
Medizin, Medizinische<br />
Klinik und Poliklinik II,<br />
Fachbereich Medizin,<br />
sind zum nächstmöglichen Zeitpunkt, zunächst auf zwei Jahre befristet, zwei<br />
Ganztagsstellen einer/eines<br />
Technischen Assistentin/Assistenten<br />
(BTA, CTA, VTA, MTA, MTLA)<br />
zu besetzen. Bei Erfüllung der tarifrechtlichen Voraussetzungen erfolgt die<br />
Eingruppierung bis BAT Vb.<br />
Aufgaben:<br />
Die Arbeitsgebiete sind sowohl in der Klinischen Forschergruppe 118 „Pathomechanismen<br />
und Therapie der Lungenfibrose“, dem von der Europäischen<br />
Kommission im 7. Rahmenprogramm geförderten Forschungsprojekt „European<br />
IPF Network: Natural course, Pathomechanisms and Novel Treatment Options in<br />
Idiopathic Pulmonary Fibrosis“ und in dem Exzellenz-Cluster „Cardiopulmonary<br />
System“ angesiedelt. Inhaltlich ist die Tätigkeit an der Aufdeckung wesentlicher<br />
Pathomechanismen der Entstehung fibrosierender Lungenerkrankungen ausgerichtet.<br />
Der vielfältige und äußerst interessante Tätigkeitsbereich innerhalb<br />
unserer Forschungslabore umfasst zellphysiologische, histologische, biochemische,<br />
molekularbiologische sowie tierexperimentelle Methoden.<br />
• Ein Schwerpunkt liegt in der Betreuung und Durchführung tierexperimenteller<br />
Versuchs ansätze der akuten respiratorischen Insuffizienz und pulmonalen<br />
Fibrose, sowie der Durchführung lungenphysiologischer Messungen und der<br />
histologischen Aufarbeitung von Lungengewebe aus den tierexperimentellen<br />
Ansätzen.<br />
• Ein weiterer Schwerpunkt umfasst molekularbiologische Arbeiten: RNA/DNA<br />
Isolierung, (RT)-PCR, Genotypisierungen, Laser-gestütztes „cell picking“ aus<br />
Gefrierschnitten und Nachweis der Expression von Surfactant, Gerinnungs- und<br />
Fibrinolysekomponenten mittels TaqMan real-time PCR, Vektor-Klonierungen,<br />
Sequenzierungen zur Überprüfung der korrekten Insertion sowie Transfektionen<br />
(in vitro und in vivo) unter Verwendung verschiedener Transfektionsverfahren.<br />
Biochemische Analysen umfassen SDS-PAGE, Western Blotting, ELISA, Enzymographien,<br />
Aktivitäts- und Gerinnungsassays.<br />
Voraussetzungen:<br />
Erwünscht sind eine abgeschlossene Ausbildung zur/zum BTA, CTA, MTA,<br />
MTLA, VTA oder eine ähnliche Ausbildung. Die Bereitschaft zur Mitarbeit in<br />
einem jungen engagierten Team sowie das Interesse, sich in wissenschaftliche<br />
Aufgabenbereiche einzuarbeiten. Aufgrund der ständig wechselnden und breit<br />
gefächerten Versuchsansätze ist ein hohes Maß an Flexibilität und Selbständigkeit<br />
erforderlich. Zumindest für eine der beiden Positionen sind weitreichende<br />
Erfahrungen auf tierexperimenteller Ebene, die zur selbstständigen Durchführung<br />
von physiologischen Untersuchungen befähigen, ausdrücklich erwünscht. Für<br />
die andere Position sind fundierte molekularbiologische Kenntnisse vorteilhaft.<br />
Eine kompetente Einarbeitung durch unsere erfahrenen Mitarbeiter ist für uns<br />
selbstverständlich.<br />
Ihre Bewerbung richten Sie bitte mit den üblichen Unterlagen an:<br />
Herrn Prof. Dr. Andreas Günther, Medizinische Klinik II,<br />
Klinikstraße 36, 35392 Gießen<br />
E-Mail: Andreas.Guenther@innere.med.uni-giessen.de.<br />
Bewerbungen Schwerbehinderter werden - bei gleicher Eignung - bevorzugt.<br />
Wir bitten, Bewerbungen nur in Kopie vorzulegen, da diese nach<br />
Abschluss des Verfahrens nicht zurückgesandt werden.<br />
Am Universitätsklinikum Tübingen, Abteilung für Molekulare Pathologie, Ärztlicher<br />
Direktor Prof. Dr. med. R. Kandolf ist im Rahmen eines DFG geförderten<br />
Projektes des SFB-Transregio 19 „Inflammatorische Kardiomyopathie, Molekulare<br />
Pathogenese und Therapie“ zunächst für die Dauer von 4 Jahren ab 1.<br />
Juli 2008 eine Stelle zu besetzen:<br />
Wissenschaftliche/n Mitarbeiter/in (E13/14)<br />
Schwerpunkt des Projektes ist die Evaluierung von Pathogenitätsdeterminanten<br />
im Rahmen kardialer Remodellierungsprozesse im Mausmodell der Coxsakkievirus<br />
B3-Myokarditis (J Mol Med. 2008, 86:49-60) u.a. durch Anwendung<br />
von siRNA-Strategien. Zudem unterliegt dem Mitarbeiter die Koordinierung<br />
der Kooperationen innerhalb des SFB-TR19 bezüglich der Charakterisierung<br />
pathobiochemischer/immunologischer Prozesse (z.B Signaltransduktion) in<br />
verschiedenen Modellsystemen der Virusmyokarditis. Das Projekt wird von<br />
einer MTA unterstützt.<br />
Qualifikation:<br />
abgeschlossenes Hochschulstudium und Promotion (Biologie, Biochemie,<br />
Medizin) und fundierte Kenntnisse in: Molekularer <strong>Zellbiologie</strong>, Immunologie<br />
und/oder Virologie/Pathologie. Gute Englischkenntnisse.<br />
Ihre aussagekräftige Bewerbung mit Lebenslauf und wissenschaftlichem<br />
Werdegang richten Sie bitte an: Prof. Dr. med. Karin Klingel<br />
Tel.: 07071 29 84925, E-mail: karin.klingel@med.uni-tuebingen.de<br />
Abteilung für Molekulare Pathologie, Liebermeisterstr. 8, 72076 Tübingen<br />
http://www.sfb-transregio-19.de/<br />
http://www.medizin.uni-tuebingen.de/mol-pathologie/<br />
Am Institut für Biophysik der Technisch-<br />
Naturwissenschaftlichen Fakultät der<br />
Universität Linz, Österreich ist die Stelle<br />
eines/r<br />
wissenschaftlichen Mitarbeiters/in<br />
mit Doktorat im Forschungs- und Lehrbetrieb gemäß § 94 Abs. 2 Z 2 UG 2002<br />
nach dem Angestelltengesetz im vollen Beschäftigungsausmaß für 4 Jahre<br />
zu besetzen.<br />
Molekularbiologen, Biochemiker, Biophysiker, Biologen<br />
Erfordernisse:<br />
abgeschlossenes Studium der Molekularbiologie, Biologie, Biochemie oder<br />
Biophysik.<br />
Sonstige Kenntnisse:<br />
Erfahrung in der Führung von Zellkulturen und Überexpression/ Reinigung von<br />
Proteinen aus E.coli und Hefe erwünscht.<br />
Mitarbeit in Forschung und Lehre und Administration wird erwartet.<br />
Nähere Auskünfte erteilt Univ. Prof. Dr. Peter Pohl,<br />
Tel.: +43-732-2468-9269.<br />
Johannes Kepler Universität Linz, Institut für Biophysik<br />
Altenbergerstraße 69, A-4040 Linz<br />
peter.pohl@jku.at<br />
64 | 9. Jahrgang | Nr. 3/2008 LABORWElT
Curacyte Discovery GmbH ist ein Tochterunternehmen der<br />
Curacyte AG und hat ihren Sitz in der Bio City in Leipzig, einem<br />
Biotechnologie-Biomedizin Zentrum, das Industrie, Wissenschaft<br />
und Forschung kombiniert. Curacyte beschäftigt sich<br />
mit der Entdeckung und Entwicklung neuer Medikamente in<br />
Bereichen akuter und kritischer Erkrankungen.<br />
Curacyte Discovery sucht eine/n<br />
Leiter/in der Chemie<br />
in geschäftsführender Funktion<br />
Der Arbeitsort ist Leipzig. Ein multidisziplinäres Team von ca. 25<br />
Mitarbeitern (Chemikern, Biologen und technisches Personal)<br />
wird an diese Funktion berichten.<br />
Bewerber/innen sollten über eine abgeschlossene Hochschulausbildung<br />
und Promotion in Chemie oder Biochemie, sowie<br />
eine mehrjährige Industrieerfahrung verfügen.<br />
Industrieerfahrung und spezielles Know-How sollte einschließen:<br />
• Organische und Peptid-Synthese<br />
• Computergestützte Chemie und molekulares Modelling<br />
• Quantitative Struktur-Aktivitäts-Beziehung (QSAR)<br />
• Medizinal Chemie<br />
• Verbinden biologischer Charakterisierung mit der<br />
chemischen Optimierung von Wirksubstanzen<br />
Neben starken Führungsqualitäten und guten Englisch-<br />
Kenntnissen wird von dem/der Kandidaten/in erwartet, den<br />
Forschungsbetrieb durch stringentes und ergebnisorientiertes<br />
Projektmanagement zu leiten, sowie die interdisziplinäre<br />
Kommunikation im Team zu fördern.<br />
Bitte senden Sie Ihren Lebenslauf mit Foto, Zeugnissen sowie<br />
Angaben hinsichtlich eines möglichen Beginns Ihrer neuen<br />
Tätigkeit und Gehaltserwartungen an:<br />
Curacyte Discovery GmbH<br />
Frau Daniela Pohlmann<br />
Deutscher Platz 5d, 04103 Leipzig<br />
e-mail: daniela.pohlmann@curacyte.com<br />
Service Stellenmarkt<br />
Guava Technologies ist der führende Hersteller kapillarer Flowzytometer für die Biotech-<br />
und Pharmaindustrie. Für den Ausbau unseres Direktvertriebs suchen wir zum<br />
baldmöglichsten Termin einen<br />
Vertriebsrepräsentant für Nord-Ostdeutschland<br />
als Mitglied eines jungen und dynamischen Vertriebsteams mit<br />
e nachgewiesener Erfahrung und Erfolg im Vertrieb erklärungsbedürftiger Investitionsgüter<br />
e wissenschaftlicher Ausbildung in Zellbilogie/Molekularbiologie<br />
HOCHSCHULE BIBERACH<br />
HOCHSCHULE BIBERACH<br />
BIBERACH HOCHSCHULE UNIVERSITY OF APPLIED SCIENCES<br />
BIBERACH UNIVERSITY OF APPLIED SCIENCES<br />
HOCHSCHULE<br />
BIBERACH<br />
BIBERACH UNIVERSITY OF APPLIED BIBERACH<br />
SCIENCES<br />
An der Hochschule Biberach sind in der Fakultät Pharma-<br />
BIBERACH An zeutische der Hochschule Biotechnologie UNIVERSITY Biberach zum OF sind 01.09.2008 APPLIED in der Fakultät SCIENCES Pharma-<br />
BIBERACH An<br />
zeutische<br />
der Hochschule UNIVERSITY folgende zwei<br />
Professorenstellen Biotechnologie<br />
Biberach OF APPLIED SCIENCES<br />
zu besetzen zum<br />
sind<br />
01.09.2008<br />
in der Fakultät<br />
folgende<br />
Pharma-<br />
An<br />
zwei<br />
An Professorenstellen<br />
zeutische der Hochschule Biotechnologie Biberach<br />
der Hochschule zu Biberach besetzen<br />
zum sind 01.09.2008 in der Fakultät folgende Pharma- zwei<br />
sind in der Fakultät Pharma-<br />
Professorenstellen zeutische Biotechnologie zu besetzen zum 01.09.2008 folgende zwei<br />
zeutische<br />
Professorenstellen W2 Biotechnologie -Professur<br />
zu besetzen<br />
zum 01.09.2008 folgende zwei<br />
Professorenstellen zu besetzen<br />
W2 -Professur<br />
(Kennziffer W2 PBT 08)<br />
(Kennziffer W2<br />
-Professur<br />
-Professur PBT 08)<br />
(Kennziffer PBT 08)<br />
(Kennziffer PBT 08)<br />
für das Lehrgebiet Entwicklung und Steuerung biotechnologischer<br />
für das Lehrgebiet Entwicklung und Steuerung biotechnologischer<br />
für<br />
Produktionsprozesse.<br />
Produktionsprozesse.<br />
das Lehrgebiet (Kennziffer Entwicklung<br />
Diese Professur<br />
Diese Professur<br />
und PBT Steuerung<br />
wird i. d. 08) R.<br />
wird i. d. R.<br />
biotechnologischer<br />
zunächst drei Jahre<br />
für auf zunächst drei Jahre<br />
auf<br />
Produktionsprozesse. das Probe Lehrgebiet wahrgenommen Entwicklung<br />
Probe wahrgenommen<br />
Diese<br />
und<br />
und<br />
Professur<br />
bei und entsprechender Steuerung<br />
bei entsprechender<br />
wird i. d. R. biotechnologischer<br />
Eignung<br />
Eignung<br />
zunächst<br />
und<br />
und<br />
drei<br />
VorlieVorlie-<br />
Jahre<br />
für gen das der Lehrgebiet weiteren Voraussetzungen Entwicklung und anschließend Steuerung entfristet. biotechnologischer<br />
gen<br />
auf Produktionsprozesse. Probe<br />
der weiteren<br />
wahrgenommen Diese<br />
Voraussetzungen<br />
und Professur bei<br />
anschließend<br />
entsprechender wird i. d. R.<br />
entfristet.<br />
Eignung zunächst und drei Vorlie- Jahre<br />
Produktionsprozesse. Diese Professur wird i. d. R. zunächst drei Jahre<br />
Bewerber/innen gen auf Probe der weiteren wahrgenommen sollten Voraussetzungen über und einen bei überdurchschnittlichen anschließend entsprechender entfristet. Eignung Hochschulab- und Vorlie-<br />
Bewerber/innen<br />
auf<br />
schlussgen Probe<br />
der und weiteren<br />
wahrgenommen<br />
eine sollten Voraussetzungen Promotion über<br />
und<br />
einen<br />
bei<br />
sowie überdurchschnittlichen über anschließend<br />
entsprechender<br />
mehrjährige entfristet.<br />
Eignung<br />
einschlägige Hochschulab-<br />
und VorlieErfahschlussgen<br />
Bewerber/innen der<br />
rungen in und<br />
weiteren<br />
der eine<br />
sollten Voraussetzungen<br />
Bioprozesstechnik Promotion<br />
über einen<br />
sowie<br />
überdurchschnittlichen<br />
für über<br />
anschließend<br />
eukaryontische mehrjährige<br />
entfristet.<br />
Zellen einschlägige<br />
Hochschulab-<br />
verfügen. Erfahrungenschluss<br />
Bewerber/innen<br />
Bewerber/innen in<br />
und<br />
der<br />
eine sollten<br />
Bioprozesstechnik<br />
Promotion über einen sowie überdurchschnittlichen<br />
sollten über einen für<br />
über<br />
überdurchschnittlichen eukaryontische<br />
mehrjährige<br />
Zellen<br />
einschlägige HochschulabHochschulab-<br />
verfügen.<br />
Erfah-<br />
Die/der rungenschluss in und zukünftige der eine Bioprozesstechnik Promotion Stelleninhaber/in sowie für über eukaryontische soll mehrjährige Vorlesungen Zellen einschlägige und verfügen. praktische Erfah-<br />
Die/der<br />
schluss und<br />
Übungen rungen in zukünftige<br />
eine Promotion<br />
in der den Bioprozesstechnik Lehrgebieten Stelleninhaber/in<br />
sowie über<br />
der für Zellkulturtechnik, eukaryontische soll<br />
mehrjährige<br />
Vorlesungen Zellen<br />
einschlägige<br />
Bioprozessentwick-<br />
und verfügen. praktische<br />
Erfah-<br />
Übungen<br />
rungen Die/der in zukünftige<br />
lung und in der<br />
verwandten den<br />
Bioprozesstechnik<br />
Lehrgebieten<br />
Stelleninhaber/in<br />
Disziplinen der<br />
für<br />
Zellkulturtechnik,<br />
eukaryontische soll Vorlesungen Zellen<br />
übernehmen. Bioprozessentwick-<br />
und verfügen. praktische<br />
lung<br />
Übungen Die/der zukünftige<br />
Die/der und zukünftige in<br />
in<br />
verwandten<br />
den Lehrgebieten Stelleninhaber/in<br />
Stelleninhaber/in Disziplinen<br />
der Zellkulturtechnik, soll Vorlesungen<br />
übernehmen.<br />
Bioprozessentwick-<br />
und praktische<br />
soll Vorlesungen und praktische<br />
lung Übungen und in in verwandten den Lehrgebieten Disziplinen der Zellkulturtechnik, übernehmen. Bioprozessentwick-<br />
Übungen<br />
lung und in<br />
in<br />
verwandten<br />
den Lehrgebieten W2 Disziplinen -Professur<br />
der Zellkulturtechnik,<br />
übernehmen.<br />
Bioprozessentwicklung<br />
und in verwandten Disziplinen übernehmen.<br />
W2 -Professur<br />
(Kennziffer W2 PBT 07)<br />
(Kennziffer W2<br />
-Professur<br />
-Professur PBT 07)<br />
(Kennziffer PBT 07)<br />
(Kennziffer PBT 07)<br />
für das Lehrgebiet Physik, Mathematik und Statistik. Diese Professur<br />
für ist das Lehrgebiet Physik, Mathematik und Statistik. Diese Professur<br />
ist<br />
für<br />
zunächst<br />
zunächst<br />
das Lehrgebiet<br />
bis 31.12.2015<br />
bis 31.12.2015<br />
Physik, (Kennziffer Mathematik<br />
auf Probe PBT /Zeit<br />
auf Probe /Zeit<br />
und Statistik.<br />
geplant. 07) Die<br />
geplant. Die<br />
Diese<br />
Vollzeitstelle<br />
Vollzeitstelle<br />
Professur<br />
kann<br />
kann<br />
ist für zunächst das grundsätzlich Lehrgebiet<br />
grundsätzlich<br />
bis 31.12.2015 Physik, auch in Teilzeit Mathematik<br />
auch in Teilzeit<br />
auf Probe<br />
besetzt<br />
besetzt<br />
/Zeit und werden. Statistik.<br />
werden.<br />
geplant. Die Diese Vollzeitstelle Professur<br />
für das Lehrgebiet Physik, Mathematik und Statistik. Diese Professur<br />
Bewerber/innen kann ist zunächst grundsätzlich bis sollten 31.12.2015 auch über in Teilzeit einen auf Probe überdurchschnittlichen besetzt /Zeit werden. geplant. Die Hochschulab-<br />
Vollzeitstelle<br />
Bewerber/innen<br />
ist<br />
schluss kann<br />
zunächst<br />
grundsätzlich<br />
bis<br />
sollten<br />
31.12.2015<br />
und eine Promotion auch über in Teilzeit einen<br />
auf Probe<br />
sowie überdurchschnittlichen besetzt<br />
/Zeit<br />
über mehrjährige werden.<br />
geplant. Die<br />
Hochschulab-<br />
Vollzeitstelle<br />
einschlägige Erfahschluss<br />
kann Bewerber/innen grundsätzlich<br />
und eine<br />
sollten<br />
Promotion<br />
auch über in Teilzeit einen<br />
sowie<br />
überdurchschnittlichen besetzt<br />
über mehrjährige<br />
werden.<br />
einschlägige<br />
HochschulabrungenErfahrungenschluss<br />
Bewerber/innen in<br />
in<br />
und<br />
einem<br />
einem<br />
eine sollten angewandten<br />
angewandten<br />
Promotion über einen sowie<br />
Bereich überdurchschnittlichen<br />
Bereich<br />
über<br />
der<br />
der<br />
mehrjährige<br />
oben genannten<br />
oben genannten<br />
einschlägige HochschulabFachrichtunFachrichtunErfahgen<br />
Bewerber/innen verfügen. sollten über einen überdurchschnittlichen Hochschulabgenrungenschluss<br />
verfügen.<br />
in und einem eine angewandten Promotion sowie Bereich über der mehrjährige oben genannten einschlägige FachrichtunErfahschluss und eine Promotion sowie über mehrjährige einschlägige Erfah-<br />
Die/der genrungen verfügen. in zukünftige einem angewandten Stelleninhaber/in Bereich soll der oben Vorlesungen genannten und Fachrichtun- praktische<br />
Die/der<br />
rungen<br />
Übungen gen verfügen.<br />
in<br />
zukünftige<br />
einem angewandten<br />
Stelleninhaber/in<br />
Bereich<br />
soll<br />
der oben<br />
Vorlesungen<br />
genannten<br />
und<br />
Fachrichtun-<br />
in den Lehrgebieten der Physik, Mathematik, Statistik praktische sowie in<br />
Übungen<br />
gen Die/der verfügen. zukünftige<br />
den Vertiefungsfächern in den Lehrgebieten<br />
Stelleninhaber/in<br />
Bioinformatik der Physik,<br />
soll<br />
oder Mathematik,<br />
Vorlesungen<br />
verwandten Ingenieurwissen-<br />
Statistik<br />
und praktische<br />
sowie in<br />
den<br />
Übungen Die/der zukünftige<br />
schaften Vertiefungsfächern<br />
in den Lehrgebieten Stelleninhaber/in<br />
übernehmen. Bioinformatik<br />
der Physik, soll<br />
oder<br />
Mathematik, Vorlesungen<br />
verwandten Ingenieurwissen-<br />
Statistik und praktische sowie in<br />
Die/der zukünftige Stelleninhaber/in soll Vorlesungen und praktische<br />
schaften<br />
den Übungen Vertiefungsfächern in den Lehrgebieten<br />
übernehmen.<br />
Bioinformatik der Physik, oder Mathematik, verwandten Ingenieurwissen-<br />
Statistik sowie in<br />
Übungen in den Lehrgebieten der Physik, Mathematik, Statistik sowie in<br />
Beide schaften den Vertiefungsfächern Professuren übernehmen. sind Bioinformatik für den Studiengang oder verwandten Pharmazeutische IngenieurwissenBiotech- Beide<br />
den<br />
nologieschaften Vertiefungsfächern<br />
Professuren in übernehmen.<br />
der gleichnamigen sind<br />
Bioinformatik<br />
für den Fakultät Studiengang<br />
oder verwandten<br />
vorgesehen, Pharmazeutische<br />
Ingenieurwissen-<br />
den die Hochschule Biotechnologieschaften<br />
Beide Professuren<br />
im Wintersemester in<br />
übernehmen.<br />
der gleichnamigen<br />
sind für den<br />
2006/2007 Fakultät<br />
Studiengang<br />
in ihr Studienprogramm vorgesehen,<br />
Pharmazeutische<br />
den die aufgenommen<br />
Hochschule<br />
Biotech-<br />
im<br />
nologie Beide Professuren<br />
hat. Wintersemester<br />
in der gleichnamigen sind für den<br />
Eine detaillierte 2006/2007<br />
Fakultät Studiengang<br />
Beschreibung in ihr des Studienprogramm<br />
vorgesehen, Pharmazeutische den die<br />
jeweiligen Lehrgebiets aufgenommen<br />
Hochschule Biotech-<br />
Beide Professuren sind für den Studiengang Pharmazeutische Biotech- erhalten<br />
hat.<br />
im nologie Wintersemester in der gleichnamigen<br />
Sie vom Eine Dekan, detaillierte<br />
2006/2007 Fakultät<br />
Herrn Beschreibung<br />
in ihr<br />
Prof. Dr. Hannemann, des<br />
Studienprogramm vorgesehen, den die<br />
jeweiligen unter Tel. Lehrgebiets<br />
aufgenommen<br />
Hochschule<br />
nologie in der gleichnamigen Fakultät vorgesehen, den 0 73 die 51/582-450.<br />
Hochschule erhalten<br />
Sie<br />
hat. im Wintersemester<br />
vom<br />
Eine<br />
Dekan,<br />
detaillierte 2006/2007<br />
im Wintersemester Herrn<br />
Beschreibung in ihr<br />
2006/2007 Prof. Dr. Hannemann,<br />
des Studienprogramm jeweiligen<br />
in ihr Studienprogramm unter Tel.<br />
Lehrgebiets aufgenommen<br />
0 73aufgenommen 51/582-450.<br />
erhalten<br />
Es Sie hat. wird vom Eine vorausgesetzt, Dekan, detaillierte Herrn Beschreibung Prof. dass Dr. die/der Hannemann, des zukünftige jeweiligen unter Stelleninhaber/in Tel. Lehrgebiets 0 73 51/582-450. erhalten beim<br />
Es<br />
hat.<br />
Auf- Sie wird vom<br />
Eine<br />
und vorausgesetzt, Dekan,<br />
detaillierte Beschreibung<br />
Ausbau Herrn sowie Prof. bei dass der Dr. die/der<br />
des<br />
Fortentwicklung Hannemann, zukünftige<br />
jeweiligen<br />
unter des Stelleninhaber/in Tel.<br />
Lehrgebiets erhalten<br />
Studiengangs 0 73 51/582-450. Phar- beim<br />
Auf-<br />
Sie Es wird vom<br />
mazeutische und<br />
vorausgesetzt, Dekan,<br />
Ausbau<br />
Herrn<br />
Biotechnologie sowie<br />
Prof.<br />
bei<br />
dass<br />
der<br />
Dr. die/der<br />
tatkräftig Fortentwicklung<br />
Hannemann, zukünftige unter<br />
mitarbeitet. des<br />
Stelleninhaber/in Tel.<br />
Dazu Studiengangs<br />
0 73 51/582-450.<br />
gehören Phar-<br />
beim<br />
auch<br />
die mazeutische<br />
Auf- Es wird und vorausgesetzt, Ausbau<br />
Es wird Konzeption vorausgesetzt, Biotechnologie<br />
sowie bei dass der die/der<br />
und Übernahme dass tatkräftig<br />
Fortentwicklung zukünftige<br />
die/der von zukünftige mitarbeitet.<br />
des Stelleninhaber/in<br />
Lehrveranstaltungen Stelleninhaber/in Dazu<br />
Studiengangs<br />
gehören<br />
Phar- beim<br />
aus auch beim dem<br />
die<br />
mazeutische Auf- und Ausbau<br />
Bereich Auf- Konzeption<br />
Biotechnologie sowie bei der<br />
und anderer Ausbau Professorenstellen. und sowie Übernahme<br />
tatkräftig Fortentwicklung<br />
bei der Fortentwicklung von<br />
mitarbeitet. des<br />
Darüber Lehrveranstaltungen<br />
Dazu Studiengangs gehören Phar-<br />
hinaus des Studiengangs wird die aktive aus<br />
auch<br />
Phar- dem Mit-<br />
Bereich<br />
die mazeutische Konzeption Biotechnologie<br />
arbeitmazeutische in anderer der Selbstverwaltung Biotechnologie Professorenstellen.<br />
und Übernahme tatkräftig von mitarbeitet.<br />
tatkräftig und in Darüber<br />
Lehrveranstaltungen Dazu gehören<br />
Gremien mitarbeitet. hinaus erwartet. Dazu wird die gehören aktive<br />
aus auch dem<br />
auch Mitarbeit<br />
Bereich die Konzeption<br />
die Konzeption in<br />
anderer<br />
der Selbstverwaltung<br />
Professorenstellen. und Übernahme von<br />
und Übernahme und in<br />
Darüber Lehrveranstaltungen<br />
von Gremien<br />
hinaus<br />
Lehrveranstaltungen erwartet.<br />
wird die aktive aus dem Mit-<br />
aus dem<br />
arbeit Bereich Weitere in allgemeine anderer der Selbstverwaltung Professorenstellen. Informationen und zur in Dienstaufgabe, Darüber Gremien hinaus erwartet. den wird Bewerbungs- die aktive Mit- und<br />
Bereich<br />
arbeit Einstellungsvoraussetzungen Weitere in allgemeine<br />
anderer<br />
der Selbstverwaltung<br />
Professorenstellen.<br />
Informationen sowie und zur die in Dienstaufgabe,<br />
Darüber<br />
Gremien<br />
hinaus<br />
detaillierten erwartet. Stellenausschreibungen<br />
den<br />
wird<br />
Bewerbungsdie<br />
aktive Mit-<br />
und<br />
arbeit<br />
Einstellungsvoraussetzungen<br />
Weitere in allgemeine der Selbstverwaltung Informationen<br />
finden Sie unter http://www.fh-biberach.de/service/Stellenanzeigen.<br />
sowie<br />
und zur<br />
die<br />
in Dienstaufgabe, Gremien<br />
detaillierten<br />
erwartet.<br />
Stellenausschreibungen<br />
den Bewerbungs- und<br />
finden<br />
Einstellungsvoraussetzungen Weitere allgemeine Informationen<br />
Weitere Sie allgemeine unter http://www.fh-biberach.de/service/Stellenanzeigen.<br />
sowie zur die Dienstaufgabe, detaillierten Stellenausschreibungen<br />
den Bewerbungs- und<br />
Informationen zur Dienstaufgabe, den Bewerbungs- und<br />
finden Einstellungsvoraussetzungen Sie unter http://www.fh-biberach.de/service/Stellenanzeigen.<br />
sowie die detaillierten Stellenausschreibungen<br />
Einstellungsvoraussetzungen<br />
finden Sie unter http://www.fh-biberach.de/service/Stellenanzeigen.<br />
sowie die detaillierten Stellenausschreibungen<br />
Bewerbungen finden Sie unter mit http://www.fh-biberach.de/service/Stellenanzeigen.<br />
den üblichen Unterlagen werden unter Angabe der<br />
Bewerbungen Kennziffer bis 13.06.2008 mit den üblichen erbeten Unterlagen an die Personalabteilung werden unter Angabe der Hoch- der<br />
Kennziffer<br />
Bewerbungen<br />
schule Biberach, bis 13.06.2008<br />
mit den üblichen<br />
Karlstr. erbeten<br />
Unterlagen<br />
11, 88400 an Biberach, die Personalabteilung<br />
werden unter Angabe<br />
Tel. 0 73 51/5der 82-120. Hoch-<br />
der<br />
Kennziffer Bewerbungen<br />
Bewerbungen schule Biberach,<br />
bis 13.06.2008 mit den üblichen<br />
mit Karlstr.<br />
erbeten Unterlagen<br />
den üblichen 11, 88400<br />
an<br />
Unterlagen Biberach,<br />
die Personalabteilung werden unter Angabe<br />
werden Tel. 0unter 73 51/5<br />
der<br />
Angabe 82-120.<br />
Hoch- der<br />
Kennziffer<br />
der<br />
schule Biberach, bis 13.06.2008 Karlstr. erbeten 11, 88400 an Biberach, die Personalabteilung Tel. 0 73 51/5der 82-120. Hoch-<br />
Kennziffer<br />
schule Biberach,<br />
bis 13.06.2008<br />
Karlstr.<br />
erbeten<br />
11, 88400<br />
an<br />
Biberach,<br />
die Personalabteilung<br />
Tel. 0 73 51/5<br />
der<br />
82-120.<br />
Hochschule<br />
Biberach, Karlstr. 11, 88400 Biberach, Tel. 0 73 51/5 82-120.<br />
Bitte richten Sie Ihre Bewerbung gerne per E-Mail an:<br />
Dr. Michael Liebler • Guava Technologies • Kanalstrasse 19/1 • 72631 Aichtal • mliebler@guavatechnologies.com • www.guavatechnologies.com<br />
LABORWElT 9. Jahrgang | Nr. 3/2008 | 65
Service Produktwelt<br />
Olympus Deutschland GmbH<br />
High-Content-Screening<br />
für die Forschung<br />
scan® ist eine modulare, Mikroskop-basierte<br />
<strong>Imaging</strong>-Plattform, die Olympus in Zusammenarbeit<br />
mit dem EMBL (Heidelberg) speziell<br />
für die vollautomatische Bilderfassung und die<br />
Datenanalyse biologischer Proben entwickelt<br />
hat. scan® unterstützt diverse Probenformate,<br />
wie Multiwell-Platten, Objektträger oder Cus-<br />
tom-Arrays. Der modulare Aufbau des Systems<br />
ermöglicht einen flexiblen Einsatz sowohl für<br />
Routinezwecke als auch in hochentwickelten<br />
Applikationen, zum Beispiel in der Assay-Entwicklung<br />
und dem High-Content-Screening.<br />
Ein leistungsstarkes Analysemodul ermöglicht<br />
komplexe Bildanalysen und anspruchsvolle<br />
Datenauswertungen; unterschiedliche<br />
Partikel- und Objekterkennungsfunktionen<br />
können miteinander kombiniert werden und<br />
steigern dadurch die Effizienz der Bildanalyse.<br />
Anschließend werden die zuvor detektierten<br />
Objekte einer eingehenden zytometrischen<br />
Analyse unterzogen. Dabei können komplexe<br />
Entwürfe für Multiparameter-Datenanalysen<br />
problemlos durch Wahl von Ausschluss- und<br />
Klassifikationsparametern erstellt werden.<br />
Olympus Deutschland GmbH<br />
Andrea Rackow<br />
Wendenstr. 14-18<br />
20097 Hamburg<br />
Tel.: +49-(0)40-23773-4612<br />
Fax: +49-(0)40-230817<br />
mikroskopie@olympus.de<br />
SBMC 2008<br />
Nachwuchspreise<br />
Als besonderes Highlight der diesjährigen Systembiologie-Konferenz<br />
SBMC (Dresden, 22.–24<br />
Mai 2008) vergibt die MTZ-Stiftung, gemeinsam<br />
mit dem Bundesforschungsministerium und<br />
dem Projektträger Jülich erstmals den mit 5.000<br />
Euro dotierten „MTZ-Award for medicallyoriented<br />
Systems Biology“. Die Auszeichnung<br />
wird am zweiten Veranstaltungstag an drei<br />
junge Wissenschaftler für ihre herausragenden<br />
Promotionen verliehen. Der Preis soll künftig<br />
in jährlichem Turnus vergeben werden, um<br />
talentierten Systembiologen mehr öffentliche<br />
Aufmerksamkeit zuteil werden zu lassen.<br />
evocatal GmbH<br />
Fluoreszenz-Markierung von Biomolekülen<br />
evoglow®, so heißt eine neue Art fluoreszierender<br />
Proteine (FPs), die – im Gegensatz<br />
zu den bekannten Leuchtproteinen wie GFP<br />
und davon abgeleiteten Fluorophoren – auch<br />
unter anaeroben Bedingungen in vivo leuchten.<br />
Die von der Firma evocatal entwickelten<br />
Leuchtproteine eignen sich somit auch für<br />
Untersuchungen in Biofilmen, anaeroben<br />
Mikroorganismen oder Zellverbänden. Diese<br />
spielen in vielen biotechnologischen und<br />
biomedizinischen Prozessen eine zentrale<br />
Rolle, waren aber bislang nahezu unzugänglich<br />
für bildgebende Verfahren wie zum<br />
Beispiel die Fluoreszenzmikroskopie.<br />
Die evoglow®-Serie, die in Kürze auf den<br />
Markt kommen wird, eröffnet nunmehr<br />
neue Möglichkeiten auf diesem Gebiet,<br />
beispielsweise durch selektive Anfärbung<br />
einzelner Zellen oder Zellkompartimente<br />
oder aber durch den Einsatz in Expressionsstudien<br />
in Form von Transkriptions- oder<br />
Translationsfusionsproteinen.<br />
Ein weiterer Vorteil: Dadurch dass die<br />
Leuchtintensitäten sowie die Anregungs-<br />
und Emissionsspektren der evoglow®-<br />
Proteine im selben Bereich wie jene von<br />
GFP und seiner Varianten liegen, können<br />
Porvair Sciences Ltd.<br />
Untersuchung von Wirkstoffen in biologischen<br />
Matrices<br />
Porvair Sciences hat einen Anwendungsbericht<br />
veröffentlicht, der die operationalen<br />
Vorteile des Mikroplatten-basierten Microlute<br />
Festphasenextraktion (SPE)-Systems<br />
bei der Analyse pharmazeutischer Stoffe in<br />
biologischen Flüssigkeiten beleuchtet.Unter<br />
Verwendung von Microlute mit SPE-Wells<br />
mit geringer Sorbens-Menge wurden Analyte<br />
unterschiedlicher Wasserlöslichkeit untersucht<br />
– von fettlöslichen Steroiden bis zu wasserlöslichen<br />
Nukleosid-Analoga. Dabei zeigte sich,<br />
dass bei Probenaufarbeitung mit Microlute<br />
die SPE, das Priming, Waschen und die Elution<br />
signifikant eingegrenzt werden, woraus eine<br />
zur Detektion und Analyse vergleichbare<br />
instrumentelle Anordnungen verwendet<br />
werden.<br />
evocatal GmbH<br />
Dr. Michael Puls<br />
Merowingerplatz 1a<br />
40225 Düsseldorf<br />
Tel.: +49- (0) 211-1576095-2<br />
m.puls@evocatal.de<br />
schnellere Verarbeitung und gesteigerte Sensitivität<br />
resultierten.<br />
Die 96 wells von Microlute werden aus einem<br />
einzigen Formteil aus qualitativ hochwertigem<br />
Kunststoff konstruiert. Dadurch werden<br />
durch das Einstecken einzelner SPE-Kartuschen<br />
hervorgerufene Verformungen vermieden.<br />
Durch Einsatz eines unternehmenseigenen<br />
Verfahrens zum Beschicken mit schlammförmig<br />
vorliegendem Sorbens konnte Porvair Kanalisierungseffekte<br />
ausschalten, die häufig die<br />
Leistung der mit Trockenpulver beladenen SPE-<br />
Kolonnen begrenzen. Jede einzelne Vertiefung<br />
der Microlute-Platte wurde mit seiner eigenen<br />
Abflussrille versehen – die Gewähr für 100%igen<br />
Probentransfer und Null-Überkreuzkontamination.<br />
Microlute bietet eine hohe Produktivität<br />
auf allen robotergestützten Systemen zur Probenhandhabung<br />
und -bearbeitung.<br />
Porvair Sciences Ltd.<br />
Unit 6, Shepperton Business Park<br />
Govett Avenue<br />
Shepperton, Middlesex TW17 8BA UK<br />
Tel.: + 44-(0)1932-240255<br />
int.sales@porvair.com<br />
www.porvair-sciences.com<br />
66 | 9. Jahrgang | Nr. 3/2008 LABORWElT
PromoCell GmbH<br />
Fluoreszenz-Markierung von Biomolekülen<br />
PromoCell bietet eine große Auswahl qualitativ<br />
hochwertiger Fluoreszenzfarbstoffe,<br />
die den blau-grünen bis nahen Infrarot-Bereich<br />
des Lichtspektrums abdecken und zur<br />
schnellen, einfachen und effizienten Kopplung<br />
an Proteine, Nukleinsäuren und Antikörper<br />
geeignet sind. Die aktuell lieferbaren<br />
PromoFluor-Farbstoffe (z.B. PromoFluor-488,<br />
PromoFluor-555, PromoFluor-590, PromoFluor-647,<br />
PromoFluor-680, PromoFluor-700<br />
oder PromoFluor-750) sind kostengünstige<br />
und qualitativ hochwertige Alternativen<br />
zu etablierten Fluorophoren wie etwa den<br />
Alexa Fluo®- und Cy-Farbstoffen. Sie zeigen<br />
eine außergewöhnliche Photostabilität,<br />
hohe Fluoreszenzquantenausbeute, eine<br />
starke Absorption sowie gute Wasserlöslichkeit.<br />
Die PromoFluor-Farbstoffe sind als<br />
Carboxylsäuren, NHS-Ester und Maleimide<br />
sowie als Konjugate mit Aminogruppen,<br />
Biotin, Avidin, Streptavidin und Phalloidin<br />
verfügbar und für Immunfluoreszenz-,<br />
FISH-, FACS- sowie Microarray-Experimente<br />
geeignet.<br />
Gebrauchsfertige „PromoFluor Labeling-<br />
Kits“ zur Markierung von Proteinen und Antikörpern<br />
sowie mit den PromoFluor-Farbstoffen<br />
markierte Sekundär-Antikörper stehen<br />
ebenfalls zur Verfügung. Des weiteren bietet<br />
das Unternehmen R- und B-Phycoerythrin<br />
In Vitro Systems & Services GmbH<br />
Die Objektträgerflasche mit dem Klick – lumox<br />
slide flask<br />
Die Neuentwicklung von In Vitro Systems &<br />
Services ist Objektträger und Zellkulturflasche<br />
in einem. Die Basis dieses Zellkultursystems<br />
ist ein gasdurchlässiger Folienobjektträger<br />
mit sehr geringer Autofluoreszenz<br />
und hoher Transparenz.<br />
Aufgrund der hervorragenden optischen<br />
Eigenschaften der lumox Folie eignet sich<br />
die lumox slide flask besonders für zellba-<br />
sowie Allophycocyanin – ebenfalls als Streptavidin-<br />
und Biotin-Konjugate.<br />
PromoCell GmbH<br />
Sickingenstr. 63/65<br />
69126 Heidelberg<br />
Tel.: +49-(0)6221-649 34-0<br />
Fax: +49-(0)6221-64934-40<br />
info@promokine.de<br />
www.promokine.de<br />
sierte Fluoreszenz-Assays in der hochauflösenden<br />
Mikroskopie. Eine hohe Sensitivität<br />
bei Fluoreszenzmessungen ermöglicht eine<br />
hintergrundfreie Bilddarstellung.<br />
Die Gasdurchlässigkeit der lumox Folie<br />
(TC-Qualität) sorgt für optimalen O 2 - und<br />
CO 2 -Austausch und bietet allen Zelltypen<br />
eine adäquate Wachstumsfläche.<br />
Nach der Kultivierung kann der lumox<br />
Folienobjektträger von dem Flaschengehäuse<br />
abgezogen werden. Dadurch sind die<br />
Zellen für die mikroskopische Analyse leicht<br />
zugänglich.<br />
Weitere Varianten des objektträgerbasierten<br />
Zellkultursystems sind im 1, 2, 4 und<br />
8-well Format erhältlich.<br />
In Vitro Systems & Services GmbH<br />
Rudolf-Wissell-Str. 28<br />
37079 Göttingen<br />
Tel.: +49-(0)551-500-97-329<br />
Fax: +49-(0)551-500-97-311<br />
customerservice@ivss.de<br />
www.ivss.de<br />
Eppendorf AG<br />
Guava Technologies Inc.<br />
Service Produktwelt<br />
Nachweis aktivierter<br />
Transkriptionsfaktoren<br />
Zwei neue Kits der Eppendorf Biochip<br />
Systems GmbH weisen in einer einzigen<br />
Messung bis zu 12 aktivierte Transkriptionsfaktoren<br />
aus Kernextrakten nach.<br />
Dazu wird Doppelstrang-DNA mit jeweils<br />
spezifischen Bindungssequenzen für die<br />
Transkriptionsfaktoren auf eine beschichtete<br />
Glasoberfläche gespottet. Nach Inkubation<br />
mit einem Kernproteinextrakt<br />
werden mittels Antikörpern die gebundenen<br />
Transkriptionsfaktoren nachgewiesen, die<br />
spezifisch für den aktivierten Status sind.<br />
Die Detektion erfolgt mit dem Eppendorf<br />
Silverquant®-System oder mittels fluoreszierender<br />
Sekundär-Antikörper. Allerdings ist<br />
der Nachweis mit dem Silverquant®-System,<br />
der auf der Silbernitratreduktion mit Hilfe<br />
eines Anti-Biotin-Nanogold-Konjugates<br />
beruht, deutlich sensitiver als die etablieren<br />
Fluoreszenzfärbungen. Der TF Chip MAPK-<br />
Kit detektiert acht zentrale Transkriptionsfaktoren<br />
des MAPK-Pathways. Der TF Chip<br />
Stem Cell-Kit ermöglicht den Nachweis von<br />
12 aktivierten Transkriptionsfaktoren, die als<br />
Pluripotenz- und Differenzierungsmarker für<br />
Stammzellen fungieren.<br />
Eppendorf AG<br />
Bettina Doerler<br />
doerler.b@eppendorf.de<br />
www.eppendorf-biochip.com<br />
Informationsschrift<br />
Guava Technologies hat eine Broschüre zur<br />
firmeneigenen Laser-basierten, Mikrokapillartechnologie<br />
veröffentlicht, die deren<br />
Vorteile bei der Detektion von Säugerzellen,<br />
Bakterien und Mikroperlen darlegt. Neben<br />
zahlreichen Daten präsentiert das Unternehmen<br />
auch einen Vergleich mit anderen<br />
Systemen zur Zellzählung.<br />
Guava Technologies Inc. (Europe)<br />
Stamford, UK<br />
Tel.: +44-(0)1780-764390<br />
mtaylor@guavatechnologies.com<br />
www.guavatechnologies.com<br />
LABORWElT 9. Jahrgang | Nr. 3/2008 | 67
Service Produktwelt<br />
PE LAS GmbH<br />
ELISAs im Kit-Format<br />
PerkinElmer hat sein Angebot an Assays für das<br />
Wirkstoff-Screening um die neue AlphaLISA TM -<br />
Plattform erweitert. AlphaLISA TM basiert auf<br />
der ALPHAScreen TM -Technologie und erlaubt<br />
die Bestimmung kleinster Biomarker-Mengen<br />
sowie von biologischen Therapeutika. Die<br />
für das Hochdurchsatz-Screening geeignete<br />
Technologie bietet sich besonders für Marker<br />
an, für die keine kommerziellen Kits existieren<br />
und für komplexe Proben wie Serum<br />
oder Plasma, wie sie in präklinischen Studien<br />
verwendet werden. Die ab Mai verfügbaren<br />
ersten Analyte umfassen P24, Amyloid-beta<br />
40, Amyloid-beta 42, TNF-alpha, IgG, EPO, VEGF<br />
und Insulin. PerkinElmer bietet für AlphaLISA TM<br />
auch einen Assay-Entwicklungs Service an, um<br />
den Wechsel vom traditionellen ELISA-Format<br />
auf die neue Plattform zu erleichtern.<br />
PE LAS (Germany) GmbH<br />
Dr. Michael Lässle<br />
Ferdinand-Porsche-Ring 17<br />
63110 Rodgau<br />
Tel.: +49-(0)6198-587692<br />
Fax: +49-(0)6198-587561<br />
michael.lassle@perkinelmer.com<br />
Fujifilm Europe GmbH<br />
<strong>Imaging</strong>-System für<br />
Proteinanalysen<br />
Mit „Starion“ hat Fujifilm Life Science das erste<br />
Modell der neuesten Generation Laser-basierter<br />
Infrarot-<strong>Imaging</strong>-Systeme für Proteomics-<br />
Anwendungen vorgestellt. Der für das Western<br />
Blotting oder Multiplexed Western ausgestattete<br />
Laser-Scanner erlaubt durch Exzitationswel-<br />
lenlängen von 685nm und 785nm einen Einsatz<br />
von Fluoreszenzfarbstoffen, die im Nah-Infrarot-<br />
Bereich emittieren. Hohe Auflösung und ein dynamischer<br />
Bereich über fünf Größenordnungen<br />
tragen zur Leistungsfähigkeit des Systems bei.<br />
Mit einem weiteren Laser kann Starion auch als<br />
Phosphor-Imager eingesetzt werden.<br />
FUJIFILM Europe GmbH<br />
European Life Science Division<br />
Tel.: +49-(0)211-5089-144<br />
lifescience@fujifilm.de<br />
www.fujifilm.de/lifescience<br />
m2p-Labs GmbH<br />
Hochdurchsatz-Fermentation<br />
Forschen und Entwickeln wird leichter. Mit<br />
Hilfe des neuen Mikrofermentation-Systems<br />
BioLector können jetzt wesentlich mehr<br />
Fermentationen im Screening und in der<br />
frühen Bioprozessentwicklung automatisiert<br />
durchgeführt werden. Neben der einfachen<br />
Handhabung von Mikrotiterplatten kombiniert<br />
die Technologie Hochdurchsatz mit<br />
online-Monitoring aller essentiellen Fermentationsparameter<br />
unter verfahrenstechnisch<br />
definierten Bedingungen. Die nicht-invasive<br />
optische Messtechnik detektiert Biomasse-<br />
und Proteinkonzentrationen sowie pH und pO 2<br />
parallel in bis zu 96 Mikroreaktoren.<br />
ProBioGen AG<br />
Lymphknotenmodell zur in vitro-Testung<br />
Die ProBioGen AG hat mit dem künstlichen<br />
humanen Lymphknoten (human Artificial<br />
Lymph Node, „human ALN“) ein neues<br />
in vitro-Testsystem entwickelt, das die<br />
Vorhersage von Immunreaktionen auf biopharmazeutische<br />
Wirkstoffe ermöglicht.<br />
Mit Hilfe des ALN kann das Wirkungs- und<br />
Nebenwirkungsprofil eines humanen Pharmazeutikums<br />
vorhergesagt werden.<br />
Das komplexe Gewebemodell basiert auf<br />
humanen Blutzellen definierter Spender,<br />
die in einer dreidimensionalen Kulturmatrix<br />
immunkompetente Gewebestrukturen<br />
ausbilden, sogenannte Mikro-Organoide.<br />
Im Laufe der Kultivierung können Prozess-<br />
und immunologisch relevante Parameter<br />
,wie etwa Zytokinprofile, erfasst werden.<br />
Im Anschluss kann zudem die zelluläre Zusammensetzung<br />
der Organoide und ihre<br />
Differenzierung immunhistologisch sowie<br />
im Durchflusscytometer und via Genexpressionanalyse<br />
ermittelt werden. Dies erlaubt<br />
sowohl das immunogene Risiko als auch das<br />
Wirksamkeitspotential eines Medikamentes<br />
frühzeitig zu bestimmen.<br />
Modelle wie der von ProBioGen entwickelte<br />
humane ALN, liefern neue Einsichten<br />
in die Wirkweise eines Pharmazeutikums<br />
und können für die Evaluierung produktbezogener<br />
Risiken sowie für die Erstellung von<br />
Die hohe Informationsdichte der Daten<br />
garantiert eine bessere Beschreibung der<br />
zu untersuchenden biologischen Systeme<br />
und reduziert den Arbeitsaufwand spürbar.<br />
Erstmals ist es somit möglich, auf Basis<br />
kinetischer „real-time“-Daten, Fermentationen<br />
bereits im Mikromaßstab von 200µL<br />
bis 1000µL quantitativ zu beurteilen und<br />
den Prozess auf einer breiten Wissenbasis<br />
zu gestalten.<br />
Die Skalierbarkeit der Ergebnisse im Mikromaßstab<br />
auf größere Laborsysteme konnte für<br />
etablierte Expressionssysteme wie Bakterien<br />
und Hefen bereits gezeigt werden und ebnet<br />
somit den direkten Übertrag auf größere<br />
Reaktoreinheiten. Der BioLector findet Anwendung<br />
im Klonscreening, der Medien- und<br />
Prozessparameter-Optimierung sowie der<br />
Aufklärung von Wachstums- und Produktbildungskinetiken.<br />
m2p-labs GmbH<br />
Forckenbeckstr. 6<br />
52074 Aachen<br />
Tel.: +49-(0)241-608513121<br />
www.m2p-labs.com<br />
Wirkstoffprofilen herangezogen werden.<br />
Zusätzlich verhilft der ALN entscheidend<br />
die Anzahl notwendiger Tierversuche zu<br />
reduzieren.<br />
ProBioGen AG<br />
Dr. Gabriele Schneider<br />
Tel.: +49-(0)30-924006-0<br />
info@probiogen.de<br />
www.probiogen.de<br />
68 | 9. Jahrgang | Nr. 3/2008 LABORWElT
Mai bis Juli 2008<br />
Veranstaltungskalender<br />
19.-21.05.08<br />
1 st European Conference for Clinical Nanomedicine,<br />
Basel (CH)<br />
Info: Beat Löffler, European Foundation<br />
for Clinical Nanomedicine<br />
(Tel.: +41-61-695-9395,<br />
E-Mail: loeffler@clinam.org,<br />
Web: www.clinam.org)<br />
22.-23.05.08<br />
Biotech 2008 , Zürich (CH)<br />
Info: Birgit Camenisch, Universität Zürich<br />
(Tel.: +41-589-345-733,<br />
E-Mail: birgit.camenisch@zhaw.ch,<br />
Web: www.biotech2008.ch)<br />
22.-25.05.08<br />
Analysis of Microbial Cells at the Single-Cell<br />
Level, Bad Schandau<br />
Info: (Web: http://qbab.dbs.aber.ac.uk/sc2008)<br />
03.–04. Juni 2008<br />
European Downstream<br />
Technology Forum, Göttingen<br />
Um die neuesten Entwicklungen im Feld<br />
des Downstream Processing zu diskutieren,<br />
treffen sich Europas Top-Experten im Sartorius<br />
College in Göttingen. Von Disposables<br />
über Prozessintegration bis hin zu neuen<br />
Separationstechnologien reicht dabei<br />
die Themenpalette. Registrierung unter:<br />
www.sartorius-stedim.com/downstream<br />
23.-24.05.08<br />
4. Glycan-Forum, Berlin<br />
Info: Thomas Ruttkowski, P&R Kongresse GmbH<br />
(E-Mail: info@pr-kongresse.de,<br />
Web: www.glycostrukturfabrik.de)<br />
26.-30.05.08<br />
Modification of Large DNA Constructs<br />
like BACs, Cosmids and the E.coli Genome,<br />
Heidelberg<br />
Info: Gene Bridges GmbH<br />
(E-Mail: contact@genebridges.com,<br />
Web: www.genebridges.com)<br />
03.–04. Juni 2008<br />
mAb Workshop, Heidelberg<br />
Einen hervorragenden Überblick über<br />
Stand und Entwicklung beim Design und<br />
Engineering, der Analytik, Formulierung<br />
und Produktion sowie klinischen Anwendung<br />
monoklonaler Antikörper gibt der<br />
2. EUFEPS + EAPB Workshop zum Thema.<br />
Information: www.eufeps.org<br />
28.05.-01.06.08<br />
Crossing Frontiers in Biomaterials and<br />
Regenerative Medicine – WBC 2008,<br />
Amsterdam (NL)<br />
Info: European Society for Biomaterials (ESB)<br />
(E-Mail: wbc2008-info@ics-online.nl,<br />
Web: www.wbc2008.nl)<br />
28.05.08<br />
Fortschritte in der Laboratoriumsmedizin:<br />
Schwerpunkt Hämatologie, Berlin<br />
Info: Priv.-Doz. Dr. Andreas Lun, Charité<br />
(Fax: +49-30-8445-4152,<br />
E-Mail: andreas.lun@charite.de)<br />
03.-04.06.08<br />
2 nd EUFEPS and EAPB Workshop on<br />
Monoclonal Antibodies, Heidelberg<br />
Info: EUFEPS Secretariat, Stockholm<br />
(Tel.: +46-8-7235-000,<br />
Fax: +46-8-4113-217,<br />
E-Mail: conferences@eufeps.org,<br />
Web: www.eufeps.org)<br />
03.-04.06.08<br />
European Downstream Technology Forum<br />
2008, Göttingen<br />
Info: Beate Sommerfeld,<br />
Sartorius College Göttingen<br />
(Tel.: +49-551-308-3318,<br />
E-Mail: beate.sommerfeld@sartorius-stedim.com,<br />
Web: www.sartorius-stedim.com/downstream)<br />
Service Kalender<br />
06.06.08<br />
„Mikrosystemtechnik für Analytik und<br />
Diagnostik“, Mainz<br />
Info: mst-Netzwerk Rhein-Main e.V.<br />
(Web: www.mst-rhein-main.de)<br />
22.-26.06.08<br />
TERMIS-EU Meeting 2008, Porto (P)<br />
Info: Ariana Santos, 3B’s Research Group<br />
(E-Mail: termiseu2008@dep.uminho.pt,<br />
Web: www.termis.org/eu2008)<br />
24.-27.06.08<br />
1 st Global Conference on GMO Analysis,<br />
Como (IT)<br />
Info: European Commission/Institute for Health and<br />
Consumer Protection<br />
(E-Mail: gmo-global-conference@jrc.it,<br />
Web: http://gmoglobalconference.jrc.it)<br />
06.-09.07.08<br />
2 nd International Congress on Stem Cells<br />
and Tissue Formation 2008, Dresden<br />
Info: Hilke Marina Petersen, Center for Regenerative<br />
Therapies Dresden/SFB 655 Cells into Tissues<br />
(Web: www.stemcellcongress-dresden.de)<br />
07.-11.07.08<br />
qPCR-Workshop, Freising<br />
Info: Dr. Michael Pfaffl, Tataa Biocenter<br />
(Web: www.tataa.gene-quantification.info)<br />
09.-10.07.08<br />
Medizin Innovativ 2008, Nürnberg<br />
IInfo: Forum MedTech Pharma e.V.<br />
(Web: www.medizin-innovativ.de)<br />
9.–10. Juli 2008<br />
Medizin Innovativ, Nürnberg<br />
Mehr als 90 Aussteller und ein hochkarätiges<br />
Vortragsprogramm locken jetzt nach<br />
Nürnberg. Wer mehr über Biomateria lien,<br />
Diagnostik, Pharma-Biotech, Bildgebung<br />
etc. erfahren möchte, kann sich unter<br />
www.medizin-innovativ.de anmelden.<br />
LABORWElT 9. Jahrgang | Nr. 3/2008 | 69
Ausblick<br />
Endlich! Entscheidungen<br />
für die Forschung<br />
Thomas Gabrielczyk<br />
Gleich zwei richtungsweisende Entscheidungen haben Politiker im April getroffen, die das<br />
Forschungsabarometer in Deutschland abrupt auf „schön“ haben springen lassen. Die eine<br />
– zur Forschung an embryonalen Stammzellen – war mit kontroversen Diskussionen und viel<br />
Öffentlichkeit verbunden, die andere Weichenstellung – zum geplanten Gendiagnostikgesetz<br />
– wurde von der breiten Masse noch gar nicht als solche wahrgenommen.<br />
Im April entschieden die Abgeordneten des<br />
Deutschen Bundestages mit großer Mehrheit,<br />
dass deutsche Stammzellforscher straffrei und<br />
zusammen mit ihren Kollegen in Europa und<br />
der Welt das Geheimnis der menschlichen<br />
Entwicklung an geeigneten humanen embryonalen<br />
Stammzellen untersuchen dürfen. Sie<br />
verschoben dazu den Stichtag für den Import<br />
der Stammzellen auf den 1. Mai 2007 und gewährten<br />
den Forschern damit den Zugriff auf<br />
die besten in einem ethisch balancierten Rahmen<br />
verfügbaren embryonalen Stammzellen.<br />
Zusätzlich entschieden Politiker, dass ein<br />
hohes Datenschutzniveau bei prädiktiven<br />
gendiagnostischen Tests möglich ist, ohne die<br />
klinische oder Grundlagen-Forschung einzuschränken.<br />
Denn zur Freude klinischer Forscher<br />
soll das künftige Gendiagnostikgesetz weder<br />
pharmakogenetische Tests an zugelassenen<br />
Arzneimitteln noch genetische Untersuchungen<br />
im Rahmen von Biobankprojekten<br />
betreffen. Über die pharmakogenetischen<br />
Tests soll wie bisher das Bundesinstitut für Arzneimittel<br />
und Medizinprodukte entscheiden,<br />
und auch die Biobanken fallen weiter unter<br />
bestehendes Recht, sagt die Biotechnologin<br />
und gesundheitspolitische Sprecherin der SPD,<br />
Carola Reimann.<br />
Landauf, landab redet deshalb auch kein Forscher<br />
davon, dass es besser gewesen wäre, den<br />
Stichtag ganz abzuschaffen oder dass die Beschränkung<br />
auf Gentests angesichts der Fortschritte<br />
in den Proteomics und Metabonomics<br />
Impressum<br />
LABORWELT (ISSN 1611-0854)<br />
erscheint zweimonatlich im<br />
BIOCOM Verlag GmbH<br />
Stralsunder Straße 58–59<br />
13355 Berlin, Germany<br />
Tel./Fax: 030/264921-0 / 030/264921-11<br />
laborwelt@biocom.de<br />
www.biocom.de<br />
Redaktion<br />
Dipl.-Biol. Thomas Gabrielczyk<br />
Tel.: 030/264921-50<br />
Anzeigenleitung<br />
Oliver Schnell<br />
Tel. 030/264921-45,<br />
o.schnell@biocom.de<br />
Leserservice<br />
Angelika Werner<br />
Tel. 030/264921-40<br />
Bildtechnik und Layout<br />
Heiko Fritz<br />
Graphik-Design<br />
Michaela Reblin<br />
Druck:<br />
Druckhaus Humburg GmbH, 28325 Bremen<br />
Für einen regelmäßigen Bezug von LABORWELT<br />
ist eine kostenlose Registrierung unter www.<br />
biocom.de oder per Fax erforderlich. Namentlich<br />
gekennzeichnete Beiträge stehen in der<br />
inhaltlichen Verantwortung der Autoren.<br />
eigentlich viel zu eng ist. Statt dessen werden<br />
emsig Forschungsanträge geschrieben, neue<br />
Schwerpunkte mit induzierten pluripotenten<br />
Stammzellen (ipS) gestartet wie etwa an der<br />
Medizinischen Hochschule Hannover, an der<br />
bislang nur mit Affenstammzellen gearbeitet<br />
wurde, und EU-Stammzellprojekte unter deutscher<br />
Federführung initiiert.<br />
Toxizitätsmodelle aus<br />
embryonalen Stammzellen<br />
Koordiniert vom Stammzellexperten Jürgen<br />
Hescheler hob etwa Mitte April in Köln das<br />
15,5 Mio.-Euro-Programm ESNATS ab. Mit<br />
neuen Stammzelllinien der schwedischen<br />
Cellartis AB wollen 21 Forschungsgruppen,<br />
sieben KMU und drei große Pharma-Unternehmen<br />
Toxizitätssignaturen in aus embryonalen<br />
Stammzellen differenzierten Zellen finden,<br />
validieren und in einem automatisierbaren<br />
Assay vereinen. Im Fokus dabei: pharmakogenetische<br />
und Protein-Toxizitätsmarker, die<br />
während der Neurogenese, Spermatogenese,<br />
in embryonalen Stammzellen des Menschen<br />
und in aus diesen differenzierten Hepatozyten<br />
detektiert werden. Dass sich deutsche Forscher<br />
kurz nach dem Beschluss des Bundestages mit<br />
einem solch ambitionierten Projekt der internationalen<br />
Szene präsentieren, lässt hoffen.<br />
Und es zeigt, was politischer Rückhalt möglich<br />
machen kann.<br />
Alle Beiträge sind urheberrechtlich geschützt.<br />
Ohne schriftliche Genehmigung des BIOCOM<br />
Verlages darf kein Teil in irgendeiner Form<br />
reproduziert oder mit elektronischen Systemen<br />
verarbeitet, vervielfältigt oder verbreitet werden.<br />
Dieser Ausgabe liegt eine Beilage der<br />
Invitrogen GmbH und der BIOCOM bei.<br />
Die Druckauflage von 20 000 Exemplaren<br />
ist IVW geprüft (Stand I/08):<br />
© BIOCOM Verlag GmbH, Berlin<br />
® BIOCOM ist eine geschützte Marke der<br />
BIOCOM AG, Berlin<br />
BIOCOM<br />
Inserentenverzeichnis<br />
Applied Biosystems …………………………… 13,25<br />
BD Biosciences GmbH …………………………… 27<br />
Biometra GmbH ………………………………………… 7<br />
Biocrates Life Sciences GmbH ……………… 33<br />
Curacyte AG ……………………………………………… 65<br />
DASGIP GmbH ………………………………………… 35<br />
Guava Technologies Ltd. ………………………… 65<br />
Invitrogen GmbH ……………………………Beilage<br />
In Vitro Systems & Services GmbH ……… 23<br />
GE Healthcare Europe GmbH ………………… 41<br />
Molecular Devices Ltd. …………………………… 39<br />
Mole AS …………………………………………………………9<br />
New England Biolabs GmbH …………………U4<br />
Nikon GmbH ……………………………………………… 15<br />
Porvair Sciences Ltd ………………………………… 39<br />
Olympus Deutschland GmbH …………………11<br />
Provendis GmbH ……………………………………… 39<br />
Roche Diagnostics GmbH ……………………… U2<br />
Forum MedTech Pharma e.V ………………… U3<br />
Sigma-Aldrich Chemie GmbH ………………… 5<br />
USB Europe GmbH …………………………………… 17<br />
Technologiezentrum Dortmund …………… 31<br />
Zinsser Analytik ………………………………………… 45<br />
Vorschau Heft 4/2008<br />
Thema<br />
Proteinanalytik<br />
Die Juli-Ausgabe von LABORWELT steht ganz<br />
im Zeichen der Proteinanalytik. Neue Ansätze,<br />
die Lokalisation von Proteinen zu erfassen,<br />
Modifikationen zu detektieren, Proteine zu<br />
angeln, auf Arrays oder Filtermembranen<br />
nachzuweisen, sind nur einige unserer Themen<br />
des nächsten Heftes.<br />
Marktübersicht: Leuchtproteine<br />
Werbekunden bietet diese Ausgabe mit einer<br />
garantierten Auflage von 20.000 Exemplaren<br />
(IVW-geprüft) eine optimale Plattform<br />
für ihre Produkt- und Imageanzeigen. Reservieren<br />
Sie Ihren Werbeplatz in LABORWELT<br />
4-08 bis spätestens 4. Juli. Ergänzend zu<br />
dem Themenschwerpunkt Proteinanalytik<br />
veröffentlichen wir eine Marktübersicht<br />
„Leuchtproteine und in vivo-<strong>Imaging</strong>“. Weitere<br />
Informationen zu Ihrer möglichen Teilnahme<br />
gibt Oliver Schnell (Tel: +49-30-264921-45,<br />
eMail: o.schnell@biocom.de).<br />
70 | 9. Jahrgang | Nr. 3/2008 LABORWElT
������������������������<br />
�����������������<br />
����<br />
��������������������������������������������������������<br />
�������������������<br />
�������������������������������<br />
�����������������������������������<br />
����������������<br />
���������������������<br />
�������������������<br />
����������������������<br />
��������������������������������<br />
��������������������������<br />
������������������������<br />
������������������<br />
����������������������<br />
�������������<br />
�����������������������������������������������������������������<br />
���������������������������������������������������������������������������������������
Unparalleled<br />
Antibody Quality<br />
Translates into reproducible results in all tested applications.<br />
As scientists we understand your needs – our entire focus is your experimental success.<br />
The combination of our experienced PhD level development scientists overseeing antibody<br />
development together with novel antibody production technologies results in the highest<br />
possible product quality. All product development and production follows rigorous<br />
in-house testing on a wide range of assay applications. Moreover, technical support is<br />
provided by the same scientists who produce the antibodies and know them best.<br />
Because of this, Cell Signaling Technology's phospho-antibodies are cited more<br />
than all other competing products combined.<br />
...from Cell Signaling Technology<br />
Unparalleled product quality, validation and technical support<br />
:: Our commitment to quality means reproducible results in all tested applications.<br />
:: Our antibodies are optimized in a wide range of assay applications by our in-house<br />
clinical applications group – optimization is not left up to you, the user.<br />
:: In-house product development and production means technical support by the same<br />
scientists who develop the antibodies and know them best – this translates into a<br />
thorough, fast and accurate response.<br />
:: Our total protein antibodies meet the same quality standards as our activation-state<br />
antibodies – purchasing both from Cell Signaling Technology ensures product quality,<br />
support and validation.<br />
www.neb-online.de<br />
in Deutschland und Österreich exklusiv von<br />
� New England Biolabs GmbH<br />
Frankfurt/Main Deutschland Tel.: +49(0)69-305-23140 Fax.: +49(0)69-305-23149 email: info@de.neb.com www.neb-online.de<br />
� Cell Signaling Technology Inc.<br />
Danvers, MA USA Tel. 1-877-616-CELL (2355) Fax 1-978-867-2488 email: info@cellsignal.com www.cellsignal.com<br />
Cox IV<br />
Cox IV