- Page 1 and 2: ESERCITAZIONI DI STATISTICA BIOMEDI
- Page 3 and 4: Indice Introduzione e notazione 1 1
- Page 5 and 6: Esempio . . . . . . . . . . . . . .
- Page 7 and 8: Esempio . . . . . . . . . . . . . .
- Page 9: Introduzione e notazione Queste not
- Page 12 and 13: 4 STATISTICA DESCRITTIVA E FUNZIONI
- Page 14 and 15: 6 STATISTICA DESCRITTIVA E FUNZIONI
- Page 16 and 17: 8 STATISTICA DESCRITTIVA E FUNZIONI
- Page 18 and 19: 10 STATISTICA DESCRITTIVA E FUNZION
- Page 20 and 21: 12 STATISTICA DESCRITTIVA E FUNZION
- Page 22 and 23: 14 STATISTICA CLASSICA: TEST T , TE
- Page 24 and 25: 16 STATISTICA CLASSICA: TEST T , TE
- Page 26 and 27: 18 STATISTICA CLASSICA: TEST T , TE
- Page 28 and 29: 20 STATISTICA CLASSICA: TEST T , TE
- Page 30 and 31: 22 STATISTICA CLASSICA: TEST T , TE
- Page 32 and 33: 24 STATISTICA CLASSICA: TEST T , TE
- Page 34 and 35: 26 REGRESSIONE LINEARE E NON LINEAR
- Page 36 and 37: 28 REGRESSIONE LINEARE E NON LINEAR
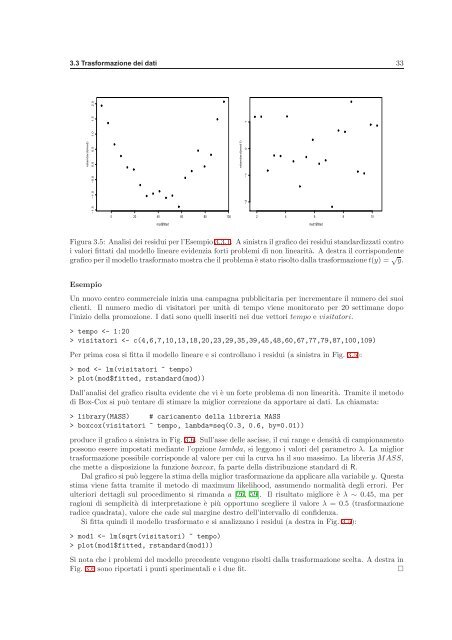
- Page 38 and 39: 30 REGRESSIONE LINEARE E NON LINEAR
- Page 42 and 43: 34 REGRESSIONE LINEARE E NON LINEAR
- Page 44 and 45: 36 REGRESSIONE LINEARE E NON LINEAR
- Page 46 and 47: 38 REGRESSIONE LINEARE E NON LINEAR
- Page 48 and 49: 40 REGRESSIONE LINEARE E NON LINEAR
- Page 50 and 51: 42 REGRESSIONE LINEARE E NON LINEAR
- Page 52 and 53: 44 REGRESSIONE LINEARE E NON LINEAR
- Page 54 and 55: 46 REGRESSIONE LINEARE E NON LINEAR
- Page 56 and 57: 48 REGRESSIONE LINEARE E NON LINEAR
- Page 58 and 59: 50 REGRESSIONE LINEARE E NON LINEAR
- Page 60 and 61: 52 REGRESSIONE LINEARE E NON LINEAR
- Page 62 and 63: 54 REGRESSIONE LINEARE E NON LINEAR
- Page 64 and 65: 56 REGRESSIONE LINEARE E NON LINEAR
- Page 66 and 67: 58 ANALISI DELLA VARIANZA Response:
- Page 68 and 69: 60 ANALISI DELLA VARIANZA 4.4 Contr
- Page 70 and 71: 62 ANALISI DELLA VARIANZA mentre se
- Page 72 and 73: 64 ANALISI DELLA VARIANZA > cont^2
- Page 74 and 75: 66 ANALISI DELLA VARIANZA [1] 17.76
- Page 76 and 77: 68 ANALISI DELLA VARIANZA varieta e
- Page 78 and 79: 70 ANALISI DELLA VARIANZA con εijk
- Page 80 and 81: 72 ANALISI DELLA VARIANZA Levels: 1
- Page 82 and 83: 74 ANALISI DELLA VARIANZA La prima
- Page 84 and 85: 76 ANALISI DELLA VARIANZA settimana
- Page 86 and 87: 78 ANALISI DELLA VARIANZA analisi s
- Page 88 and 89: 80 ANALISI DELLA VARIANZA > abline(
- Page 90 and 91:
82 ANALISI DELLA VARIANZA Bates, au
- Page 92 and 93:
84 ANALISI DELLA VARIANZA Formula:
- Page 94 and 95:
86 ANALISI DELLA VARIANZA 123.1 129
- Page 96 and 97:
88 ANALISI DELLA VARIANZA soggetto
- Page 98 and 99:
90 ANALISI DELLA VARIANZA > atleti2
- Page 100 and 101:
92 ANALISI DELLA VARIANZA da cui si
- Page 102 and 103:
94 ANALISI DELLA VARIANZA Per sotto
- Page 104 and 105:
96 ANALISI DELLA VARIANZA k Na k 7.
- Page 106 and 107:
98 METODI NON PARAMETRICI E POTENZA
- Page 108 and 109:
100 METODI NON PARAMETRICI E POTENZ
- Page 110 and 111:
102 METODI NON PARAMETRICI E POTENZ
- Page 112 and 113:
104 METODI NON PARAMETRICI E POTENZ
- Page 114 and 115:
106 METODI NON PARAMETRICI E POTENZ
- Page 116 and 117:
108 MODELLI LINEARI GENERALIZZATI (
- Page 118 and 119:
110 MODELLI LINEARI GENERALIZZATI (
- Page 120 and 121:
112 MODELLI LINEARI GENERALIZZATI (
- Page 122 and 123:
114 MODELLI LINEARI GENERALIZZATI (
- Page 124 and 125:
116 MODELLI LINEARI GENERALIZZATI (
- Page 126 and 127:
118 MODELLI LINEARI GENERALIZZATI (
- Page 128 and 129:
120 MODELLI LINEARI GENERALIZZATI (
- Page 130 and 131:
122 MODELLI LINEARI GENERALIZZATI (
- Page 132 and 133:
124 MODELLI LINEARI GENERALIZZATI (
- Page 134 and 135:
126 MODELLI LINEARI GENERALIZZATI (
- Page 136 and 137:
128 MODELLI LINEARI GENERALIZZATI (
- Page 138 and 139:
130 MODELLI LINEARI GENERALIZZATI (
- Page 140 and 141:
132 MODELLI LINEARI GENERALIZZATI (
- Page 142 and 143:
134 ANALISI DELLA SOPRAVVIVENZA 7.2
- Page 144 and 145:
136 ANALISI DELLA SOPRAVVIVENZA S(t
- Page 146 and 147:
138 ANALISI DELLA SOPRAVVIVENZA Per
- Page 148 and 149:
140 ANALISI DELLA SOPRAVVIVENZA Lik
- Page 150 and 151:
142 ANALISI DELLA SOPRAVVIVENZA
- Page 152 and 153:
144 ANALISI MULTIVARIATA: TECNICHE
- Page 154 and 155:
146 ANALISI MULTIVARIATA: TECNICHE
- Page 156 and 157:
148 ANALISI MULTIVARIATA: TECNICHE
- Page 158 and 159:
150 ANALISI MULTIVARIATA: TECNICHE
- Page 160 and 161:
152 ANALISI MULTIVARIATA: TECNICHE
- Page 162 and 163:
154 ANALISI MULTIVARIATA: TECNICHE
- Page 164 and 165:
156 ANALISI MULTIVARIATA: TECNICHE
- Page 166 and 167:
158 ANALISI MULTIVARIATA: TECNICHE
- Page 168 and 169:
160 ANALISI MULTIVARIATA: TECNICHE
- Page 170 and 171:
162 ANALISI MULTIVARIATA: TECNICHE
- Page 172 and 173:
164 ANALISI MULTIVARIATA: TECNICHE
- Page 174 and 175:
166 ANALISI MULTIVARIATA: TECNICHE
- Page 176 and 177:
168 ANALISI MULTIVARIATA: METODI DI
- Page 178 and 179:
170 ANALISI MULTIVARIATA: METODI DI
- Page 180 and 181:
172 ANALISI MULTIVARIATA: METODI DI
- Page 182 and 183:
174 ANALISI MULTIVARIATA: METODI DI
- Page 184 and 185:
176 ANALISI MULTIVARIATA: METODI DI
- Page 186 and 187:
178 ANALISI MULTIVARIATA: METODI DI
- Page 188 and 189:
180 ANALISI MULTIVARIATA: METODI DI
- Page 190 and 191:
182 ANALISI MULTIVARIATA: METODI DI
- Page 192 and 193:
184 ANALISI MULTIVARIATA: METODI DI
- Page 194 and 195:
186 ANALISI MULTIVARIATA: METODI DI
- Page 196 and 197:
188 ANALISI MULTIVARIATA: METODI DI
- Page 198 and 199:
190 ANALISI MULTIVARIATA: METODI DI
- Page 200 and 201:
192 ANALISI MULTIVARIATA: METODI DI
- Page 202 and 203:
194 ANALISI MULTIVARIATA: METODI DI
- Page 204 and 205:
196 ANALISI MULTIVARIATA: METODI DI
- Page 206 and 207:
198 ANALISI MULTIVARIATA: METODI DI
- Page 208 and 209:
200 ANALISI MULTIVARIATA: METODI DI
- Page 210 and 211:
202 ANALISI MULTIVARIATA: METODI DI
- Page 212 and 213:
204 ANALISI MULTIVARIATA: METODI DI
- Page 214 and 215:
206 ANALISI MULTIVARIATA: METODI DI
- Page 216 and 217:
208 GEOSTATISTICA suppone che Zi as
- Page 218 and 219:
210 GEOSTATISTICA > vgm plot(vgm)
- Page 220 and 221:
212 GEOSTATISTICA semivariance 0 10
- Page 222 and 223:
214 GEOSTATISTICA Come in tutte le
- Page 224 and 225:
216 GEOSTATISTICA y 333000 332000 3
- Page 226 and 227:
218 GEOSTATISTICA y 333000 332000 3
- Page 228 and 229:
220 GEOSTATISTICA tecniche non para
- Page 230 and 231:
222 GEOSTATISTICA Dato che la trasf
- Page 232 and 233:
224 GEOSTATISTICA Practical Range w
- Page 234 and 235:
226 GEOSTATISTICA Tra gli argomenti
- Page 236 and 237:
228 GEOSTATISTICA All’interno del
- Page 238 and 239:
230 GEOSTATISTICA Frequency density
- Page 240 and 241:
232 GEOSTATISTICA > data(gambia) >
- Page 242 and 243:
234 GEOSTATISTICA parametro mediana
- Page 244 and 245:
236 GEOSTATISTICA [...] I risultati
- Page 246 and 247:
238 ANALISI GENOMICA Il primo coman
- Page 248 and 249:
240 ANALISI GENOMICA 30000 25000 20
- Page 250 and 251:
242 ANALISI GENOMICA Il codice util
- Page 252 and 253:
244 ANALISI GENOMICA Fn(x) 0.0 0.2
- Page 254 and 255:
246 ANALISI GENOMICA ATTCAGGACT AT-
- Page 256 and 257:
248 ANALISI GENOMICA assumendo come
- Page 258 and 259:
250 TECNICHE BOOTSTRAP E METODI MON
- Page 260 and 261:
252 TECNICHE BOOTSTRAP E METODI MON
- Page 262 and 263:
254 TECNICHE BOOTSTRAP E METODI MON
- Page 264 and 265:
256 TECNICHE BOOTSTRAP E METODI MON
- Page 266 and 267:
258 TECNICHE BOOTSTRAP E METODI MON
- Page 268 and 269:
260 UNA BREVE INTRODUZIONE AI COMAN
- Page 270 and 271:
262 UNA BREVE INTRODUZIONE AI COMAN
- Page 272 and 273:
264 UNA BREVE INTRODUZIONE AI COMAN
- Page 274 and 275:
266 GNU FREE DOCUMENTATION LICENSE
- Page 276 and 277:
268 GNU FREE DOCUMENTATION LICENSE
- Page 278 and 279:
270 GNU FREE DOCUMENTATION LICENSE
- Page 280 and 281:
272 HISTORY • v. 1.2.2 (aprile 20
- Page 282 and 283:
Indice analitico A agnes, 150 AIC,
- Page 284 and 285:
276 INDICE ANALITICO K kde2d, 7 ker
- Page 286 and 287:
278 INDICE ANALITICO sequenza annot
- Page 288 and 289:
280 BIBLIOGRAFIA [24] B. Efron, Boo