Identifizierung und Charakterisierung von Interaktionspartnern des ...
Identifizierung und Charakterisierung von Interaktionspartnern des ...
Identifizierung und Charakterisierung von Interaktionspartnern des ...
Sie wollen auch ein ePaper? Erhöhen Sie die Reichweite Ihrer Titel.
YUMPU macht aus Druck-PDFs automatisch weboptimierte ePaper, die Google liebt.
Ergebnisse<br />
Die Molekulargewichte der sequenzierten Proteinsegmente wurden mit Hilfe <strong>des</strong><br />
Computerprogramms DNA-Strider 1.1 sowie <strong>des</strong> durch die Internetseite<br />
http://www.ultranet.com (Gill <strong>und</strong> Hippel, 1989) zur Verfügung gestellten Programms<br />
theoretisch kalkuliert <strong>und</strong> mit den durch die elektrophoretische Auftrennung ermittelten<br />
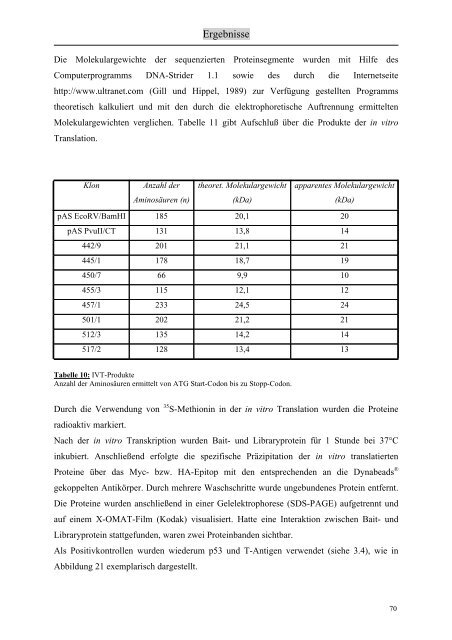
Molekulargewichten verglichen. Tabelle 11 gibt Aufschluß über die Produkte der in vitro<br />
Translation.<br />
Klon<br />
Anzahl der<br />
theoret. Molekulargewicht<br />
apparentes Molekulargewicht<br />
Aminosäuren (n)<br />
(kDa)<br />
(kDa)<br />
pAS EcoRV/BamHI 185 20,1 20<br />
pAS PvuII/CT 131 13,8 14<br />
442/9 201 21,1 21<br />
445/1 178 18,7 19<br />
450/7 66 9,9 10<br />
455/3 115 12,1 12<br />
457/1 233 24,5 24<br />
501/1 202 21,2 21<br />
512/3 135 14,2 14<br />
517/2 128 13,4 13<br />
Tabelle 10: IVT-Produkte<br />
Anzahl der Aminosäuren ermittelt <strong>von</strong> ATG Start-Codon bis zu Stopp-Codon.<br />
Durch die Verwendung <strong>von</strong> 35 S-Methionin in der in vitro Translation wurden die Proteine<br />
radioaktiv markiert.<br />
Nach der in vitro Transkription wurden Bait- <strong>und</strong> Libraryprotein für 1 St<strong>und</strong>e bei 37°C<br />
inkubiert. Anschließend erfolgte die spezifische Präzipitation der in vitro translatierten<br />
Proteine über das Myc- bzw. HA-Epitop mit den entsprechenden an die Dynabeads ®<br />
gekoppelten Antikörper. Durch mehrere Waschschritte wurde ungeb<strong>und</strong>enes Protein entfernt.<br />
Die Proteine wurden anschließend in einer Gelelektrophorese (SDS-PAGE) aufgetrennt <strong>und</strong><br />
auf einem X-OMAT-Film (Kodak) visualisiert. Hatte eine Interaktion zwischen Bait- <strong>und</strong><br />
Libraryprotein stattgef<strong>und</strong>en, waren zwei Proteinbanden sichtbar.<br />
Als Positivkontrollen wurden wiederum p53 <strong>und</strong> T-Antigen verwendet (siehe 3.4), wie in<br />
Abbildung 21 exemplarisch dargestellt.<br />
70