PDF Download - Laborwelt
PDF Download - Laborwelt
PDF Download - Laborwelt
Erfolgreiche ePaper selbst erstellen
Machen Sie aus Ihren PDF Publikationen ein blätterbares Flipbook mit unserer einzigartigen Google optimierten e-Paper Software.
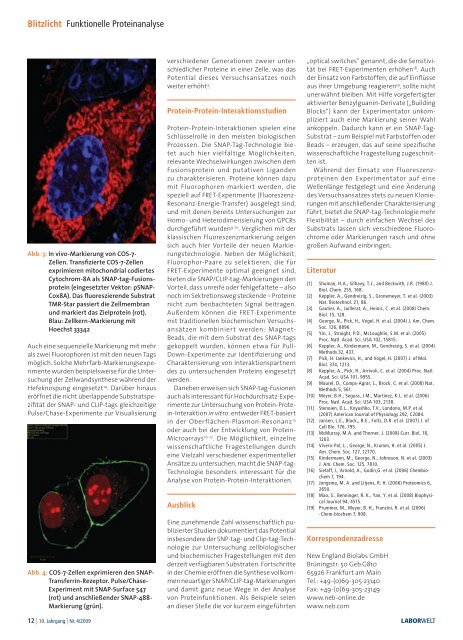
Blitzlicht Funktionelle ProteinanalyseAbb. 3: In vivo-Markierung von COS-7-Zellen. Transfizierte COS-7-Zellenexprimieren mitochondrial codiertesCytochrom-8A als SNAP-tag-Fusionsprotein(eingesetzter Vektor: pSNAP-Cox8A). Das fluoreszierende SubstratTMR-Star passiert die Zellmembranund markiert das Zielprotein (rot).Blau: Zellkern-Markierung mitHoechst 33342Auch eine sequenzielle Markierung mit mehrals zwei Fluorophoren ist mit den neuen Tagsmöglich. Solche Mehrfarb-Markierungsexperimentewurden beispielsweise für die Untersuchungder Zellwandsynthese während derHefeknospung eingesetzt 14 . Darüber hinauseröffnet die nicht überlappende Substratspezifitätder SNAP- und CLIP-tags gleichzeitigePulse/Chase-Experimente zur VisualisierungAbb. 4: COS-7-Zellen exprimieren den SNAP-Transferrin-Rezeptor. Pulse/Chase-Experiment mit SNAP-Surface 547(rot) und anschließender SNAP-488-Markierung (grün).verschiedener Generationen zweier unterschiedlicherProteine in einer Zelle, was dasPotential dieses Versuchsansatzes nochweiter erhöht 3 .Protein-Protein-InteraktionsstudienProtein-Protein-Interaktionen spielen eineSchlüsselrolle in den meisten biologischenProzessen. Die SNAP-Tag-Technologie bietetauch hier vielfältige Möglichkeiten,relevante Wechselwirkungen zwischen demFusionsprotein und putativen Ligandenzu charakterisieren. Proteine können dazumit Fluorophoren markiert werden, diespeziell auf FRET-Experimente (Fluoreszenz-Resonanz-Energie-Transfer) ausgelegt sind,und mit denen bereits Untersuchungen zurHomo- und Heterodimerisierung von GPCRsdurchgeführt wurden 9-10 . Verglichen mit derklassischen Fluoreszenzmarkierung zeigensich auch hier Vorteile der neuen Markierungstechnologie.Neben der Möglichkeit,Fluorophor-Paare zu selektieren, die fürFRET-Experimente optimal geeignet sind,bieten die SNAP/CLIP-tag-Markierungen denVorteil, dass unreife oder fehlgefaltete – alsonoch im Sektretionsweg steckende – Proteinenicht zum beobachteten Signal beitragen.Außerdem können die FRET-Experimentemit traditionellen biochemischen Versuchsansätzenkombiniert werden: Magnet-Beads, die mit dem Substrat des SNAP-tagsgekoppelt wurden, können etwa für Pull-Down-Experimente zur Identifizierung undCharakterisierung von Interaktionspartnerndes zu untersuchenden Proteins eingesetztwerden.Daneben erweisen sich SNAP-tag-Fusionenauch als interessant für Hochdurchsatz-Experimentezur Untersuchung von Protein-Protein-Interaktionin vitro: entweder FRET-basiertin der Oberflächen-Plasmon-Resonanz 15oder auch bei der Entwicklung von Protein-Microarrays 16-17 . Die Möglichkeit, einzelnewissenschaftliche Fragestellungen durcheine Vielzahl verschiedener experimentellerAnsätze zu untersuchen, macht die SNAP-tag-Technologie besonders interessant für dieAnalyse von Protein-Protein-Interaktionen.AusblickEine zunehmende Zahl wissenschaftlich publizierterStudien dokumentiert das Potentialinsbesondere der SNP-tag- und Clip-tag-Technologiezur Untersuchung zellbiologischerund biochemischer Fragestellungen mit denderzeit verfügbaren Substraten. Fortschrittein der Chemie eröffnen die Synthese vollkommenneuartiger SNAP/CLIP-tag-Markierungenund damit ganz neue Wege in der Analysevon Proteinfunktionen. Als Beispiele seienan dieser Stelle die vor kurzem eingeführten„optical switches“ genannt, die die Sensitivitätbei FRET-Experimenten erhöhen 18 . Auchder Einsatz von Farbstoffen, die auf Einflüsseaus ihrer Umgebung reagieren 19 , sollte nichtunerwähnt bleiben. Mit Hilfe vorgefertigteraktivierter Benzylguanin-Derivate („BuildingBlocks“) kann der Experimentator unkompliziertauch eine Markierung seiner Wahlankoppeln. Dadurch kann er ein SNAP-Tag-Substrat – zum Beispiel mit Farbstoffen oderBeads – erzeugen, das auf seine spezifischewissenschaftliche Fragestellung zugeschnittenist.Während der Einsatz von Fluoreszenzproteinenden Experimentator auf eineWellenlänge festgelegt und eine Änderungdes Versuchsansatzes stets zu neuen Klonierungenmit anschließender Charakterisierungführt, bietet die SNAP-tag-Technologie mehrFlexibilität – durch einfachen Wechsel desSubstrats lassen sich verschiedene Fluorochromeoder Markierungen rasch und ohnegroßen Aufwand einbringen.Literatur[1] Shuman, H.A., Silhavy, T.J., and Beckwith, J.R. (1980) J.Biol. Chem. 255, 168.[2] Keppler, A., Gendreizig, S., Gronemeyer, T. et al. (2003)Nat. Biotechnol. 21, 86.[3] Gautier, A., Juillerat, A., Heinis, C. et al. (2008) Chem.Biol. 15, 128.[4] George, N., Pick, H., Vogel, H. et al. (2004) J. Am. Chem.Soc. 126, 8896.[5] Yin, J., Straight, P.D., McLoughlin, S.M. et al. (2005)Proc. Natl. Acad. Sci. USA 102, 15815.[6] Keppler, A., Kindermann, M., Gendreizig, S. et al. (2004)Methods 32, 437.[7] Pick, H. Jankevics, H., and Vogel, H. (2007) J. of Mol.Biol. 374, 1213.[8] Keppler, A., Pick, H., Arrivoli, C. et al. (2004) Proc. Natl.Acad. Sci. USA 101, 9955.[9] Maurel, D., Comps-Agrar, L., Brock, C. et al. (2008) Nat.Methods 5, 561.[10] Meyer, B.H., Segura, J.M., Martinez, K.L. et al. (2006)Proc. Natl. Acad. Sci. USA 103, 2138.[11] Stenoien, D.L., Knyushko, T.V., Londono, M.P. et al.(2007) American Journal of Physiology 292, C2084.[12] Jansen, L.E., Black,, B.E., Foltz, D.R. et al. (2007) J. ofCell Bio. 176, 795.[13] McMurray, M.A. and Thorner, J. (2008) Curr. Biol, 18,1203.[14] Vivero-Pol, L., George, N., Krumm, H. et al. (2005) J.Am. Chem. Soc. 127, 12770.[15] Kindermann, M., George, N., Johnsson, N. et al. (2003)J. Am. Chem. Soc. 125, 7810.[16] Sielaff, I., Arnold, A., Godin,G. et al. (2006) Chembiochem7, 194.[17] Jongsma, M. A. and Litjens, R. H. (2006) Proteomics 6,2650.[18] Mao, S., Benninger, R. K., Yan, Y. et al. (2008) BiophysicalJournal 94, 4515.[19] Prummer, M., Meyer, B. H., Franzini, R. et al. (2006)-Chem-biochem 7, 908.KorrespondenzadresseNew England Biolabs GmbHBrüningstr. 50 Geb.G81065926 Frankfurt am MainTel.: +49-(0)69-305-23140Fax: +49-(0)69-305-23149www.neb-online.dewww.neb.com12 | 10. Jahrgang | Nr. 4/2009 LABORWELT