PDF Download - Laborwelt
PDF Download - Laborwelt
PDF Download - Laborwelt
Erfolgreiche ePaper selbst erstellen
Machen Sie aus Ihren PDF Publikationen ein blätterbares Flipbook mit unserer einzigartigen Google optimierten e-Paper Software.
Auch hier ermöglicht das Bandenmuster eine<br />
Aussage über die Anzahl der ursprünglich<br />
methylierten Cytosine, da nur bei diesen<br />
das Motiv so erhalten bleibt; aus dem nicht<br />
methylierten Tetranukleotid wird das Motiv<br />
TGTG, welches BstUI nicht erkennt. Wesentlich<br />
genauer, aber auch aufwendiger und<br />
kostenintensiver ist die Klonierung des zu<br />
untersuchenden Fragments und die anschließende<br />
Sequenzierung einiger Klone.<br />
Eine methodische Erleichterung ergibt sich<br />
durch die Anwendung von methylierungssensitiven<br />
Microarrays. In jüngerer Zeit wurde<br />
eine Reihe von Methoden entwickelt, um den<br />
Methylierungsstatus genomischer DNA speziell<br />
aus Tumoren mit Hilfe von Microarrays zu<br />
erfassen. Aufgrund der mangelnden Flexibilität<br />
der Untersuchungsmethoden konnten jedoch<br />
nur wenige CpG-Inseln in einigen Genen<br />
untersucht werden. Als limitierend erwiesen<br />
sich besonders der große Zeitaufwand und die<br />
hohen Kosten, wenn mit einer großen Anzahl<br />
von Sonden gearbeitet werden soll. Dies ist<br />
immer dann der Fall, wenn Sonden für eine<br />
neue Fragestellung hergestellt und validiert<br />
werden sollen.<br />
Um diese Limitierungen zu überwinden,<br />
wurde in dem hier beschriebenen neuen<br />
Ansatz mit dem flexiblen Microarraysystem<br />
Geniom der Febit Biotech GmbH gearbeitet<br />
(Abb.1). Geniom besteht aus einem Oligonukleotid-Synthesizer,<br />
der in einer lichtinduzierten,<br />
maskenfreien Synthese in situ mehr<br />
als 6.700 unterschiedliche Oligonukleotide<br />
parallel in jeweils acht Microarrays synthetisiert<br />
(erweiterbar auf 15.000 Oligos pro<br />
Array). Diese sind jeweils auf einem Biochip<br />
untergebracht und können unabhängig voneinander<br />
hybridisiert werden. Der komplette<br />
Synthesevorgang dauert wenige Stunden.<br />
Auch die Hybridisierung, Detektion und<br />
Kennziffer 24 LW 04 · www.biocom.de �<br />
Datenanalyse lassen sich mit GENIOM einfach<br />
durchführen. Dadurch, daß der gesamte<br />
Prozeß auf einem Gerät im eigenen Labor abläuft,<br />
sind schnelle Optimierungen des Arraydesigns<br />
möglich. Somit werden die eben erst<br />
gewonnenen Erkenntnisse bereits innerhalb<br />
weniger Stunden in Form eines neuen Arrays<br />
mit optimierter Oligonukleotid-Zusammensetzung<br />
umgesetzt. Geniom eignet sich daher<br />
hervorragend für neue Fragestellungen oder<br />
auch die Arbeit mit selten untersuchten Organismen,<br />
für die keine anderen Microarrays<br />
erhältlich sind.<br />
Analyse der CpG-Methylierung eines<br />
Tumorsuppressorgens<br />
Mit Hilfe von Geniom ist es gelungen, beispielhaft<br />
den Methylierungsstatus der CpG-Insel<br />
in der Promotorregion des p16 INK4A -Tumorsuppressorgens<br />
aufzuklären 4 . Diese beinhaltet<br />
bei einer Länge von etwa 650 Nukleotiden<br />
53 CpG-Dinukleotide. Die Expression des<br />
kodierten CDK-Inhibitors 2A wird bei vielen<br />
Tumortypen durch die CpG-Methylierung<br />
inhibiert. Nach Eingabe der Sequenzdaten,<br />
die die einzelnen zu untersuchenden CpGs<br />
beinhalten, errechnet die Geniom ® -Software,<br />
welche Oligonukleotide für deren Detektion<br />
synthetisiert werden müssen. Die Herausforderung<br />
an das Design von spezifischen Proben<br />
besteht bei CpG-Inseln darin, daß die Sequenzen,<br />
in welche die Dinukleotide eingebettet<br />
sind, eine geringe Komplexität aufweisen und<br />
daß die einzelnen CpGs teilweise so nahe beieinander<br />
liegen, daß der Methylierungsgrad<br />
von jeweils mehreren CpGs mit einem Satz<br />
von permutierten Oligos detektiert werden<br />
DASGIP –<br />
Parallel<br />
Bioreactors<br />
for Unparalleled<br />
Results<br />
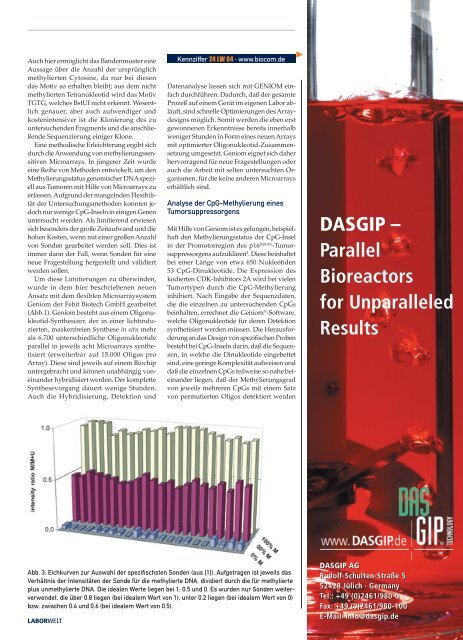
Abb. 3: Eichkurven zur Auswahl der spezifischsten Sonden (aus [1]). Aufgetragen ist jeweils das<br />
Verhältnis der Intensitäten der Sonde für die methylierte DNA, dividiert durch die für methylierte<br />
plus unmethylierte DNA. Die idealen Werte liegen bei 1; 0.5 und 0. Es wurden nur Sonden weiterverwendet,<br />
die über 0.8 liegen (bei idealem Wert von 1), unter 0.2 liegen (bei idealem Wert von 0)<br />
DASGIP AG<br />
Rudolf-Schulten-Straße 5<br />
52428 Jülich · Germany<br />
Tel.: +49 (0)2461/980-0<br />
bzw. zwischen 0.4 und 0.6 (bei idealem Wert von 0.5).<br />
Fax: +49 (0)2461/980-100<br />
LABORWELT<br />
E-Mail: info@dasgip.de<br />
7. Jahrgang | Nr. 2/2005 | 33