PDF Download - Laborwelt
PDF Download - Laborwelt
PDF Download - Laborwelt
Erfolgreiche ePaper selbst erstellen
Machen Sie aus Ihren PDF Publikationen ein blätterbares Flipbook mit unserer einzigartigen Google optimierten e-Paper Software.
A<br />
B<br />
B L I T Z L I C H T<br />
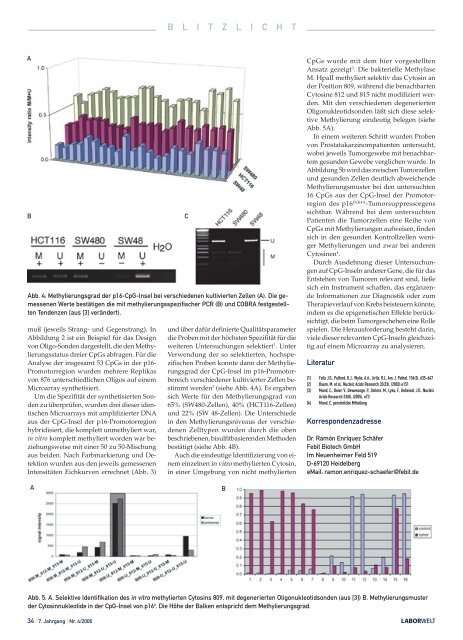
Abb. 4: Methylierungsgrad der p16-CpG-Insel bei verschiedenen kultivierten Zellen (A). Die gemessenen<br />
Werte bestätigen die mit methylierungsspezifischer PCR (B) und COBRA festgestellten<br />
Tendenzen [aus [3] verändert).<br />
muß (jeweils Strang- und Gegenstrang). In<br />
Abbildung 2 ist ein Beispiel für das Design<br />
von Oligo-Sonden dargestellt, die den Methylierungsstatus<br />
dreier CpGs abfragen. Für die<br />
Analyse der insgesamt 53 CpGs in der p16-<br />
Promotorregion wurden mehrere Replikas<br />
von 876 unterschiedlichen Oligos auf einem<br />
Microarray synthetisiert.<br />
Um die Spezifität der synthetisierten Sonden<br />
zu überprüfen, wurden drei dieser identischen<br />
Microarrays mit amplifizierter DNA<br />
aus der CpG-Insel der p16-Promotorregion<br />
hybridisiert, die komplett unmethyliert war,<br />
in vitro komplett methyliert worden war beziehungsweise<br />
mit einer 50 zu 50-Mischung<br />
aus beiden. Nach Farbmarkierung und Detektion<br />
wurden aus den jeweils gemessenen<br />
Intensitäten Eichkurven errechnet (Abb. 3)<br />
A B<br />
C<br />
und über dafür definierte Qualitätsparameter<br />
die Proben mit der höchsten Spezifität für die<br />
weiteren Untersuchungen selektiert 3 . Unter<br />
Verwendung der so selektierten, hochspezifischen<br />
Proben konnte dann der Methylierungsgrad<br />
der CpG-Insel im p16-Promotorbereich<br />
verschiedener kultivierter Zellen bestimmt<br />
werden 3 (siehe Abb. 4A). Es ergaben<br />
sich Werte für den Methylierungsgrad von<br />
65% (SW480-Zellen), 40% (HCT116-Zellen)<br />
und 22% (SW 48-Zellen). Die Unterschiede<br />
in den Methylierungsniveaus der verschiedenen<br />
Zelltypen wurden durch die oben<br />
beschriebenen, bisulfitbasierenden Methoden<br />
bestätigt (siehe Abb. 4B).<br />
Auch die eindeutige Identifizierung von einem<br />
einzelnen in vitro methylierten Cytosin,<br />
in einer Umgebung von nicht methylierten<br />
CpGs wurde mit dem hier vorgestellten<br />
Ansatz gezeigt 3 . Die bakterielle Methylase<br />
M. HpaII methyliert selektiv das Cytosin an<br />
der Position 809, während die benachbarten<br />
Cytosine 812 und 815 nicht modifiziert werden.<br />
Mit den verschiedenen degenerierten<br />
Oligonukleotidsonden läßt sich diese selektive<br />
Methylierung eindeutig belegen (siehe<br />
Abb. 5A).<br />
In einem weiteren Schritt wurden Proben<br />
von Prostatakarzinompatienten untersucht,<br />
wobei jeweils Tumorgewebe mit benachbartem<br />
gesunden Gewebe verglichen wurde. In<br />
Abbildung 5b wird das zwischen Tumorzellen<br />
und gesunden Zellen deutlich abweichende<br />
Methylierungsmuster bei den untersuchten<br />
16 CpGs aus der CpG-Insel der Promotorregion<br />
des p16 INK4A -Tumorsuppressorgens<br />
sichtbar. Während bei dem untersuchten<br />
Patienten die Tumorzellen eine Reihe von<br />
CpGs mit Methylierungen aufweisen, finden<br />
sich in den gesunden Kontrollzellen weniger<br />
Methylierungen und zwar bei anderen<br />
Cytosinen 4 .<br />
Durch Ausdehnung dieser Untersuchungen<br />
auf CpG-Inseln anderer Gene, die für das<br />
Entstehen von Tumoren relevant sind, ließe<br />
sich ein Instrument schaffen, das ergänzende<br />
Informationen zur Diagnostik oder zum<br />
Therapieverlauf von Krebs beisteuern könnte,<br />
indem es die epigenetischen Effekte berücksichtigt,<br />
die beim Tumorgeschehen eine Rolle<br />
spielen. Die Herausforderung besteht darin,<br />
viele dieser relevanten CpG-Inseln gleichzeitig<br />
auf einem Microarray zu analysieren.<br />
Literatur<br />
[1] Falls J.G., Pulford, D.J., Wylie, A.A., Jirtle, R.L. Am. J. Pathol. 154(3), 635-647<br />
[2] Baum, M. et al., Nucleic Acids Research 33(23), (2003) e151<br />
[3] Mund, C., Beier, V., Bewerunge, P., Dahms, M., Lyko, F., Hoheisel, J.D., Nucleic<br />
Acids Research 33(8), (2005), e73<br />
[4] Mund, C. persönliche Mitteilung<br />
Korrespondenzadresse<br />
Dr. Ramón Enríquez Schäfer<br />
Febit Biotech GmbH<br />
Im Neuenheimer Feld 519<br />
D-69120 Heidelberg<br />
eMail: ramon.enriquez-schaefer@febit.de<br />
Abb. 5: A. Selektive Identifikation des in vitro methylierten Cytosins 809, mit degenerierten Oligonukleotidsonden (aus [3]) B. Methylierungsmuster<br />
der Cytosinnukleotide in der CpG-Insel von p16 4 . Die Höhe der Balken entspricht dem Methylierungsgrad.<br />
34 | 7. Jahrgang | Nr. 4/2006 LABORWELT