Öffnen - eDiss - Georg-August-Universität Göttingen
Öffnen - eDiss - Georg-August-Universität Göttingen
Öffnen - eDiss - Georg-August-Universität Göttingen
Erfolgreiche ePaper selbst erstellen
Machen Sie aus Ihren PDF Publikationen ein blätterbares Flipbook mit unserer einzigartigen Google optimierten e-Paper Software.
Methoden - Kapitel 3.4.<br />
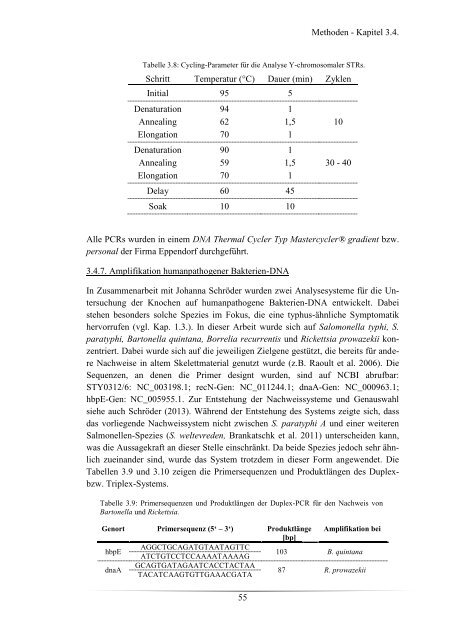
Tabelle 3.8: Cycling-Parameter für die Analyse Y-chromosomaler STRs.<br />
Schritt Temperatur (°C) Dauer (min) Zyklen<br />
Initial 95 5<br />
Denaturation<br />
Annealing<br />
Elongation<br />
Denaturation<br />
Annealing<br />
Elongation<br />
94<br />
62<br />
70<br />
90<br />
59<br />
70<br />
1<br />
1,5<br />
1<br />
1<br />
1,5<br />
1<br />
Delay 60 45<br />
Soak 10 10<br />
10<br />
30 - 40<br />
Alle PCRs wurden in einem DNA Thermal Cycler Typ Mastercycler® gradient bzw.<br />
personal der Firma Eppendorf durchgeführt.<br />
3.4.7. Amplifikation humanpathogener Bakterien-DNA<br />
In Zusammenarbeit mit Johanna Schröder wurden zwei Analysesysteme für die Untersuchung<br />
der Knochen auf humanpathogene Bakterien-DNA entwickelt. Dabei<br />
stehen besonders solche Spezies im Fokus, die eine typhus-ähnliche Symptomatik<br />
hervorrufen (vgl. Kap. 1.3.). In dieser Arbeit wurde sich auf Salomonella typhi, S.<br />
paratyphi, Bartonella quintana, Borrelia recurrentis und Rickettsia prowazekii konzentriert.<br />
Dabei wurde sich auf die jeweiligen Zielgene gestützt, die bereits für andere<br />
Nachweise in altem Skelettmaterial genutzt wurde (z.B. Raoult et al. 2006). Die<br />
Sequenzen, an denen die Primer designt wurden, sind auf NCBI abrufbar:<br />
STY0312/6: NC_003198.1; recN-Gen: NC_011244.1; dnaA-Gen: NC_000963.1;<br />
hbpE-Gen: NC_005955.1. Zur Entstehung der Nachweissysteme und Genauswahl<br />
siehe auch Schröder (2013). Während der Entstehung des Systems zeigte sich, dass<br />
das vorliegende Nachweissystem nicht zwischen S. paratyphi A und einer weiteren<br />
Salmonellen-Spezies (S. weltevreden, Brankatschk et al. 2011) unterscheiden kann,<br />
was die Aussagekraft an dieser Stelle einschränkt. Da beide Spezies jedoch sehr ähnlich<br />
zueinander sind, wurde das System trotzdem in dieser Form angewendet. Die<br />
Tabellen 3.9 und 3.10 zeigen die Primersequenzen und Produktlängen des Duplexbzw.<br />
Triplex-Systems.<br />
Tabelle 3.9: Primersequenzen und Produktlängen der Duplex-PCR für den Nachweis von<br />
Bartonella und Rickettsia.<br />
Genort Primersequenz (5‘ – 3‘) Produktlänge Amplifikation bei<br />
[bp]<br />
hbpE<br />
AGGCTGCAGATGTAATAGTTC<br />
ATCTGTCCTCCAAAATAAAAG<br />
103 B. quintana<br />
dnaA<br />
GCAGTGATAGAATCACCTACTAA<br />
TACATCAAGTGTTGAAACGATA<br />
87 R. prowazekii<br />
55