Therya agosto 2012.indb - AMMAC: Acerca de la Asociación ...
Therya agosto 2012.indb - AMMAC: Acerca de la Asociación ...
Therya agosto 2012.indb - AMMAC: Acerca de la Asociación ...
Create successful ePaper yourself
Turn your PDF publications into a flip-book with our unique Google optimized e-Paper software.
Cervantes, Castañeda<br />
L. c. sheldoni y L. c. texianus. Asimismo, estas estimaciones resultaron menores a sus<br />
correspondientes valores <strong>de</strong> heterocigosidad esperada. No se <strong>de</strong>tectaron alelos fijos en<br />
<strong>la</strong>s especies <strong>de</strong> liebre bajacalifornianas, pero si se i<strong>de</strong>ntificaron 11 alelos exclusivos: uno<br />
en L. insu<strong>la</strong>ris (HK); cuatro en L. c. xanti (MDH-2, ME-1, PAP y LGG); dos en L. c. sheldoni<br />
(ME-2 y ALB); dos en L. c. martirensis (LDH-1 y G6PDH); y dos en L. c. texianus (ME-2<br />
y LA). Por su parte, L. callotis y S. floridanus presentaron diversos alelos fijos (Tab<strong>la</strong> 3).<br />
El valor promedio <strong>de</strong>l índice <strong>de</strong> fijación <strong>de</strong> Wright’s (Fst) para los taxones estudiados<br />
fue <strong>de</strong> 0.449 (Tab<strong>la</strong> 4), mientras que el Fst calcu<strong>la</strong>do únicamente con <strong>la</strong>s subespecies <strong>de</strong><br />
L. californicus y L. insu<strong>la</strong>ris fue notablemente menor (0.192). Del total <strong>de</strong> los 26 loci, el<br />
valor <strong>de</strong> chi-cuadrada <strong>de</strong>l Fst para 22 <strong>de</strong> ellos resultó ser estadísticamente significativo (P<br />
< 0.05). Los otros cuatro loci (SOD-2, MDH-1, GPI y GOT-2) no mostraron diferencias<br />
significativas (Tab<strong>la</strong> 4).<br />
La matriz <strong>de</strong> distancias genéticas <strong>de</strong> <strong>la</strong>s liebres <strong>de</strong> <strong>la</strong> penínsu<strong>la</strong> <strong>de</strong> Baja California<br />
basada en el índice <strong>de</strong> Cavalli-Sforza y Edwards (1967), indicó que el menor valor <strong>de</strong><br />
distancia genética correspondió a <strong>la</strong>s pob<strong>la</strong>ciones <strong>de</strong> L. insu<strong>la</strong>ris y L. c. texianus, seguido<br />
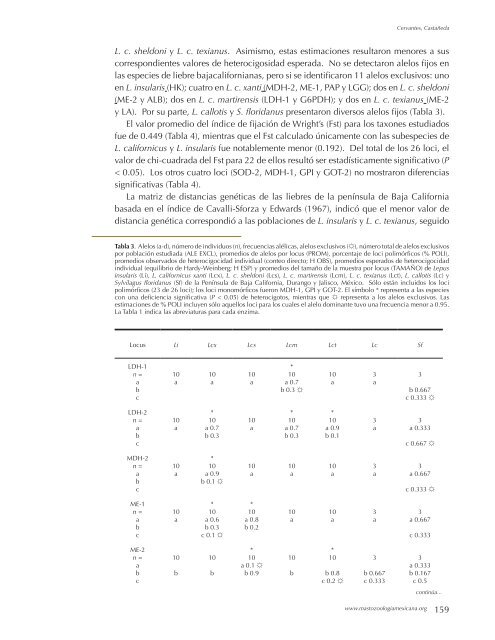
Tab<strong>la</strong> 3. Alelos (a-d), número <strong>de</strong> individuos (n), frecuencias alélicas, alelos exclusivos (☼), número total <strong>de</strong> alelos exclusivos<br />
por pob<strong>la</strong>ción estudiada (ALE EXCL), promedios <strong>de</strong> alelos por locus (PROM), porcentaje <strong>de</strong> loci polimórficos (% POLI),<br />
promedios observados <strong>de</strong> heterocigocidad individual (conteo directo; H OBS), promedios esperados <strong>de</strong> heterocigocidad<br />
individual (equilibrio <strong>de</strong> Hardy-Weinberg; H ESP) y promedios <strong>de</strong>l tamaño <strong>de</strong> <strong>la</strong> muestra por locus (TAMAÑO) <strong>de</strong> Lepus<br />
insu<strong>la</strong>ris (Li), L. californicus xanti (Lcx), L. c. sheldoni (Lcs), L. c. martirensis (Lcm), L. c. texianus (Lct), L. callotis (Lc) y<br />
Sylvi<strong>la</strong>gus floridanus (Sf) <strong>de</strong> <strong>la</strong> Penínsu<strong>la</strong> <strong>de</strong> Baja California, Durango y Jalisco, México. Sólo están incluidos los loci<br />
polimórficos (23 <strong>de</strong> 26 loci); los loci monomórficos fueron MDH-1, GPI y GOT-2. El símbolo * representa a <strong>la</strong>s especies<br />
con una <strong>de</strong>ficiencia significativa (P < 0.05) <strong>de</strong> heterocigotos, mientras que ☼ representa a los alelos exclusivos. Las<br />
estimaciones <strong>de</strong> % POLI incluyen sólo aquellos loci para los cuales el alelo dominante tuvo una frecuencia menor a 0.95.<br />
La Tab<strong>la</strong> 1 indica <strong>la</strong>s abreviaturas para cada enzima.<br />
Locus Li Lcx Lcs Lcm Lct Lc Sf<br />
LDH-1<br />
n =<br />
a<br />
b<br />
c<br />
LDH-2<br />
n =<br />
a<br />
b<br />
c<br />
MDH-2<br />
n =<br />
a<br />
b<br />
c<br />
ME-1<br />
n =<br />
a<br />
b<br />
c<br />
ME-2<br />
n =<br />
a<br />
b<br />
c<br />
10<br />
a<br />
10<br />
a<br />
10<br />
a<br />
10<br />
a<br />
10<br />
b<br />
10<br />
a<br />
*<br />
10<br />
a 0.7<br />
b 0.3<br />
*<br />
10<br />
a 0.9<br />
b 0.1 ☼<br />
*<br />
10<br />
a 0.6<br />
b 0.3<br />
c 0.1 ☼<br />
10<br />
b<br />
10<br />
a<br />
10<br />
a<br />
10<br />
a<br />
*<br />
10<br />
a 0.8<br />
b 0.2<br />
*<br />
10<br />
a 0.1 ☼<br />
b 0.9<br />
*<br />
10<br />
a 0.7<br />
b 0.3 ☼<br />
*<br />
10<br />
a 0.7<br />
b 0.3<br />
10<br />
a<br />
10<br />
a<br />
10<br />
b<br />
10<br />
a<br />
*<br />
10<br />
a 0.9<br />
b 0.1<br />
10<br />
a<br />
10<br />
a<br />
*<br />
10<br />
b 0.8<br />
c 0.2 ☼<br />
3<br />
a<br />
3<br />
a<br />
3<br />
a<br />
3<br />
a<br />
3<br />
b 0.667<br />
c 0.333<br />
www.mastozoologiamexicana.org 159<br />
3<br />
b 0.667<br />
c 0.333 ☼<br />
3<br />
a 0.333<br />
c 0.667 ☼<br />
3<br />
a 0.667<br />
c 0.333 ☼<br />
3<br />
a 0.667<br />
c 0.333<br />
3<br />
a 0.333<br />
b 0.167<br />
c 0.5<br />
continúa...