You also want an ePaper? Increase the reach of your titles
YUMPU automatically turns print PDFs into web optimized ePapers that Google loves.
RADAR CIENTÍFICO<br />
Estudo da Universidade de Leipzig:<br />
Comparação da sensibilidade do diagnóstico<br />
de testes de anticorpos baseados<br />
em nucleoproteína e glicoproteína<br />
do SARS-CoV-22<br />
A Universidade de Leipzig avaliou a<br />
sensibilidade de sete ensaios de sorologia<br />
para SARS-CoV-2 comercialmente<br />
disponíveis. Amostras de soro foram<br />
obtidas de 57 pacientes com resultados<br />
de PCR positivos, a maioria dos quais<br />
teve a doença de forma leve a moderada<br />
(nenhum em estado grave ou que<br />
necessitou de cuidados hospitalares).<br />
Em 16 pacientes foram coletadas duas<br />
amostras, 25 amostras de sangue foram<br />
coletadas 2 a 3 vezes após um resultado<br />
de PCR positivo e 48 amostras de sangue<br />
foram retiradas após 4 semanas de<br />
um resultado de PCR positivo.<br />
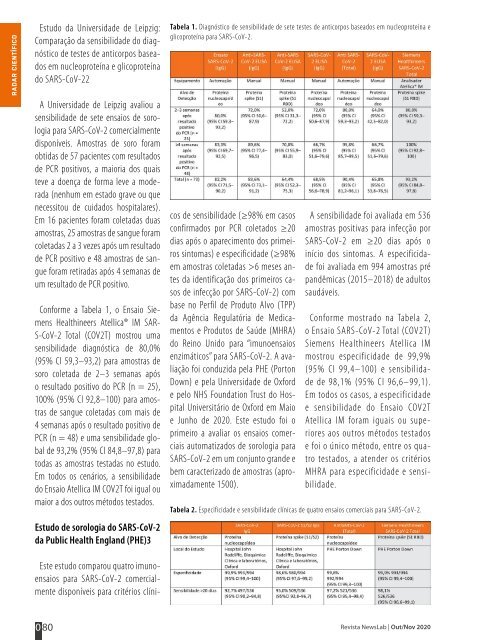
Conforme a Tabela 1, o Ensaio Siemens<br />
Healthineers Atellica® IM SAR-<br />
S-CoV-2 Total (COV2T) mostrou uma<br />
sensibilidade diagnóstica de 80,0%<br />
(95% CI 59,3–93,2) para amostras de<br />
soro coletada de 2–3 semanas após<br />
o resultado positivo do PCR (n = 25),<br />
100% (95% CI 92,8–100) para amostras<br />
de sangue coletadas com mais de<br />
4 semanas após o resultado positivo de<br />
PCR (n = 48) e uma sensibilidade global<br />
de 93,2% (95% CI 84,8–97,8) para<br />
todas as amostras testadas no estudo.<br />
Em todos os cenários, a sensibilidade<br />
do Ensaio Atellica IM COV2T foi igual ou<br />
maior a dos outros métodos testados.<br />
Estudo de sorologia do SARS-CoV-2<br />
da Public Health England (PHE)3<br />
Tabela 1. Diagnóstico de sensibilidade de sete testes de anticorpos baseados em nucleoproteína e<br />
glicoproteína para SARS-CoV-2.<br />
cos de sensibilidade (≥98% em casos<br />
confirmados por PCR coletados ≥20<br />
dias após o aparecimento dos primeiros<br />
sintomas) e especificidade (≥98%<br />
em amostras coletadas >6 meses antes<br />
da identificação dos primeiros casos<br />
de infecção por SARS-CoV-2) com<br />
base no Perfil de Produto Alvo (TPP)<br />
da Agência Regulatória de Medicamentos<br />
e Produtos de Saúde (MHRA)<br />
do Reino Unido para “imunoensaios<br />
enzimáticos” para SARS-CoV-2. A avaliação<br />
foi conduzida pela PHE (Porton<br />
Down) e pela Universidade de Oxford<br />
e pelo NHS Foundation Trust do Hospital<br />
Universitário de Oxford em Maio<br />
e Junho de 2020. Este estudo foi o<br />
primeiro a avaliar os ensaios comerciais<br />
automatizados de sorologia para<br />
SARS-CoV-2 em um conjunto grande e<br />
bem caracterizado de amostras (aproximadamente<br />
1500).<br />
A sensibilidade foi avaliada em 536<br />
amostras positivas para infecção por<br />
SARS-CoV-2 em ≥20 dias após o<br />
início dos sintomas. A especificidade<br />
foi avaliada em 994 amostras pré<br />
pandêmicas (2015–2018) de adultos<br />
saudáveis.<br />
Conforme mostrado na Tabela 2,<br />
o Ensaio SARS-CoV-2 Total (COV2T)<br />
Siemens Healthineers Atellica IM<br />
mostrou especificidade de 99,9%<br />
(95% CI 99,4–100) e sensibilidade<br />
de 98,1% (95% CI 96,6–99,1).<br />
Em todos os casos, a especificidade<br />
e sensibilidade do Ensaio COV2T<br />
Atellica IM foram iguais ou superiores<br />
aos outros métodos testados<br />
e foi o único método, entre os quatro<br />
testados, a atender os critérios<br />
MHRA para especificidade e sensibilidade.<br />
Tabela 2. Especificidade e sensibilidade clínicas de quatro ensaios comerciais para SARS-CoV-2.<br />
Este estudo comparou quatro imunoensaios<br />
para SARS-CoV-2 comercialmente<br />
disponíveis para critérios clíni-<br />
0 80<br />
<strong>Revista</strong> NewsLab | Out/Nov 2020