BULETINUL ИЗВЕСТИЯ JOURNAL
BULETINUL ИЗВЕСТИЯ JOURNAL
BULETINUL ИЗВЕСТИЯ JOURNAL
You also want an ePaper? Increase the reach of your titles
YUMPU automatically turns print PDFs into web optimized ePapers that Google loves.
Buletinul AŞM. Ştiinţele vieţii. Nr. 3(318) 2012<br />
Genetica, Biologia moleculară şi Ameliorarea<br />
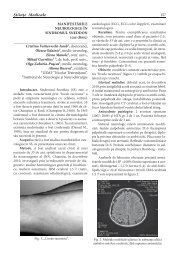
Figura 1. Modelul structurii transpozonului Activator (4565 p. b.) al Zea mays cu<br />
indicarea poziţiei primerilor utilizaţi în studiu. TIR (terminal inverted repeats) – repetiţii<br />
terminale inversate, ORFa (open reading frame) – cadru deschis citirii<br />
A fost utilizată tehnica “single primer PCR” (reacţia polimerazei în lanţ cu un<br />
singur primer), care s-a desfăşurat în 8 cicluri: denaturarea 95 0 C – 1 min., alinierea<br />
40 0 C – 2 min., extensia 72 0 C – 1 min., urmate de 35 cicluri: denaturarea 95 0 C– 0,5 min.,<br />
alinierea 65 0 C – 1 min., extensia 72 0 C – 1 min..<br />
Amestecul reactiv pentru PCR a inclus 5 pM primer, 5-10 ng ADN, soluţie tampon:<br />
66 mM tris-HCl (pH-8,4), 16 mM (NH 4 ) 2 SO 4 , 2,5 mM MgCl 2 , 0,1% Tween 20, 7 %<br />
glicerol, 100 μg · ml -1 BSA (Bovin Serum Albumin), cîte 0,2 mM de fi ecare dNTP,<br />
1,25 U Taq DNA polymeraza (Fermantas).<br />
Amplimerii au fost separaţi prin electroforeză (5-8 V/cm) în gelul de agaroză de<br />
2%, cu bromură de etidiu, în soluţia tampon de migrare tris-borat-EDTA (pH 8,3),<br />
vizualizate prin diafonoscopie în unde UV scurte (302 nm) şi fotografi ate (DigimaxS600/<br />
KenoxSamsung).<br />
Pentru calcularea dimensiunilor amplimerilor, s-a utilizat marcherul de referinţă<br />
“100 bp Ladder DNA marker” ( Axygen biosciences), softurile Paint Shop Pro 7 şi<br />
Microsoft Offi ce Excel 2003 (curba exponenţială y = a*e^bx).<br />
Rezultate şi discuţii<br />
Spectrele polimorfe de ADN ale genomurilor studiate, obţinute în rezultatul “single<br />
primer PCR” sînt prezentate în fi gura2 şi tabelul 1.<br />
Numărul fragmentelor polinucleotidice amplifi cate, de fi ecare primer, pentru<br />
diferite genomuri nu diferă semnifi cativ. Astfel primerul E16 a asigurat amplifi carea<br />
a 3-4 amplimeri specifi ci pentru fi ecare din genomurile analizate, D3: 2 - 5 şi 7 - 8<br />
fragmente, E17: 4 - 5 fragmente (excepţie Allium cepa L.), E18: 4 - 7 fragmente, E19:<br />
6 - 8 fragmente, E20: 5 - 8 fragmente, E21: 6 - 9 fragmente, E22: 7 - 11 fragmente. Se<br />
observă că, primerul E22 a determinat amplifi carea unui număr mai mare de fragmente<br />
pentru toate genomurile analizate.<br />
Fragmentele sintetizate de acelaşi primer pe ADN-ul genomic al diferitor specii<br />
diferă după mărime, ponderea fragmentelor de dimensiuni identice fi ind neglijabilă în<br />
raport cu numărul total de fragmente polimorfe. De exemplu, primerul E20 determină<br />
sinteza a 32 fragmente polimorfe şi doar a două fragmente identice, ca mărime (847<br />
p. b.), pentru Magnolia sp. şi Anethum graveolens L.. Sînt sau nu, aceste fragmente,<br />
identice în ceea ce priveşte succesiunea nucleotidelor, rămîne de a fi stabilit prin<br />
cercetări de secvenţiere.<br />
114