Genotüübi mõju NK rakkude käitumisele rakukultuuris
Genotüübi mõju NK rakkude käitumisele rakukultuuris
Genotüübi mõju NK rakkude käitumisele rakukultuuris
Create successful ePaper yourself
Turn your PDF publications into a flip-book with our unique Google optimized e-Paper software.
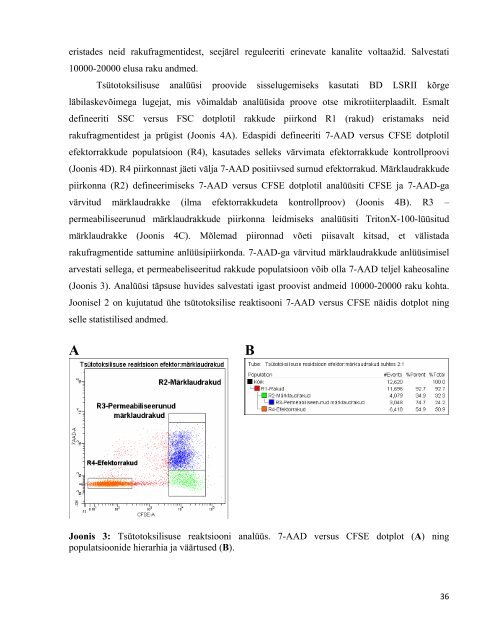
eristades neid rakufragmentidest, seejärel reguleeriti erinevate kanalite voltaažid. Salvestati10000-20000 elusa raku andmed.Tsütotoksilisuse analüüsi proovide sisselugemiseks kasutati BD LSRII kõrgeläbilaskevõimega lugejat, mis võimaldab analüüsida proove otse mikrotiiterplaadilt. Esmaltdefineeriti SSC versus FSC dotplotil <strong>rakkude</strong> piirkond R1 (rakud) eristamaks neidrakufragmentidest ja prügist (Joonis 4A). Edaspidi defineeriti 7-AAD versus CFSE dotplotilefektor<strong>rakkude</strong> populatsioon (R4), kasutades selleks värvimata efektor<strong>rakkude</strong> kontrollproovi(Joonis 4D). R4 piirkonnast jäeti välja 7-AAD positiivsed surnud efektorrakud. Märklaud<strong>rakkude</strong>piirkonna (R2) defineerimiseks 7-AAD versus CFSE dotplotil analüüsiti CFSE ja 7-AAD-gavärvitud märklaudrakke (ilma efektor<strong>rakkude</strong>ta kontrollproov) (Joonis 4B). R3 –permeabiliseerunud märklaud<strong>rakkude</strong> piirkonna leidmiseks analüüsiti TritonX-100-lüüsitudmärklaudrakke (Joonis 4C). Mõlemad piironnad võeti piisavalt kitsad, et välistadarakufragmentide sattumine anlüüsipiirkonda. 7-AAD-ga värvitud märklaud<strong>rakkude</strong> anlüüsimiselarvestati sellega, et permeabeliseeritud <strong>rakkude</strong> populatsioon võib olla 7-AAD teljel kaheosaline(Joonis 3). Analüüsi täpsuse huvides salvestati igast proovist andmeid 10000-20000 raku kohta.Joonisel 2 on kujutatud ühe tsütotoksilise reaktisooni 7-AAD versus CFSE näidis dotplot ningselle statistilised andmed.ABJoonis 3: Tsütotoksilisuse reaktsiooni analüüs. 7-AAD versus CFSE dotplot (A) ningpopulatsioonide hierarhia ja väärtused (B).36