Isolierung und Charakterisierung von Bakterien aus ...
Isolierung und Charakterisierung von Bakterien aus ...
Isolierung und Charakterisierung von Bakterien aus ...
Erfolgreiche ePaper selbst erstellen
Machen Sie aus Ihren PDF Publikationen ein blätterbares Flipbook mit unserer einzigartigen Google optimierten e-Paper Software.
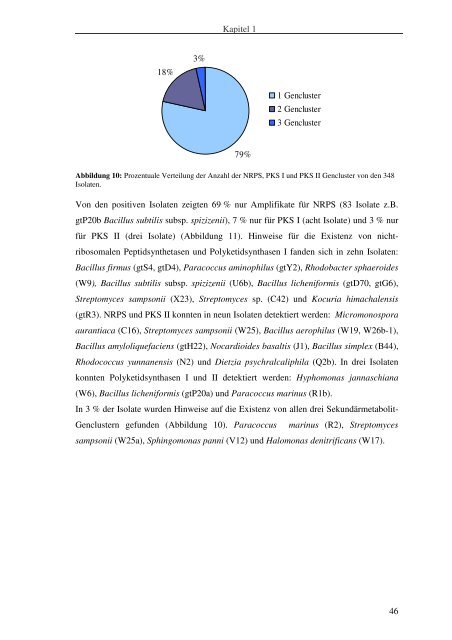
18%<br />
3%<br />
Kapitel 1<br />
79%<br />
1 Gencluster<br />
2 Gencluster<br />
3 Gencluster<br />
Abbildung 10: Prozentuale Verteilung der Anzahl der NRPS, PKS I <strong>und</strong> PKS II Gencluster <strong>von</strong> den 348<br />
Isolaten.<br />
Von den positiven Isolaten zeigten 69 % nur Amplifikate für NRPS (83 Isolate z.B.<br />
gtP20b Bacillus subtilis subsp. spizizenii), 7 % nur für PKS I (acht Isolate) <strong>und</strong> 3 % nur<br />
für PKS II (drei Isolate) (Abbildung 11). Hinweise für die Existenz <strong>von</strong> nicht-<br />
ribosomalen Peptidsynthetasen <strong>und</strong> Polyketidsynthasen I fanden sich in zehn Isolaten:<br />
Bacillus firmus (gtS4, gtD4), Paracoccus aminophilus (gtY2), Rhodobacter sphaeroides<br />
(W9), Bacillus subtilis subsp. spizizenii (U6b), Bacillus licheniformis (gtD70, gtG6),<br />
Streptomyces sampsonii (X23), Streptomyces sp. (C42) <strong>und</strong> Kocuria himachalensis<br />
(gtR3). NRPS <strong>und</strong> PKS II konnten in neun Isolaten detektiert werden: Micromonospora<br />
aurantiaca (C16), Streptomyces sampsonii (W25), Bacillus aerophilus (W19, W26b-1),<br />
Bacillus amyloliquefaciens (gtH22), Nocardioides basaltis (J1), Bacillus simplex (B44),<br />
Rhodococcus yunnanensis (N2) <strong>und</strong> Dietzia psychralcaliphila (Q2b). In drei Isolaten<br />
konnten Polyketidsynthasen I <strong>und</strong> II detektiert werden: Hyphomonas jannaschiana<br />
(W6), Bacillus licheniformis (gtP20a) <strong>und</strong> Paracoccus marinus (R1b).<br />
In 3 % der Isolate wurden Hinweise auf die Existenz <strong>von</strong> allen drei Sek<strong>und</strong>ärmetabolit-<br />
Genclustern gef<strong>und</strong>en (Abbildung 10). Paracoccus marinus (R2), Streptomyces<br />
sampsonii (W25a), Sphingomonas panni (V12) <strong>und</strong> Halomonas denitrificans (W17).<br />
46