Isolierung und Charakterisierung von Bakterien aus ...
Isolierung und Charakterisierung von Bakterien aus ...
Isolierung und Charakterisierung von Bakterien aus ...
Erfolgreiche ePaper selbst erstellen
Machen Sie aus Ihren PDF Publikationen ein blätterbares Flipbook mit unserer einzigartigen Google optimierten e-Paper Software.
Kapitel 1<br />
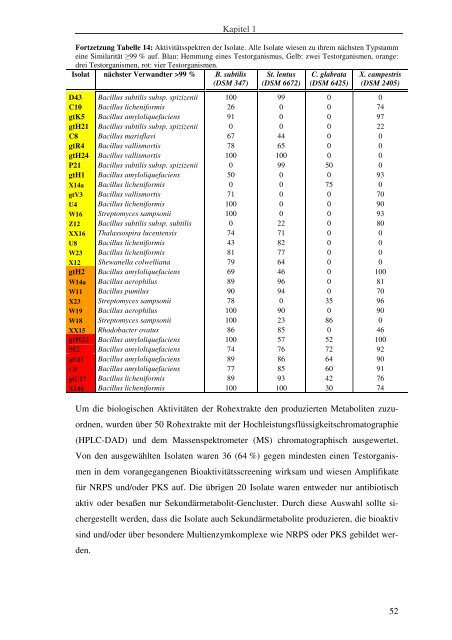
Fortzetzung Tabelle 14: Aktivitätsspektren der Isolate. Alle Isolate wiesen zu ihrem nächsten Typstamm<br />
eine Similarität ≥99 % auf. Blau: Hemmung eines Testorganismus, Gelb: zwei Testorganismen, orange:<br />
drei Testorganismen, rot: vier Testorganismen.<br />
Isolat nächster Verwandter >99 % B. subtilis<br />
(DSM 347)<br />
St. lentus<br />
(DSM 6672)<br />
C. glabrata<br />
(DSM 6425)<br />
X. campestris<br />
(DSM 2405)<br />
D43 Bacillus subtilis subsp. spizizenii 100 99 0 0<br />
C10 Bacillus licheniformis 26 0 0 74<br />
gtK5 Bacillus amyloliquefaciens 91 0 0 97<br />
gtH21 Bacillus subtilis subsp. spizizenii 0 0 0 22<br />
C8 Bacillus marisflavi 67 44 0 0<br />
gtR4 Bacillus vallismortis 78 65 0 0<br />
gtH24 Bacillus vallismortis 100 100 0 0<br />
P21 Bacillus subtilis subsp. spizizenii 0 99 50 0<br />
gtH1 Bacillus amyloliquefaciens 50 0 0 93<br />
X14a Bacillus licheniformis 0 0 75 0<br />
gtV3 Bacillus vallismortis 71 0 0 70<br />
U4 Bacillus licheniformis 100 0 0 90<br />
W16 Streptomyces sampsonii 100 0 0 93<br />
Z12 Bacillus subtilis subsp. subtilis 0 22 0 80<br />
XX16 Thalassospira lucentensis 74 71 0 0<br />
U8 Bacillus licheniformis 43 82 0 0<br />
W23 Bacillus licheniformis 81 77 0 0<br />
X12 Shewanella colwelliana 79 64 0 0<br />
gtH2 Bacillus amyloliquefaciens 69 46 0 100<br />
W14a Bacillus aerophilus 89 96 0 81<br />
W11 Bacillus pumilus 90 94 0 70<br />
X23 Streptomyces sampsonii 78 0 35 96<br />
W19 Bacillus aerophilus 100 90 0 90<br />
W18 Streptomyces sampsonii 100 23 86 0<br />
XX15 Rhodobacter ovatus 86 85 0 46<br />
gtH22 Bacillus amyloliquefaciens 100 57 52 100<br />
M2 Bacillus amyloliquefaciens 74 76 72 92<br />
gtI11 Bacillus amyloliquefaciens 89 86 64 90<br />
C4 Bacillus amyloliquefaciens 77 85 60 91<br />
gtU17 Bacillus licheniformis 89 93 42 76<br />
X14b Bacillus licheniformis 100 100 30 74<br />
Um die biologischen Aktivitäten der Rohextrakte den produzierten Metaboliten zuzu-<br />
ordnen, wurden über 50 Rohextrakte mit der Hochleistungsflüssigkeitschromatographie<br />
(HPLC-DAD) <strong>und</strong> dem Massenspektrometer (MS) chromatographisch <strong>aus</strong>gewertet.<br />
Von den <strong>aus</strong>gewählten Isolaten waren 36 (64 %) gegen mindesten einen Testorganis-<br />
men in dem vorangegangenen Bioaktivitätsscreening wirksam <strong>und</strong> wiesen Amplifikate<br />
für NRPS <strong>und</strong>/oder PKS auf. Die übrigen 20 Isolate waren entweder nur antibiotisch<br />
aktiv oder besaßen nur Sek<strong>und</strong>ärmetabolit-Gencluster. Durch diese Auswahl sollte si-<br />
chergestellt werden, dass die Isolate auch Sek<strong>und</strong>ärmetabolite produzieren, die bioaktiv<br />
sind <strong>und</strong>/oder über besondere Multienzymkomplexe wie NRPS oder PKS gebildet wer-<br />
den.<br />
52