Download - Fakultät 06
Download - Fakultät 06
Download - Fakultät 06
Sie wollen auch ein ePaper? Erhöhen Sie die Reichweite Ihrer Titel.
YUMPU macht aus Druck-PDFs automatisch weboptimierte ePaper, die Google liebt.
Inhaltsverzeichnis<br />
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
1. Einleitung und Motivation ................................................................................................. 6<br />
1.1. Geschichte und Bedeutung der Chemotaxisforschung ................................................ 6<br />
1.2. Mechanismus von Zellbewegung und Chemotaxis ..................................................... 7<br />
1.3. Zellbewegung durch 3-dimensionale Matrizes ........................................................... 9<br />
1.4. Analytische Verfahren ............................................................................................... 11<br />
1.4.1. Entwicklung analytischer Verfahren .................................................................. 11<br />
1.4.2. Anforderungsprofil an neue Systeme. ................................................................ 14<br />
1.5. Diffusion in Flüssigkeiten ......................................................................................... 16<br />
2. Zielsetzung der Arbeit ...................................................................................................... 17<br />
3. Material und Methoden .................................................................................................... 18<br />
3.1. Das µ-Slide Chemotaxis 3D ...................................................................................... 18<br />
3.1.1. Physikalische Eigenschaften .............................................................................. 18<br />
3.1.2 Zellkultur in µ-Slides ......................................................................................... 20<br />
3.2. Zellkultur ................................................................................................................... 21<br />
3.2.1. Kultivierung und Differenzierung von HL-60 Zellen ........................................ 21<br />
3.2.2. Kultivierung von HT-1080 Zellen ...................................................................... 22<br />
3.3. Mikroskopietechniken ............................................................................................... 24<br />
3.3.1. Zellmikroskopie ................................................................................................. 24<br />
3.3.1.1. Nieder- und Hochaufgelöster Phasenkontrast ............................................. 24<br />
3.3.1.2. Fluoreszenzmarkierung mit LifeAct-tag GFP2/RFP .................................. 24<br />
3.3.2. Diffusionsstudien ............................................................................................... 25<br />
3.3.2.1. Konfokale Laser Scanning Mikroskopie .................................................... 25<br />
3.3.2.2. Fluoreszenz-Korrelations Spektroskopie .................................................... 26<br />
3.4. Verwendete Gelmatrizes ............................................................................................ 29<br />
3.4.1. Collagen ............................................................................................................. 29<br />
3.4.2. Synthetische Hydrogele ..................................................................................... 29<br />
3.5. Chemotaxis mit HL-60 und HT-1080 ........................................................................ 32<br />
3.5.1. Durchführung der Experimente .......................................................................... 32<br />
3.5.2. Auswertung und Datenanalyse ........................................................................... 34<br />
4. Ergebnisse ........................................................................................................................ 36<br />
4.1. Errichtung und Messung von definierten Konzentrationsprofilen ............................ 36<br />
im µ-Slide Chemotaxis 3D. .................................................................................................. 36<br />
4.1.1. Untersuchung von Sonden-Matrix Interaktionen ................................................... 36<br />
4.1.2. LSM - Messungen mit Alexa 488 und FITC-Dextran ....................................... 40<br />
4.1.3. FCS-Messungen ................................................................................................. 46<br />
4.2. Zellkultur und Migration im Chemotaxis 3D µ-Slide ................................................... 52<br />
1.Einleitung und Motivation 4
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
4.2.1. HT-1080 Zellen in Gelmatrizes .......................................................................... 52<br />
4.2.1.1. Chemotaxis in Collagen I Matrizes: ........................................................... 52<br />
4.2.1.3. Chemotaxis in synthetischen Gelmatrizes .................................................. 55<br />
4.2.2. HL-60 Zellen in Gelmatrizes ............................................................................. 58<br />
4.2.3. Migrationsanalyse durch Aktinfärbung mit LifeAct-tag GFP2/RFP ................. 63<br />
5. Diskussion und Ausblick .................................................................................................. 65<br />
5.1. Bewertung der Ergebnisse ......................................................................................... 65<br />
5.1.1. Diffusionsmessungen ......................................................................................... 65<br />
5.1.2. Chemotaxis-experimente mit HT-1080 und HL-60 Zellen ................................ 66<br />
5.2 Weiterführende Ansätze ............................................................................................. 72<br />
6. Zusammenfassung ............................................................................................................ 73<br />
7. Abbildungsverzeichnis: .................................................................................................... 74<br />
8. Tabellenverzeichnis .......................................................................................................... 78<br />
9. Abkürzungsverzeichnis .................................................................................................... 79<br />
10. Anhang ......................................................................................................................... 80<br />
11. Literaturverzeichnis ...................................................................................................... 88<br />
Selbstständigkeitserklärung ...................................................................................................... 91<br />
1.Einleitung und Motivation 5
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
1. Einleitung und Motivation<br />
1.1. Geschichte und Bedeutung der Chemotaxisforschung<br />
Erste Beobachtungen und Erkenntnisse bezüglich der Fortbewegung zellulärer Organismen<br />
wurden schon während der Entwicklung von Mikroskopen im 17. Jahrhundert durch den<br />
niederländischen Mikroskopbauer und Naturforscher Antoni van Leeuwenhoek gewonnen. In<br />
seinen Studien beobachtete er Bakterien und Protozoen, sowie menschliche und tierische<br />
Spermatozoen. Erheblich später, vor ungefähr 130 Jahren, befasste sich die Wissenschaft,<br />
vertreten durch die beiden deutschen Forscher Theodor Wilhelm Engelmann und Wilhelm<br />
Pfeffer erstmals gezielt und eingehender mit der Chemotaxis von Bakterien. Ihre Studien<br />
eröffneten den Zugang zu einem neuen Forschungsgebiet, das in den folgenden Jahre<br />
wichtige Erkenntnisse vor allem im Bereich der Medizin ermöglichte.<br />
Heute ist bekannt, dass Chemotaxis im menschlichen Körper unter Anderem wichtige Stufen<br />
der embryonalen Entwicklung beeinflusst [Singer & Kupfer, 1987], die Bekämpfung einer<br />
Infektion durch das Immunsystem koordiniert und eine entscheidende Rolle bei der<br />
Verbreitung und Versorgung bösartiger Tumore spielt [Schiffmann , 1982] [Downey, 1994].<br />
Die Forschungsarbeit auf dem Gebiet der Chemotaxis stellt inzwischen eine der wichtigsten<br />
Datensammlungen dar, um neue Therapieansätze zur Bekämpfung von malignen Tumoren<br />
oder bakteriellen Infektionen zu entwickeln.<br />
Es sollte diesbezüglich also nicht verwundern, dass inzwischen verschiedenste Ansätze zur<br />
detaillierten Beobachtung chemotaktischer aktiver Zellen und deren Reaktion auf negative<br />
oder positive Stimuli in der Forschung etabliert sind. Bevor die wichtigsten unter ihnen kurz<br />
vorgestellt werden, gilt es aber zuerst einen kurzen Blick auf die Bewegungsmechanismen<br />
von Zellen zu werfen.<br />
1.Einleitung und Motivation 6
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
1.2. Mechanismus von Zellbewegung und Chemotaxis<br />
Bei der Betrachtung und Analyse zellulärer Bewegungen ist zwischen den unterschiedlichen<br />
Bewegungsprofilen grundlegend zu unterscheiden. Chemokinese stellt die Fähigkeit zu<br />
willkürlicher und ungerichteter Bewegung, als Reaktion auf die Zugabe eines bestimmten<br />
Stoffes dar. [Becker, 1977]. Erfolgt die Ausrichtung des Bewegungsprofiles von Zellen gezielt<br />
und in Abhängigkeit eines zugegebenen Stoffes spricht man von Chemotaxis. [Engelmann,<br />
1881] [Pfeffer, 1884/1888]. Die Bewegung kann dabei auf den Stoff zu erfolgen<br />
(Chemoattraktant), oder von ihm fort (Chemorepellent). Dabei wird noch unterschieden, ob<br />
die chemotaktische Reaktion durch Stoffgradienten in der flüssigen Phase (Chemotaxis) oder<br />
bezüglich der Anzahl der Adhäsionsmoleküle an einer Oberfläche induziert wird<br />
(Haptotaxis). [Carter, 1967].<br />
Abbildung 1 gibt einen Überblick über die unterschiedlichen Formen der Fortbewegung<br />
eukaryontischer Zellen.<br />
Abbildung 1: Die verschiedenen Arten stimulierter Zellbewegungen eukaryontischer Zellen in<br />
schematischer Darstellung [Kőhidai L., 20<strong>06</strong>]<br />
Es bestehen grundlegende Unterschiede in der Bewegungsmechanik von Prokaryonten und<br />
Eukaryonten. Diese Arbeit befasst sich allerdings ausschließlich mit der Chemotaxis von<br />
Eukaryonten.<br />
Eukaryontischen Zellen steht zur „Erkundung“ ihres Umfelds ein umfangreiches Netzwerk an<br />
1.Einleitung und Motivation 7
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Oberflächenrezeptoren zur Verfügung. Im Idealfall, bei einer mittleren Chemoattraktand<br />
Konzentration, die in etwa der Dissoziations-Konstante des Rezeptor-Ligand Komplexes<br />
entspricht, kann so über die Zelllänge ein Gradient von etwa 2% „erfühlt“ werden. [Devreotes<br />
et al., 1988]. So vielfältig dabei stimulierende und hemmende Mechanismen auch sein<br />
mögen, läuft die dadurch ausgelöste Reaktion in den meisten Fällen nach einem simultanen<br />
Schema ab:<br />
– Protrusion/Fortsatzbildung (Lamellipodien)<br />
– Adhäsion der Zellfront.<br />
– Translokation/Umlagerung der Zellkörpers<br />
– Retraktion/Ablösen des rückwärtigen Zellteils<br />
Innerhalb der Zelle kommt es während der Bewegung zu einer Umlagerung des Aktingerüsts.<br />
Die chemotaktisch induzierte Ausrichtung einer Zelle und die Ausbildung einer Zellfront<br />
sowie eines rückwärtigen Zellteils bezeichnet man als Polarisation. [Zigmond et al.,<br />
1981],[Weiner, 2002]. Die Polarisation eines neutrophilen Granulozyten, ausgelöst durch den<br />
chemotaktischen Stimulus aus einer Mikropipette ist in Abbildung 2 gezeigt.<br />
Abbildung 2: Polarisation eines neutrophilen Granulozyten als Reaktion auf eine Stimulation mit N-<br />
Formylmethionyl-Lencyl-Phenylalanin (fMLP) aus einer Mikropipettenspitze (weisser Punkt), nach (A) 5s,<br />
(B) 30s, (C) 81s, (D) 129s. Der weiße Balken in (A) entspricht 5µm. [Weiner et al., 1999]<br />
1.Einleitung und Motivation 8
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
1.3. Zellbewegung durch 3-dimensionale Matrizes<br />
Unter natürlichen Bedingungen bewegen sich wandernde Zellen in vivo durch eine aus<br />
mehreren Komponenten bestehende Matrix. Bei dem Versuch den Vorgang der Chemotaxis<br />
auf in vitro Modelle zu übertragen, beschränkten sich die meisten Modelle der vergangenen<br />
Jahre darauf, die Bewegung von Zellen über eine beschichtete Oberfläche zu analysieren.<br />
Diese Art Versuchsansatz hat den Vorteil, dass die Bewegung der Zellen abhängig von einem<br />
überschaubaren Maß an einstellbaren Parametern erfolgt und sich hauptsächlich über die<br />
Fähigkeit zur Adhäsion und De-adhäsion definiert. Inzwischen hat man festgestellt dass<br />
zwischen dem „Kriechen“ von Zellen entlang einer Oberfläche und der Bewegung durch eine<br />
vernetzte, 3-dimensionale Matrix wichtige Unterschiede bestehen.<br />
Das morphologische Erscheinungsbild von Zellen in einer 3-dimensionalen Matrix<br />
unterscheidet sich grundlegend von adhärenten Zellen auf einer beschichteten Oberfläche.<br />
Adhärente Zellen besitzen einen großen Durchmesser und weitreichende Fortsätze<br />
(Pseudopodien). Zellen, die komplett von einer Gelmatrix umschlossen werden, erscheinen<br />
kleiner, rundlich und bilden deutlich kürzere Pseudopodien aus. Je nach Beschaffenheit der<br />
Matrix und der Größe der Zelle findet die Fortbewegung entweder durch eine morphologische<br />
Anpassungsreaktion (lange spindelförmige Verformung der Zellen), eine Ummodellierung der<br />
umgebenden Matrix, zum Beispiel durch Matrix-metalloproteasen (MMPs) , oder eine<br />
Mischung aus beiden Prozessen statt. Der Ablauf und die Geschwindigkeit dieses Prozesses<br />
variiert in Abhängigkeit des untersuchten Zelltyps und der Beschaffenheit von dessen<br />
natürlicher Umgebung. Dieser Umstand resultiert in der Notwendigkeit einer weiteren<br />
Einteilung chemotaktisch aktiver Zellen.<br />
„langsame Zellen“<br />
Die Geschwindigkeit mit der sich eine Zelle durch ein Gel bewegt ist im weitesten Sinne<br />
umgekehrt proportional zu ihrer Größe. Um sich zu bewegen müssen große, langsame Zellen<br />
die umgebende Gelmatrix „umzuformen“. Meist geschieht das unter Verwendung einer<br />
vielfältigen Palette an Proteasen, mit denen die Quervernetzung der zu durchwandernden<br />
Struktur aufgelöst werden kann. Derartige Zellen bewegen sich mit Geschwindigkeiten im<br />
Bereich von 0,5 – 1µm pro Minute. Als „langsam migrierend“ zu klassifizierende Zellen sind<br />
unter Anderem diverse Endothelzellen, mit etwa 10-20µm/h, oder Fibroblasten mit ungefähr<br />
1.Einleitung und Motivation 9
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
30-40µm/h einzuordnen. [Martin et al., 2001] [Even-Ram et al., 2005]. Besonders<br />
hervorzuheben sind hierbei HUVEC-Zellen (human umbilicar vascular endothelial cells), die<br />
bei der Tumorforschung und Angiogenese eine zentrale Rolle spielen oder die humane<br />
Fibrosarkom Zelllinie HT-1080, die im Rahmen dieser Arbeit stellvertretend für Versuche mit<br />
langsamen Zellen betrachtet werden soll.<br />
„schnelle Zellen“<br />
Zellen des Immunsystem die, anhand ihrer natürlichen Bestimmung, schnell am Einsatzort<br />
sein müssen und außerdem Gewebe aller Art durchdringen, migrieren zwischen 10 und 40-<br />
fach so schnell wie oben beschriebene Zellen. Aufgrund der, in der Regel geringeren Größe,<br />
bewegen sich schnelle Zellen, wie zum Beispiel Leukozyten ohne Matrixummodellierung und<br />
nur durch eine morphologische Anpassungsreaktion durch die Gelmatrix. [Friedl et al., 2000]<br />
Innerhalb der Zellen zeigt sich eine höhere Tendenz zur Polarisation und das Ausbilden und<br />
Ablösen von Adhäsionspunkten findet deutlich beschleunigt statt. [Even-Ram et al., 2005]<br />
So liegen die Literaturangaben für die Migrationsgeschwindigkeit von beispielsweise T-<br />
Lyphozyten bei etwa 15 µm/min [Lefort et al., 2010] und die neutrophiler Granulozyten bei<br />
etwa 24-27 µm/min [Foxman et al. 1999]. Ambivalent dazu können auch Zellen der HL-60<br />
Leukämie Zellinie betrachtet werden, die zu eingeschränkt funktionellen Neutrophilen<br />
differenziert werden können und in dieser Arbeit stellvertretend für schnelle Zelllinien<br />
verwendet werden sollen.<br />
1.Einleitung und Motivation 10
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
1.4. Analytische Verfahren<br />
Die fortlaufende Veröffentlichung neuer Erkenntnisse auf dem Gebiet der Zellmigration und<br />
Chemotaxis erfordert die stetige Entwicklung neuer, dem Fokus des Interesses angepasster<br />
Methoden. Im Folgenden soll ein kurzer Überblick über die Entwicklung der<br />
Analysemethoden in den vergangenen Jahren gegeben werden.<br />
1.4.1. Entwicklung analytischer Verfahren<br />
Eines der ersten Systeme zur Analyse des chemotaktischen Potentials verschiedener<br />
Substanzen und zugleich der Pionier einer andauernden Entwicklung sogenannter 2-Kammer<br />
Systeme, war die von Steve Boyden entwickelte Boyden-Kammer [Boyden, 1962]. Das<br />
zugrunde liegende Prinzip dieser Systeme sind 2 Flüssigkeitsreservoirs, von denen eines mit<br />
dem zu untersuchenden Stoff und das andere mit Referenzlösung gefüllt ist. Im Fall der<br />
ursprünglichen Boyden-Kammer werden die beiden Flüssigkeiten durch eine Membran<br />
getrennt, deren Porengröße so gewählt wurde, dass darauf liegende Zellen sie nur durch<br />
aktive Migration überwinden können. Später wurde das System dahingehend modifiziert, dass<br />
man die poröse Membran durch eine Gelmatrix ersetzte, welche die Zellen durchwandern<br />
mussten, um in Richtung der höheren Chemoattraktant Konzentration zu gelangen.<br />
Der Aufbau einer modifizierten Boyden-Kammer ist in Abbildung 3 schematisch dargestellt:<br />
Abbildung 3: Modifizierte Boyden-Kammer mit Gelmatrix. C0 entspricht Medium ohne Chemoattraktant,<br />
C100 Medium mit Chemoattraktant.<br />
Ausgewertet wird in beiden Verfahren rein statistisch durch die Ermittlung der Zellzahl auf<br />
der Unterseite der Membran (für adhärente Zellen) und/oder in den beiden Reservoiren (für<br />
Suspensionskulturen).<br />
1.Einleitung und Motivation 11
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Vorteile des Verfahrens liegen klar bei der Vermeidung umständlicher bildgebender Verfahren.<br />
Eine Beobachtung der Zellen während des Migrationsvorganges und damit eine Analyse von<br />
Veränderungen von Morphologie, Geschwindigkeit oder Direktionalität der Bewegung sind<br />
nach dieser Methode jedoch nicht möglich. Je nach Zelltyp muss die Porengröße der<br />
Membran neu gewählt werden. Dieser Umstand macht das Verfahren für Zellen mit wenig<br />
bekannten Parametern ungeeignet.<br />
Eine Weiterentwicklung der von Boyden eingeführten 2-Kammer Techniken und zugleich<br />
einen großen Schritt in der Entwicklung der Chemotaxis-Assays stellt die sogenannte<br />
Zigmond-Kammer dar, mit der es erstmals möglich war, die Zellen während der Migration<br />
unter dem Mikroskop zu beobachten und die Anzahl der migrierenden Zellen festzuhalten<br />
[Zigmond, 1977]. Bei der Zigmond-Kammer sind die Kammer mit Hungermedium und die<br />
Kammer mit Chemoattraktant über einen schmalen Spalt verbunden, der zwischen der Brücke<br />
der beiden Kammern und einem darüber liegenden Deckglas entsteht. Durch<br />
Diffusionsprozesse entsteht innerhalb dieses Spalts, in welchen die Zellen zur<br />
Versuchsdurchführung eingebracht werden, ein Stoffgradient, der von den Zellen detektiert<br />
werden kann.<br />
Der Aufbau der Zigmond-Kammer ist in Abbildung 4 skizziert<br />
Abbildung 4: Aufbauskizze einer Zigmond-Kammer [Zigmond, 1977]<br />
Es besteht die Möglichkeit den Spalt zwischen den Kammern mit einer Agarschicht zu füllen,<br />
durch welche sich die Zellen bei der Migration bewegen müssen und dadurch eine Art 3D-<br />
Chemotaxis-Assay durchzuführen. Daraus abgeleitete Methoden, wie die sogenannte Dunn-<br />
Kammer folgen einem ähnlichen Prinzip bei leicht unterschiedlichem Aufbau. Der Gradient<br />
zwischen beiden Kammern kann für lange Zeit stabil gehalten werden. Durch das Aufbringen<br />
des Deckglases mit den Zellen kann es jedoch bei unvorsichtiger Handhabung zu einer<br />
Durchmischung der Lösung kommen. Es bedarf daher einiger Erfahrung reproduzierbare<br />
Versuchsbedingungen bereitzustellen.<br />
1.Einleitung und Motivation 12
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Die moderne Kunststofftechnologie und hochpräzise Fertigungsmethoden haben es in den<br />
letzten Jahren ermöglicht, Kanäle mit einer Höhe von wenigen µm in Kunststoffträger zu<br />
prägen. Jeon et al. veröffentlichte vor einigen Jahren eine Methode, bei der in einem µ-Kanal<br />
durch ein Netzwerk aus verbundenen µ-Kanälen Stoffflüsse unterschiedlicher<br />
Chemoattraktant-Konzentrationen zu einem statischen Gradienten angeordnet werden können<br />
[Jeon et al., 2002]. Die Methode ist in Abbildung 5 am Beispiel von Neutrophilen<br />
schematisch dargestellt.<br />
Abbildung 5: (A) Schematische Darstellung der Kanalanordnung zur Generierung eines Stoffgradienten<br />
und (B) 3-Dimensionale Ansicht des Beobachtungsbereiches mit Zellen in einem Stoffgradienten. [Jeon et<br />
al., 2002]<br />
Ein großer Vorteil dieser Methode liegt darin, dass der Gradient durch den anliegenden Fluss<br />
von Anfang des Experiments an zur Verfügung steht und durch die fehlende Möglichkeit<br />
eines Konzentrationsausgleichs statisch bleibt. Das System ist jedoch relativ aufwendig und<br />
teuer was einer weitläufigen Verbreitung bisher im Wege steht.<br />
Neben den genannten Verfahren gibt es noch Untersuchungsmethoden mit Zellen in<br />
Agarosegelen oder unter Verwendung eines chemotaktischen Stimulus aus einer<br />
Mikropipettenspitze. Jede von ihnen bietet speziellen Bedürfnissen angepasste Vor- und<br />
Nachteile. Es lassen sich aber unabhängig davon einige Punkte hervorheben, die bei der<br />
Entwicklung neuer Analysemethoden heute fast immer eine Rolle spielen.<br />
1.Einleitung und Motivation 13
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
1.4.2. Anforderungsprofil an neue Systeme.<br />
Das anzustrebende Eigenschaftsprofil für neue Chemotaxis-Assays setzt sich in erster Linie<br />
aus den Vorzügen bestehender Methoden zusammen.<br />
Folgende Eigenschaften sollen dabei vereint werden:<br />
– gute mikroskopische Eigenschaften (Live Cell Imaging)<br />
– kostengünstig<br />
– anwendungsfreundlich<br />
– reproduzierbar<br />
– stabile Versuchsbedingungen<br />
Der Trend zur Annäherung der in vitro Chemotaxis an die Bedingungen in vivo erfordert von<br />
neuen Untersuchungsmethoden außerdem, wie bereits erwähnt, die Möglichkeit zur Analyse<br />
von 3-dimensionaler Zellmigration in einer vernetzten Struktur unter Verwendung einer<br />
Gelmatrix.<br />
Die ibidi GmbH entwickelt seit einigen Jahren Objektträger mit integrierten µ-Kanälen auf<br />
Basis modernster Kunststofftechnologie. Die Systeme arbeiten mit Kanälen einer Höhe von<br />
wenigen µm in Objektträgern aus hochwertigem Kunststoff und bieten dadurch einige<br />
Vorteile:<br />
– stabil und bruchresistent<br />
– kostengünstig<br />
– gute optische Eigenschaften<br />
– geringer Einsatz an Zellen/Medien notwendig<br />
Vor etwa 1 Jahr wurde das µ-Slide Chemotaxis 3D vorgestellt. Das Produkt ist eine<br />
Weiterentwicklung des µ-Slide Chemotaxis, einem Objektträger mit einem integrierten 2-<br />
Kammersystem, das durch einen µ-Kanal verbunden ist und in welchem Zellen einem<br />
Stoffgradienten ausgesetzt werden können. (siehe auch Punkt 3.1.1.) Mit dem µ-Slide 3D ist<br />
es möglich, den Kanal mit einer Gelmatrix zu füllen und die Migration der Zellen damit in die<br />
3. Dimension zu erweitern.<br />
1.Einleitung und Motivation 14
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Durch die geringe Höhe der µ-Kanäle kann die Diffusion zwischen den beiden Kammern nur<br />
sehr langsam stattfinden und der erzeugte Gradient, in welchem sich die Zellen bewegen<br />
sollen, bleibt lange Zeit annähernd konstant.<br />
Die Betrachtung der Diffusion in den µ-Slides soll in dieser Arbeit eine zentrale Rolle spielen.<br />
Aus diesem Grunde soll im Folgenden kurz auf einige Zusammenhänge der Stoffdiffusion in<br />
Flüssigkeiten eingegangen werden.<br />
1.Einleitung und Motivation 15
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
1.5. Diffusion in Flüssigkeiten<br />
Die treibende Kraft bei der Bewegung von Teilchen im Raum ist die Brownsche<br />
Molekularbewegung. Liegt eine ungleichmäßige Konzentrationsverteilung eines gelösten<br />
Stoffes in einem Flüssigkeitsvolumen vor, tritt ausgelöst durch die Brownsche<br />
Molekularbewegung nach einem bestimmten Zeitraum ein Konzentrationsausgleich auf.<br />
Die differentielle Steilheit eines vorliegenden Konzentrationsunterschieds wird als Gradient<br />
(gradC) bezeichnet. Der durch den Vorgang dieses Konzentrationsausgleichs auftretende<br />
Teilchenstrom (j) wird durch das 1. Ficksche Gesetz beschrieben:<br />
j = -DgradC<br />
Zur Beschreibung instationärer, wie zum Beispiel zeitlich veränderlicher Diffusion zwischen<br />
endlichen Reservoiren wird das 2. Ficksche Gesetz herangezogen, welches auch als<br />
Diffusionsgleichung bezeichnet wird und die zeitliche und räumliche Entwicklung des<br />
Gradienten beschreibt.<br />
.<br />
D bezeichnet dabei den Diffusionskoeffizienten des gelösten Stoffes in m 2 /s. Er gibt an,<br />
welche Fläche in einer bestimmten Zeit durchdrungen wird und ist abhängig von der Größe<br />
des diffundierenden Moleküls (R0), der Temperatur (T) und der dynamischen Viskosität (µ)<br />
der umgebenden Flüssigkeit.<br />
Zellen können unterschiedliche Stoffkonzentrationen über mehrere Größenordnungen hinweg<br />
erkennen. Die Orientierung erfolgt dabei auf Grundlage des relativen Stoffgradienten.<br />
Der relative Gradient wird als gradC/C beschrieben und bezieht sich auf den<br />
Konzentrationsunterschied unabhängig von der Absolutkonzentration. Wie sensitiv Zellen auf<br />
einen Gradienten reagieren können hängt davon ab, wie nahe die Absolutkonzentration des<br />
vorhandenen Chemoattractant an der Dissoziationskonstante des Rezeptor-Ligand Komplexes<br />
der Zellen liegt. Abhängig von der verwendeten Zelllinie können Zellen einen relativen<br />
Gradienten von etwa 1-2% über die Länge des Zellkörpers aufspüren. [Mato et al., 1975],<br />
[Tranquillo et al., 1988], [Fisher et al., 1989]<br />
1.Einleitung und Motivation 16
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
2. Zielsetzung der Arbeit<br />
Aus dem Wissen um die Diffusionsvorgänge in Flüssigkeiten und deren Abhängigkeit von der<br />
Molekülgröße, leitet sich die Fragestellung ab, ob die Wahl des verwendeten Chemoattraktant<br />
Einfluss auf die Etablierung des Diffusionsprofils im µ-Slide Chemotaxis 3D hat. Zudem soll<br />
untersucht werden, ob durch die, mit Einführung des µ-Slide Chemotaxis 3D neu geschaffene<br />
Möglichkeit, eine Gelmatrix in den Kanalbereich des µ-Slides einzubringen, eine<br />
Diffusionsbarriere erzeugt.<br />
Durch konfokale mikroskopische Aufnahmen mit diffundierenden Fluoreszenzfarbstoffen soll<br />
dazu eine Analyse der Entwicklung des Diffusionsprofils abhängig von der Versuchsdauer,<br />
der verwendeten Matrix und der diffundierenden Stoffe durchgeführt werden. Anhand der<br />
gewonnenen Daten soll eine Einschätzung abgegeben werden, ob, und in welchem zeitlichen<br />
Rahmen sich das µ-Slide Chemotaxis 3D für Chemotaxis Experimente mit Zellen in einer<br />
Gelmatrix eignet.<br />
Parallel dazu soll versucht werden, anhand ausgewählter Zelllinien, stellvertretend für<br />
langsam und schnell migrierende Zellen, 3D Chemotaxis Experimente zu etablieren und die<br />
erzeugten Daten mit bestehenden 2D Chemotaxis Daten zu vergleichen.<br />
2.Zielsetzung der Arbeit 17
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
3. Material und Methoden<br />
Eine Liste aller, im Rahmen der vorliegenden Arbeit verwendeten Geräte, Chemikalien und<br />
Verbrauchsmaterialien befindet sich in Anhang H auf Seite 101f.<br />
3.1. Das µ-Slide Chemotaxis 3D<br />
3.1.1. Physikalische Eigenschaften<br />
Das Chemotaxis 3D µ-Slide der Firma ibidi ist ein, speziell für chemotaktische<br />
Untersuchungen in 3-dimensionaler Umgebung entwickelter Objektträger aus optisch<br />
hochwertigem, biokompatiblem Kunststoff. In den Objektträger mit standardmikroskopischen<br />
Maßen sind nummerierte 2-Kammer Systeme für 3 separate Chemotaxis Experimente<br />
eingebettet. Der Aufbau des Systems ist in Abbildung 6 schematisch dargestellt.<br />
Abbildung 6: Schematische Darstellung des µ-Slide Chemotaxis 3D in der Draufsicht. Der Nummern von<br />
1-3 bezeichnen die 3 unabhängigen 2-Kammer Systeme. Die Buchstaben A und B stellen die Füllstutzen<br />
des Mittelkanals, C-F diejenigen der seitlichen Kammern dar. [Ibidi GmbH, 2010]<br />
Jedes dieser 3 Module setzt sich aus 2 Reservoiren zusammen die über einen, parallel der<br />
Längsseiten verlaufenden, 70µm hohen und 1mm breiten Mikrokanal verbunden sind. Die<br />
beiden Reservoire und der dazwischenliegende Kanal können durch Einfüllstutzen an den<br />
äußeren Ecken beziehungsweise den beiden begrenzenden Enden des Kanals einzeln gefüllt<br />
werden. Nach dem Einfüllen der betreffenden Lösungen werden die Stutzen durch<br />
eingepasste Stöpsel steril verschlossen. Das Fassungsvermögen des Kanals beträgt 6µl das der<br />
beiden Reservoirs jeweils 60 µl. Die zu untersuchenden Zellen befinden sich entweder<br />
adhärent auf dem Boden des Kanals oder eingebettet in eine eingefüllte Gelmatrix.<br />
3.Material und Methoden 18
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Wird eines der beiden Reservoirs mit Chemotaxin und das verbleibende mit Referenzlösung<br />
befüllt, so stellt sich ein definierter Stoffgradient über den Querschnitt des Kanals ein. Durch<br />
den geringen Querschnitt des Kanals soll der Gradient für lange Zeit konstant bleiben. Der<br />
Boden, der beide Reservoire sowie den Kanal nach unten abschließt besteht aus einer dünnen<br />
Kunststofffolie, die in ihren optischen Eigenschaften mit, in der Mikroskopie verwendeten<br />
Deckgläsern, vergleichbar ist. Das System wurde für die Verwendung mit inversen<br />
Mikroskopen konzipiert.<br />
A<br />
B<br />
Abbildung 7: Schematische Schnittdarstellung des µ-Slide Chemotaxis 3D entlang der x-Achse mit (A)<br />
Zellen in einer 3D-Gelmatrix und (B) einer 2D-Zellschicht auf der Kanaloberfläche. [Ibdi GmbH, 2010]<br />
3.Material und Methoden 19
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
3.1.2 Zellkultur in µ-Slides<br />
Durch die Kunststofffolie, die den Boden des Slides bildet kann Gasaustausch mit der<br />
Umgebungsluft beziehungsweise der CO2-angereicherten Atmossphäre eines Brutschrankes<br />
stattfinden. Der Kunststoff des Slides kann bei ausreichender Equilibrierung in einer<br />
beheizten Atmosphäre, in der eingefüllten Matrix gelöste Gase aufnehmen. Der Entstehung<br />
von Luftblasen und störenden Gaseinschlüssen wird somit entgegengewirkt. Während der<br />
chemotaktischen Untersuchungen können Zellen mit minimalem Aufwand über ein Heiz- und<br />
Begasungssystem mit optimierten Überlebensfaktoren versorgt werden. Dadurch können in<br />
den µ-Slides Bedingungen wie in einem Brutschrank eingestellt werden. Die Zellen bleiben<br />
länger vital und können auch über eine Zeitspanne von mehreren Tagen hinweg beobachtet<br />
werden. Die Höhe des Kanals entspricht mit 70µm in etwa dem Schärfebereich von häufig<br />
verwendeten Objektiven von 4-facher bis 10-facher Vergrößerung. Die verwendete<br />
extrazelluläre Matrix beeinflusst Morphologie und Migrationsverhalten der Zellen, so dass die<br />
Eigenschaften 3-dimensionaler Zellmigration untersucht werden können, ohne dass sich die<br />
Zellen dabei aus der Schärfeebene gängiger Objektive (10x/20x) bewegen. Die hydrophobe<br />
Oberfläche des Kunststoffes wurde mit einer Beschichtung aus Collagen, Fibronektin oder<br />
Poly-L-Lysin versehen, um adhärenten Zellen eine Kontaktfläche bereitzustellen.<br />
Im Bezug auf Biokompatibilität und Eignung zur Zellkultur zeichnet sich das µ-Slides<br />
Chemotaxis 3D durch folgende Eigenschaften aus.<br />
– Gasaustausch möglich<br />
– geringe Volumina nötig<br />
– beschichtbar und biokompatibel<br />
3.Material und Methoden 20
3.2. Zellkultur<br />
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
3.2.1. Kultivierung und Differenzierung von HL-60 Zellen<br />
Kultivierung der Zellen<br />
Für chemotaktische Experimente standen HL-60 Leukämie-Zellen vom Wildtyp und<br />
transfiziert mit LifeAct-tag GFP zur Verfügung. Beide Varianten wurden in 75 cm 2<br />
Zellkulturflaschen (roter Verschluss) mit RPMI-1640 Medium mit L-Glutamin der Firma C-C<br />
Pro kultiviert. Die genaue Zusammensetzung des Mediums ist in Anhang F auf Seite 99<br />
dargestellt. Dem Medium wurden nachträglich 10% Foetales Kälberserum (FCS) und 1%<br />
Penicilin-Streptomycin Lösung hinzugefügt. Soweit nicht anders dargestellt, kann im<br />
Folgenden davon ausgegangen werden, dass RPMI 1640 Medium immer mit den<br />
beschriebenen Zusätzen verwendet wurde. Die Zellen wurden in einer bestehenden<br />
Suspensionskultur der Passage 3 übernommen und in einem Brutschrank bei 37° C und 5%<br />
CO2 stabil auf einer Zellzahl von etwa 1x10 6 Zellen/ml gehalten. Die Zelldichte wurde dazu<br />
im 2-Tagesrythmus mit dem Phasenkontrastmikroskop überprüft. Anschließend wurden die<br />
Zellen mit einer Neubauer Kammer improved ausgezählt und abhängig von der Zelldichte in<br />
einem Verhältnis von 1:4 bis 1:6 verdünnt und mit frischem Medium versorgt. Ein<br />
Unterschied zwischen dem Wildtyp und der transfizierten Variante war dabei nicht zu<br />
erkennen.<br />
Differenzierung der Zellen<br />
HL-60 Zellen können durch Zugabe polarer Stoffe zu eingeschränkt funktionalen<br />
neutrophilen Granulozyten differenziert werden. Zur Differenzierung wurden Zellen einer<br />
Dichte von 1x10 6 Zellen/ml in 3,5 ml RPMI 1640 Medium mit 1,3% DMSO aufgenommen.<br />
Die Zellen teilen sich während der Differenzierung kaum, so dass für nachfolgende<br />
Berechnungen von einer Absolutmenge von 3,5 x 10 6 Zellen ausgegangen werden konnte.<br />
Für die Differenzierung wurden Petrischalen der Firma BD mit einem Durchmesser von 3,5<br />
cm und der Oberflächenbeschichtung tissue culture treat verwendet. Die Beschichtung diente<br />
dazu, die Anzahl adhärenter Zellen so niedrig wie möglich zu halten, da diese beim<br />
Entnehmen der Flüssigkeit nicht erfasst wurden und dadurch nicht für die anschließenden<br />
Chemotaxis Versuche verwendet werden konnten. Die Differenzierung der Zellen lieferte die<br />
besten Ergebnisse nach einem Zeitraum von 4-6 Tagen, was sich mit den Vorgaben aus der<br />
Literatur deckt. [Collins, 1987] Der Erfolg der Differenzierung wurde anhand der<br />
3.Material und Methoden 21
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Morphologie der Zellen und dem Anteil adhärenter Zellen an der Gesamtzellmenge überprüft.<br />
Differenzierte Neutrophilen-ähnliche Zellen sind segmentkernig, etwas kleiner als<br />
undifferenzierte Zellen und adhärent. Die Morphologischen Unterschiede zwischen<br />
differenzierten und undifferenzierten Zellen sind in Abbildung 8 dargestellt.<br />
Abbildung 8:Morphologisches Erscheinungsbild von (A) undifferenzierten HL-60 Zellen und (B) HL-60<br />
Zellen nach 6 Tagen in 60mM DMSO. [Wright-Giemsa Färbung, Collins, 1978]<br />
Es wurden durchwegs bessere Ergebnisse erzielt, wenn die Zellen während der<br />
Differenzierung nach 2-3 Tagen durch Zentrifugieren bei 1500 RPM für 5min vom<br />
übersäuerten Medium getrennt und anschließend in frischem Medium resuspendiert wurden.<br />
Nach demselben Prinzip wurden die Zellen nach erfolgter Differenzierung vom Medium<br />
getrennt, mit 5ml PBS gewaschen und in 175 µl Hanks Buffered Salt Solution aufgenommen,<br />
um eine finale Konzentration von 1,7x10 7 Zellen zu erhalten. Ausgehend von dieser Lösung<br />
konnten Chemotaxis-Experimente an den Zellen durchgeführt werden.<br />
3.2.2. Kultivierung von HT-1080 Zellen<br />
Für die Chemotaxisexperimente an HT-1080 Fibrosarkom-Zellen wurden anfangs Zellen vom<br />
Wildtyp verwendet. Etwa ab der Hälfte der durchgeführten Versuche wurde mit einer mit<br />
LifeAct-tag RFP transfizierten Variante gearbeitet. Hierbei ist anzumerken, dass die<br />
transfizierten Zellen etwa um den Faktor 1,5 erhöhte Generationszeiten aufweisen. Das<br />
Vorgehen bei der Kultivierung und das Verhalten in Chemotaktisexperimenten waren jedoch<br />
für beide Zellinien identisch.<br />
HT-1080 Wildtyp Zellen wurden in Passage 11 übernommen, die LifeAct-tag RFP<br />
transfizierten Zellen wurden ab Passage 2 weitergeführt. Beide Zelllinien wurden in 75 cm 2<br />
3.Material und Methoden 22
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Zellkulturflaschen (gelber Verschluss, Cell+) in DMEM-Medium (C-C-Pro) mit 10% FCS<br />
kultiviert.<br />
Die genaue Zusammensetzung des Mediums findet sich in Anhang G auf Seite 100. Im<br />
Folgenden darf davon ausgegangen werden, dass DMEM soweit nicht anders beschrieben<br />
immer mit 10% FCS verwendet wurde. Die Kultivierung der Zellen erfolgte in einem<br />
Brutschrank bei 37°C und 5% CO2. Im Abstand von 2 Tagen wurde die Konfluenz der Zellen<br />
unter dem Phasenkontrastmikroskop überprüft. Das Wachstum von HT-1080 Zellen ist<br />
weitgehend unabhängig von Zell-Zell Kontakten [Collins, 1978]. Um die Zellzahl dennoch<br />
stabil zu halten, wurde versucht die Zellen stets so lange in einer Flasche zu kultivieren bis<br />
die Zellschicht zu mindestens 80% optisch konfluent war. Anschließend wurden die Zellen in<br />
den Kulturflaschen mit 10 ml PBS gewaschen Zellen und nach Absaugen des PBS durch<br />
Trypsinierung mit 2ml Trypsin-EDTA vom Flaschenboden gelöst. Die gelösten Zellen wurden<br />
mit 10 ml DMEM versetzt und anschließend, abhängig von der Zelldichte in einem Verhältnis<br />
etwa 1:4 mit frischem Medium verdünnt. Auf diese Weise wurde die Zellzahl bei etwa 1x10 5<br />
bis 1x10 6 Zellen stabil gehalten.<br />
Die überschüssigen Zellen wurden durch 5-minütiges Zentrifugieren bei 1500 RPM wieder<br />
vom Medium getrennt und abhängig von der vorhandenen Gesamtzellmenge in etwa 500µl<br />
DMEM resuspendiert um eine finale Zellzahl von ungefähr 1,7x10 7 Zellen für<br />
Chemotaxisexperimente zu erhalten.<br />
Die mit LifeAct transfizierten Zellen wurden aufgrund des langsameren Wachstums in der<br />
Regel nur im Verhältnis 1:2 verdünnt. Zusätzlich wurde hier dem Medium noch G418-Sulphat<br />
in einer Konzentration von 375µg/ml zugefügt, um den Selektionsdruck auf die transfizierten<br />
Zellen aufrecht zu erhalten.<br />
3.Material und Methoden 23
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
3.3. Mikroskopietechniken<br />
3.3.1. Zellmikroskopie<br />
3.3.1.1. Nieder- und Hochaufgelöster Phasenkontrast<br />
Ein Großteil der Bilder, die als Rohdaten zur Auswertung der Experimente dienten, wurde<br />
nach dem Phasenkontrastverfahren aufgenommen. Dazu wurde das inverse<br />
Fluoreszenzmikroskop Nikon Eclipse Ti mit den Objektiven Plan Fluor 4x/0.13 und Planfluor<br />
10x/0.3 verwendet. Die Bilddaten wurden mit der 5Mhz CCD-Kamera MicroMax der Firma<br />
Princeton Instruments mit einer Farbtiefe von 16 bit generiert. Die Belichtungszeit der<br />
Aufnahmen betrug im Schnitt etwa 30ms unter einer Leistung der Dia-Lampe von 30 Watt bei<br />
6V Spannung. Um Detailaufnahmen von Zellen zu erzeugen wurde das Objektiv Plan ApoVC<br />
60x/1.4 mit Immersionsöl verwendet. Mikroskop und Peripherie wurden etwa 1 Stunde vor<br />
Versuchsbeginn eingeschaltet, um Einflüsse auf die Versuchsdaten durch Abwärme zu<br />
normalisieren.<br />
3.3.1.2. Fluoreszenzmarkierung mit LifeAct-tag GFP2/RFP<br />
Um bei den chemotaktischen Experimenten einen Blick in die Vorgänge im inneren der Zelle<br />
zu ermöglichen wurden Zellen verwendet, die mit dem sogenannten LifeAct-tag GFP2/RFP<br />
transfiziert waren. Das LifeAct-tag ist ein 17 Aminosäuren langes Protein, dass es ermöglich<br />
Fluoreszenzmarker wie GFP2 oder RFP an die Aktinfilamente (F-Aktin) in Zellen anzulagern,<br />
ohne die Dynamik des Aktinmoleküls dadurch zu beeinträchtigen. [Riedl, 2008]<br />
Abbildung 9 zeigt schematisch den Aufbau eines Aktin-Filaments das mit LifeAct markiert<br />
wurde.<br />
Abbildung 9: Schematischer Aufbau von F-Aktin mit LifeAct-tag GFP2/RFP [modifiziert aus<br />
Produktflyer, Ibidi GmbH, 2011]<br />
3.Material und Methoden 24
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Die angehängten fluoreszierenden Proteine GFP2 und RFP besitzen Anregungsmaxima bei<br />
Wellenlängen von 483nm (GFP2) und 555nm (RFP) sowie Emissionsmaxima bei 5<strong>06</strong>nm<br />
(GFP2) und 584nm (RFP).<br />
Für die Aufnahmen wurde das unter 3.3.1.1. beschriebene inverse Fluoreszenzmikroskop<br />
Nikon Eclipse Ti mit einem Plan ApoVC 60x/1.4 Objektiv verwendet. Als Anregungsquelle<br />
wurde eine Nikon Intensilight Quecksilberhochdrucklampe verwendet. Das verwendete<br />
Filterset für Versuche mit GFP2 war FiTC mit einem Anregungsspektrum von 465-495 nm<br />
und einem Emissionsspektrum von 515-555 nm. Für RFP wurde TxRed mit einem<br />
Anregungsspektrum von 540-580 nm und einem Emissionsspektrum von 600-660 nm<br />
verwendet. Die Belichtungszeit war in allen Fällen 500ms. Die Zellen konnten nur über einen<br />
sehr begrenzten Zeitraum verfolgt werden, weil die auftretenden Bleichungseffekte sonst zu<br />
stark waren. Von HT-1080-Zellen wurden Aufnahmen in einem Intervall von 10 Minuten, für<br />
HL-60 Zellen in einem Intervall von 2 Minuten aufgezeichnet.<br />
3.3.2. Diffusionsstudien<br />
Um die Entwicklung eines Stoffgradienten im µ-Slide Chemotaxis 3D untersuchen zu<br />
können, wurde mit fluoreszierenden Farbstoffen gearbeitet.<br />
Verwendet wurden dazu die Fluoreszenzfarbstoffe Alexa 488 (Invitrogen) und FITC-Dextran<br />
(Sigma-Aldrich). Das Molekulargewicht von Alexa entspricht mit 643 Da in etwa demjenigen<br />
von fMLP (437.55 Da), womit es stellvertretend für Chemotattraktant kleiner Molekülgröße<br />
betrachtet werden kann.<br />
FITC-Dextran soll mit einem Molekulargewicht von etwa 18 kDa stellvertretend für<br />
Experimente mit Chemoattraktant großer Molekülgröße verwendet werden.<br />
Um den Verlauf des Farbstoffgradienten über den Kanalquerschnitt unabhängig von<br />
gestreutem Licht und der Hintergrundfluoreszenz des umgebenden Materials zu gestalten,<br />
mussten dazu mikroskopische Verfahren gewählt werden, die es ermöglichen, ein horizontales<br />
Konzentrationsprofil durch einen Schnitt mit möglichst geringer z-Koordinate zu erstellen.<br />
3.3.2.1. Konfokale Laser Scanning Mikroskopie<br />
Die Konfokale Laser Scanning Mikroskopie ist eine besondere Form der von Marvin Minsky<br />
in den 1950er Jahren entwickelten Konfokal-Mikroskopie zur Anwendung mit<br />
Fluoreszierenden Proben. Die Besonderheit der konfokalen Mikroskopie liegt darin, dass im<br />
Gegensatz zu konventionellen Mikroskopen immer nur sehr kleiner Bereich des Präparats<br />
3.Material und Methoden 25
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
durch einen beugungsbegrenzten Lichtfleck beleuchtet wird. Das Gesamtbild wird durch<br />
Rasterscannung des Beobachtungsbereichs erzeugt. Durch die Verwendung eines Lasers als<br />
Lichtquelle, lässt sich kohärentes Licht einer definierten Wellenlänge erzeugen, dass genutzt<br />
wird, um fluoreszierende Partikel im untersuchten Medium anzuregen. Das Licht wird dabei<br />
über ein Objektiv in die Probe hineinfokussiert. Die Partikel im Bereich des Anregungsfokus<br />
werden zur Fluoreszenz angeregt und das emittierte Licht wird über dasselbe Objektiv<br />
zurückgeleitet und wird über eine Lochblende (pinhole) auf einen Detektor geführt.<br />
Anregendes und emittiertes Licht werden dabei über einen Strahlteiler aufgetrennt, so dass<br />
nur Emissionslicht auf den Detektor trifft. Anregungsfokus und Detektionsfokus liegen nach<br />
diesem Prinzip übereinander, also konfokal, woher sich auch der Name ableitet.<br />
In Abbildung 10 ist das Prinzip des Verfahrens schematisch dargestellt.<br />
Abbildung 10: Schematische Darstellung des konfokalen Prinzips. Bei der LSM wird als Lichtquelle ein<br />
Laser verwendet.[aus: Commons: Bilder zum Funktionsweise des konfokalen Prinzips, Stand September<br />
2011]<br />
Das Verfahren hat den Vorteil, dass vorwiegend Partikel angeregt werden, die im<br />
Anregungsfokus liegen. Licht von Partikeln außerhalb des Anregungsfokus wird nicht auf die<br />
Lochblende fokussiert, so dass ein Großteil davon vom Detektor nicht erfasst wird. Dadurch<br />
lässt sich eine sehr geringe Schärfentiefe erzielen, wodurch sich das Auflösungsvermögen in<br />
z-Richtung deutlich erhöht. So können relativ präzise Aussagen über die ortsabhängige<br />
Verteilung der Fluoreszenzintensität in einer Probe gewonnen werden.<br />
3.3.2.2. Fluoreszenz-Korrelations Spektroskopie<br />
Die Fluoreszenz-Korrelations Spektroskopie wurde in den 1970er Jahren von Watt W. Webb,<br />
Elliot Elson und Douglas Magde als Verfahren zur Messung von Diffusionskonstanten<br />
3.Material und Methoden 26
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
entwickelt. In einem sehr kleinen Volumen, meist realisiert durch ein konfokales Mikroskop,<br />
wird die Fluktuation der Fluoreszenzintensität gemessen. Durch Laserlicht werden dabei<br />
einzelne fluoreszierende Partikel, die in das Beobachtungsvolumen eindiffundieren angeregt.<br />
Als Detektoren werden hier heute zumeist Avalanche-Photodioden eingesetzt, mit denen es<br />
möglich ist, einzelne Photonen zu erfassen. Die Abtastrate der Detektoren liegt in der Regel<br />
deutlich unter der Zeit, welche die Partikel zum durchqueren des Beobachtungsvolumens<br />
benötigen, so dass Signale einzelner Partikel bei aufeinanderfolgenden Abtastvorgängen<br />
mehrfach erfasst werden. Die durch FCS generierten Daten erfassen dadurch die<br />
Fluoreszenzintensität als Funktion der Zeit. Die gemessenen Intensitäten sind zeitlich<br />
korreliert, woher sich auch der Name des Verfahrens ableitet. Um die Mehrfachdetektion<br />
einzelner Teilchen in die Funktion einzubeziehen, wird sie nach einer festgelegten Formel mit<br />
sich selbst korreliert, man spricht hier von sogenannter Autokorrelation.<br />
Gibt man der zugehörigen Software die Diffusionskonstante und den Strukturparameter der<br />
diffundierenden Partikel als bereits definierte Werte an, kann durch die Messung die absolute<br />
Fluoreszenzintensität am Messpunkt zum vorliegenden Zeitpunkt bestimmt werden. Über<br />
einen Proportionalitätsfaktor lässt sich daraus die Molarität der Lösung am Messpunkt<br />
berechnen. Im Gegensatz zur Laser Scanning Mikroskopie kann der Beobachtungsbereich<br />
durch die FCS nicht gerastert werden und die Messung erfolgt an einzelnen Punkten. Die<br />
Aussagekraft der FCS-Messungen bleibt allerdings auf einen bestimmten<br />
Konzentrationsbereich fluoreszierender Partikel beschränkt, weshalb der<br />
Konzentrationsbereich der verwendeten Farbstoffe vor Beginn der Experimente eingegrenzt<br />
werden muss.<br />
Für die unter Punkt 4.1.3 beschriebenen Versuche wurde die Farbstoffkonzentration anhand<br />
eines bestehenden Verdünnungsgraphen so festgelegt, dass sowohl die Lösung für die hohe<br />
Konzentration C100, wie auch die Lösung für die niedrige Konzentration C0 in einem<br />
Konzentrationsbereich liegen, in dem ein linearer Zusammenhang zwischen der Anzahl der<br />
Fluoreszenzsignale der FCS-Messung [N] und der Molarität der Lösung [M] angenommen<br />
werden kann.<br />
3.Material und Methoden 27
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Abbildung 11 zeigt die verwendeten Verdünnungsgraphen, die durch Messungen an<br />
Verdünnungsreihen einzelner wichtiger Fluoreszenzfarbstoffe durch Mitarbeiter des<br />
Lehrstuhls für Biophysik von Herrn Prof. Dr. Joachim Rädler an der Ludwig Maximilians<br />
Universität München entstanden sind.<br />
Abbildung 11: Verdünnungsgraphen verschiedener Fluorophore. Der Bereich zwischen den beiden<br />
gestrichelten Linien deutet das Spektrum zwischen C0 [1nM] und C100 [50nM] des verwendeten<br />
Farbstoffes Alexa 488 an. [zur Verfügung gestellt vom Lehrstuhl für Biophysik, Dr. J. Raedler, LMU-<br />
München, 2011]<br />
Die Konzentration für C100 wurde auf 50nM festgelegt, die Konzentration für C0 auf 1nM, um<br />
eine klare Abhebung von der Autofluoreszenz der zur Verdünnung verwendeten PBS zu<br />
erhalten. Der lineare Verlauf des Verdünnungsgraphen für Alexa 488 für den Bereich<br />
zwischen den beiden verwendeten Konzentrationen ist in Abbildung 11 durch die<br />
gestrichelten roten Linien dargestellt.<br />
3.Material und Methoden 28
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
3.4. Verwendete Gelmatrizes<br />
3.4.1. Collagen<br />
Bei einem Großteil der in dieser Arbeit vorgestellten Chemotaxisexperimente wurde eine<br />
Matrix aus bovinem Typ I Collagen (Sigma-Aldrich) verwendet. Die Collagen-Lösung wurde<br />
in der kommerziell erhältlichen Form einer Stammlösung mit 3mg/ml bei 4°C gelagert. Für<br />
die Chemotaxis Experimente wurden Gele nach firmeneigenem Protokoll der Firma Ibidi<br />
gefertigt. Die vorgegebene Zusammensetzung ist so eingestellt, dass der pH-Wert der Lösung<br />
mit 7.4 bis 7.6 nahe dem physiologischen pH der verwendeten Zellen liegt. In den meisten<br />
Fällen wurde eine Gelmatrix mit 1.5mg/ml und DMEM für HT-1080 Zellen und 1.0 mg/ml<br />
und RPMI 1640/MEM für HL-60 Zellen verwendet. Eine vollständige Auflistung aller zum<br />
Einsatz gekommenen Gelzusammensetzungen findet sich in Anhang D.<br />
Nach dem Pipettieren der Bestandteile wurde die Lösung gründlich vermischt, damit es trotz<br />
der hohen Viskosität des Collagens zu einer gleichmäßigen Verteilung der Zellen in der<br />
Lösung kam. Anschließend wurden 6 µl der Collagen-Lösung in die Kanäle des µ-Slides<br />
Chemotaxis 3D eingefüllt und im Brutschrank bei 37°C und 5% CO2 für etwa 45 Minuten<br />
polymerisiert. Abschließend wurde die Regelmäßigkeit der Faserstruktur des Collagens unter<br />
dem Phasenkontrastmikroskop überprüft.<br />
3.4.2. Synthetische Hydrogele<br />
Die Möglichkeit zur Verwendung komplett synthetischer Matrizes in den µ-Slides<br />
Chemotaxis 3D wurde anhand von 2 kommerziell erhältlichen Hydrogelen überprüft.<br />
3D-Matrix der Firma Qgel:<br />
Bei der 3-D Matrix der Firma Qgel handelt es sich um ein PEG Gerüst, dessen<br />
zellphysiologische Eigenschaften aus vorgefertigten Sets an Zusatzkomponenten ausgewählt<br />
werden können.<br />
Getestet wurde eine PEG-Matrix mit RGD-Peptiden als Bindungsmotive und<br />
Quervernetzungen, die von MMP-Proteasen der Zellen abgebaut werden können.<br />
Eine Migration durch das Gel ist wegen einer Maschenweite im Nanometer-Bereich nur durch<br />
die Spaltung dieser Quervernetzungen möglich.<br />
Um Qgel in den µ-Slides nutzen zu können, wurde die, vom Hersteller angegebene<br />
Verdünnung „Soft IV“ verwendet, um eine vorzeitige Polymerisation der Matrix zu<br />
3.Material und Methoden 29
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
verhindern. Die Herstellung des Gels ist im Folgenden stichpunktartig beschrieben:<br />
- Mischen des lyophilisierten Pulvers mit 600µl des mitgeliefertern Buffer A.<br />
- Vortexen der Lösung für etwa 10 Sekunden.<br />
- Hinzufügen von 150 µl Zellsuspension mit einer Zellzahl von 1.5x10 7 /ml.<br />
- Vortexen für 10-15 Sekunden zur vollständigen Homogenisierung der Lösung.<br />
- Einfüllen von 6µl Gelsuspension in den Kanal des µ-Slide.<br />
- Polymerisation des Gels für 30-45 Minuten im Brutschrank bei 37°C und 5% CO2.<br />
Die finale Zellkonzentration in den Kanälen betrug 3x10 6 Zellen/ml. Eine vollständige<br />
Tabelle zu den unterschiedlichen Verdünnungen und den mechanischen Eigenschaften des<br />
Gels findet sich in Anhang A auf Seite 95.<br />
Cellendes Maleimid-BSA 3D-Matrix:<br />
Das verwendete Hydrogel der Firma Cellendes ist eine Matrix aus BSA-Protein mit<br />
funktionellen Maleimid-Gruppen.<br />
Abbildung 12: Schematische Darstellung des BSA-Proteins mit funktionellen Maleimid-Gruppen<br />
[modifiziert aus: Cellendes – Hydrogel User Guide Version 3.0, 2011]<br />
In der untersuchten Ausführung wurden diese Maleimid-Gruppen mit Thiol-Gruppen eines<br />
Polyethylen-glycol Proteinkomplexes quervernetzt. Der Proteinkomplex enthält eine Sequenz,<br />
welche die Zellen bei der Migration durch MMP-Proteasen spalten können.<br />
Abbildung 13: Schematische Darstellung des PEG-Crosslinkers mit funktionellen Thiol-Gruppen und<br />
spaltbarer Peptidsequenz. [Cellendes – Hydrogel User Guide Version 3.0, 2011]<br />
3.Material und Methoden 30
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Um den Zellen die notwenigen Bindungsmotive zu liefern, wurde den Gelen RGD-Peptid in<br />
einer Konzentration von 0.1mM bis 1mM beigemischt. Die Polymerisationszeit des Gele lag<br />
im Bereich weniger Minuten, weswegen zur Verwendung in den µ-Slides immer mit der<br />
niedrigsten verfügbaren Konzentration von 2.5mg/ml Maleimid-BSA gearbeitet wurde. Die<br />
Polymerisationszeit des Gels war außerdem abhängig vom pH-Wert der Lösung. Vom<br />
Hersteller wurden aus diesem Grund zwei Puffer mit den pH-Werten 5.5 und 7.4 mitgeliefert.<br />
Obwohl damit eine leichte Abweichung vom physiologischen pH-Wert der Zellen eintrat,<br />
wurde für die vorgestellten Versuche immer bei einem pH-Wert von 5.5 gepuffert, um die<br />
Polymerisationszeit zu verlängern.<br />
Aus den Pipettierschemata des Herstellers, in denen dieser Fall nicht beschrieben war, wurden<br />
die Mengenangaben für Gele der höchsten Verdünnung unter Verwendung von RGD-Peptid<br />
errechnet. Die Zusammensetzung ist für eine RGD-Konzentration von 1mM im Folgenden<br />
beispielhaft dargestellt.<br />
Maleimid-BSA 2.5mg/ml mit 1mM RGD-Peptid<br />
Wasser 6.25µl<br />
10xCBpH5.5 2.5µl<br />
Maleimid-BSA 2.5µl<br />
RGD-Peptid (3mM) 10µl<br />
Zellsuspension 5.0µl<br />
CD-Link 3.75µl<br />
Total 30µl<br />
Um mehrere Kanäle befüllen zu können, wurde die Lösung vor Hinzufügen des CD-Link<br />
gleichmäßig aufgeteilt und einzeln fertiggestellt. Nach Einfüllen des CD-Link wurde die<br />
Lösung mit der verwendeten Pipettenspitze schnell durchmischt und unmittelbar danach in<br />
die Kanäle des µ-Slides eingefüllt. Nach etwa 5-10 Minuten im Brutschrank bei 37°C und 5%<br />
CO2 waren die Gele bereits vollständig polymerisiert. Die vollständigen Verdünnungstabellen<br />
des Herstellers sind in Anhang B und C auf den Seiten 95 und 96 zu finden.<br />
3.Material und Methoden 31
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
3.5. Chemotaxis mit HL-60 und HT-1080<br />
3.5.1. Durchführung der Experimente<br />
Zur Chemotaxis-Analyse wurden 6 µl der jeweiligen Zellen in einer Gelmatrix in die Kanäle<br />
des µ-Slides Chemotaxis 3D gefüllt. Nach der Polymerisationszeit des Gels wurde<br />
Chemoattraktant in die Reservoire der Slides gefüllt. Das genaue Vorgehen bei dem Befüllen<br />
der Slides ist in Anhang E auf Seite 97 beschrieben. Das Befüllschema war hier jeweils<br />
abhängig davon, welche Parameter bei dem Experiment untersucht wurden.<br />
Zur Ermittlung der notwendigen Konzentration an Chemoattaktand wurden die linken<br />
Reservoire der Kammern mit einer steigenden Chemoattraktand Konzentration befüllt. Die<br />
Aufteilung der verschiedenen Chemoattraktant-Konzentrationen ist in Abbildung 14<br />
schematisch dargestellt.<br />
Abbildung 14: Schematische Darstellung der Verteilung der Chemoattraktant Konzentrationen auf die<br />
Kammern des µ-Slides Chemotaxis 3D zur Ermittlung der einzusetzenden Dosis.<br />
Um bei einer festgelegten Konzentration von Chemoattraktant die Chemotaxis-Analyse gegen<br />
zufällig auftretende Effekte abzusichern, wurde ein Vergleich mit einer Positiv- und einer<br />
Negativkontrolle durchgeführt. Die Positivkontrolle beinhaltet die Verwendung einer Lösung<br />
mit hoher Konzentration an Chemoattraktant in beiden Reservoirs. Die Negativkontrolle wird<br />
mit chemoattraktant-freiem Medium in beiden Reservoirs durchgeführt.<br />
3.Material und Methoden 32
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Das Füllschema der Slides für Experimente mit Positiv- und Negativkontrolle ist in<br />
Abbildung 15 dargestellt.<br />
Abbildung 15: Schematische Darstellung der Verteilung der Chemoattraktand-Konzentrationen für<br />
Chemotaxis Versuche mit Kontrollexperimenten.<br />
Für HT-1080 Zellen wurde bei der Zusammenstellung der Gele für das Chemotaxis-<br />
Experiment und die Negativkontrolle DMEM ohne FCS verwendet. Die Positiv-Kontrolle<br />
enthielt DMEM mit 60% FCS, um nach einer Verdünnung im Rahmen des, in Anhang D<br />
gezeigten Pipettierschemas, auch im Kanalbereich an eine finale Konzentration von 10% FCS<br />
zu erhalten.<br />
Die verwendete Gelmatrix schließt Konvektionsströmungen zwischen den Reservoirs aus.<br />
Dadurch kann unmittelbar nach Aushärten des Gels ein Reservoir mit Chemoattraktant gefüllt<br />
werden ohne dass es zu Vermischung im Kanalbereich kommt. Dieses Vorgehen wird als „fast<br />
Method“ bezeichnet.<br />
In einem Großteil der beschriebenen Versuche wurden jedoch zuerst beide Reservoirs mit<br />
60µl des Hungermediums C0 gefüllt. Anschließend wurden 30µl einer Chemoattraktant-<br />
Lösung mit doppelter Konzentration auf einen der beiden Einfüllstutzen des linken Reservoirs<br />
pipettiert. Durch Absaugen von 30µl Flüssigkeit aus dem anderen Stutzen mit einer<br />
Pipettenspitze wurde das Chemoattraktant in das Reservoir gesaugt. Der Gradient sollte so<br />
schonender und mit geringer zeitlicher Verzögerung etabliert werden. Das Vorgehen nach<br />
dieser Methode wird als „slow method“ bezeichnet. Ausführliche Beschreibungen der beiden<br />
Methoden finden sich in Anhang E, auf Seite 82f.<br />
Um die Bedingungen für die Zellen während der Experimente möglichst optimal zu halten,<br />
wurden die Slides während der Laufzeit des Experimentes unter dem Mikroskop in eine, von<br />
der Firma Ibidi eigens dafür entwickelte Heizplatte mit ebenfalls beheizbarer Abdeckhaube<br />
gelegt. Die Temperatur der Bodenplatte war auf 41.5°C die Temperatur der Abdeckhaube auf<br />
43.5°C eingestellt, was einer Temperatur der Atmosphäre innerhalb dieser Kammer von etwa<br />
3.Material und Methoden 33
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
37°C entsprach. Durch einen Gasmischer kombiniert mit einem System zur Erhöhung der<br />
Luftfeuchte, wurde Luft mit 5% CO2 in die Heizkammern geleitet. Die Bilddaten wurden auf<br />
dem unter 3.3.1.1. beschriebenen Phasenkontrastmikroskop generiert.<br />
Zur genauen Positionierung der Kammern und um Daten für Experimente in mehreren<br />
Kammern parallel erzeugen zu können, wurde ein software-gesteuerter automatischer Tisch<br />
mit programmierbarer Positionsliste genutzt. Die Zeitrafferaufnahmen wurden unter<br />
Verwendung der, auf dem Programm ImageJ basierenden Software, Micromanager Version<br />
1.4 erzeugt. Für HT-1080 Zellen war der Beobachtungszeitraum in der Regel über 24<br />
Stunden, bei einem Aufnahmeintervall von 10 Minuten. HL-60 Zellen wurden 3 Stunden lang<br />
bei einem Aufnahmeintervall von 1 Minute beobachtet.<br />
3.5.2. Auswertung und Datenanalyse<br />
Zell-Tracking<br />
Die Bilddaten aus den Versuchen wurden mit dem Manual-Tracking Plugin des Programms<br />
ImageJ ausgewertet. Dazu wurde die Bilderserie des Experiments in den Zwischenspeicher<br />
des Programms geladen. Bei Verwendung des Manual-Tracking Plugin wurden für jedes Bild<br />
nacheinander die Zentren der einzelnen Zellen mit dem Cursor markiert, so dass die Software<br />
nach Abschluss einer Bilderserie daraus die Spur der Zellbewegungen darstellen konnte.<br />
Das Ergebnis des Manual Tracking ist in Abbildung 16 beispielhaft dargestellt.<br />
Abbildung 16: Vergleich der Phasenkontrastbilder unmittelbar nach Start des Experiments und nach 50<br />
Minuten mit farbig markierten Zellspuren.<br />
3.Material und Methoden 34
4. Ergebnisse<br />
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
4.1. Errichtung und Messung von definierten Konzentrationsprofilen<br />
im µ-Slide Chemotaxis 3D.<br />
Die Aussagekraft von Zellmigrationsdaten ist stark von der Entwicklung des zur Verfügung<br />
stehenden Lockstoffgradienten abhängig. Die Untersuchung der zeitlichen Veränderung des<br />
Gradienten beziehungsweise dessen Langzeitstabilität ist daher von maßgeblicher Bedeutung.<br />
Anhand einer Versuchsreihe soll untersucht werden, ob sich das Diffusionsprofil im µ-Slide<br />
Chemotaxis unabhängig von der Größe der diffundierenden Partikel sowie dem<br />
Vernetzungsgrad der verwendeten Matrix verhält. Die Ermittlung des Konzentrationsprofils<br />
wurde durch ortsabhängige Integration und graphische Aufbereitung der Fluoreszenz-Signale<br />
realisiert.<br />
4.1.1. Untersuchung von Sonden-Matrix Interaktionen<br />
Bevor Untersuchungen an den Diffusionsprofilen durchgeführt wurden, wurden die<br />
verwendeten Farbstoffe auf mögliche Wechselwirkungen mit den verwendeten Collagen<br />
Matrizes und dem Kunststoff der Slides geprüft. Auf diese Weise sollte ausgeschlossen<br />
werden, dass andere Faktoren als eine physische Diffusionsbarriere in Form der<br />
Strukturfasern der verwendeten Gelmatrix eine Einschränkung der Diffusion bewirken<br />
können. Zu diesem Zweck wurde untersucht, ob sich die verwendeten Fluoreszenz-Sonden,<br />
nach Eindiffusion in Collagen-Matrizes unterschiedlicher Vernetzungsgrade, wieder<br />
rückstandslos aus den Gelen waschen lassen.<br />
Im Folgenden werden Durchführung und Ergebnisse dieses Vorexperimentes kurz<br />
beschrieben.<br />
Für den Versuch wurden Collagen-Gele der Stärken 0,5mg/ml, 1mg/ml und 1,5mg/ml nach<br />
dem unter Punkt 3.4.1. beschriebenen Pipettierschema hergestellt und in Angiogenese-Slides<br />
der Firma Ibidi gefüllt. Die Gele wurden in die, in den Boden der Probenkammern des<br />
Angiogeneseslides eingebetteten 10µl Reservoirs eingebracht. Zur Polymerisation des<br />
Collagens wurden die Gele etwa 15 Minuten bei 37°C im Brutschrank inkubiert und<br />
anschließend mit 50µl PBS in den darüber liegenden Reservoiren überschichtet, um ein<br />
Austrocknen der Matrix zu vermeiden.<br />
4. Ergebnisse 36
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Zur vollständigen Vernetzung des Collagens wurden die Slides anschließend wieder für 30<br />
Minuten in den Inkubator gestellt.<br />
Die auszuwertenden Bilddaten wurden mit dem EVOS fl. Fluoreszenz-Mikroskop der Firma<br />
AMG generiert. Die Wellenlänge des anregenden Lichts betrug 470nm, die des detektierten<br />
emittierten Lichts 525nm. Als Referenzwert für die anschließenden Messungen wurde die<br />
Hintergrundfluoreszenz des Collagen-Gels festgelegt. Zur Bestimmung der<br />
Hintergrundfluoreszenz wurde die Detektionszeit und die Intensität des anregenden Lichtes<br />
variiert bis unter dem integrierten Fluorit-Objektiv (10x/0.3) ein deutlicher Unterschied der<br />
Emissionsintensität zwischen der Messung im Collagen-Gel und einer Referenzmessung in<br />
einem leeren Reservoir erkennbar war. Die Werte betrugen im untersuchten Fall 500ms<br />
Belichtungszeit bei 50% der, zur Verfügung stehenden Anregungsintensität des Mikroskops.<br />
Über Verdünnungsreihen der beiden verwendeten Fluoreszenz-Farbstoffe wurden diejenigen<br />
Konzentrationen ermittelt, bei denen die, als 8-bit Grauwert auf einer Skala von 0 (Schwarz)<br />
bis 255 (Weiß) dargestellte Emissionsintensität etwa im mittleren Bereich lag.<br />
Für FiTC-Dextran ergab sich in Folge dessen eine Konzentration von 1µg/ml, für Alexa 488<br />
wurde eine 100nM Lösung gewählt.<br />
4. Ergebnisse 37
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Die Entwicklung der Fluoreszenzfarbstoffkonzentration im Gel wurde im Anschluss an die<br />
Referenzmessung in 3 Schritten bestimmt:<br />
1.Schritt: Überschichten der Gele mit Fluoreszenz-Farbstoff und anschließender<br />
30-minütiger Diffusionszeit.<br />
2.Schritt: Erste Messung unmittelbar nach Austausch der Farbstofflösung mit PBS.<br />
3.Schritt: Drei weitere Messungen nach jeweils 15-minütiger Diffusionszeit und<br />
Austausch des PBS.<br />
Abbildung 17: Schematische Darstellung der Versuchsdurchführung in einer Kammer des<br />
Angiogeneseslide. Die schwarzen Pfeile deuten die Farbstoffdiffusion in das und aus dem Gel (brauner<br />
Bereich) an.<br />
Durch den Austausch lediglich der überschichteten Flüssigkeit kann der Farbstoff nur durch<br />
erneute Diffusion wieder aus dem Gel gelöst werden. Ein Auslösen oder Ausbrechen von<br />
gebundenen Molekülen durch mechanische Scherkräfte oder Konvektionsströme kann somit<br />
ausgeschlossen werden.<br />
Die Fokusebene wurde anhand der Bilddarstellung im Phasenkontrast so positioniert, dass die<br />
Struktur der Collagenfasern zu erkennen war. Auf diese Weise wurde sichergestellt, dass die<br />
Messungen der Fluoreszenzintensität innerhalb der Gelmatrix durchgeführt wurden. Die<br />
Grauwerte der gemessenen Bilder wurden über das jeweilige Gesamtbild gemittelt und<br />
tabellarisch aufgetragen.<br />
4. Ergebnisse 38
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Abbildung 18 zeigt, dass die gemessene Intensität nach drei Waschvorgängen für alle<br />
Gelkonzentrationen wieder auf, beziehungsweise teilweise sogar leicht unter der<br />
Hintergrundfluoreszenz des Collagens liegt.<br />
Die Form der beiden Graphen legt nahe, dass auch die Größe der verwendeten Sonde dabei<br />
keine Rolle spielt.<br />
Abbildung 18: Verlauf der gemessenen Fluoreszenz-Intensität während der Versuche für (A) Alexa 488<br />
und (B) FiTC-Dextran in Collagen Matrizes unterschiedlicher Stärke. Die Messungen wurden immer<br />
unmittelbar vor den Waschvorgängen durchgeführt.<br />
Auf Grundlage der vorliegenden Messergebnisse wurde in den folgenden Versuchen davon<br />
ausgegangen, dass es zu keinen Interaktionen zwischen der Matrix und dem verwendeten<br />
Farbstoffen kommt.<br />
4. Ergebnisse 39
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
4.1.2. LSM - Messungen mit Alexa 488 und FITC-Dextran<br />
Bestimmung des Messprinzips:<br />
Um das Profil und die Stabilität eines Stoffgradienten im µ-Slide Chemotaxis 3D zu<br />
untersuchen, wurden definierte Konzentrationen der Fluoreszenzfarbstoffe Alexa 488<br />
(stellvertretend für kleine Chemotaxine) und FiTC-Dextran (stellvertretend für große<br />
Chemotaxine) in die Reservoire eines Chemotaxis 3D-Slides eingebracht und die<br />
Konzentrationsentwicklung über den Kanalbereich in x-Richtung durch Messungen der<br />
Fluoreszenzintensität ermittelt.<br />
Der quadratische Bildausschnitt der Laser Scanning Mikroskops hatte mit dem verwendeten<br />
Apochromat 63x/1.4 Öl - Objektiv näherungsweise eine Seitenlänge von 146,25µm. Um den<br />
Kanal in seinem vollen Durchmesser von 1mm vermessen zu können, und zudem noch Werte<br />
für die Grenzregionen zu beiden Reservoire zu erhalten wurden 15 Bildausschnitte in x-<br />
Richtung aufgenommen und anschließend zusammengefügt. Durch die „tile scan“ Funktion<br />
der Software funktioniert das Verfahren des Tisches in passenden Schritten und das<br />
Zusammenfügen des Gesamtbildes automatisch. Dabei wird von einem festgelegten<br />
Mittelpunkt in gleichem Abstand in negativer wie positiver Richtung der jeweiligen<br />
Koordinatenachsen gemessen. Um die Statistik der Messung etwas zu erhöhen wurden in y-<br />
Richtung ebenfalls 2 Bildausschnitte aufgenommen. Die Größe des Gesamtbildes einer<br />
gemessenen Bilderserie ergab sich somit zu etwa 2193,73µm x 292,5 µm.<br />
Um die Reproduzierbarkeit der Messungen zu den unterschiedlichen Zeiten zu gewährleisten<br />
wurde die Bilderreihe auf die Kanalmitte in x-Richtung und die fertigungsbedingte Fließlinie<br />
des Plastiks, die die Mitte des Kanals in y-Richtung markiert, zentriert. Obwohl das Laser<br />
Scanning Mikroskop bei dem verwendeten Pinhole-Durchmesser von 117 Å eine optische<br />
Schnittdicke von ~ 1µm ermöglicht, kommt es dennoch zu störenden Streueffekten von<br />
benachbarten fluoreszierenden Partikeln. Die Menge an unter- beziehungsweise<br />
übergelagerten Fluoreszenz-Partikeln ist abhängig von der z-Position der Betrachtungsebene.<br />
Um den durch Streueffekte entstehenden Messfehler einheitlich zu gestalten und die<br />
Vergleichbarkeit der zeitlich varianten Messungen zu gewährleisten, wurde die optische<br />
Schnittebene für alle gemessenen Proben 20µm unterhalb der oberen Grenzfläche des Kanals<br />
festgelegt. An der Grenzfläche zwischen der Flüssigkeit im Kanal des Slides und dem darüber<br />
liegenden Kunststoff tritt ein Sprung im Brechungsindex auf. Der resultierende Unterschied<br />
im Interferenzmuster kann detektiert und visualisiert werden. Durch eine entsprechende<br />
4. Ergebnisse 40
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Veränderung der z-Koordinate kann die Betrachtungsebene auf die Grenzfläche zwischen den<br />
Materialien eingestellt werden. Ein programmierbarer definierter Schritt von 20 µm in den<br />
Innenbereich des Kanals stellt, bei einer Gesamthöhe des Kanals von 70 µm, die Lage der<br />
Messebene im mittleren Kanalbereich sicher.<br />
Abbildung 19: Lage der konfokalen Schnittebene in einer Kammer der Chemotaxis 3D µ-Slide. Im<br />
Querschnitt des Kanals ist die Platzierung der Gelmatrix (braune Struktur) angedeutet.<br />
C100 steht stellvertretend für eine hohe Konzentration an Fluoreszenzfarbstoff, C0 für eine geringe.<br />
Die Konzentration der Fluoreszenz-Farbstoffe wurde nach Anhaltspunkten bestimmt wie im<br />
Vorexperiment unter 4.1.1. beschrieben. Die Grauwertdarstellung der Fluoreszenzintensität<br />
sollte möglichst wenige unter- beziehungsweise überbelichtete Pixel enthalten, um einen<br />
Informationsverlust zu vermeiden. Für FITC-Dextran wurde C100 auf 0.1mg/ml und C0 auf<br />
0.01mg/ml festgelegt. Alexa 488 wurde unter C100 auf 1µM verdünnt und unter C0<br />
entsprechend auf 0.1µM.<br />
Die Proben wurden mit kohärentem Licht von 488nm Wellenlänge aus einem Argon/2 Laser<br />
mit 25% Output einer Maximalleistung von 30mW und einem Tube Current von 4.9 A<br />
angeregt. Der Lichtquelle war ein Transmissionsfilter mit 50% Durchgang nachgestellt. Nach<br />
Anregung des Farbstoffes wurde das emittierte Licht durch einen Emissionsfilter auf 505nm<br />
Wellenlänge gefiltert und anschließend detektiert. Das Bildfeld wurde mit einer Pixelzeit von<br />
6.4µs zeilenweise 2-fach abgetastet. Einem Gesamtprofil von 30 Bildern steht mit den<br />
Fahrzeiten des automatischen Tisches ein Zeitaufwand von etwa 4 Minuten entgegen. Wird<br />
der Wechsel des Slides und die nachfolgende Kalibrierung der Messposition beaufschlagt,<br />
ergibt sich eine Zeitauflösung von etwa 15 Minuten für parallele Messungen in mehreren<br />
Slides.<br />
4. Ergebnisse 41
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Ein Beispiel für die gewonnenen Rohdaten und einen von ImageJ daraus erstellten Plot des<br />
Intensitätsverlaufes ist in Abbildung 20 gezeigt.<br />
Abbildung 20: (A) Bilddaten einer LSM-Profilmessung bestehend aus 15*2 Einzelbildern und (B)<br />
zugehöriges Intensitätsprofil. Das Wellenmuster entsteht durch die Beugung des Lichts am Pinhole.<br />
Um das in Abbildung 20 erkennbare Wellenmuster auszugleichen wurde im Anschluss an die<br />
Messungen durch die Mittelwert-Funktion in ImageJ der durchschnittliche Grauwert der<br />
beiden in y-Richtung übereinanderliegenden Bildsegmente bestimmt, für jedes der 15<br />
Segmente in x-Richtung tabellarisch aufgetragen und graphisch als Funktion von Grauwert<br />
und x-Position relativ zur Kanalmitte dargestellt.<br />
4. Ergebnisse 42
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
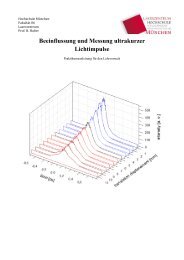
In Abbildung 21 ist das Messprinzip zusammengefasst schematisch dargestellt.<br />
Abbildung 21: Schematische Darstellung der Datenanalyse durch LSM-Messungen. Die kurz-gestrichelten<br />
Linien deuten die Dimension des Mittelkanals an. Die einzelnen Graphen bezeichnen den Verlauf des<br />
Gradienten zu unterschiedlichen Zeitpunkten an einer festgelegten Messposition. Ref. low und Ref. high<br />
stellen Referenzmessungen in Kammern mit C100 und C0 dar. In der Grafik dargestellt sind beispielhaft die<br />
Messdaten des Alexa 488 Farbstoffes in einem 1.0mg/ml Collagen-Gel. (vgl. Abb.22 auf Seite 44)<br />
4. Ergebnisse 43
Messergebnisse:<br />
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Für beide vewendeten Farbstoffe wurden Messungen mit einer Collagenmatrix von 1,5mg/ml<br />
und 1,0 mg/ml sowie ohne Gelmatrix durchgeführt.<br />
Die Ergebnisse der Messungen sind in Abbildung 21 dargestellt:<br />
Abbildung 22: Konzentrationsprofile der Farbstoffe Alexa 488 in (A) 0mg/ml, (B) 1.0mg/ml, (C) 1.5mg/ml<br />
und FITC-Dextran in (D) 0mg/ml, (E) 1.0mg/ml und (F) 1.5mg/ml CollagenI – Matrix.<br />
4. Ergebnisse 44
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Für jeden der beiden Farbstoffe wurde jeweils nur eine Referenzmessung zu C100<br />
beziehungsweise C0 durchgeführt. Durch Pipettierungenauigkeit und eine leicht variable<br />
Messposition in den jeweiligen Slides ist die Aussagekraft der Referenzlösung allerdings auf<br />
die Kammer beschränkt in welcher die zugehörige Messung unternommen wurde. Es hätten<br />
Referenzmessungen für jeden Slide und für jede seiner 3 Kammern durchgeführt werden<br />
müssen. Aussagen über den Zeitraum der Etablierung wie über den genauen Ablauf konnten<br />
deshalb mit dieser Methode nur ungenau und qualitativ getroffen werden.<br />
Erkennbar ist, dass sich die Konzentrationsprofile ausgehend von der 2-Stunden Messung im<br />
Beobachtungsbereich weitestgehend linear verlaufen. Unterschiede zwischen den<br />
unterschiedlichen Ansätzen sind kaum vorhanden. Das zeigt sich auch an der Analyse der<br />
Entwicklung des Gradienten über die Zeit.<br />
Die in Abbildung 23 gezeigten Werte wurden über eine Anpassung der Steigung der<br />
Konzentrationsprofile aus Abbildung 22 im linear abfallenden Bereich zwischen -500µm und<br />
+500µm von der Kanalmitte ermittelt.<br />
Abbildung 23: Die Gradienten von (A) Alexa 488 und (B) FiTC-Dextran aufgetragen über die Zeit von 0-<br />
36h.<br />
Nach anfänglichem steilem Anstieg und einem leichten „Einschwingen“ bleiben die<br />
4. Ergebnisse 45
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Gradienten nahezu konstant. Auch die Analyse des Gradienten zeigt einen, im Rahmen des<br />
Messfehlers vergleichbaren Verlauf der Ansätze unterschiedlicher Gelkonzentration.<br />
Um detaillierte Informationen über Etablierung und Verlauf des Gradienten zu erhalten<br />
wurden auf ein anderes Verfahren mit geringerer Fehleranfälligkeit zurückgegriffen das<br />
bezüglich der notwenigen Referenzmessungen einfacher zu handhaben ist und dadurch auch<br />
eine höhere Zeitauflösung erlaubt.<br />
4.1.3. FCS-Messungen<br />
Durch die Fluoreszenz-Korrelations Spektroskopie (FCS) lassen sich die Fluoreszenzsignale<br />
einzelner fluoreszierender Moleküle an Messpunkten mit ~ 1 µm Schichtdicke bestimmen.<br />
Durch die Integration der Signale in Abhängigkeit von der Diffusionszeit, die bestimmt, wie<br />
lange einzelne Moleküle im Durchschnitt im Beobachtungsfenster der laufenden Messung<br />
verbleiben, lässt sich eine sehr präzise Aussage über die Konzentration eines fluoreszierenden<br />
Stoffes an einem bestimmten Punkt im Untersuchungsbereich formulieren. Die Messungen<br />
sollen bei minimaler Fehleranfälligkeit detailliert Auskunft über den zeitlichen Verlauf und<br />
die Etablierung des Gradienten geben.<br />
Bestimmung des Messprinzips:<br />
Der verwendete Farbstoff war in allem Ansätzen Alexa 488 mit den in Punkt 3.3.2.2.<br />
beschriebenen Konzentrationen.<br />
Den folgenden Messungen von Konzentrationsprofilen wurde ein eigens erstellter<br />
Verdünnungsgraph zugrunde gelegt, um für Messkurven und Kalibrierungswerte gleiche<br />
Voraussetzungen bezüglich eventuell auftretender Messfehler oder Alterungsprozesse des<br />
Fluoreszenzfarbstoffes zu schaffen. Dafür wurde der Konzentrationsgradient zwischen C100<br />
und C0 in 11 Stufen zu Schritten von je 5nM Konzentrationsunterschied unterteilt. Alle<br />
Verdünnungen wurden aus der Stammlösung zu C100 erstellt und in eine Kammer eines<br />
Chemotaxis 3D µ-Slides gefüllt. Gemessen wurde in der Kanalmitte bei einer z-Verschiebung<br />
von 35µm relativ zur Kanaldecke.<br />
4. Ergebnisse 46
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Abbildung 24: Verdünnungsgraph für Alexa 488 zwischen C0 und C100 der eingesetzten<br />
Konzentrationen. Aufgetragen sind die eingesetzte Molarität gegen die Anzahl der gemessenen<br />
Fluoreszenz Signale.<br />
Der in Abbildung 24 dargestellte Verdünnungsgraph zeigt im Bereich des gesamten<br />
verwendeten Konzentrationsspektrums einen linearen Zusammenhang zwischen dem<br />
gemessenen FCS-Signal und der eingesetzten Molarität. Der ebenfalls linear ansteigende<br />
Fehleraufschlag resultiert aus dem erzwungenen Nulldurchgang des Graphen, der<br />
Abweichung in der zugrunde gelegten Steigung auslöst. Auf Grundlage dieser Messungen<br />
wird im Folgenden aus den FCS-Messwerten auf die Molarität der Lösung in Abhängigkeit<br />
der x-Position im Slide geschlossen.<br />
Für alle unternommenen Messungen wurden 5% Maximaloutput des unter 4.1.2.<br />
beschriebenen Argon/2 Lasers mit einer Wellenlänge von 488 nm verwendet. Als<br />
Fixparameter wurden eine Diffusionszeit von 46µs und ein Struktur-Parameter von 10.2<br />
angenommen. Der verwendete Einstellungsparameter Datensatz „AC-Standard_488 BP 505-<br />
550“ enthielt als Vorgabe außerdem einen Emissionsfilter für Wellenlängen zwischen 505 und<br />
550 nm. Analog zu den LSM-Messungen wurde die Messebene wieder anhand des<br />
Unterschiedes im Brechungsindex zwischen Flüssigkeit im Slide und der Kunststoffschicht<br />
darüber ermittelt. Der definierte Schritt in z-Richtung betrug 35µm in den Innenraum des<br />
Kanalbereiches, so dass sich die Messebene in etwa in der Mitte des 70µm hohen Kanals<br />
4. Ergebnisse 47
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
befand. Obwohl für die FCS-Messungen nur Signale einzelnen Moleküle integriert werden<br />
und die punktförmigen Messungen mit ihrer geringen Schichtdicke kaum anfällig für<br />
Störgrößen sind, spielt eine standardisierteGeben Sie hier eine Formel ein. und<br />
reproduzierbare Messposition eine wichtige Rolle. Durch auftretende physikalische Druck-<br />
oder Temperaturunterschiede der Lösungen während des Einfüllens oder nach dem Verschluss<br />
der Stöpsel kann es zu Konvektionseinflüssen kommen. Einflüsse durch lokale<br />
Konzentrationsverschiebungen in y- oder z-Richtung wurden durch die festgelegte<br />
Messposition ausgeschlossen.<br />
Die y-Position der Messpunkte wurde erneut auf die Fließlinie des Plastiks gelegt, die in der<br />
Draufsicht die horizontale Mitte des Kanals darstellt. In x-Richtung wurden die Messpunkte<br />
ausgehend von der Kanalmitte festgelegt, die bei einer Kanalbreite von 1mm durch einen<br />
definierten 500µm-Schritt vom rechten Rand bestimmt wurde.<br />
Gemessen wurde an Punkten 7 verschiedener x-Koordinaten von +/- 1000, +/-500 und +/-250<br />
µm Entfernung, sowie am Kanalmittelpunkt, wie in Abbildung 25 dargestellt.<br />
Abbildung 25: Positionierung der FCS – Messpunkte in einer Kammer des µ-Slide. Innerhalb des<br />
vergrößerten Ausschnittes ist eine Bilderserie zu sehen, die aus 7 x 2 Einzelbildern einer LSM-Messung<br />
besteht und die Fließlinie des Plastiks in der horizontalen Kanalmitte zeigt. (horizontaler Streifen). Die<br />
äußeren Bilder zeigen den Grenzverlauf zwischen dem Kanal (heller Bereich) und den angrenzenden<br />
Reservoirs (dunkler Bereich). Die roten Querstriche deuten die Position der Messpunkte an.<br />
4. Ergebnisse 48
Messergebnisse:<br />
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Zur Untersuchung der Einflussnahme einer verwendeten Gelmatrix, wurden die Messungen<br />
unter standardisierten Bedingungen für eine im Chemotaxis 3D µ-Slide eingebettete Matrix<br />
mit 1.5mg/ml Collagen beziehungsweise einem 2.5mg/ml Maleimid-Polymer der Firma<br />
Cellendes, sowie ohne Gelmatrix durchgeführt. Die Ergebnisse der Messungen sind in<br />
Abbildung 26 dargestellt.<br />
Abbildung 26: Graphen der FCS-Messungen für die Ansätze (A) ohne Gel, (B) mit 1,5mg/ml Collagen I<br />
und (C) 2,5mg/ml Maleimid-BSA Matrix der Firma Cellendes über einen Zeitrahmen von 15 min bis 48h.<br />
4. Ergebnisse 49
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Verlauf über. Nach 24 Stunden ist ein Rückgang des Gradienten in Folge des<br />
Konzentrationsausgleichs zu beobachten. Trotz leichter Unterschiede in der Steigung der<br />
Graphen innerhalb der ersten Stunden, gleicht sich der Verlauf der Gradienten über den<br />
gesamten Zeitraum zu beobachten.<br />
4. Ergebnisse 51
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
4.2. Zellkultur und Migration im Chemotaxis 3D µ-Slide<br />
4.2.1. HT-1080 Zellen in Gelmatrizes<br />
HT-1080 Zellen wurden schon erfolgreich zur Gewinnung von 2-dimensionalen<br />
Migrationsdaten langsam migrierender Zellen in µ-Slides eingesetzt. Im Folgenden soll<br />
gezeigt werden, dass sie sich ebenfalls zur Gewinnung von 3D-Migrationsdaten im<br />
Chemotaxis 3D µ-Slide eignen und ein Vergleich zwischen den vorhandenen 2D-Daten dem<br />
neu erstellten 3D-Datensatz durchgeführt werden. Anschließend soll untersucht werden, ob<br />
sich hochvernetzte synthetische Gelmatrizes zur Verwendung mit HT-1080 Zellen eignen.<br />
4.2.1.1. Chemotaxis in Collagen I Matrizes:<br />
Für die Versuche mit Matrizes aus bovinem Collagen I wurden HT-1080 Zellen vom Wildtyp<br />
zwischen der 30. und 50. Passage verwendet. Die verwendete Matrix enthielt 1,5mg/ml<br />
Collagen und wurde nach dem unter Punkt 3.4.1. beschriebenen Standard-Protokoll erstellt.<br />
Die Zellen wurden zu einer finalen Konzentration von 3x10 6 der Collagen Lösung<br />
beigemischt und wie in Punkt 3.5.1 beschrieben in die Mittelkanäle der Slides gefüllt.<br />
Während der etwa 45-Minütigen Polymerisationszeit des Collagens wurden die Slides in 10<br />
Minütigen Intervallen um 180° um die x-Achse gedreht, um ein Absinken der Zellen auf den<br />
Kanalboden und dadurch die Adhäsion der Zellen an feste Oberflächen zu vermeiden.<br />
Adhärente Zellen bewegen sich nur entlang einer Oberfläche und erlauben keine Aussage<br />
über Zellmigration durch Gelmatrizes.<br />
Nach dem Aushärten der Gelmatrix wurde dem linken Reservoir des Chemotaxis 3D-Slides<br />
DMEM mit 10% Foetalem Kälber Serum (FCS) als Chemoattraktant hinzugefügt. Das rechte<br />
Reservoir wurde als Referenz genutzt und mit DMEM ohne FCS gefüllt (+/-). Um die<br />
Signifikanz des Versuches zu erhöhen, wurden die beiden anderen Kammern der jeweiligen<br />
Slides ebenfalls mit Zellen in einer Gelmatrix gefüllt.<br />
Die Reservoirs einer Kammer wurden hier als sogenannte Positivkontrolle auf beiden Seiten<br />
mit DMEM gefüllt, dass 10% FCS enthielt (+/+). Die Reservoirs der verbleibenden Kammer<br />
enthielten als Negativkontrolle entsprechend nur DMEM (-/-).<br />
4. Ergebnisse 52
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Anschließend wurden die Zellen unter einem Nikon Eclipse Ti Phasenkontrastmikroskop in<br />
einer Zeitraffer Aufnahme für 24 Stunden aufgezeichnet. Die Aufnahmen wurden wie unter<br />
Punkt 3.5.2. beschrieben ausgewertet. Abbildung 28 zeigt beispielhaft den<br />
Chemotaxisversuch (+/-), sowie die Positiv (+/+) und Negativkontrolle (-/-) eines<br />
Experiments.<br />
Abbildung 28: Mit dem Chemotaxis and Migration Tool erstellter Plot der Zellbewegungen des<br />
Chemotaxis-Experiments (A), der negativ-Kontrolle (B) und der positiv-Kontrolle (C).<br />
Zellen deren Position sich im Vergleich zur Ausgangsposition nach links verlagert hat sind grün<br />
eingefärbt, Zellen die nach rechts gewandert sind rot.<br />
4. Ergebnisse 53
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Dabei wurden nur Zellen berücksichtigt, deren längliche, kompaktere Morphologie ohne<br />
sichtbaren Adhäsionsfilm auf eine Bewegung durch das Gel und nicht entlang der Oberfläche<br />
hindeutete. Aus den in Abbildung 28 dargestellten Migrationsmustern lässt sich für das<br />
Chemotaxis-Experiment (+/-) im Vergleich zu den Kontrollexperimenten schon eine starke<br />
linksgerichtete Tendenz der Zellbewegung erkennen. Aus den gemessenen Daten wurden im<br />
Chemotaxis und Migration Tool der Firma Ibidi rechnerisch einige Parameter ermittelt, die<br />
eine genauere Abschätzung erlauben, in wie weit es sich bei dem aufgetretenen<br />
Bewegungsmuster um Chemotaxis handelt.<br />
Tabelle 1 zeigt einen Auszug der Wertetabelle zu dem in Abbildung 28 dargestellten<br />
Chemotaxis Versuch. Die Beschreibung der Parameter findet sich unter Punkt 3.5.2.<br />
Konz. Chemoattraktant p-Value x-FMI Y-FMI Directionality Velocity [µm/min] Tracks<br />
10 % FCS 2,1079*10 -8 -0,3384 0,058 0.3947 0,455 29<br />
Tabelle 1: Auszug der gemessenen Chemotaxis Parameter des in Abbildung 29 dargestellten Experiments.<br />
Anhand dieser Parameter können die durchgeführten Versuche gut untereinander verglichen<br />
und auf ihre Reproduzierbarkeit geprüft werden. In Abbildung 29 sind die FMI von drei<br />
Chemotaxis Experimenten mit HT-1080 Zellen in1,5 mg/ml Collagen Gelen parallel (x-FMI)<br />
und senkrecht (y-FMI) zum Chemoattraktant-Gradienten gezeigt.<br />
Abbildung 29: Schematischer Vergleich der gemessenen FMI parallel und senkrecht zum<br />
Konzentrationsgradienten für den Chemotaxisversuch (+/-) aus drei Experimenten, sowie Negativ- (-/-)<br />
und Positiv-Kontrolle (+/+) zu je zwei Experimenten.<br />
Für alle 3 durchgeführten Experimente waren die Werte für den x-FMI deutlich negativ<br />
während y-FMI immer nahe 0 war. Die an zwei Experimenten durchgeführten Negativ- und<br />
Positiv-kontrollen zeigen sowohl für den x-FMI wie auch den y-FMI sehr kleine Werte an.<br />
4. Ergebnisse 54
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
4.2.1.3. Chemotaxis in synthetischen Gelmatrizes<br />
Durch Matrix-Metalloproteasen (MMP) auf ihrer Oberfläche sind HT-1080 Zellen fähig, sich<br />
durch vernetzte Strukturen mit definierten Verbindungsmolekülen zu schneiden, deren<br />
Maschenweite ansonsten zu klein für eine Bewegung der Zelle durch das Gel wäre. Vor<br />
diesem Hintergrund sollen im Folgenden kurz die Migrationseigenschaften dieser Zellen<br />
durch hochvernetzte Hydrogele getestet werden, deren gute optische Eigenschaften eine<br />
Qualitätsverbesserung mikroskopischer Aufnahmen versprechen. In Anbetracht der<br />
Parameter-Entkopplung kann eine Zusammensetzung der Matrix aus komplett synthetischen<br />
Materialen mit bekannten Mengenangaben auch helfen, die Abhängigkeit der Zellmigration<br />
von einzelnen Matrixparametern besser zu verstehen.<br />
Getestet wurden zwei kommerziell erhältliche Hydrogele unterschiedlicher Hersteller, deren<br />
Zusammensetzung und Eigenschaften, soweit bekannt, in Punkt 3.4.2.beschrieben sind. Als<br />
Chemoattraktant wurde in allen Fällen 10% FCS in DMEM verwendet. Zum Einfüllen der<br />
Chemoattraktant-Lösung wurden in allen Fällen die unter Punkt 3.5.2. auf Seite 33<br />
beschriebene Slow method verwendet.<br />
3D-Matrix der Firma Qgel<br />
Wie unter Punkt 3.4.2. auf Seite 29 beschrieben, wurden 3x10 6 HT-1080 Zellen der Qgel 3D-<br />
Matrix hinzugefügt und in die Mittelkanäle von Chemotaxis 3D-Slides gefüllt. Das Aushärten<br />
der Gelmatrix nahm etwa 10 Minuten in Anspruch. Die Zusammensetzung des Gels war<br />
größtenteils vom Hersteller vorgegeben. Getestet wurden die Varianten mit und ohne RGD-<br />
Peptid in der höchst möglichen Verdünnung.<br />
4. Ergebnisse 55
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Abbildung 30 zeigt Zellen im Qgel Hydrogel im Vergleich zu einer 1,5mg/ml Collagen I<br />
Matrix.<br />
Abbildung 30: Vergleich der Morphologie von HT-1080 Zellen in (A) einem Hydrogel der Firma Qgel und<br />
(B) einem Collagen I Gel.<br />
In der Aufnahme des Hydrogels sind keine Fasern zu erkennen und der Hintergrund erscheint<br />
einheitlicher. Die Zellen im Hydrogel zeigen eine rundlichere Morphologie und bilden<br />
deutlich weniger beziehungsweise kürzere Fortsätze aus. Die Zellen migrieren durch das<br />
Hydrogel, zeigen aber keine Vorzugsrichtung in Reaktion auf einen Lockstoffgradienten wie<br />
nachfolgende Wertetabelle zeigt.<br />
Chemoattraktant p-Value ǁ(x)-FMI ⊥(y)-FMI Directness Velocity [µm/min] Tracks<br />
10% FCS 0.5738 -0.<strong>06</strong>0 0.019 0.2936 0.429 29<br />
40 % FCS 0.2258 0.058 0.024 0.1617 0.486 26<br />
Tabelle 2: Migrationsparameter von HT-1080 Zellen in Qgel.<br />
Alle gezeigten Parameter deuten darauf hin, dass die Bewegung der Zellen nicht<br />
chemotaktisch erfolgt. Die Geschwindigkeit der Zellen entspricht mit einem mittleren Wert<br />
von 27.45 µm/h in etwa der Geschwindigkeit, die bei der Migration durch Collagen-Matrizes<br />
erreicht werden konnte. Es war mit der zur Verfügung stehenden Menge der Gelmatrix jedoch<br />
nicht möglich, genug Versuche durchzuführen, um Zellen in diesem Gel zur gerichteten<br />
Migration zu bringen.<br />
4. Ergebnisse 56
Cellendes-Hydrogele:<br />
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Getestet wurde hier ein Hydrogel mit 2,5mg/ml einer Maleimid-BSA Matrix. Die Versuche<br />
wurden ebenfalls mit 3x10 6 Zellen durchgeführt, wie unter Punkt 3.5.1. auf Seite 33ff<br />
beschrieben. RGD-Peptid musste manuell hinzugefügt werden, um den Zellen<br />
Bindungsmotive für zur Migration zur Verfügung zu stellen. Das Aushärten der Matrix<br />
erfolgte innerhalb weniger Minuten, so dass es nicht möglich war, das Gel ohne mechanische<br />
Verformungen in die Kanäle der Chemotaxis 3D Slides zu füllen.<br />
Abbildung 31: HT-1080 Zellen in einer 2,5mg/ml Maleimid-BSA Matrix der Firma Cellendes.<br />
In dem rot eingekreisten Bereich sind deutlich mechanische Verformungen der Matrix in Folge der<br />
Eintretenden Polymerisation während des Einfüllvorgangs in den Slide zu sehen.<br />
Zwischen der Konzentration an RGD Peptid und der Beweglichkeit der Zellen konnte eine<br />
positive Proportionalität bis zur möglichen Maximalkonzentration festgestellt werden, wie<br />
Tabelle 3 zeigt.<br />
RGD-Konz. Gesamt Beweglich Unbeweglich Tot<br />
0.2mM 26 4 22 5<br />
0.5mM 74 15 59 4<br />
1.0mM 60 24 36 6<br />
Tabelle 3: Zellzahlen geordnet nach Vitalität in Maleimid-BSA Matrizes der Firma Cellendes mit<br />
unterschiedlichen RGD-Konzentrationen.<br />
4. Ergebnisse 57
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Die Morphologie der Zellen in der Maleimid-BSA Matrix der Firma Cellendes gleicht in etwa<br />
derjenigen, die in Qgel beobachtet wurde. Die wenigen Ausläufer sind relativ kurz und die<br />
allgemeine Form der Zellen ist tendenziell eher rundlich. Die Zellen zeigen auch hier<br />
ungerichtete Bewegung durch das Gel ohne erkennbare Vorzugsrichtung in Reaktion auf<br />
einen chemotaktischen Stimulus. Die Bewegung durch das Gel unter Einsatz von MMP<br />
scheint aber möglich.<br />
4.2.2. HL-60 Zellen in Gelmatrizes<br />
Anhand der schnellen HL-60 Zellen soll die Eignung des µ-Slide für Chemotaxisexperimente<br />
getestet werden, die sich im Bereich weniger Stunden abspielen. Es soll ermittelt werden, ob<br />
die Zeit bis zur vollständigen Etablierung des Gradienten (vgl. 4.1.3. Seite 50f) erkennbare<br />
Auswirkungen auf das Migrationsverhalten der Zellen hat. Im Anschluss soll daraus ein<br />
angemessener Beobachtungzeitraum für Chemotaxis-Assays mit schnellen Zellen bestimmt<br />
werden.<br />
Im Vorfeld der Experimente mit HL-60 Zellen stand die Optimierung der<br />
Migrationsparameter für diese Zellen in Gelmatrizes. Die unter 3.2.1. beschriebene<br />
Differenzierung der Zellen gestaltete sich mit wechselhaftem Erfolg und in Abhängigkeit der<br />
Sorgfalt mit der das Differenzierungsmedium gewechselt wurde. Standardisierte<br />
Voraussetzungen sind folglich nicht für jedes unternommene Experiment als gegeben<br />
anzunehmen.<br />
Die Migration von HL-60 Zellen läuft der Literatur zu Folge hauptsächlich durch plastische<br />
Verformung der umgebenden Matrixfasern und Ausnutzung von Hohlräumen in der Matrix ab<br />
[Friedl et al., 2000]. Zur Migration von Neutrophilen finden sich Konzentrationen von<br />
1,8mg/ml Collagen in den verwendeten Matrizes [Grinell, 1982]. Für HL-60 Zellen in<br />
Rattenschwanz Collagen I Gelen wurden die besten Ergebnisse allerdings für eine<br />
Konzentration von 1mg/ml erzielt. An Zellen in stärker vernetzten Matrizes konnte in den<br />
meisten Fällen nur ein Zittern der Zelle innerhalb eines bestimmten Radius beobachtet<br />
werden. Bewegungen über Strecken, die das Maß einer Zelllänge [~15µm] überschreiten<br />
konnten kaum beobachtet werden.<br />
Die im Folgenden dargestellten Experimente wurden ausnahmslos mit einer Konzentration<br />
von 1mg/ml Collagen durchgeführt.<br />
4. Ergebnisse 58
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Als Chemoattraktant wurde in allen untersuchten Fällen N-Formylmethionyl-Lencyl-<br />
Phenylalanin (fMLP) verwendet. Zur Menge des verwendeten Chemoattraktant variieren die<br />
Literaturangaben stark in Abhängigkeit der verwendeten Untersuchungsmethode. Zur<br />
Ermittlung der idealen Konzentration wurden für C100 verschiedene Konzentrationen getestet.<br />
Tabelle 4 soll auf die wichtigsten kurz Bezug nehmen.<br />
Für alle aufgeführten chemotaktischen Untersuchungen wurde das Chemoattraktant in das<br />
links vom Mittelkanal gelegene Reservoir eingefüllt.<br />
Eine dadurch ausgelöste gerichtete Zellbewegung ist folglich in Form einer Wanderung in<br />
negativer x-Richtung zu erwarten.<br />
Die Bewegung von Einzelzellen wurden wie in Punkt 3.5.2. beschrieben mit Hilfe des<br />
„manual tracking“ Plugins der Software ImageJ verfolgt und in Tabelle 4 dargestellte Werte<br />
anschließend durch das Chemotaxis and Migration Tool der Firma Ibidi generiert.<br />
Konzentration p-Value ǁ(x)-FMI ⊥(y)-FMI Directness Velocity[µm/min] Tracks<br />
1µM 0,763 -0,014 0,026 0,153 4,85 21<br />
200nM 0,024 -0,048 -0,035 0,148 5,46 24<br />
10nM 0,092 -0,038 -0,013 0,139 4,39 22<br />
5nM 0,022 -0,097 -0,013 0,192 1,68 7<br />
Tabelle 4: Gemessene Chemotaxis-Parameter einer Versuchsreihe mit unterschiedlichen Chemoattraktant<br />
Konzentrationen.<br />
Tracks bezeichnet hier die Anzahl verfolgter Zellen. Diese dient auch als Maß für die<br />
statistische Signifikanz der Werte.<br />
Die gemessenen Werte zeigen tendenziell eine Zunahme des Verhältnisses der Beträge von x<br />
und y-FMI. Die Konzentration von 1µM ausgenommen, überschreitet der p-wert bei keinem<br />
der Experimente den Wert 0.01. Die Direktionalität der Zellenmigration ist für alle Ansätze<br />
weitestgehend einheitlich. Die Geschwindigkeit der Zellen liegt in dem Versuch mit 5nM<br />
fMLP um den Faktor 15, mit höheren fMLP-Konzentrationen in etwa um den Faktor 5<br />
unterhalb des Erwartungswertes von 22-27µm/min für vergleichbare neutrophile<br />
Granulozyten. [Foxman et al. 1999]<br />
4. Ergebnisse 59
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Die Entwicklung der FMI spricht für einen ansteigenden chemotaktischen Effekt mit<br />
Rückgang der Chemoattraktant Konzentration. (Tendenz in negative x-Richtung zu wandern<br />
nimmt zu) Die Geschwindigkeit und allgemeine Beweglichkeit der Zellen bei Zugabe von nur<br />
5nM fMLP waren zu gering um verwertbare Daten zu produzieren. Für weiterführende<br />
Untersuchungen wurde deshalb eine fMLP-Konzentration von 10nM als Chemoattraktant<br />
gewählt.<br />
An dieser Stelle sei angemerkt, dass die in der Tabelle 4 gelisteten Werte chemotaktische<br />
Stimuli andeuten, aber weit hinter einer erwarteten Reaktion zurückbleiben.<br />
Es war im Rahmen des praktischen Zeitkontingents dieser Arbeit leider nur möglich ein<br />
einziges Chemotaxis Experiment an HL-60-Zellen durchzuführen, dessen Ergebnisse deutlich<br />
genug ausfallen, um daraus die andeutungsweise weiterführende Zusammenhänge abzuleiten.<br />
Es ist vor Abschluss der Versuchsreihen nicht gelungen, dessen Reproduzierbarkeit zu<br />
gewährleisten. Im Folgenden soll es unabhängig davon einer kurzen Analyse und einer<br />
Abschätzung der Zusammenhänge zum Diffusionsprofil im µ-Slide unterzogen werden.<br />
HL-60 Zellen wurden nach einem Differenzierungszeitraum von 5 Tagen in einer Collagen<br />
Matrix in Chemotaxis 3D µ-Slides eingebettet und etwa 30 Minuten nach Befüllen der<br />
Reservoire mit Chemoattraktant über einen Zeitraum von 3 Stunden beobachtet. Die<br />
Schrittweite der Zeitraffer Aufnahmen wurde auf 1 Minute festgelegt. Als Chemoattraktant<br />
wurde fMLP mit einer Konzentration von 10nM verwendet.<br />
Die ermittelten Parameter sind in Tabelle 5 aufgelistet.<br />
Chemoattraktant p-Value ǁ(x)-FMI ⊥(y)-FMI Directness Velocity [µm/min] Tracks<br />
10nM fMLP 2.2542*10 -7 -0.189 0.040 0.2471 7.243 26<br />
Tabelle 5: Chemotaxis Parameter des zum Vergleich mit den Diffusionsmessungen herangezogenen<br />
Experiments.<br />
Um festzustellen, ob sich die untersuchten Zellen innerhalb der Gelmatrix bewegen, muss die<br />
Morphologie während der Migration betrachtet werden. Die Form der Zellen ist im ruhenden<br />
Zustand meist rundlich und während der Bewegung langgezogen und spindelförmig.<br />
4. Ergebnisse 60
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Abbildung 32 zeigt eine Zelle während der Bewegung in Richtung des chemotaktischen<br />
Stimulus aus dem linken Reservoir des Slides.<br />
Abbildung 32: Bilderreihe von Phasenkontrastaufnahmen zur Darstellung der morphologischen<br />
Anpassung der Zelle während der Migration durch eine Gelmatrix.<br />
Die Anfangs rundliche Form des Zellkörpers zieht sich während des Bewegungsschrittes in<br />
die Länge und nimmt eine spindelförmige Form an. Nach der Bewegung ist der Zellkörper<br />
wieder komprimiert und rundlich. Die dunkleren Fortsätze an dem Zellkörper sind Ausläufer<br />
der Zelle, mit welchen Adhäsionspunkte zur Matrix hergestellt oder gelöst werden. In<br />
Abbildung 33 sind die Wegstrecken zu sehen, welche die Zellen über den<br />
Beobachtungszeitraum zurückgelegt haben. Die schwarzen Punkte stellen die Endpositionen<br />
der Zellkörper dar.<br />
Abbildung 33: Bewegungsmuster von HL-60 Zellen in einem Collagen I Gel mit 1,0mg/ml als Reaktion<br />
auf einen Stimulus mit 10nM fMLP.<br />
4. Ergebnisse 61
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Um die Vorzugsrichtung zu verdeutlichen, sind Zellen deren Position sich relativ zum Start<br />
des Experiments in positiver x-Richtung verschoben hat rot gefärbt, Zellen mit negativer<br />
x-Verschiebung schwarz. Der gemittelte Massenschwerpunkt der Kreise, welche die<br />
Endpositionen der Zellen darstellen, ist als blaues Kreuz dargestellt. Anhand der<br />
Farbmarkierung der Spuren ist eine eindeutige Vorzugsrichtung erkennbar, was auch die<br />
Positionierung des Massenschwerpunktes unterstreicht, der eine deutliche Auslenkung in<br />
negativer x-Richtung erfahren hat.<br />
In Abbildung 34 ist die Entwicklung der x und y Koordinaten des Massenschwerpunkts aller<br />
Zellen über die Zeit gezeigt.<br />
Abbildung 34: Verlagerung der Position des Massenschwerpunkts in x und in y Richtung in Abhängigkeit<br />
der Zeit.<br />
Während die y-Koordinate trotz des Anstiegs innerhalb der letzten Stunde weitestgehend<br />
konstant um den Nullpunkt verläuft, zeigt sich für die x-Koordinate ein linearer Abfall in den<br />
negativen Bereich über das gesamte Zeitfenster. Bezogen auf die Zellen stehen diese Werte<br />
für eine Bewegung die gleichermaßen in positive wie negative y-Richtung aber mit einer<br />
eindeutigen Vorzugsrichtung in negativer x-Richtung abläuft, die die Wahrscheinlichkeit eines<br />
zufällig auftretenden Effekts minimiert.<br />
4. Ergebnisse 62
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
4.2.3. Migrationsanalyse durch Aktinfärbung mit LifeAct-tag GFP2/RFP<br />
Neben Wildtyp Zellen wurden von den jeweiligen Zellkulturen auch LifeAct-tag GFP2/RFP<br />
transfizierte Stämme kultiviert und in Chemotaxis-Experimenten untersucht. Das LifeAct-tag<br />
lagert Moleküle des Green fluorescent protein (GFP) beziehungsweise des Red fluorescent<br />
protein (RFP) an die Aktinmoleküle der Zellen an. Die Migration der Zellen findet, wie in<br />
Punkt 1.3. beschrieben, durch Umlagerungen des Aktingerüsts innerhalb der Zelle statt.<br />
Durch den Einsatz fluoreszenzmarkierten Aktins ist es möglich die Umlagerung und<br />
Ausrichtung des Aktins während der Migration zu beobachten.<br />
Abbildung 35 zeigt die Aktinstrukturen einer HT-1080 Zelle mit LifeAct-tag RFP.<br />
Abbildung 35: HT-1080-Zelle mit LifeAct-tag RFP.<br />
In der Abbildung 35 ist eine Anhäufung des gefärbten Aktins im Zentrum des Zellkörpers zu<br />
erkennen. Die Fortsätze mit denen sich die Zelle in dem umgebenden Gel orientiert und<br />
festhält sind ebenfalls deutlich erkennbar.<br />
Für HT-1080 Zellen war während der durchgeführten Chemotaxisversuche keine Verlagerung<br />
der Fluoreszenzintensität in Bewegungsrichtung zu erkennen, obwohl die Zellen in ihrem<br />
Migrationsverhalten und der Reaktion auf den chemotaktischen Stimulus der Wildtyp-<br />
Variante gleichen.<br />
Erkennbar waren nur eine permanente dynamische Umlagerung der Aktinstrukturen im<br />
Inneren der Zelle während der Bewegung und eine deutliche Intensitätsanreicherung an dem<br />
hinteren Ende der Zellen beim Lösen von Verankerungen aus dem Gel.<br />
4. Ergebnisse 63
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Abbildung 36 zeigt Bilddaten eines HT-1080 Chemotaxis-Experiments im Phasenkontrast<br />
und als Fluoreszenzaufnahme.<br />
Abbildung 36: Vergleich zwischen (A) Phasenkonstrast und (B) Fluoreszenzaufnahme von HT-1080 mit<br />
Life Act-tag RFP in einer Collagenmatrix.<br />
Für HL-60 Zellen konnten wegen der undefinierten Migrationsparameter keine<br />
Fluoreszenzdaten von gerichteter Migration generiert werden.<br />
Man erkennt aber eine deutliche Anreicherung markierter Strukturen am Frontende der Zelle<br />
in Richtung einer zufälligen, ungerichteten Bewegung, wie in Abbildung 37 zu erkennen ist.<br />
Abbildung 37: Fluoreszenzaufnahme einer HL-60 Zelle mit LifeAct-tag GFP2 während einer<br />
ungerichteten Bewegung.<br />
Die Fortsätze mit denen das Gel durchdrungen wird (rote Pfeile), um Adhäsionspunkte für die<br />
folgende Bewegung zu schaffen weisen ebenfalls eine starke Fluoreszenzintensität auf.<br />
4. Ergebnisse 64
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
5. Diskussion und Ausblick<br />
5.1. Bewertung der Ergebnisse<br />
5.1.1. Diffusionsmessungen<br />
Obwohl die LSM Messungen durch fehlende Referenzmessungen in ihrer Aussagekraft<br />
eingeschränkt wurden lassen sich daraus einige wichtige Erkenntnisse ableiten.<br />
Durch die Verwendung der Slow-method , liegt die Konzentration C100 nicht von Anfang an<br />
am linken Rand des Kanals an. In Folge interner Diffusionsvorgänge im Reservoir von C100<br />
kommt es dadurch zu einer Schrittweisen Etablierung des Gradienten. Dieser Vorgang kann<br />
anhand der deutlich flacher verlaufenden Kurven der 1-Stunden Messungen in Abbildung 22,<br />
unter Punkt 4.1.2. auf Seite 44 abgeschätzt werden Diese Beobachtung lässt vermuten, dass<br />
die Etablierung des Gradienten schon innerhalb weniger Stunden abläuft. Alle weiteren<br />
gemessenen Konzentrationsprofile sind annähernd deckungsgleich In Folge des 2. Fick’schen<br />
Gesetzes müsste sich in Folge der Diffusion mit der Zeit ein Konzentrationsausgleich und<br />
damit ein Abflachen des linearen Bereichs der Kurvensteigungen aus Abbildung 22 um einen<br />
Drehpunkt bei etwa 50% von C100 einstellen. Damit ergibt sich für den erwarteten Verlauf der<br />
Konzentrationsprofile ein Muster, wie schematisch in Abbildung 38 dargestellt.<br />
Abbildung 38: Schematische Darstellung der erwarteten Konzentrationsverläufe.<br />
Aufgetragen sind Erwartungswerte (A) nach vollständiger Etablierung des Gradienten (t~12h) bis (D)<br />
kurz vor Einstellung des Konzentrationsgleichgewichts.<br />
5. Diskussion und Ausblick 65
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Es ist wahrscheinlich, dass sich die Konzentration nicht exakt um den beschriebenen<br />
Drehpunkt einpendelt, weil die beiden Reservoire C0 und C100 in Folge interner<br />
Diffusionsvorgänge keine optimale Quelle beziehungsweise Senke darstellen, aber ein<br />
Abflachen der Kurvensteigung sollte zu beobachten sein. Dieser Rückgang ist im Rahmen des<br />
Messfehlers der gesammelten Daten nicht erkennbar. Referenzmessungen in allen Kammern<br />
wären für jeden Slide notwendig gewesen. Der damit verbundene zeitliche Aufwand war<br />
jedoch zu groß für das zur Verfügung stehende Zeitkontingent.<br />
Obwohl die Messungen am LSM für eine detaillierte Messung der Entwicklung des<br />
Konzentrationsprofils im zellbiologisch relevanten Zeitraum von 48 Stunden zu störanfällig<br />
sind, konnte mit ihrer Hilfe gezeigt werden, dass der Gradient im Chemotaxis 3D-Slide nach<br />
anfänglicher Etablierung für mindestens 36 Stunden konstant bleibt und unabhängig von der<br />
verwendeten Gelmatrix verläuft. Ähnlicher Ergebnisse bei der Verwendung von Farbstoffen<br />
stark unterschiedlicher Molekülgrößen legen nahe, dass der Durchmesser der diffundierenden<br />
Partikel keinen Einfluss auf die Form des Gradienten hat.<br />
Durch FCS Messungen konnten schließlich Etablierung und Verlauf des Gradienten detailliert<br />
gezeigt werden. Auch hier ist erkennbar, dass die Verwendung unterschiedlich stark vernetzter<br />
Gelmatrizes keine Rolle spielt. Es konnte gezeigt werden, dass, wie anhand der LSM<br />
Ergebnisse vermutet, die Etablierung des Gradienten zu einem Großteil schon innerhalb der<br />
ersten 1-2 Stunden stattfindet. In Folge des Konzentrationsausgleichs nach dem 2. Fick’schen<br />
Gesetz konnte eine leichte Abnahme des Gradienten nach 24 Stunden festgestellt werden, die<br />
sich aber in einem von den Zellen vermutlich nicht wahrnehmbaren Bereich abspielt.<br />
Für Chemotaxisexperimente ergibt sich damit ein Zeitraum von 1-48 Stunden, in welchem,<br />
bezogen auf den Gradienten, einheitliche Bedingungen angenommen werden können.<br />
5.1.2. Chemotaxis-experimente mit HT-1080 und HL-60 Zellen<br />
Parallel durchgeführte Experimente mit HT-1080 Zellen zeigen deutlich und reproduzierbar,<br />
dass die Zellen fähig sind in den µ-Slides Chemotaxis 3D über einen Zeitraum von mehr als<br />
24-Stunden, als Reaktion auf einen chemotaktischen Stimulus, durch eine Gelmatrix zu<br />
migrieren. Um bestimmten zu können, ob Unterschiede im Migrationsverhalten der Zellen im<br />
Gel im Vergleich zur 2-dimensionalen Migration auf einer Oberfläche auftreten, wurden die<br />
unter Punkt 4.2.1.1. beschriebenen Daten mit einem bestehenden Datensatz von 2d-<br />
Migrationsdaten verglichen.<br />
Abbildung 39 stellt dazu die Verlagerung des Massenschwerpunkts (COM) in x- und in y-<br />
5. Diskussion und Ausblick 66
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Richtung, den FMI in x- und in y- Richtung und die Migrationsgeschwindigkeit von jeweils 3<br />
Experimenten in 2D und in 3D gegenüber. Die Werte für die Verschiebung des<br />
Massenschwerpunkts in den 3D-Migrationsexperimenten wurden mit dem Faktor -1<br />
multipliziert, um eine bessere Vergleichbarkeit der Ergebnisse zu erhalten.<br />
Abbildung 39: Schematischer Vergleich von (A,B) FMI, (C,D) p-Value (Rayleigh Test), (E,F) Verschiebung<br />
des Massenschwerpunkts und (G,H) Geschwindigkeit der Zellen in (A,C,E,G) 2-dimensionalen und<br />
(B,D,F,H) 3-dimensionalen Chemotaxisexperimenten.<br />
Für alle Versuche wurden zusätzlich die Kontrollexperimente mit aufgenommen.<br />
5. Diskussion und Ausblick 67
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Die FMI parallel zum Gradienten sind für alle Chemotaxis Experimente (+/-) betragsmäßig in<br />
einem Bereich um 0,3. Die FMI senkrecht zum Gradienten sowie alle Kontrollmessungen<br />
bewegen sich im weitesten Sinne um den Nullpunkt. Zwischen 2D und 3D Versuchsansätzen<br />
bestehen hier keine nennenswerten Unterschiede. Die Verlagerung des Massenschwerpunkts<br />
weist für die Versuche in 3D leicht höhere Werte, aber auch eine höhere Streuung der<br />
Ergebnisse auf. Die unterschiedlichen Migrationsmodi scheinen auch hier keinen<br />
entscheidenden Einfluss zu haben. Die Geschwindigkeit der Zellen war in 2D und 3D<br />
Experimenten annähernd exakt gleich. Nur die Zellen der Positivkontrolle in 2D bewegen<br />
sich merklich schneller. Die Negativkontrolle weist in beiden Fällen leicht niedrigere<br />
Migrationsgeschwindigkeiten auf. Der Mittelwert der Geschwindigkeit aus allen betrachteten<br />
Experimenten entspricht mit ~25,7µm/h in etwa dem in der Literatur angegebenen<br />
Wertebereich für Fibroblasten von 30-40 µm/h. [Even-Ram, 2005]<br />
Die Verwendung von synthetischen Hydrogelen im µ-Slide Chemotaxis 3D ist möglich.<br />
Eine gerichtete Migration der Zellen in den Gelen hängt aber von zu vielen Parametern ab, als<br />
dass sie im Rahmen dieser Arbeit noch hätte erfolgreich gezeigt hätte werden können.<br />
Die optische Qualität der Ergebnisse ist gut und spricht weiteren Experimenten mit<br />
synthetischen Matrizes eine nicht unbedeutende Rolle zu.<br />
Obwohl für HL-60 Zellen mit Abschluss der Versuchsreihen noch keine reproduzierbaren<br />
Chemotaxis-Daten vorlagen, konnte anhand von Einzelergebnissen angedeutet werden, dass<br />
die Etablierung des Gradienten keinen Einfluss auf das Migrationsverhalten der Zellen<br />
ausübt. Bei Analyse des FMI in Abhängigkeit von der Zeitkoordinate, wie in Abbildung 34<br />
dargestellt, zeigt sich ein konstant hoher Wert für den Betrag des ǁ(x)-FMI und ein Wert nahe<br />
0 bezogen auf den ⊥(y)-FMI. Dabei fällt ein stark verrauschter Bereich während der ersten 30<br />
Minuten der Messung auf.<br />
5. Diskussion und Ausblick 68
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Interessant stellt sich diese Beobachtung im Vergleich mit dem in Abbildung 27 unter Punkt<br />
4.1.3 auf Seite 50 gezeigten Verlauf der Steigung des Konzentrationsprofils in den ersten<br />
Stunden während der Etablierung dar.<br />
Abbildung 40: Vergleich zwischen (A) der Entwicklung des Gradienten über die Zeit während der<br />
Etablierung und (B) dem Verlauf der FMI der HL-60 Zellen während dieser Zeit.<br />
Nach dem anfänglichen Rauschen, das in etwa der Zeitspanne des steilen Anstiegs des<br />
Gradienten entspricht, bleibt das Verhalten des FMI, ähnlich wie der Verlauf des Gradienten<br />
tendenziell konstant.<br />
Analysiert man die Entwicklung des FMI anhand des Gradientenverlaufes, ist es möglich,<br />
zwischen dem steilen Anstieg des Chemoattraktant-Gradienten innerhalb der ersten Stunden<br />
und dem anfänglichen „Rauschen“ des FMI einen Zusammenhang zu sehen. Ein gesicherte<br />
Behauptung lässt sich hier aber nicht aufstellen, weil das auftretende Rauschen auch durch<br />
eine hohe Varianz der akkumulierten Migrationsstrecke der Zellen innerhalb der ersten<br />
5. Diskussion und Ausblick 69
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Minuten zustande kommt, die in die Formel für die Berechnung des FMI mit einfließt. Nach<br />
etwa 30 Minuten gestaltet sich der Verlauf des FMI aber konstant, was darauf schließen lässt,<br />
dass auch Experimente mit schnellen Zellen und über einen kurzen Zeitraum keiner<br />
anfänglichen Wartezeit bedürfen.<br />
Anhand der Morphologie der Zellen kann geschlossen werden, dass die Migration der Zellen<br />
innerhalb der Matrix stattfindet und nicht entlang der Kanaldecke oder des Bodens.<br />
Die spindelförmige Form während der Bewegung und der rundliche Zellkörper ohne den, bei<br />
adhärenten Zellen sichtbaren Adhäsionsfilm um die Zelle, sind Zeichen dafür, dass die<br />
Bewegung der Zellen durch Anpassung an die Gelmatrix erfolgt.<br />
Obwohl es bis zum Ende des Versuchszeitraumes nicht gelungen ist, Migrationsexperimente<br />
von HL-60 in Gelmatrizes mit durchwegs eindeutigen Ergebnissen zu reproduzieren,<br />
gestaltete sich die Arbeit mit dieser Zellinie vielversprechend. Es ist zu erwarten, dass durch<br />
die gewonnenen Erkenntnisse die Möglichkeit besteht, mit etwas zusätzlichem Aufwand<br />
reproduzierbare Chemotaxis-Assays mit HL-60 Zellen zu entwerfen, die stellvertretend für<br />
Versuche mit schnellen Zellen gesehen werden können.<br />
Die Versuche mit dem LifeAct-tag haben einen ersten Blick in die Vorgänge im Inneren der<br />
Zellen während der gerichteten Migration ermöglicht. Dabei scheint sich die Dynamik der<br />
Umlagerung des Aktingerüsts zwischen HT-1080 Zellen und HL-60 Zellen merklich zu<br />
unterscheiden, wie die Betrachtung der unterschiedlich starken Polarisation der Zellen zeigt.<br />
Es wäre möglich, dass dieser Effekt auf die, unter Punkt 1.3. beschriebenen, unterschiedlichen<br />
Migrationsmodi der beiden Zelllinien zurückzuführen ist, wonach langsame, integrin-<br />
vermittelt migrierende Zellen wie HT-1080, weit weniger polarisieren, als schnelle,<br />
vermutlich Integrin-unabhängig agierende Zellen wie HL-60 [Even-Ram, 2005]. Das geringe<br />
Volumen der vorhandenen Daten erlaubt allerdings keine weitere Einschätzung dieser<br />
Beobachtung.<br />
Die Probleme bei der Bereitstellung der richtigen Migrationsparameter für HL-60 Zellen und<br />
der kurze Zeitraum, in welchem transfizierte HT-1080 Zellen für Versuche zur Verfügung<br />
standen, führten dazu, dass das Potential dieser Methode im Rahmen dieser Arbeit nicht<br />
annähernd ausgeschöpft werden konnte. Die unter Punkt 4.2.3. vorgestellten Ergebnisse<br />
vermitteln jedoch einen ungefähren Eindruck, auf welcher Ebene Chemotaxis Experimente in<br />
naher Zukunft ausgewertet werden können.<br />
5. Diskussion und Ausblick 70
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Zusammenfassend lässt sich aussagen, dass die Daten der Chemotaxis-Experimente mit<br />
HT-1080 und HL-60 Zellen die Daten aus den Diffusionsmessungen bestätigen. Die schnelle<br />
Etablierung und der anschließend über mehrere Stunden konstante Verlauf des Gradienten<br />
erfüllen die Anforderungen für Chemotaxisexperimente mit schnellen und mit langsamen<br />
Zellen. Das µ-Slide Chemotaxis 3D liefert die Grundlage dafür, neue Erkenntnisse im Bereich<br />
der Chemotaxisforschung zu gewinnen, die sich aus der Arbeit mit Zellen in Gelmatrizes<br />
ergeben.<br />
5. Diskussion und Ausblick 71
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
5.2 Weiterführende Ansätze<br />
Im Rahmen dieser Diplomarbeit wurden weiterhin einige Methoden untersucht, die ebenfalls<br />
Verbesserungspotential für zukünftige Chemotaxis-Experimente in sich tragen.<br />
Softwaregestütztes Zell-tracking:<br />
So wurde unter anderem eine auf Math-Lab basierende Software zum automatischen Tracking<br />
der Zellen bei der Auswertung der Bilddaten von Migrationsexperimenten getestet.<br />
Das Time Lapse Analyser (TLA) getaufte Programm der Arbeitsgruppe um H.Kestler aus der<br />
<strong>Fakultät</strong> für Informatik der Universität Ulm, erkennt anhand der Kontraste zum Hintergrund<br />
die Position der Zellkörper und erstellt daraus für alle vorhandenen Zellen ein<br />
Bewegungsprofil. Der mühsame, sehr zeitaufwändige Prozess des manuellen Zell-tracking<br />
könnte dadurch überflüssig werden. Genauere Aussagen über die Funktionalität der Software<br />
können allerdings mangels Rechnerleistung und der notwenigen Einarbeitungszeit hier nicht<br />
getroffen werden.<br />
Digitalholographische Mikroskopie:<br />
Bei einem Besuch am Lehrstuhl für Biophysik der Universität Münster wurde durch die<br />
Arbeitsgruppe von Herrn Dr. Björn Kemper zur Gewinnung realistischer 3D-Bilddaten ein<br />
Verfahren der Digitalholographischen Mikroskopie vorgestellt. In Kombination mit dem µ-<br />
Slide Chemotaxis 3D kann 3-dimensionale Struktur der Zellen während der Migration<br />
dadurch erstaunlich genau dargestellt werden.<br />
3-dimensionales Zell-tracking:<br />
Der Trend zu Chemotaxis-Experimenten in Gelmatrizes legt es des Weiteren nahe sich mit<br />
möglichen Verfahren zu Zell-Tracking in 3D auseinanderzusetzen. Durch die geringe z-<br />
Komponente des Mittelkanals im µ-Slide Chemotaxis 3D ist die Bewegung der Zellen in z-<br />
Richtung stark eingeschränkt. Die Analyse der Migration erfolgt aber trotzdem ausschließlich<br />
in 2 Dimensionen und berücksichtigt Bewegungen entlang der z-Achse nicht. Durch<br />
sogenanntes z-stacking ist es möglich zu einem Zeitpunkt mehrere Bilder unterschiedlicher z-<br />
Ebenen aufzunehmen. Die Wanderung der Zelle über einen bestimmten Bereich der z-Achse<br />
entspricht einem Übertritt in eine andere Schärfeebene des verwendeten Objektivs. Analysiert<br />
man anschließend die gewonnenen Bilddaten, kann die z-Position der Zelle anhand der<br />
Schärfe der Einzelaufnahmen in den Bildstapeln gefolgert werden.<br />
5. Diskussion und Ausblick 72
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
6. Zusammenfassung<br />
Anhand der Ergebnisse der konfokalen Fluoreszenzmikroskopie-Techniken konnte gezeigt<br />
werden, dass sich ein Stoffgradient im µ-Slide Chemotaxis 3D innerhalb kurzer Zeit etabliert<br />
und anschließend mindestens 48 Stunden annähernd konstant bleibt.<br />
Die Wahl der verwendeten Gelmatrix, genau wie die Größe des diffundierenden Stoffes<br />
spielen dabei keine Rolle, so lange die Größe der Stoffpartikel deutlich kleiner ist, als die<br />
Maschenweite der Matrix.<br />
Dies ist für die meisten Chemotaxine in Verbindung mit kommerziell erhältlichen Hydrogelen<br />
der Fall.<br />
Anhand mehrerer Experimente an HT-1080-Zellen konnte gezeigt werden, dass sich der µ-<br />
Slide Chemotaxis 3D gut zur Gewinnung 3-dimensionaler Migrationsdaten an langsamen<br />
Zelllinien eignet.<br />
Der Vergleich der HT-1080 3D-Daten mit vorhandenen 2D-Chemotaxis Daten und den aus<br />
der Literatur bekannten Werten liefern, abgesehen von der Migrationsgeschwindigkeit der<br />
Zellen in der Positivkontrolle keine nennenswerten Unterschiede.<br />
HL-60 Zellen konnte Chemotaxis in GelMatrizes im Rahmen dieser Arbeit nicht<br />
reproduzierbar nachgewiesen werden. Daten aus Einzelexperimenten weisen jedoch darauf<br />
hin, dass dies möglich ist und legen eine Eignung des µ-Slide Chemotaxis 3D für<br />
Experimente mit schnellen Zellen nahe.<br />
Versuche mit LifeAct-tag RFP/GFP2 ermöglichten leider nur wenige Einblicke in die<br />
Aktindynamik der Zellen während der Migration, zeigen aber das Potential der Methode in<br />
Kombination mit den guten optischen Eigenschaften der µ-Slides.<br />
6.Zusammenfassung 73
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
7. Abbildungsverzeichnis:<br />
Abbildung 1: Die verschiedenen Arten stimulierter Zellbewegungen eukaryontischer Zellen<br />
in schematischer Darstellung [Kőhidai L., 20<strong>06</strong>] ...................................................................... 7<br />
Abbildung 2: Polarisation eines neutrophilen Granulozyten. [Weiner et al., 1999] ................. 8<br />
Abbildung 3: Modifizierte Boyden-Kammer mit Gelmatrix. ................................................. 11<br />
Abbildung 4: Aufbauskizze einer Zigmond-Kammer [Zigmond, 1977] ................................ 12<br />
Abbildung 5: Schematische Darstellung eines µ-fluidischen Chemotaxis-Systems [Jeon et<br />
al., 2002] ................................................................................................................................... 13<br />
Abbildung 6: Schematische Darstellung des µ-Slide Chemotaxis 3D in der Draufsicht. [Ibidi<br />
GmbH, 2010]............................................................................................................................ 18<br />
Abbildung 7: Schematische Schnittdarstellung des µ-Slide Chemotaxis 3D entlang der x-<br />
Achse. [Ibdi GmbH, 2010] ....................................................................................................... 19<br />
Abbildung 8:Morphologisches Erscheinungsbild von undifferenzierten HL-60 Zellen und<br />
HL-60 Zellen nach 6 Tagen in 60mM DMSO. [Wright-Giemsa Färbung, Collins, 1978] ...... 22<br />
Abbildung 9: Schematischer Aufbau von F-Aktin mit LifeAct-tag GFP2/RFP [modifiziert<br />
aus Produktflyer, Ibidi GmbH, 2011] ....................................................................................... 24<br />
Abbildung 10: Schematische Darstellung des konfokalen Prinzips.[aus: Commons: Bilder<br />
zum Funktionsweise des konfokalen Prinzips, Stand September 2011] .................................. 26<br />
Abbildung 11: Verdünnungsgraphen verschiedener Fluorophore. [zur Verfügung gestellt vom<br />
Lehrstuhl für Biophysik, Dr. J. Raedler, LMU-München, 2011] ............................................. 28<br />
Abbildung 12: Schematische Darstellung des BSA-Proteins mit funktionellen Maleimid-<br />
Gruppen [modifiziert aus: Cellendes – Hydrogel User Guide Version 3.0, 2011] ................... 30<br />
Abbildung 13: Schematische Darstellung des PEG-Crosslinkers mit funktionellen Thiol-<br />
Gruppen und spaltbarer Peptidsequenz. [Cellendes – Hydrogel User Guide Version 3.0, 2011]<br />
.................................................................................................................................................. 30<br />
7.Abbildungsverzeichnis: 74
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Abbildung 14: Schematische Darstellung der Verteilung der Chemoattraktant<br />
Konzentrationen auf die Kammern des µ-Slides Chemotaxis 3D zur Ermittlung der<br />
einzusetzenden Dosis. .............................................................................................................. 32<br />
Abbildung 15: Schematische Darstellung der Verteilung der Chemoattraktand-<br />
Konzentrationen für Chemotaxis Versuche mit Kontrollexperimenten. .................................. 33<br />
Abbildung 16: Vergleich der Phasenkontrastbilder unmittelbar nach Start eines<br />
Chemotaxisexperiments und nach 50 Minuten mit farbig markierten Zellspuren. .................. 34<br />
Abbildung 17: Schematische Darstellung der Versuchsdurchführung zur Matrix-Interaktion<br />
von Fluoreszenzfarbstoffen in einer Kammer des Angiogeneseslide ...................................... 38<br />
Abbildung 18: Verlauf der gemessenen Fluoreszenz-Intensität während der Versuche für<br />
Alexa 488 und FiTC-Dextran in Collagen Matrizes unterschiedlicher Stärke. ....................... 39<br />
Abbildung 19: Lage der konfokalen Schnittebene in einer Kammer der Chemotaxis 3D µ-<br />
Slide.......................................................................................................................................... 41<br />
Abbildung 20: Bilddaten einer LSM-Profilmessung bestehend aus 15*2 Einzelbildern und<br />
zugehöriges Intensitätsprofil. ................................................................................................... 42<br />
Abbildung 21: Schematische Darstellung der Datenanalyse durch LSM-Messungen ........... 43<br />
Abbildung 22: Konzentrationsprofile der Farbstoffe Alexa 488 und FITC-Dextran in einer<br />
CollagenI – Matrix. .................................................................................................................. 44<br />
Abbildung 23: Die Gradienten von Alexa 488 und FiTC-Dextran aufgetragen über die Zeit<br />
von 0-36h. ................................................................................................................................ 45<br />
Abbildung 24: Verdünnungsgraph für Alexa 488 zwischen C0 und C100 der eingesetzten<br />
Konzentrationen. ...................................................................................................................... 47<br />
Abbildung 25: Positionierung der FCS – Messpunkte in einer Kammer des µ-Slide. ........... 48<br />
Abbildung 26: Graphen der FCS-Messungen über einen Zeitrahmen von 15 min bis 48h.... 49<br />
7.Abbildungsverzeichnis: 75
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Abbildung 27: Entwicklung des Gradienten aus den FCS-Messungen .................................. 50<br />
Abbildung 28: Mit dem Chemotaxis and Migration Tool erstellter Plot von Zellbewegungen<br />
.................................................................................................................................................. 53<br />
Abbildung 29: Schematischer Vergleich der gemessenen FMI parallel und senkrecht zum<br />
Konzentrationsgradienten für den Chemotaxisversuch (+/-) aus drei Experimenten, sowie<br />
Negativ- (-/-) und Positiv-Kontrolle (+/+) zu je zwei Experimenten. ...................................... 54<br />
Abbildung 30: Vergleich der Morphologie von HT-1080 Zellen in einem Hydrogel der Firma<br />
Qgel und einem Collagen I Gel. ............................................................................................... 56<br />
Abbildung 31: HT-1080 Zellen in einer 2,5mg/ml Maleimid-BSA Matrix der Firma<br />
Cellendes. ................................................................................................................................. 57<br />
Abbildung 32: Bilderreihe von Phasenkontrastaufnahmen zur Darstellung der<br />
morphologischen Anpassung der Zelle während der Migration durch eine Gelmatrix. .......... 61<br />
Abbildung 33: Bewegungsmuster von HL-60 Zellen in einem Collagen I Gel mit 1,0mg/ml<br />
als Reaktion auf einen Stimulus mit 10nM fMLP. ................................................................... 61<br />
Abbildung 34: Verlagerung der Position des Massenschwerpunkts in x und in y Richtung in<br />
Abhängigkeit der Zeit. ............................................................................................................. 62<br />
Abbildung 35: HT-1080-Zelle mit LifeAct-tag RFP. ............................................................. 63<br />
Abbildung 36: Vergleich zwischen Phasenkonstrast und Fluoreszenzaufnahme von HT-1080<br />
mit Life Act-tag RFP in einer Collagenmatrix. ........................................................................ 64<br />
Abbildung 37: Fluoreszenzaufnahme einer HL-60 Zelle mit LifeAct-tag GFP2 während einer<br />
ungerichteten Bewegung. ......................................................................................................... 64<br />
Abbildung 38: Schematische Darstellung der erwarteten Konzentrationsverläufe. ............... 65<br />
Abbildung 39: Schematischer Vergleich von FMI, p-Value (Rayleigh Test), Verschiebung des<br />
Massenschwerpunkts und Geschwindigkeit der Zellen in 2-dimensionalen und 3-<br />
dimensionalen Chemotaxisexperimenten. ................................................................................ 67<br />
7.Abbildungsverzeichnis: 76
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Abbildung 40: Vergleich zwischen der Entwicklung des Gradienten über die Zeit während<br />
der Etablierung und dem Verlauf der FMI der HL-60 Zellen während dieser Zeit. ................ 69<br />
7.Abbildungsverzeichnis: 77
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
8. Tabellenverzeichnis<br />
Tabelle 1: Auszug der gemessenen Chemotaxis Parameter des in Abbildung 29 dargestellten<br />
Experiments. ............................................................................................................................. 54<br />
Tabelle 2: Tabelle: Migrationsparameter von HT-1080 Zellen in Qgel. ................................. 56<br />
Tabelle 3: Tabelle: Migrationsparameter von HT-1080 Zellen einer Maleimid-BSA Matrix der<br />
Firma Cellendes. ...................................................................................................................... 57<br />
Tabelle 4: Gemessene Chemotaxis-Parameter von HL- 60 Zellen in einer Versuchsreihe mit<br />
unterschiedlichen Chemoattraktant Konzentrationen. ............................................................. 59<br />
Tabelle 5: Chemotaxis Parameter von HL 60 Zellen des zum Vergleich mit den<br />
Diffusionsmessungen herangezogenen Experiments. .............................................................. 60<br />
8.Tabellenverzeichnis 78
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
9. Abkürzungsverzeichnis<br />
BSA Bovines Serum Albumin<br />
CCD charged coupled device<br />
COM center of mass<br />
DMEM Dulbeccos’ Modified Eagle Medium<br />
DMSO Dimethyl Sulfoxid<br />
EDTA Ethylendiamintetraacetat<br />
FCS Foetales Kälber Serum<br />
FCS Fluoreszenz Korrelations Spektroskopie<br />
FITC fluorescein isothiocyanate<br />
FMI forward migration index<br />
fMLP N-Formylmethionyl-Lencyl-Phenylalanin<br />
GFP green fluorescent protein<br />
HUVEC human umbilical cord vascular endothelial cell<br />
LSM Laser Scanning Mikroskop<br />
MMP Matrix-Metallo Protease<br />
PBS phosphate buffered saline<br />
PMN Polymorphonukleare Neutrophile<br />
RFP red fluorescent protein<br />
RGD Einheitsbuchstabencode für: Arginin-Glycin-Asparaginsäure<br />
RPM revolutions per minute (entspricht U/min)<br />
RPMI Roswell Park Memorial Institute<br />
9.Abkürzungsverzeichnis 79
10. Anhang<br />
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Anhang A: Verdünnungstabelle Qgel mit Angabe physikalischer Parameter (Ref 1001)<br />
[Qgel, 3D-Matrix Produkt Information, p03v00, April 2010]<br />
Anhang B: Pipettierschema der Cellendes 3D-Matrix nach Gelstärke<br />
[Cellendes, Hydrogel Userguide, v3.0, 2011]<br />
10. Anhang 80
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Anhang C: Pipettierschema der Cellendes 3D-Matrix in Abhängigkeit der<br />
Zusatzpeptidkonzentration. [Cellendes, Hydrogel Userguide, v3.0, 2011]<br />
Anhang D: Pipettierschemata für unterschiedliche Medien aus dem Standardprotokoll zum<br />
Erstellen von Gelmatrizes unterschiedlicher Dichte aus Collagen bovine.<br />
[Ibidi GmbH, Application Note 26, 2011]<br />
10. Anhang 81
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Anhang E: Standardprotokoll zur Vorbereitung von Chemotaxis Experimenten im µ-Slide<br />
Chemotaxis 3D. [Ibidi GmbH, Application Note 17, 2011]<br />
Standardprotokoll zum Befüllen des µ-Slide Chemotaxis 3D:<br />
- Slides und Medium sollten zur Gas Equillibrierung<br />
am Vortag schon in den Brutschrank mit einer<br />
Atmosphäre von etwa 37 °C und 5% CO2 gestellt<br />
werden.<br />
Verschlossene Gefäße sollten dazu leicht geöffnet<br />
werden.<br />
- Zuerst wird die Gelmatrix in den Slide gefüllt (vgl.<br />
Anhang D).<br />
Dazu werden die Einfüllstutzen C,D,E und F jeweils<br />
mit den mitgelieferten Stöpsel verschlossen<br />
- Anschließend werden 6µl der Gelsuspension auf<br />
Reservoir A des Kanals pipettiert<br />
- Die Pipettenspitze muss dann so auf Reservoir B<br />
gesetzt werden, dass eine schlüssige Verbindung<br />
besteht. Von dort werden 6 µl Volumen abgesaugt,<br />
so dass sich der ganze Kanal mit Flüssigkeit füllt.<br />
- Nach Einfüllen des Gels werden die Stöpsel aus den<br />
Stutzen C-E entfernt und die Reservoirs A und B<br />
verschlossen<br />
- Die Polymerisation des Gels im System erfolgt<br />
druckentlastet ohne Stöpsel auf den seitlichen<br />
Reservoirs.<br />
Um die Verdunstung von Flüssigkeit zu vermeiden<br />
müssen die Slides während dieser Zeit in eine<br />
feuchte Umgebung, wie zum Beispiel eine<br />
Petrischale mit angefeuchteten Tüchern gelegt<br />
werden.<br />
- Nach der matrixabhängigen Polymerisationszeit<br />
werden die seitlichen Reservoirs mit<br />
Chemoattraktant – Lösung befüllt.<br />
Dabei wird je nach Bedarf zwischen zwei<br />
Vorgehensweisen unterschieden:<br />
10. Anhang 82
Fast method:<br />
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
- Chemoattraktant-Lösung wird direkt in eines der<br />
beiden Reservoirs und Referenzlösung in das Andere<br />
gefüllt.<br />
- Dabei wird zuerst das Reservoir mit Hungermedium<br />
befüllt und mit Stöpseln verschlossen und<br />
anschließend das Reservoir mit Chemoattraktant, um<br />
ein Eindringen von Chemoattraktant-Lösung ins Gel<br />
durch Druckunterschiede zu verhindern.<br />
Slow method:<br />
- Zuerst werden beide Reservoirs mit Chemoattraktantfreier<br />
Lösung gefüllt und die Stutzen E und F<br />
anschließend verschlossen.<br />
- 30 µl Chemoattraktant-Lösung mit der doppelten<br />
Konzentration der gewünschten Endkonzentration<br />
werden auf Reservoir C pipettiert.<br />
- Mit einer geeigneten Pipettenspitze werden 30 µl<br />
Flüssigkeit aus Reservoir D gesaugt, um eine<br />
langsame Verteilung des Chemoattraktant innerhalb<br />
der Kammer zu erreichen.<br />
10. Anhang 83
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Anhang F: Zusammensetzung des RPMI 1640 Mediums (FM-32L,C-C-Pro)<br />
10. Anhang 84
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Anhang G: Zusammensetzung des DMEM Mediums (FM-58L,C-C-Pro)<br />
10. Anhang 85
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Anhang H: Tabelle der verwendeten Geräte, Chemikalien und Verbrauchsmaterialien.<br />
Geräte<br />
Brutschrank, Modell Nr. 3336 Forma Scientific<br />
Brutschrank, HERAcell Heraeus<br />
Fluoreszenzmikroskop, Evos fl. Advanced Microscopy Group<br />
Fluoreszenzmikroskop, Eclipse Ti Nikon Co.<br />
Gasmischer, The Brick<br />
Kolbenhubpipetten, 10µl/20µl/100µl/200µl/1000µl Gilson Inc.<br />
Laser Scanning Mikroskop LSM 510 an Axiovert 200 Carl Zeiss<br />
Objektträgerheizsystem, TC02 ibidi GmbH<br />
Sterilbank HERAsafe HS12 Heraeus<br />
Zählkammer (Neubauer improved) Paul Marienfeld GmbH & Co. KG<br />
Zellkulturmikroskop LSM 40 Carl Zeiss<br />
Zentrifuge, Universal 320 Andreas Hettich GmbH & Co. KG<br />
Chemikalien<br />
Collagen I (3mg/ml), PurCol Advanced Biomatrix Inc.<br />
Dimethylsulfoxid Sigma-Aldrich<br />
Dulbeccos' modified Eagle Medium, FM 58L C-C-Pro<br />
FiTC-Dextran Sigma-Aldrich<br />
Fluoreszenzfarbstoff, Alexa 488 Invitrogen<br />
fMLP Sigma-Aldrich<br />
G418 Sulfat PAA Laboratories GmbH<br />
Reinstwasser, MilliQ EMD Millipore Co.<br />
Minimal Essential Medium Sigma-Aldrich<br />
Maleimid-BSA, 3D-Life Cellendes GmbH<br />
Natriumkarbonat (7.5%) Sigma-Aldrich<br />
Natronlauge 1M Sigma-Aldrich<br />
Phosphat buffered saline C-C-Pro<br />
Polyethylenglycol, 3D-Life Cellendes GmbH<br />
Qgel, Ref. 1001 Qgel SA<br />
Qgel, Buffer A Qgel SA<br />
RGD-Peptid, 3D-Life Cellendes GmbH<br />
RPMI 1640 Medium, FM 32L C-C-Pro<br />
Thioglycerol, 3D-Life Cellendes GmbH<br />
Trypsin-EDTA Invitrogen<br />
Verbrauchsmaterialien<br />
µ-Slide Angiogenese ibidi GmbH<br />
µ-Slide Chemotaxis 3D ibidi GmbH<br />
Gewebekulturflaschen 75cm^2 Sarstedt AG & Co.<br />
Pipettenspitzen, Eppendorf Greiner Bio One GmbH<br />
Serotologische Einmalpipetten Sarstedt AG & Co.<br />
Zellkultur-Petrischalen 3,5cm, tissue culture treat Falcon, Becton-Dickinson<br />
10. Anhang 86
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Zellkultur-Petrischalen 10cm Sarstedt AG & Co.<br />
Zentrifugenröhrchen 1,5ml Eppendorf AG<br />
Zentrifugenröhrchen 15ml/50ml Falcon, Becton-Dickinson<br />
10. Anhang 87
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
11. Literaturverzeichnis<br />
Adler, J. 1975. Chemotaxis in bacteria. Annu Rev Biochem 44: 341-56<br />
Becker, EL. 1977. Stimulated neutrophil locomotion: chemokinesis and chemotaxis. Arch<br />
Pathol Lab Med 101(10): 509-13.<br />
Bretscher, M. S. Moving membrane up to the front of<br />
migrating cells. Cell 85: 465–467<br />
Bray, D., White, J.G. Cortical flow in animal cells. Science 239: 883-88.<br />
Carter, S.B. 1967. Haptotaxis and the mechanism of cell motility. Nature 213: 256-60.<br />
Collins, S.J., Gallo, R., Gallager R. 1977. Continous growth and differentiation of human<br />
myeloid leukemic cells in suspension culture. Nature 270: 347<br />
Collins,S.J. , Ruscetti, F.W., Gallager, R.E., Gallo R.C. 1978. Terminal differentiation of<br />
human promyelocytic leukemia cells induced by dimethyl sulfoxide and other polar<br />
compounds. Proc. Natl. Acad. Sci. USA 75: 2458-62.<br />
Collins, S.J. 1987. The HL-60 promyelocytic leukemia cell line: proliferation, differentiation<br />
and cellular oncogene expression. Blood 70: 1233-1244<br />
Engelmann, T.W. 1881. Pfüger's Arch. Gesamte Physiol. Menschen Tiere 25.<br />
Even-Ram, S., Yamada, K.M. 2005. Cell migration in 3D matrix . Current Opinion in Cell<br />
Biology 17:524–532<br />
Foxman, E.F., E.J. Kunkel und E.C. Butcher. 1999. Integration conflicting chemotactic<br />
signals. The role of memory in leukocyte navigation. J Cell Biol 147: 577-88.<br />
Friedl, P., Bröcker, E.-B. 2000. The biology of cell locomotion within 3-dimensional<br />
extracellular Matrix. Cell Mol Life Sci 57: 41–64.<br />
11.Literaturverzeichnis 88
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Fisher, P.R., Merkl, R. und Gerisch, G. 1989. Quantitative analysis of cell motility and<br />
chemotaxis in Dictyostelium discoideum by using an image processing system and a<br />
novel chemotaxis chamber providing stationary chemical gradients.<br />
J cell Biol 108: 973-84<br />
Grinell, F. 1982. Migration of human neutrophils<br />
in hydrated Collagen lattices J. Cell Sci. 58, 95-108<br />
Jeon, N.L., Baskaran, H., Dertinger, S.K.W., Whitesides, G.M., Van de Water, L. and<br />
Toner, M. 2002. Neutrophil chemotaxis in linear and complex gradients of interleukin-<br />
8 formed in a microfabricated device. Nature biotechnology 20: 826-30.<br />
Kirkman-Brown, J.C., Sutton K.A. und H.M. Florman. 2003. How to attract sperm. Nat Cell<br />
Biol 5: 93-6<br />
Kőhidai L. 1995. Method for determination of chemoattraction in Tetrahymena pyriformis.<br />
Curr Microbiol 30: 251–3.<br />
Lefort, C. T., Kim, M. 2010. Human T Lymphocyte Isolation, Culture and Analysis of<br />
Migration In Vitro. J. Vis. Exp. 40, e2017, DOI: 10.3791/2017<br />
Mato, J.M., Losada, A., Nanjundiah, V., Konijn, T. 1975. Signal input for a chemotactic<br />
response in the cellular slime mold Dictyostelium discoideum. Procl. Nat. Acad. Sci.<br />
USA 72: 4991-93.<br />
Martin, T.A., R. Mansel und W.G. Jiang 2001. Hepatocyte growth factor modulates vascular<br />
endothelial-cadherin expression in human endothelial cells. Clin cancer Res 7: 734-7.<br />
Pfeffer, W. 1884/1888. Untersuch. Bot. Inst. Tübingen 1+2. 363-482/582-661.<br />
Rasheed, S., W. A. Nelson-Rees, et al. 1974. Characterization of a newly derived human sar-<br />
coma cell line (HT-1080). Cancer 33: 1027-33.<br />
11.Literaturverzeichnis 89
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Riedl, J., Crevenna, A.H., Kessenbrock, K., Haochen, Yu, J., Neukirchen, D., Bista, M.,<br />
Bradke, F., Jenne, D., A Holak, T., Werb Z., Sixt, M. and Wedlich-Soldne, R. 2008.<br />
Lifeact: a versatile marker to visualize F-actin. Nature methods 5: 605-7.<br />
Roussos, ET.,Condeelis, JS., Patsialou A. 2011. Chemotaxis in Cancer. Nat Rev Cancer 11(8):<br />
573-87.<br />
Schiffman, E. 1982. Leukocyte chemotaxis. Ann Rev Physiol 44: 553-68.<br />
Singer, S. J., Kupfer, A. 1986. The directed migration of eukaryotic cells. Ann Rev Cell Biol.<br />
2: 337-65.<br />
Tranquillo, R.T., Lauffenburger, T.A., Zigmond, S.H. 1988. A stochastic model for leukocyte<br />
random motility and chemotaxis based on receptor binding fluctuations. J. Cell Biol.<br />
1<strong>06</strong>: 303-9<br />
Weiner, O.D.,Servant G, Welch MD, Mitchison TJ, Sedat JW, Bourne HR. 1999. Spatial<br />
control of actin polymerization during neutrophil chemotaxis. Nat Cell Biol 1:75-<br />
81.<br />
Weiner, O.D. 2002. Regulation of cell polarity during eukaryotic chemotaxis: the<br />
chemotactic compass . Current Opinion in Cell Biology 14:196–202 .<br />
Zigmond, S.H. et al. 1981. Cell Polarity : An examination of its behavioral expression and its<br />
consequences for polymorphonuclear leukocyte chemotaxis . The journal of cell<br />
biology 89: 585-92.<br />
Devreotes, P.N., Zigmond, SH, 1988. Chemotaxis in eucaryotic cells: A focus on leukocytes<br />
and dictyostelium. Ann Rev Cell Biol. 4: 649-86<br />
Zigmond, S.H. 1977. Ability of polymorphonuclear leukocytes to orient in gradients of<br />
chemotactic factors. J Cell Biol 75: 6<strong>06</strong>-16.<br />
11.Literaturverzeichnis 90
Jürschick, Johannes – Chemotaxis in µ-Slides mit 3-dimensionalen Gelmatrizes<br />
Selbstständigkeitserklärung<br />
Ich erkläre hiermit, dass ich die vorliegende Arbeit selbsständig und nur unter Verwendung<br />
der angegebenen Literatur und Hilfsmittel angefertigt habe:<br />
Ort, Datum Unterschrift<br />
Selbstständigkeitserklärung 91