PDF Download - Laborwelt
PDF Download - Laborwelt
PDF Download - Laborwelt
Erfolgreiche ePaper selbst erstellen
Machen Sie aus Ihren PDF Publikationen ein blätterbares Flipbook mit unserer einzigartigen Google optimierten e-Paper Software.
Abb. 3: Genomische DNAs aus (v.l.n.r.) Tomatenblättern<br />
(100 mg), Hefe (Saccharomyces, 1 ml),<br />
Zellkultur (Affennieren, 10 7 Zellen), Blut (human,<br />
100 µl), Lunge (Ratte, 150 mg), Phage Lambda<br />
(2,5 ml, 8 x 10 11 /ml), Waldboden (0,5 g). M: Marker<br />
Abb. 4 : Isolierung von pUC13-DNA aus 10 ml E.<br />
coli SF8-Kulturen. Aufgetragen wurden gleiche<br />
proportionale Volumina der Präparationen.<br />
M: Marker; 1: ‚PrestoSpin D Plasmid Mini Spin‘-<br />
Säulen-Kit, Eluat; 2: Säulendurchlauf; 3: Gesamt-<br />
Plasmid-DNA im Rohlysat; 4: Plasmid-DNA mit<br />
einem gravitationsgetriebenen Durchfluß-Säulen-Kit<br />
(Anionenaustauscher) eines anderen Anbieters<br />
gereinigt.<br />
mM) von multivalenten Kationen, etwa<br />
Mg 2+ , an negativ geladene Oberflächen. Untersuchungsergebnisse<br />
weisen darauf hin,<br />
daß die Mg 2+ -Komplexierung für die stabile<br />
Bindung der negativ geladenenen Nukleinsäuren<br />
an negativ geladene Oberflächen verantwortlich<br />
ist 2 . Der Komplex wird aufgelöst,<br />
wenn komplexierende Agenzien wie<br />
EDTA die Mg 2+ -Ionen entfernen. Als Folge<br />
gehen gebundene Nukleinsäuren in Lösung.<br />
In den ‚PrestoSpin D‘-Kits von Molzym (zu<br />
beziehen über Omnilab Life Science,<br />
www.ols-biotech.de) laufen alle durch verschiedene<br />
Verfahren erhaltene Zellysate<br />
durch dasselbe Reinigungsverfahren nach<br />
obigem Muster (Abb. 2).<br />
Tonminerale als<br />
Nukleinsäure-Bindematrix<br />
Bindematrix für die ‚PrestoSpin D‘-Kits sind<br />
Tonminerale. Sie zeichnen sich in Verbindung<br />
mit dem Bindemechanismus der Kationen-Komplexierung<br />
durch eine hohe Bindekapazität<br />
von mehr als 28 µg/mg aus.<br />
‚PrestoSpin D‘-Spin-Säulen bestehen aus einem<br />
Gemisch aus hochgereinigtem Sand<br />
und Ton. Die Nukleinsäure-Bindekapazität<br />
der Säulen ist mit mehr als 80 µg eine der<br />
bisher höchsten im Mini Spin-Säulenformat.<br />
Die hohe Porosität der Säulenmatrix erlaubt<br />
B L I T Z L I C H T<br />
eine einfache Reinigung von Gesamt-Nukleinsäuren<br />
oder DNA auch aus stark viskosen<br />
Lösungen. So können größere Mengen an<br />
Ausgangsmaterial in den Nukleinsäure-Reinigungsvorgang<br />
eingehen.<br />
Reinigung genomischer DNA<br />
Die ‚PrestoSpin D‘-Kits erlauben die Reinigung<br />
von Gesamt-Nukleinsäuren (Presto-<br />
Spin D-Universal-Kit) beziehungsweise<br />
DNA aus einer Vielzahl von Organismen<br />
und Materialien (Abb. 3). Mit dem Presto-<br />
Spin D-Universal-Kit kann sowohl genomische<br />
DNA aus einer Vielzahl von biologischen<br />
Materialien – unter anderem Bakteriophagen,<br />
Bakterien, Hefen, Pilzen, tierischen<br />
und pflanzlichen Geweben, Zellkulturen,<br />
Blut, Böden und Sedimenten (Abb. 3)<br />
– als auch Plasmid-DNA im Mini- und Midi-<br />
Format gereinigt werden. Die Ausbeuten betragen,<br />
je nach Ausgangsmaterial, bis zu 80<br />
µg genomische DNA und 50 µg Plasmid-<br />
DNA. Die Präparation hochreiner DNA aus<br />
Zellysaten beträgt nur 10 bis 25 Minuten.<br />
Die Lysisprotokolle sind darauf ausgelegt,<br />
bei der Zelldesintegration freiwerdende Nukleinsäuren<br />
effizient vor dem Verdau endogener<br />
Nukleasen zu schützen. Dies wird<br />
durch den Einsatz von hochchaotropem Guanidinhydrochlorid<br />
erreicht. Nach einem vereinheitlichten<br />
Protokoll werden Lysate auf<br />
eine Spin-Säule gegeben, wo die DNA-Bindung<br />
durch die Kationen-Komplexierung<br />
erfolgt. Nieder- und hochmolekulare Verunreinigungen<br />
(unter anderem Proteine, Polysaccharide,<br />
Nukleotide, Aminosäuren, Salze)<br />
werden mit einem speziellen alkoholhaltigen<br />
Puffer durch kurze Zentrifugation ausgewaschen.<br />
In einem zweiten Waschschritt mit 70%<br />
Ethanol erfolgt die Beseitigung von Salzen,<br />
wonach die reine DNA mit üblichem TE-Puffer<br />
eluiert wird (10 mM Tris, 1 mM EDTA).<br />
Optional kann RNA durch kurzzeitige Inkubation<br />
mit RNase A in der Säule vor den<br />
Waschschritten degradiert werden. Für jedes<br />
der oben genannten biologischen Materialien<br />
gibt es zudem je ein Spezialkit zur Isolierung<br />
genomischer beziehungsweise von Plasmid-DNA.<br />
Reinigung von Plasmid-DNA<br />
Die Architektur und die hohe Bindekapazität<br />
der Ton-Sand-Matrix in den ‚Mini Spin‘-<br />
Säulen hat die Entwicklung eines Reinigungs-Kits<br />
ermöglicht, mit dem Plasmid-<br />
DNA im Midi-Maßstab (bis 50 µg) isoliert<br />
werden kann (Abb. 4). ‚High copy‘-Plasmid-<br />
Vektoren können aus 10 bis 20 ml Kulturen<br />
in einem Verfahren gereinigt werden, das gegenüber<br />
den ansonsten in diesem Maßstab<br />
üblichen gravitationsgetriebenen Durchfluß-Säulen<br />
(Anionenaustauscher) deutlich<br />
schneller ist (etwa 45 Minuten gegenüber<br />
mehr als 2 Stunden). Das Verfahren ist sehr<br />
schonend, so daß die Plasmid-DNA fast aus-<br />
schließlich in der Supercoil-Konformation<br />
isoliert wird. Mit dem ‚PrestoSpin D Plasmid<br />
Mini Spin‘-Säulen-Kit besteht weiterhin<br />
die Option, Aufreinigungen aus bis zu 100<br />
ml Kulturvolumen durchzuführen. Dies erlaubt<br />
die Reinigung von ‚low copy‘-Plasmiden,<br />
etwa für Screening-Zwecke (Abb. 5),<br />
hochmolekularen rekombinanten Plasmiden<br />
mit erniedrigter Kopiezahl (Abb. 6) sowie<br />
von Cosmiden und BACs.<br />
Literatur<br />
[1] Hesselink, FT (1983) In: Adsorption from solution at the solid/liquid<br />
interface, GD Parfitt, CH Rochester (ed.), pp. 377-412, Academic Press,<br />
London.<br />
[2] Lorenz, MG (1998) In: Microbial interactions in agriculture and forestry,<br />
NS Subbarao, YR Dommergues (ed.), pp. 19-44, Oxford and IBH Publishing<br />
Co., New Delhi.<br />
Korrespondenzadresse<br />
Prof. Dr. Michael Lorenz<br />
Molzym GmbH & Co.KG<br />
Leobener Strasse<br />
D-28359 Bremen<br />
Tel./Fax: +49-(0)421-218-8780 / -8781<br />
eMail: info@molzym.com<br />
www.molzym.com<br />
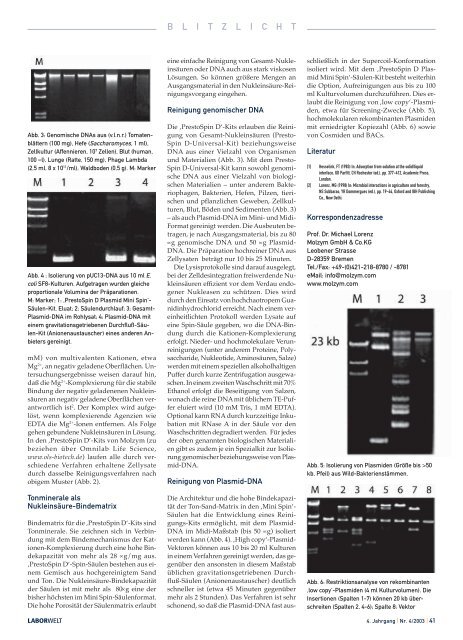
Abb. 5: Isolierung von Plasmiden (Größe bis >50<br />
kb, Pfeil) aus Wild-Bakterienstämmen.<br />
Abb. 6: Restriktionsanalyse von rekombinanten<br />
‚low copy‘-Plasmiden (4 ml Kulturvolumen). Die<br />
Insertionen (Spalten 1-7) können 20 kb überschreiten<br />
(Spalten 2, 4-6); Spalte 8: Vektor<br />
LABORWELT 4. Jahrgang | Nr. 4/2003 | 41