Der GMOfinder - DLR Online: Deutsche Lebensmittel Rundschau
Der GMOfinder - DLR Online: Deutsche Lebensmittel Rundschau
Der GMOfinder - DLR Online: Deutsche Lebensmittel Rundschau
Erfolgreiche ePaper selbst erstellen
Machen Sie aus Ihren PDF Publikationen ein blätterbares Flipbook mit unserer einzigartigen Google optimierten e-Paper Software.
642 Sonderthema: Mikrobiologische Methoden in der <strong>Lebensmittel</strong>analytik «<br />
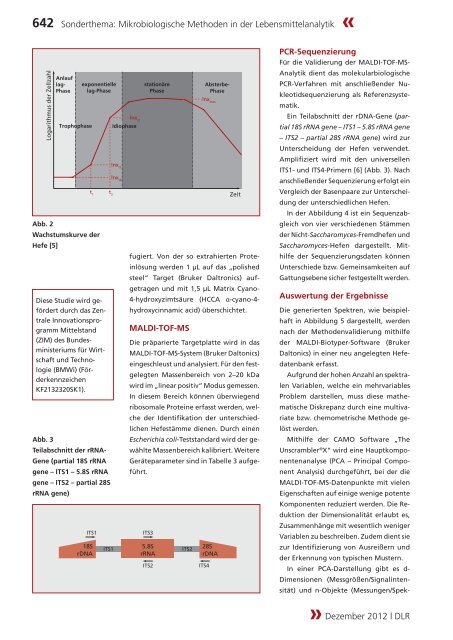
Logarithmus der Zellzahl<br />
Anlauf<br />
lag-<br />
Phase<br />
Trophophase<br />
exponentielle<br />
lag-Phase<br />
t 1<br />
Abb. 2<br />
Wachstumskurve der<br />
Hefe [5]<br />
Diese Studie wird gefördert<br />
durch das Zentrale<br />
Innovationsprogramm<br />
Mittelstand<br />
(ZIM) des Bundesministeriums<br />
für Wirtschaft<br />
und Technologie<br />
(BMWi) (Förderkennzeichen<br />
KF2132320SK1).<br />
Abb. 3<br />
Teilabschnitt der rRNA-<br />
Gene (partial 18S rRNA<br />
gene – ITS1 – 5.8S rRNA<br />
gene – ITS2 – partial 28S<br />
rRNA gene)<br />
ITS1<br />
18S<br />
rDNA<br />
lnx t2<br />
Idiophase<br />
lnx t1<br />
lnx t0<br />
t 2<br />
ITS1<br />
stationäre<br />
Phase<br />
fugiert. Von der so extrahierten Proteinlösung<br />
werden 1 µL auf das „polished<br />
steel“ Target (Bruker Daltronics) aufgetragen<br />
und mit 1,5 µL Matrix Cyano-<br />
4-hydroxyzimtsäure (HCCA α-cyano-4-<br />
hydroxycinnamic acid) überschichtet.<br />
MALDI-TOF-MS<br />
Absterbe-<br />
Phase<br />
lnx max<br />
Zeit<br />
Die präparierte Targetplatte wird in das<br />
MALDI-TOF-MS-System (Bruker Daltonics)<br />
eingeschleust und analysiert. Für den festgelegten<br />
Massenbereich von 2–20 kDa<br />
wird im „linear positiv“ Modus gemessen.<br />
In diesem Bereich können überwiegend<br />
ribosomale Proteine erfasst werden, welche<br />
der Identifikation der unterschiedlichen<br />
Hefestämme dienen. Durch einen<br />
Escherichia coli-Teststandard wird der gewählte<br />
Massenbereich kalibriert. Weitere<br />
Geräteparameter sind in Tabelle 3 aufgeführt.<br />
ITS3<br />
5.8S<br />
rRNA<br />
ITS2<br />
ITS2<br />
28S<br />
rDNA<br />
ITS4<br />
PCR-Sequenzierung<br />
Für die Validierung der MALDI-TOF-MS-<br />
Analytik dient das molekularbiologische<br />
PCR-Verfahren mit anschließender Nukleotidsequenzierung<br />
als Referenzsystematik.<br />
Ein Teilabschnitt der rDNA-Gene (partial<br />
18S rRNA gene – ITS1 – 5.8S rRNA gene<br />
– ITS2 – partial 28S rRNA gene) wird zur<br />
Unterscheidung der Hefen verwendet.<br />
Amplifiziert wird mit den universellen<br />
ITS1- und ITS4-Primern [6] (Abb. 3). Nach<br />
anschließender Sequenzierung erfolgt ein<br />
Vergleich der Basenpaare zur Unterscheidung<br />
der unterschiedlichen Hefen.<br />
In der Abbildung 4 ist ein Sequenzabgleich<br />
von vier verschiedenen Stämmen<br />
der Nicht-Saccharomyces-Fremdhefen und<br />
Saccharomyces-Hefen dargestellt. Mithilfe<br />
der Sequenzierungsdaten können<br />
Unterschiede bzw. Gemeinsamkeiten auf<br />
Gattungsebene sicher festgestellt werden.<br />
Auswertung der Ergebnisse<br />
Die generierten Spektren, wie beispielhaft<br />
in Abbildung 5 dargestellt, werden<br />
nach der Methodenvalidierung mithilfe<br />
der MALDI-Biotyper-Software (Bruker<br />
Daltonics) in einer neu angelegten Hefedatenbank<br />
erfasst.<br />
Aufgrund der hohen Anzahl an spektralen<br />
Variablen, welche ein mehrvariables<br />
Problem darstellen, muss diese mathematische<br />
Diskrepanz durch eine multivariate<br />
bzw. chemometrische Methode gelöst<br />
werden.<br />
Mithilfe der CAMO Software „The<br />
Unscrambler ® X“ wird eine Hauptkomponentenanalyse<br />
(PCA – Principal Component<br />
Analysis) durchgeführt, bei der die<br />
MALDI-TOF-MS-Datenpunkte mit vielen<br />
Eigenschaften auf einige wenige potente<br />
Komponenten reduziert werden. Die Reduktion<br />
der Dimensionalität erlaubt es,<br />
Zusammenhänge mit wesentlich weniger<br />
Variablen zu beschreiben. Zudem dient sie<br />
zur Identifizierung von Ausreißern und<br />
der Erkennung von typischen Mustern.<br />
In einer PCA-Darstellung gibt es d-<br />
Dimensionen (Messgrößen/Signalintensität)<br />
und n-Objekte (Messungen/Spek-<br />
» Dezember 2012 | <strong>DLR</strong>