PDF Download - Laborwelt
PDF Download - Laborwelt
PDF Download - Laborwelt
Sie wollen auch ein ePaper? Erhöhen Sie die Reichweite Ihrer Titel.
YUMPU macht aus Druck-PDFs automatisch weboptimierte ePaper, die Google liebt.
B L I T Z L I C H T<br />
Oligonukleotid-Arrays<br />
Sequenzoptimierte Longmer-<br />
Oligonukleotide für DNA-Microarrays<br />
CHRIS SEIDEL 1 , SAJEEV BATRA 1 , DR. RALF HUCH 2 UND DR. PETER SCHÜSSLER 3 ,<br />
1<br />
OPERON INC., ALAMEDA (CA), USA, 2 QIAGEN GMBH, HILDEN, 3 OPERON EUROPE, KÖLN<br />
DNA-Microarrays ermöglichen<br />
die parallele Expressionsanalyse<br />
einer großen Anzahl von<br />
Genen in einem Experiment. Dabei<br />
können die zu untersuchenden<br />
Gene durch Oligonukleotide<br />
oder PCR-Fragmente auf<br />
dem DNA-Microarray repräsentiert<br />
werden.<br />
Bei der Genexpressionsanalyse<br />
mit Hilfe von PCR-Fragment-<br />
Microarrays (cDNA-Arrays)<br />
kommt es aufgrund von Kreuzhybridisierungen<br />
zwischen homologen<br />
DNA-Sequenzen häufig<br />
zu Schwierigkeiten, die Expressionsmuster<br />
verwandter<br />
Gene zu unterscheiden. Ebenso<br />
titativ erfaßt werden kann.<br />
In der hier beschriebenen Untersuchung<br />
werden 70mer-<br />
Microarrays mit PCR-Fragment-Microarrays<br />
hinsichtlich<br />
ihrer Fähigkeit verglichen, die<br />
durch Hitzeschock induzierte<br />
differentielle Genexpression zu<br />
analysieren.<br />
Experimentelles<br />
Vorgehen<br />
Die DNA-Microarrays zur Untersuchung<br />
des Hitzeschock-<br />
Effekts auf die Genexpression<br />
bei der Bäckerhefe (Saccharomyces<br />
cerevisiae, Stamm S288C)<br />
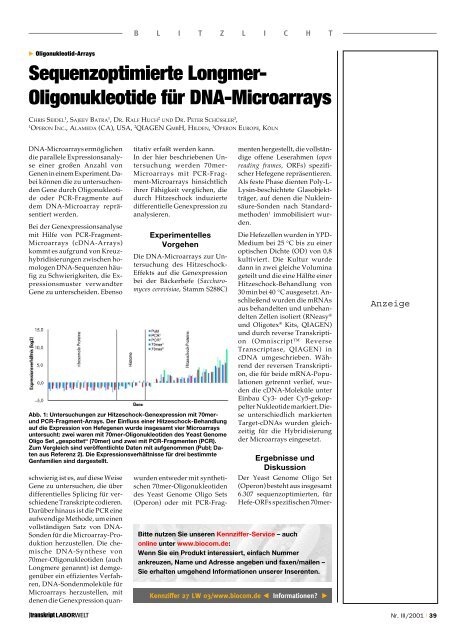
Abb. 1: Untersuchungen zur Hitzeschock-Genexpression mit 70merund<br />
PCR-Fragment-Arrays. Der Einfluss einer Hitzeschock-Behandlung<br />
auf die Expression von Hefegenen wurde insgesamt vier Microarrays<br />
untersucht: zwei waren mit 70mer-Oligonukleotiden des Yeast Genome<br />
Oligo Set „gespottet“ (70mer) und zwei mit PCR-Fragmenten (PCR).<br />
Zum Vergleich sind veröffentlichte Daten mit aufgenommen (Publ; Daten<br />
aus Referenz 2). Die Expressionsverhältnisse für drei bestimmte<br />
Genfamilien sind dargestellt.<br />
schwierig ist es, auf diese Weise<br />
Gene zu untersuchen, die über<br />
differentielles Splicing für verschiedene<br />
Transkripte codieren.<br />
Darüber hinaus ist die PCR eine<br />
aufwendige Methode, um einen<br />
vollständigen Satz von DNA-<br />
Sonden für die Microarray-Produktion<br />
herzustellen. Die chemische<br />
DNA-Synthese von<br />
70mer-Oligonukleotiden (auch<br />
Longmere genannt) ist demgegenüber<br />
ein effizientes Verfahren,<br />
DNA-Sondenmoleküle für<br />
Microarrays herzustellen, mit<br />
denen die Genexpression quanwurden<br />
entweder mit synthetischen<br />
70mer-Oligonukleotiden<br />
des Yeast Genome Oligo Sets<br />
(Operon) oder mit PCR-Fragmenten<br />
hergestellt, die vollständige<br />
offene Leserahmen (open<br />
reading frames, ORFs) spezifischer<br />
Hefegene repräsentieren.<br />
Als feste Phase dienten Poly-L-<br />
Lysin-beschichtete Glasobjektträger,<br />
auf denen die Nukleinsäure-Sonden<br />
nach Standardmethoden<br />
1 immobilisiert wurden.<br />
Die Hefezellen wurden in YPD-<br />
Medium bei 25 °C bis zu einer<br />
optischen Dichte (OD) von 0,8<br />
kultiviert. Die Kultur wurde<br />
dann in zwei gleiche Volumina<br />
geteilt und die eine Hälfte einer<br />
Hitzeschock-Behandlung von<br />
30 min bei 40 °C ausgesetzt. Anschließend<br />
wurden die mRNAs<br />
aus behandelten und unbehandelten<br />
Zellen isoliert (RNeasy ®<br />
und Oligotex ® Kits, QIAGEN)<br />
und durch reverse Transkription<br />
(Omniscript Reverse<br />
Transcriptase, QIAGEN) in<br />
cDNA umgeschrieben. Während<br />
der reversen Transkription,<br />
die für beide mRNA-Populationen<br />
getrennt verlief, wurden<br />
die cDNA-Moleküle unter<br />
Einbau Cy3- oder Cy5-gekoppelter<br />
Nukleotide markiert. Diese<br />
unterschiedlich markierten<br />
Target-cDNAs wurden gleichzeitig<br />
für die Hybridisierung<br />
der Microarrays eingesetzt.<br />
Ergebnisse und<br />
Diskussion<br />
Der Yeast Genome Oligo Set<br />
(Operon) besteht aus insgesamt<br />
6.307 sequenzoptimierten, für<br />
Hefe-ORFs spezifischen 70mer-<br />
Bitte nutzen Sie unseren Kennziffer-Service – auch<br />
online unter www.biocom.de:<br />
Wenn Sie ein Produkt interessiert, einfach Nummer<br />
ankreuzen, Name und Adresse angeben und faxen/mailen –<br />
Sie erhalten umgehend Informationen unserer Inserenten.<br />
Kennziffer 27 LW 03/www.biocom.de Informationen? <br />
Anzeige<br />
|transkript LABORWELT Nr. III/2001 | 39