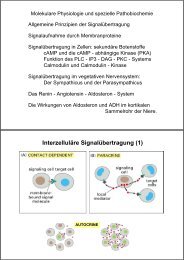
pep-2 - Lehrstuhl für Ernährungsphysiologie - TUM
pep-2 - Lehrstuhl für Ernährungsphysiologie - TUM
pep-2 - Lehrstuhl für Ernährungsphysiologie - TUM
Sie wollen auch ein ePaper? Erhöhen Sie die Reichweite Ihrer Titel.
YUMPU macht aus Druck-PDFs automatisch weboptimierte ePaper, die Google liebt.
Random Hexamers (0,5 µg/µl), 0,31 µl RNAse-Inhibitor (40 U/µl), 1µl MMLV Reverse<br />
Transcriptase Rnase H Minus, Point Mutant (200 U/µl) und 8,29 µl PCR-H2O pro Probe.<br />
Nach Zugabe des zweiten Mastermixes werden die Proben <strong>für</strong> 1 h bei 37°C und 1 min bei<br />
99°C inkubiert. Auch hier ist es wichtig den Mastermix und die Proben zuvor gut zu<br />
mischen. Es ist sinnvoll als Qualitäts-Kontrolle <strong>für</strong> die vorherige RNA-Extraktion eine<br />
Probe mitzuführen, der kein Enzym zugesetzt wurde (O-Kontroll-Probe). Dadurch lassen<br />
sich eventuelle Verunreinigungen der Proben mit DNA in den späteren LightCycler ®<br />
Amplifikationen nachweisen. Die cDNA wird bis zur Verwendung bei –80°C gelagert.<br />
2.2.8.2 Lightcycler-Amplifikation<br />
Die real-time RT-PCR (reverse transcriptase-polymerase chain reaction) ist eine sehr<br />
empfindliche Technik, um mRNA zu detektieren und zu quantifizieren. Darüber hinaus<br />
stellt die real-time RT-PCR ein wichtiges Werkzeug <strong>für</strong> die Klonierung, die Erstellung von<br />
cDNA-Bibliotheken, die Synthese von Sonden, Differential Display und <strong>für</strong> die<br />
Signalverstärkung bei in situ Hybridisierungen dar. Die Technik der real-time RT-PCR<br />
setzt sich aus zwei Stufen zusammen: zunächst muss die cDNA aus der mRNA durch die<br />
Reverse Transkription (RT) gewonnen werden und anschließend kann diese cDNA in einer<br />
spezifischen Polymerasekettenreaktion (PCR) amplifiziert werden. Die Detektion der<br />
Amplifikationsprodukte erfolgt in Echtzeit, was dieser Technik ihren Namen gab.<br />
Die Polymerasekettenreaktion besteht im wesentlichen aus drei Stufen, die <strong>für</strong> 30-50<br />
Zyklen wiederholt werden:<br />
1. Denaturierung: Hier wird der Reaktionsansatz auf ca. 95°C erhitzt, um die<br />
Trennung der doppelsträngigen DNA zu bewirken, damit der nächste Schritt<br />
erfolgen kann.<br />
2. Annealing: In diesem Schritt wird die Temperatur wieder erniedrigt (ca.55-60°C,<br />
primerabhängig). Nun lagern sich die einzelsträngigen Primer an die<br />
einzelsträngige DNA an. Primer, die an komplementären Sequenzen gebunden<br />
haben, bleiben dort gebunden, so dass an diesem Stück doppelsträngiger DNA die<br />
DNA-Polymerase ansetzen kann und die DNA komplementär vervollständigen<br />
kann. Dies geschieht im nächsten Schritt.<br />
40