Analyse avec le logiciel imagej d'un lot d'images en microscopie par ...
Analyse avec le logiciel imagej d'un lot d'images en microscopie par ...
Analyse avec le logiciel imagej d'un lot d'images en microscopie par ...
You also want an ePaper? Increase the reach of your titles
YUMPU automatically turns print PDFs into web optimized ePapers that Google loves.
in2p3-00530281, version 1 - 28 Oct 2010<br />
Pour ce faire, différ<strong>en</strong>tes tail<strong>le</strong>s d’agrégats correspondant aux dim<strong>en</strong>sions des cassures de l’ADN (10-100<br />
nm) peuv<strong>en</strong>t être exploré. Des corrélations seront recherchées <strong>en</strong>tre tail<strong>le</strong>s d'agrégats, int<strong>en</strong>sité<br />
lumineuse et tail<strong>le</strong> des foci <strong>d'un</strong>e <strong>par</strong>t, et nombre des évènem<strong>en</strong>ts physiques et des foci d'autre <strong>par</strong>t.<br />
La s<strong>en</strong>sibilité de la méthode pourra être améliorée dans un second temps, <strong>en</strong> pr<strong>en</strong>ant <strong>en</strong> compte la<br />
topologie de la distribution des agrégats dans <strong>le</strong>s évènem<strong>en</strong>ts simulés.<br />
Mise <strong>en</strong> évid<strong>en</strong>ce des dommages ADN <strong>par</strong> marqueurs<br />
moléculaires<br />
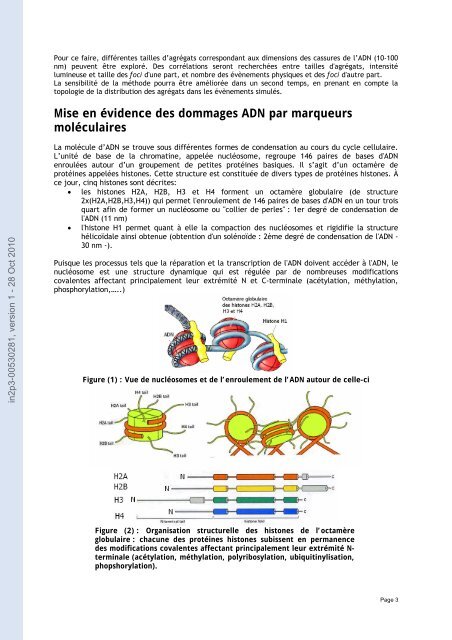
La molécu<strong>le</strong> d’ADN se trouve sous différ<strong>en</strong>tes formes de cond<strong>en</strong>sation au cours du cyc<strong>le</strong> cellulaire.<br />
L’unité de base de la chromatine, appelée nucléosome, regroupe 146 paires de bases d'ADN<br />
<strong>en</strong>roulées autour d’un groupem<strong>en</strong>t de petites protéines basiques. Il s’agit d’un octamère de<br />
protéines appelées histones. Cette structure est constituée de divers types de protéines histones. À<br />
ce jour, cinq histones sont décrites:<br />
<strong>le</strong>s histones H2A, H2B, H3 et H4 form<strong>en</strong>t un octamère globulaire (de structure<br />
2x(H2A,H2B,H3,H4)) qui permet l'<strong>en</strong>rou<strong>le</strong>m<strong>en</strong>t de 146 paires de bases d'ADN <strong>en</strong> un tour trois<br />
quart afin de former un nucléosome ou "collier de per<strong>le</strong>s" : 1er degré de cond<strong>en</strong>sation de<br />
l'ADN (11 nm)<br />
l'histone H1 permet quant à el<strong>le</strong> la compaction des nucléosomes et rigidifie la structure<br />
hélicoïda<strong>le</strong> ainsi obt<strong>en</strong>ue (obt<strong>en</strong>tion <strong>d'un</strong> solénoïde : 2ème degré de cond<strong>en</strong>sation de l'ADN -<br />
30 nm -).<br />
Puisque <strong>le</strong>s processus tels que la ré<strong>par</strong>ation et la transcription de l'ADN doiv<strong>en</strong>t accéder à l'ADN, <strong>le</strong><br />
nucléosome est une structure dynamique qui est régulée <strong>par</strong> de nombreuses modifications<br />
cova<strong>le</strong>ntes affectant principa<strong>le</strong>m<strong>en</strong>t <strong>le</strong>ur extrémité N et C-termina<strong>le</strong> (acétylation, méthylation,<br />
phosphorylation,…..)<br />
Figure (1) : Vue de nucléosomes et de l’<strong>en</strong>rou<strong>le</strong>m<strong>en</strong>t de l’ADN autour de cel<strong>le</strong>-ci<br />
Figure (2) : Organisation structurel<strong>le</strong> des histones de l’octamère<br />
globulaire : chacune des protéines histones subiss<strong>en</strong>t <strong>en</strong> perman<strong>en</strong>ce<br />
des modifications cova<strong>le</strong>ntes affectant principa<strong>le</strong>m<strong>en</strong>t <strong>le</strong>ur extrémité Ntermina<strong>le</strong><br />
(acétylation, méthylation, polyribosylation, ubiquitinylisation,<br />
phopshorylation).<br />
Page 3







![[in2p3-00383985, v1] NUCLEAR PHYSICS at GANIL ... - HAL - IN2P3](https://img.yumpu.com/19016755/1/185x260/in2p3-00383985-v1-nuclear-physics-at-ganil-hal-in2p3.jpg?quality=85)






![[tel-00752304, v1] Pions réels et virtuels dans les noyaux](https://img.yumpu.com/19016523/1/184x260/tel-00752304-v1-pions-reels-et-virtuels-dans-les-noyaux.jpg?quality=85)
